山西矿区复垦土壤中解磷细菌的筛选及鉴定
陈 倩, 刘善江*, 白 杨, 李亚星, 郜春花, 张 强, 李建华
(1 北京市农林科学院植物营养与资源研究所, 北京 100097;2 山西省农业科学院农业环境与资源研究所,山西 太原 030006)
山西煤炭资源极其丰富,占全国已探明煤炭储量的1/3[1],但在为全国经济发展做出了巨大贡献的同时,其农业生态环境也遭到了严重的破坏[2],矿区复垦土壤贫瘠,养分含量低,微生物数量少,活性低[3],而有益微生物可通过多方面的作用加速复垦进程,促进植物的生长发育[4]。磷是植物生长必需的营养元素之一,其重要性仅次于氮,约占植物干重的0.2%[5],是参与能量代谢、 核酸与细胞合成、 部分酶调控的主要成分[6],在生态系统中起着不可替代的作用。一般土壤中含磷量平均约为0.05%,但其中只有0.1%的磷能被植物所利用[7]。山西省耕地土壤严重缺磷,其速效磷含量低于5 mg/kg的耕地占总耕地面积的56.17%[8]。施入土壤的磷肥大部分会形成难溶性磷酸盐,很难被植物直接吸收利用,且化学磷肥的生产也会造成空气及水体污染[9]。解磷细菌是指能够将有机磷和难溶性无机磷转化为可溶性无机磷的细菌。解磷细菌施入土壤后会降低土壤的pH值[10],可以在植物根际形成供磷微区,从而改善植物磷素的供应,加强其它有益微生物的活动,促进植物对钾、 钙、 镁、 铁、 锌等营养元素的吸收[11]。因此,筛选高效磷细菌从而研制生物菌剂,有利于加快山西矿区复垦土壤生态恢复步伐,减少环境污染,提高作物产量。
1 材料与方法
1.1 样品与菌株
原始土样采自山西省长治、 孝义、 洪洞等地的复垦区土壤,主要取自生长期红薯、 玉米、 豆角、 苜蓿等作物根际以及撂荒地(连续几年不种作物自然恢复的土地)。对照菌株为巨大芽孢杆菌BacillusmegateriumAs1.223,由中国科学院微生物保藏中心提供,是解磷微生物肥料生产的常用菌种。
1.2 培养基
1.3 解磷菌的分离及筛选
1.3.2 复筛 通过液体发酵试验[12],以磷酸钙为磷源物质,以巨大芽孢杆菌(Bacillusmegaterium)As1.223为对照菌株,以不接种的培养基为空白对照,将磷细菌接种到无机磷液体培养基的三角瓶中,设置3个重复,置摇床上(150 r/min)保温30℃振荡培养5 d。下摇床后高速离心(5000 r/min)30 min,将上清液倾出待测。用酸度计测定上清液的pH值,采用电感耦合等离子体发射光谱法测定有效磷含量,按下式计算它们的解磷率:
X(%)=PV×10-3/W×103×100
式中:P—培养液有效磷含量(mg/L);X—解磷率(%);V—培养液体积(mL);W—发酵液中加入的磷酸钙质量(g)。
1.3.3 解磷矿粉和卵磷脂能力的测定 分别以磷矿粉和卵磷脂为磷源,测定复筛选出菌株的解磷率,方法同1.3.2。
1.3.4 磷酸酶活性的测定 取以卵磷脂为磷源培养5 d后的上清液,采用碱性及酸性磷酸酶试剂盒进行测定,试剂盒购自南京建成生物工程研究所,方法按试剂盒说明书进行。将100 mL发酵液在37℃下与基质作用15 min产生1 mg的酚定义为1个金氏单位(U)。
1.4 菌株生长试验
1.4.1 温度适应性 分别在4℃、 20℃、 28℃、 37℃、 60℃培养、 观察、 记录菌株的温度适应性,采用营养琼脂培养基,每个处理3次重复。
1.4.2 耐盐性 调整营养琼脂培养基的NaCl浓度分别为2%、 5%、 7%、 10%,每个处理3次重复,培养、 观察、 记录菌株耐盐性。
1.4.3 耐酸碱性 调整营养琼脂培养基酸度分别为pH 3、 pH 4、 pH 5、 pH 6、 pH 7、 pH 8、 pH 9、 pH 10、 pH 11、 pH 12,每个处理3次重复,培养、 观察、 记录菌株耐酸碱性。
1.5 菌种鉴定
1.5.1 菌落及菌体形态观察 将筛选出的菌株接种于营养琼脂培养基上,30℃培养48 h,观察菌落形状和颜色等特征,挑取菌体进行革兰氏染色,并将样品送至北京农业生物技术研究中心,采用扫描电镜观察细胞形态与大小。
1.5.2 16S rDNA序列测定与系统发育分析 16S rDNA基因扩增、 测序测定与系统学分析参考文献[16],引物为27f:5′-AGAGTTTGATCCTGGCTCAG-3′、 1492r:5′-TACGGTTACCTTGTTACGACTT-3′。基因在线比对采用EzTaxon数据库,系统学进化分析采用Mega4.0软件系统,邻近法构建系统发育树,Bootstrap值为1000。
1.5.3 脂肪酸组成分析 使用美国MIDI公司的Sherolock全自动细菌鉴定系统对菌株进行脂肪酸组成分析,菌株活化采用TSB培养基,由北京农业生物技术研究中心完成。
2 结果与分析
2.1 解磷菌筛选结果
2.1.1 初筛 利用无机磷及有机磷平板培养基,从不同的样品中筛选出19株解磷微生物。将得到的菌株再进行平板筛选,其中13株在以磷酸钙为磷源的无机磷培养基上形成透明圈,10株在有机磷培养基上形成透明圈,表明这些菌株可分泌一些物质,使菌落周围的磷酸钙或卵磷脂溶解,即这些菌株具有降解无机磷和有机磷的能力。D/d≥1.5的有7株(图1),这7株菌均具有溶解无机磷和有机磷的能力。在无机磷培养基上,编号为Y14的D/d值最大,达到3.28,而其他菌株的D/d值均小于1.90;在有机磷培养基上,D/d值最大的为Y11,其次为Y15和Y14,有机磷培养基的D/d值相差不大,均小于1.70。总体而言,菌株Y14在无机磷和有机磷培养基上的透明圈均较大(图2),初步判断为最佳解磷菌。
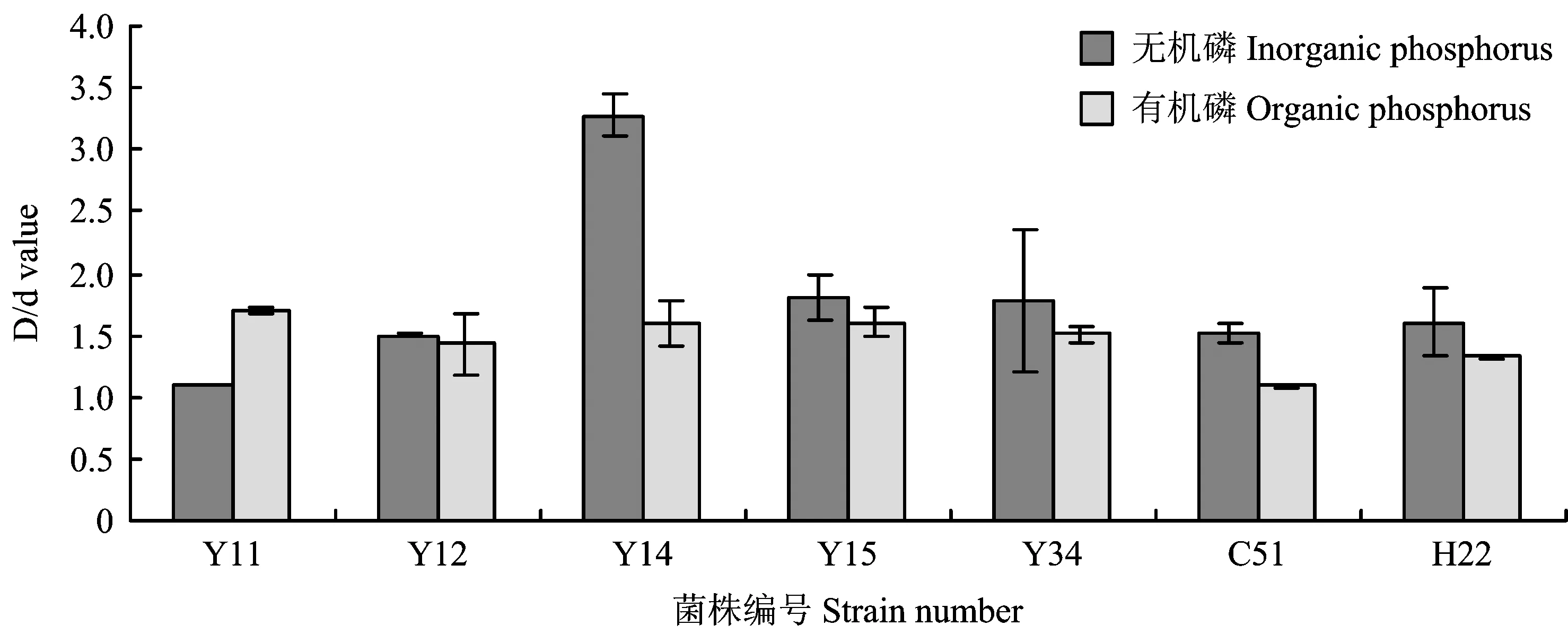
图1 解磷菌的D/d值Fig. 1 D/d value of the isolated strains

图2 菌株Y14的透明圈Fig. 2 The bacteria’s transparent turn of Y14
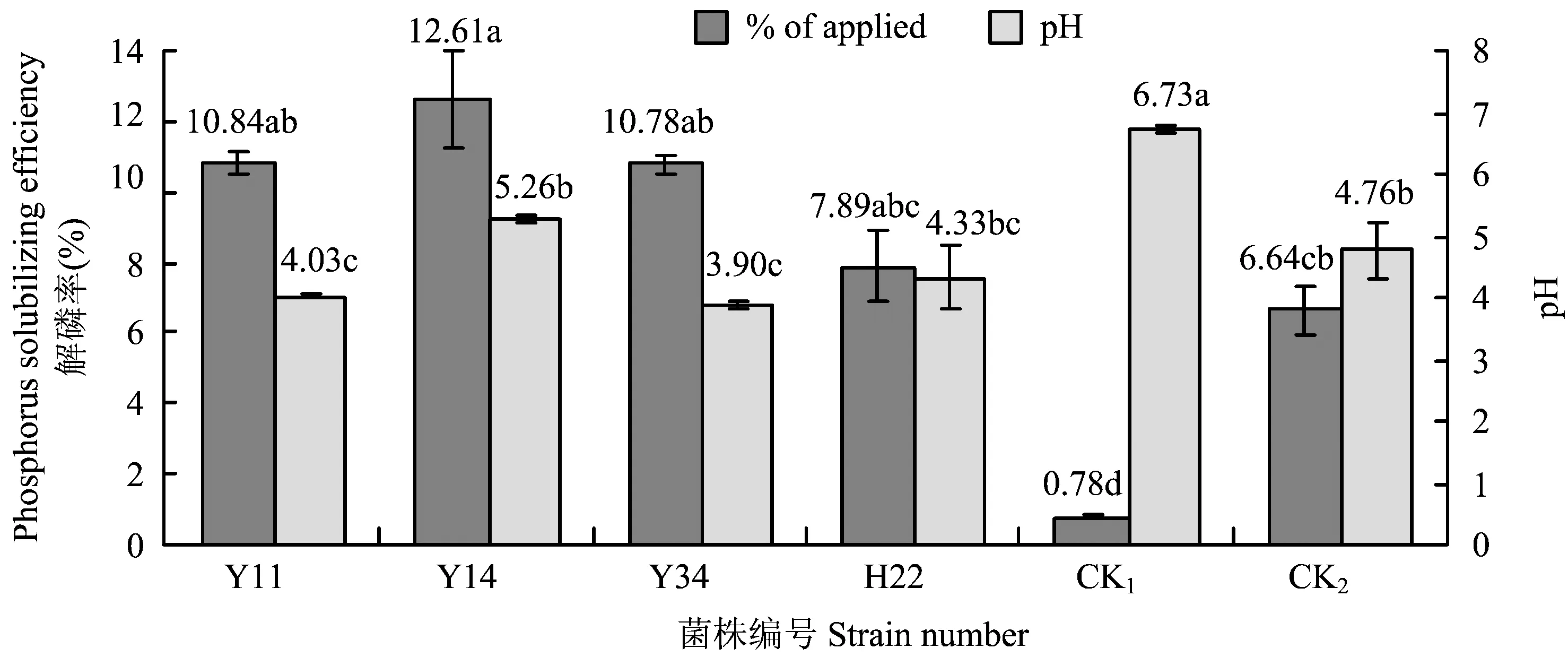
图3 解磷菌对磷酸钙的解磷率Fig. 3 Ca3(PO4)2 decomposition rates of the isolated strains
2.2 解磷菌的解磷能力
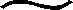
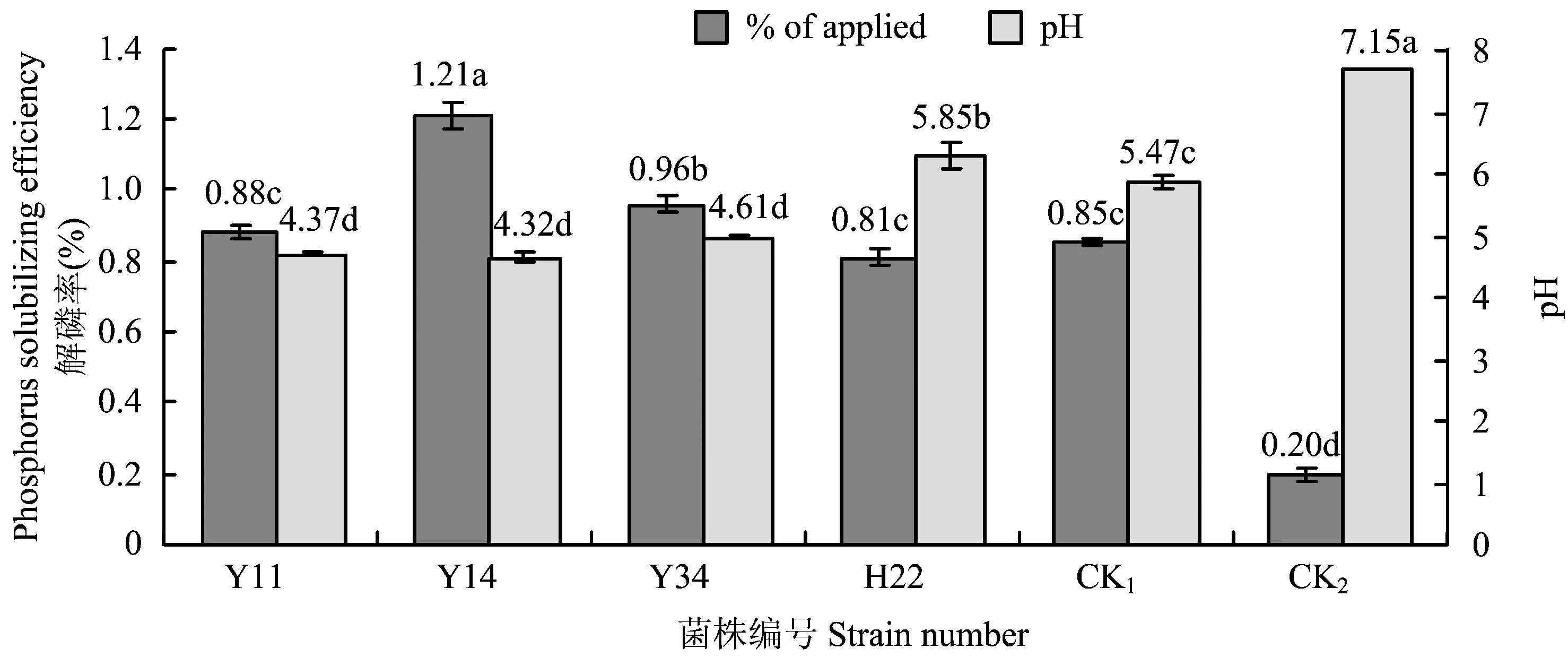
图4 解磷菌对磷矿粉的解磷率Fig. 4 Rock phosphate decomposition rates of the isolated strains
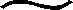
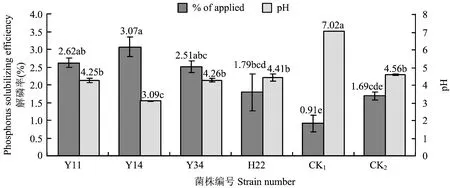
图5 解磷菌对卵磷脂的解磷率Fig. 5 Lecithin decomposition rates of the isolated strains

图6 解磷菌磷酸酶活性Fig. 6 Phosphatase activity of the isolated strains
综上所述,降解卵磷脂效果最好的菌株为Y14,其次是Y11和Y34。
卵磷脂是磷脂化合物的一种,可被微生物分泌的磷酸酶分解为甘油、 脂肪酸、 磷酸和胆碱。从图6可以看出,4株菌的酸性磷酸酶和碱性磷酸酶活性均高于不接菌的空白对照。4株菌的酸性磷酸酶活性均高于巨大芽孢杆菌As1.223,且与空白对照差异显著,酸性磷酸酶活性最高的菌株为Y14,达到28.4U/L;碱性磷酸酶活性除菌株H22外,其他3株菌均高于As1.223,菌株Y11、 Y14和Y34与空白对照相比差异达到了显著水平,菌株Y14的碱性磷酸酶活性为50.2 U/L,是4株菌中碱性磷酸酶活性最高的菌株,与As1.223差异显著。结合解磷率与磷酸酶活性的数据来看,菌株Y14的解磷率最高,其磷酸酶活性也最高,其次为Y11和Y34,解磷率和磷酸酶活性最低的菌株为H22,可以看出解磷率与磷酸酶活性具有一定的正相关关系,初步说明解磷细菌分泌胞外磷酸酶是其解有机磷的一个重要原因。
2.3 菌株生长试验

表1 解磷菌生长试验
2.4 菌种鉴定结果
2.4.2 16S rDNA基因序列分析 4株解磷菌的16S rDNA基因大小约1.5 kb,测序后,通过Contig Express软件拼接成全序列,采用EzTaxon数据库进行16S rDNA基因在线比对,并挑选数据库高同源性菌株作为参比菌进行16S rDNA系统学分析,图8与图9显示了它们的系统进化关系。比对结果显示菌株H22、 Y11、 Y34与假单胞菌属(Pseudomonassp.)有高度同源性,其中H22与PseudomonasvancouverensisATCC 700688T(AJ011507)[17]序列相似性最高,达到了99.36%,H22在系统发育树中也与ATCC 700688T聚在一起;Y11的最大相似菌株为PseudomonasmandeliiCIP 105273T(AF058286)[18],其序列相似性为99.14%,系统发育树中也与CIP 105273T聚在一起;Y34与菌株PseudomonasmandeliiCIP 105273T(AF058286)和PseudomonasfrederiksbergensisJAJ28T(AJ249382)[14]同源性最高,均达到了99.49%,而其在系统发育树中与Y11及CIP 105273T形成一个类群。菌株Y14与泛菌属(Pantoeasp.)中的Pantoeacalida1400/07T(GQ367478)[19]有99.79%的同源性,1400/07T是Y14在EzTaxon数据库中的最大相似菌株,在系统发育树中聚在一起。
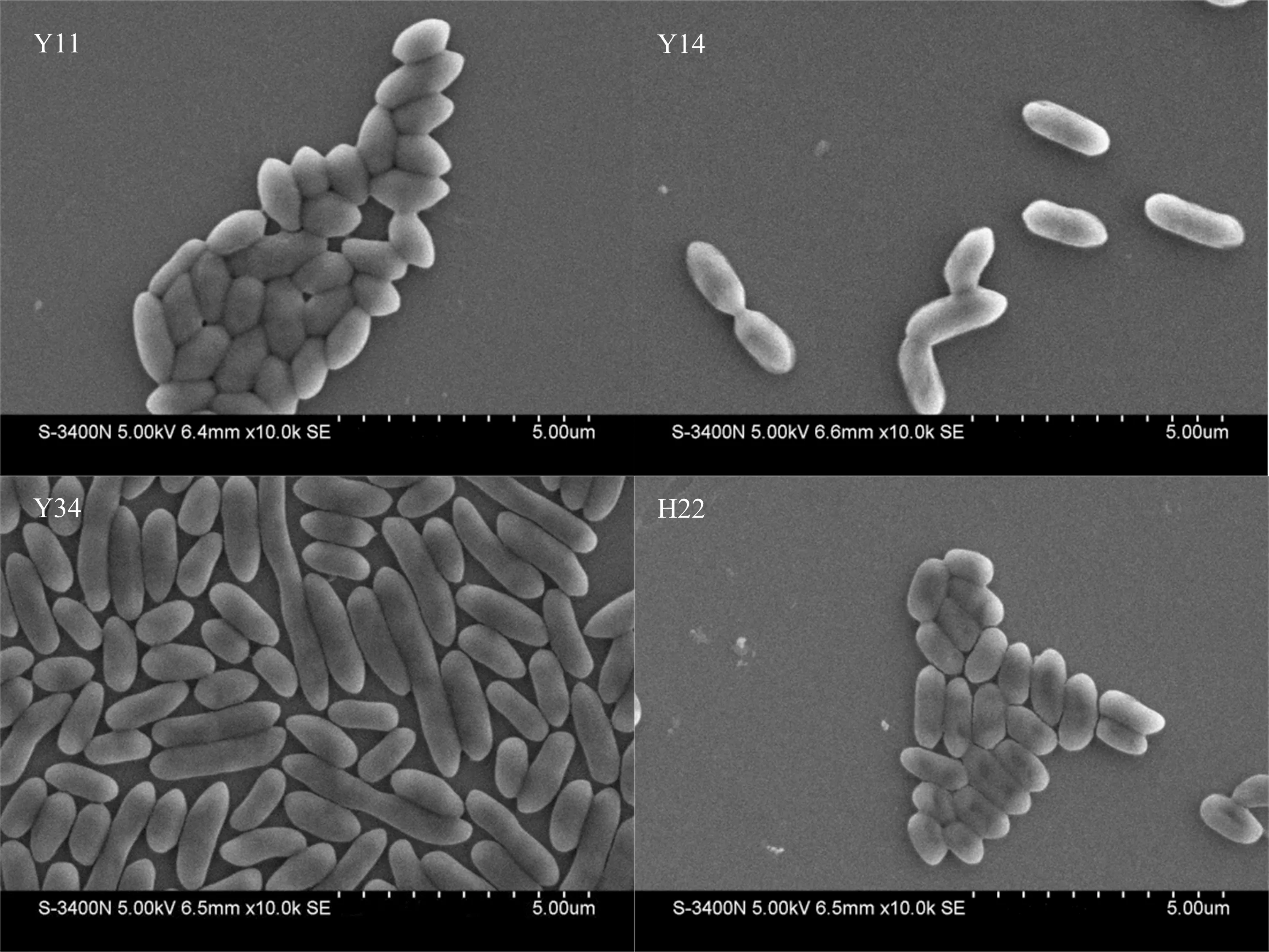
图7 扫描电镜下解磷菌的菌体形态(×10000)Fig. 7 Morphology of the isolated strains by scanning electron microscope(×10000)
2.4.3 脂肪酸组分分析 根据MIDI微生物鉴定系统分析结果,菌株Y11和Y34的细胞脂肪酸组成特征峰值大于1%的有C12 ∶0、 C16 ∶0、 C18 ∶1ω7c、 C10 ∶03-OH、 C12 ∶02-OH、 C12 ∶13-OH、 C12 ∶03-OH、 第三特征峰和C17 ∶0cyclo,与其16S rDNA相似菌株JF3835T和JAJ28T相一致。参比菌株JF3835T和JAJ28T的主要特征峰有C16 ∶0和第三特征峰,菌株Y34的细胞脂肪酸成分也集中在这两个特征峰上,分别占全细胞组分的22.1%和38.4%,菌株Y11的主要特征峰包括C16 ∶0和第三特征峰,分别为14.4%和14.8%。菌株H22的细胞脂肪酸主要成分是C16 ∶0、 C18 ∶1ω7c和第三特征峰,分别占脂肪酸总量的24.8%、 11.7%和37.5%,与ATCC 700688T和RW10T的主要特征峰相一致。菌株Y14与其16S rDNA相似菌株LMG 26277T和LMG 2603T细胞的脂肪酸组成(>1%)分布相一致,其中Y14细胞脂肪酸组成主要特征峰有C16 ∶0和C17 ∶0cyclo,分别占总量的32.5%和28.6%,参比菌株LMG 26277T的脂肪酸组成主要特征峰包括C16 ∶0,LMG 2603T的主要特征峰包括C16 ∶0和C17 ∶0cyclo(表2)。作为脂类和脂多糖的主要成分,脂肪酸主要分布在细胞膜上,不同的属、 种,甚至不同的菌株之间其脂肪酸碳链的长度、 双键的位置、 取代基团等都存在差异,据此原理进行的脂肪酸分析不仅可以进行大量菌株的比较分析,而且可以提供菌株的有关描述特征,广泛应用于细菌的分类鉴定中[20]。
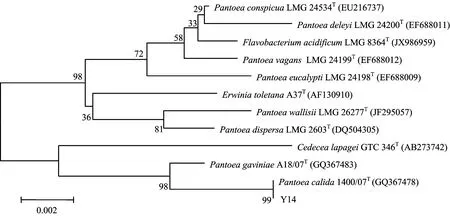
图9 菌株Y14最大相似性种系统发育树Fig. 9 Phylogenetic tree of max similar species representing the strain Y14

表2 解磷菌及近缘种脂肪酸的组成比较(%,>1%)
根据菌株的形态特征、 16S rDNA基因比对结果和细胞脂肪酸组分分析结果,参照《Bergey's Manual of Systematic Bacteriology》[25]和《常见细菌系统鉴定手册》[26],菌株H22、 Y11和Y34被初步鉴定为假单胞菌Pseudomonassp.,菌株Y14被初步鉴定为泛菌Pantoeasp.。
3 讨论

一般认为,磷细菌对有机磷的分解是通过分泌胞外酶而实现的。其中对磷素代谢起重要作用的是磷酸酶,其分酸性和碱性两种,产生酸性磷酸酶的生物有微生物、 原生动物和植物等,产生碱性磷酸酶的生物有微生物、 藻类、 蚯蚓等[32],磷酸酯酶对溶解无机难溶磷酸盐也有一定的效果。在以卵磷脂为磷源的液体发酵试验中磷酸酶活性与解磷率基本成正相关关系,这与他人研究结果相一致[33],培养液中有效磷含量最高的菌株为Y14,其磷酸酶活性也最高,碱性磷酸酶活性达到了50.2 U/L,高于钟传青等[32]报道的786 μmol/(L·h)(18.5 U/L)以及游银伟等[33]报道的20.0 U/L。

4 结论
参考文献:
[1] 白中科. 山西矿区土地复垦科学研究与试验示范十八年回顾[J]. 山西农业大学学报, 2004, 24(4):313-317.
Bai Z K. Scientific researches and experimentation of mined land reclamation of Shanxi during 1986-1994[J]. Journal of Shanxi Agricultural University, 2004, 24(4):313-317.
[2] 李建华, 郜春花, 卢朝东. 山西省矿区土地复垦的初步探讨[J]. 山西农业科学, 2008, 36(3):69-72.
Li J H, Gao C H, Lu C D. Preliminary approach to mining land reclamation in Shanxi[J]. Journal of Shanxi Agricultural Sciences, 2008, 36(3):69-72.
[3] 钱奎梅, 王丽萍, 李江. 矿区复垦土壤的微生物活性变化[J]. 生态与农村环境学报, 2011, 27(6):59-63.
Qian K M, Wang L P, Li J. Variation of microbial activity in reclaimed soil in mining area[J]. Journal of Ecology and Rural Environment, 2011, 27(6):59-63.
[4] 杜慧平, 刘利军, 闫双堆. 微生物对矿山复垦地土壤基质的改良作用[J]. 山西农业科学, 2011, 39(1):43-46.
Du H P, Liu L J, Yan S D. Improvement action of microorganism on the reclamation soil in mining area[J]. Journal of Shanxi Agricultural Sciences, 2011, 39(1):43-46.
[5] Schachtman D P, Reid R J, Ayling S M. Phosphate uptake by plants from soil to cell[J]. Plant Physiology, 1998, 116(2):447-453.
[6] Rodríguez H, Fraga R. Phosphate solubilizing bacteria and their role in plant growth promotion[J]. Biotechnology Advances, 1999, 17(4):319-339.
[7] Zhu F L, Qu L Y, Hong X G, Sun X Q. Isolation and characterization of a phosphate-solubilizing halophilic bacteriumKushneriasp. YCWA18 from Daqiao Saltern on the Coast of Yellow Sea of China[J]. Evidence-Based Complementary and Alternative Medicine, 2011.
[8] 卢朝东, 郜春花, 王岗, 等.B2和B67菌株筛选及应用效果研究[J]. 中国土壤与肥料, 2007, (5):56-59.
Lu C D, Gao C H, Wang Getal. Selection of B2 and B67 bacteria and their application[J]. Soil and Fertilizer Sciences in China, 2007, (5):56-59.
[9] Vassilev N, Vassileva M. Biotechnological solubilization of rock phosphate on media containing agro-industrial wastes[J]. Applied Microbiology and Biotechnology, 2003, 61(5-6):435-440.
[10] Abd-Alla M H. Phosphatases and the utilization of organic phosphorus byRhizobiumleguminosarumbiovarviceae[J]. Letters in Applied Microbiology, 1994, 18(5):294-296.
[11] 王亚艺, 李松龄, 蔡晓剑, 郝玉兰. 青海解磷菌菌株的分离筛选[J]. 河北农业科学, 2012, 16(2):62-64.
Wang Y Y, Li S L, Cai X J, Hao Y L. Isolation and screening of phosphorus solubilizing bacterial strains in Qinghai province[J]. Journal of Hebei Agricultural Sciences, 2012, 16(2):62-64.
[12] NY412-2000. 磷细菌肥料[S].
NY412-2000. Phosphate bacteria fertilizer[S].
[13] 沈萍, 范秀容, 李广武. 微生物学实验(第三版)[M]. 北京:高等教育出版社, 1999.
Shen P, Fan X R, Li G W. Microbiology experiment (3rdedition)[M]. Beijing:High Education Press, 1999.
[14] Andersen S M, Johnsen K, Sφrensen Jetal.Pseudomonasfrederiksbergensissp. nov., isolated from soil at a coal gasification site[J]. International Journal of Systematic and Evolutionary Microbiology, 2000, 50(6):1957-1964.
[15] 李振高, 骆永明, 滕应. 土壤与环境微生物研究法[M]. 北京:科学出版社, 2008. 107-108.
Li Z G, Luo Y M, Teng Y. The methods for studying soil and environmental microbiology[M]. Beijing:Science Press, 2008. 107-108.
[16] 孙建光, 张燕春, 徐晶, 胡海燕. 高效固氮芽孢杆菌选育及其生物学特性研究[J]. 中国农业科学, 2009, 42(6):2043-2051.
Sun J G, Zhang Y C, Xu J, Hu H Y. Isolation and biological characteristic investigation on efficient nitrogen-fixing bacilli[J]. Scientia Agricultura Sinica, 2009, 42(6):2043-2051.
[17] Mohn W W, Wilson A E, Bicho P, Moore E R. Physiological and phylogenetic diversity of bacteria growing on resin acids[J]. Systematic and Applied Microbiology, 1999, 22(1):68-78.
[18] Verhille S, Baida N, Dabboussi Fetal. Taxonomic study of bacteria isolated from natural mineral waters:proposal ofPseudomonasjesseniisp. nov. andPseudomonasmandeliisp. nov[J]. Systematic and Applied Microbiology, 1999, 22(1):45-58.
[19] Popp A, Cleenwerck I, Iversen Cetal.Pantoeagaviniaesp. nov. andPantoeacalidasp. nov., isolated from infant formula and an infant formula production environment[J]. International Journal of Systematic and Evolutionary Microbiology, 2010, 60(12):2786-2792.
[20] 黄翠丽, 王敏, 成晓杰, 等. 一株产共轭亚油酸瘤胃细菌StreptococcusinfantariusRB111的筛选与鉴定[J]. 微生物学通报, 2011, 38(1):78-84.
Huang C L, Wang M, Cheng X Jetal. Screening and identification of aStreptococcusinfantariusRB111 that produces conjugated linoleic acid[J]. Microbiology China, 2011, 38(1):78-84.
[21] Burr S E, Gobeli S, Kuhnert Petal.Pseudomonaschlororaphissubsp.pisciumsubsp. nov., isolated from freshwater fish[J]. International Journal of Systematic and Evolutionary Microbiology, 2010, 60(12):2753-2757.
[22] Cámara B, Strömpl C, Verbarg Setal.Pseudomonasreinekeisp. nov.,Pseudomonasmooreisp. nov. andPseudomonasmohniisp. nov., novel species capable of degrading chlorosalicylates or isopimaric acid[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(5):923-931.
[23] Brady C L, Cleenwerck I, van der Westhuizen Letal.Pantoearodasiisp. nov.,Pantoearwandensissp. nov. andPantoeawallisiisp. nov., isolated fromEucalyptus[J]. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(7):1457-1464.
[24] Gavini F, Mergaert J, Beji Aetal. Transfer ofEnterobacteragglomerans(Beijerinck 1888) Ewing and Fife 1972 toPantoeagen. nov. asPantoeaagglomeranscomb. nov. and description ofPantoeadispersasp. nov.[J]. International Journal of Systematic and Evolutionary Microbiology, 1989, 39(3):337-345.
[25] Holt J G. Bergey’s manual of systematic bacteriology (1stedition)[M]. Baltimore:Williams & Wilkins, 1984-1989.
[26] 东秀珠. 常见细菌系统鉴定手册[M]. 北京:科学出版社, 2001.
Dong X Z. Manual of systematic determinative bacteriology[M]. Beijing:Science Press, 2001.
[27] 朱培淼, 杨兴明, 徐阳春, 等. 高效解磷细菌的筛选及其对玉米苗期生长的促进作用[J]. 应用生态学报, 2007, 18(1):107-112.
Zhu P M, Yang X M, Xu Y Cetal. High effective phosphate-solubilizing bacteria:Their isolation and promoting effect on corn seedling growth[J]. Chinese Journal of Applied Ecology, 2007, 18(1):107-112.
[28] 贺梦醒, 高毅, 胡正雪, 等. 解磷菌株B25的筛选、 鉴定及其解磷能力[J]. 应用生态学报, 2012, 23(1):235-239.
He M X, Gao Y, Hu Z Xetal. Screening, identification, and phosphate-solubilizing capability of phosphate-solubilizing bacterial strain B25[J]. Chinese Journal of Applied Ecology, 2012, 23(1):235-239.
[29] 郝晶, 洪坚平, 刘冰, 张健. 石灰性土壤中高效解磷细菌菌株的分离、 筛选及组合[J]. 应用与环境生物学报, 2006,12(3):404-408.
Hao J, Hong J P, Liu B, Zhang J. Isolation, screening and combination of highly-effective phosphorus solubilizing bacterial strains in calcareous soil[J]. Chinese Journal of Applied and Environmental Biology, 2006, 12(3):404-408.
[30] Illmer P, Schinner F. Solubilization of inorganic phosphate by microorganisms isolated from forest soils[J]. Soil Biology and Biochemistry, 1992, 24(4):389-395.
[31] 吉蓉. 土壤解磷微生物及其解磷机制综述[J]. 甘肃农业科技, 2013, (8):42-44.
Ji R. Research summary on phosphate dissolution of phosphate solubilizing microorganisms[J]. Gansu Agricultural Science and Technology, 2013, (8):42-44.
[32] 钟传青, 黄为一. P17菌株产生磷酸酶的影响因素及其定域研究[J]. 山东建筑大学学报, 2012, 27(1):32-35.
Zhong C Q, Huang W Y. Studies on the factors and domain influencing the activities of the phosphatase produced by P17 strain[J]. Journal of Shandong Architecture University, 2012, 27(1):32-35.
[33] 游银伟, 王梅, 江丽华, 等. 高效解磷菌BacillussubtilisP-1的N离子束诱变育种[J]. 西南农业学报, 2009, (4):1020-1022.
You Y W, Wang M, Jiang L Hetal. Mutant breeding of high effective phosphate-solubilizing bacteriaBacillussubtilisP-1 by nitrogen ionic beam[J]. Southwest China Journal of Agricultural Sciences, 2009, (4):1020-1022.
[34] Sundara Raoand W V B, Sinha M K. Phosphate dissolving microorganisms in the soil and rhizosphere[J]. Indian Journal of Agricultural Sciences, 1963, 33(4):272-278.
[35] Elliott J M, Mathre D E, Sands D C. Identification and characterization of rhizosphere-competent bacteria of wheat[J]. Applied and Environmental Microbiology, 1987, 53(12):2793-2799.
[36] De Freitas J R, Banerjee M R, Germida J J. Phosphate-solubillizing rhizobactera enhance the growth and yield but not phosphorus uptake of canola(BrassicanapusL.)[J]. Biology and Fertility of Soils, 1997, 24(4):358-364.
[37] Molla M A Z, Chowdhury A A. Microbial mineralization of organic phosphate in soil[J]. Plant and Soil, 1984, 78(3):393-399.
[38] Vazquez P, Holguin G, Puente M Eetal. Phosphate-solubilizing microorganisms associated with the rhizosphere of mangroves in a semiarid coastal lagoon[J]. Biology and Fertility of Soils, 2000, 30(5-6):460-468.
[39] Altomare C, Norvell W A, Björkman T, Harmand G E. Solubilization of phosphates and micronutrients by the plant-growth-promoting and biocontrol fungusTrichodermaharzianumRifai 1295-22[J]. Applied and Environmental Microbiology, 1999, 65(7):2926-2933.
[40] Chabot R, Antoun H, Cescas M P. Growth promotion of maize and lettuce by phosphate-solubilizingRhizobiumleguminosarumbiovar.Phaseoli[J]. Plant and Soil, 1996, 184(2):311-321.
[41] Jha A, Sharma D, Saxena J. Effect of single and dual phosphate-solubilizing bacterial strain inoculations on overall growth of mung bean plants[J]. Archives of Agronomy and Soil Science, 2012, 58(9):967-981.
[42] Igual J M, Valerde A, Cervantes Eetal. Phosphate-solubilizing bacteria as inoculants for agriculture:Use of updated molecular techniques in their study[J]. Agronomy for Sustainable Development, 2001, 21:561-568.
[43] Illmer P, Schinner F. Solubilization of inorganic calcium phosphates-solubilization mechanisms[J]. Soil Biology and Biochemistry, 1995, 27:257-263.

