两种技术在广东地区布鲁氏菌菌株分型中的应用与评价
陈经雕,杨杏芬,柯昌文,梁文佳,柯碧霞,刘美真,谭海玲,李柏生,张万里
布鲁氏菌(Brucella)是一种革兰阴性、兼性厌氧的细胞内寄生菌,可引起严重危害人民健康和畜牧业发展的人兽共患传染病-布鲁氏菌病(Brucellosis)(简称布病)的广泛流行。目前世界上 170 多个国家和地区有此病的存在和流行,已成为全球性的公共卫生问题。我国自1905年首次报告该病以来,几乎每个省(市、区)的人、畜中都有不同程度的存在和流行。目前,对布鲁氏菌的分子分型方法有聚合酶链反应-限制性片段长度多态性(PCR-RELP)[1]、AMOS-PCR[2]及其衍生技术、单核苷酸多态性研究(SNP)[3]、脉冲场凝胶电泳图谱分型[4](PFGE)、多位点序列分型[5](MLST)和多位点可变数目串联重复序列分型[6](MLVA)等基因分型技术,但这些分型方法对布鲁氏菌的分型能力的适用性、优越性方面,尚少见系统的评价研究。本研究通过对广东地区不同年代、地域的、不同种型的60株布鲁氏菌地方株和3株标准株(16M、544A、1330S)应用PFGE 和 MLVA两种技术进行了分子分型研究,并对两种技术在布鲁氏菌的分型能力和潜在价值进行比较和评价,现将情况报告如下。
1 材料与方法
1.1标本来源 1965-2010年广东地区各级农业兽医机构的动物或卫生医疗机构的布病病人分离株60株,其中上世纪60年代6株、80年代2株、其余52株为2005-2010年收集;地区分布为广州35株、深圳14株、珠海4株、中山、佛山、东莞各2株、江门1株;标准菌株16M、544A、1330S由中国疾病预防控制中心传染病预防控制所布病室惠赠。
1.2主要试剂和仪器 鉴定血清、噬菌体均由中国疾病预防控制中心传染病预防控制所布病室提供;引物由上海生工公司合成,Premix反应体系(TaKaRa)、限制性内切酶XhoI(Promega)、蛋白酶K(Merck公司)、Seaken Gold Aga-rose(Cam Brex)、Tris-EDTA和SDS,所有试剂均在有效期内使用。PCR仪(Eppendorf)、脉冲场凝胶电泳仪(CHEF Mapper)、GEL 2000凝胶成像系统(BIO-RAD)和毛细管电泳仪(QIAxcel Advanced)。
1.3菌株的复核鉴定 收集全省各地农业兽医机构或医疗机构初步鉴定为布鲁氏菌的动物或临床病人菌株到省疾控中心作进一步的复核鉴定,并由当地疾控中心收集病人临床及流行病学资料。根据菌落形态、染色、生长特性、诊断血清凝集试验对上送菌株进行复核鉴定为布鲁氏菌,再采用噬菌体裂解试验、染料抑菌试验(硫堇、碱性复红)、H2S产生试验、单项特异性血清凝集试验进行种、型鉴定。具体方法依据《布鲁氏菌病防治手册》[7]进行操作。
1.4脉冲场凝胶电泳(PFGE)技术 参照文献[4]并进行优化,主要的步骤有:取新鲜培养菌落,制成OD值4.8~5.2的菌悬液,包埋菌体细胞,用50U的XhoI限制性内切酶进行酶切,0.5×TBE液2 300 mL、14 ℃、120°、6 V/cm条件下电泳,脉冲时间2~25 s,电泳时间19 h,用H9812作分子量标准对照。电泳结束后用EB染色30 min,三蒸水脱色3次,每次20 min,即可读胶,用Bio Numerics(Version 5.0)软件处理图谱,选择Dice相似性系数和非加权的几何平均数(UPGMA)进行聚类分析。
1.5多位点可变数目串联重复序列分型(MLVA)技术[8]16个位点的引物参照文献[9-10],见表1。其中panel 1包括bruce 06、08、 11、12、42、43、45和55共8个位点;panel 2A 包括bruce18、19和21共3个位点,panel 2B包括 bruce04、07、09、16和30共5个位点。挑取布氏斜面培养24~48 h的菌落,提取基因组DNA。采用25 μL反应体系,其中Premix 12.5 μL、上、下游引物(10 μmol/L)各1.0 μL、模板1.0 μL、去离子水9.5 μL。反应条件:预变性96 ℃、3 min,变性96 ℃、30 s,退火60 ℃、30 s,延伸72 ℃、30 s,35个循环,最后72 ℃、5 min。每次扩增均分别设阳性、阴性对照。panel 1位点采用琼脂糖凝胶电泳确认PCR产物大小。panel 2位点采用毛细管电泳读取PCR产物大小。根据各菌株各位点的PCR扩增产物的大小(bp),通过标准菌株1330S计算得出各位点的重复单元数。得出每株菌株的16位数字编码(各数字相对应于各位点的重复单元数),即MLVA等位基因类型。使用Bio Numerics(Version 5.0)软件处理图谱,选择 Dice 相似性系数和非加权的几何平均数(UPGMA)进行聚类分析。
1.6分型能力的比较和分析 可用辛普森(Simpson′s diversity index,DI)指数作为 PFGE 和 MLVA 两种分子分型方法的指标进行分型、分辨能力比较、评价、其公式[11]:
其中N表示菌株总数 ,nj为某一型别的菌株数。
2 结 果
2.1菌株鉴定结果 60 株地方株经单项特异性血清凝集试验、噬菌体裂解试验、染料抑菌试验(硫堇、碱性复红)和 H2S 产生试验检测,并将检测结果与FAO/WHO 1985 年公布的布鲁氏菌分类表[7]比较。所有菌株中,除了 1965 年的 6 株(4 株羊种、2 株猪种)菌株因变异不能分型外,其余菌株均能鉴定到型的水平,其中羊种 3 型 54 株、猪种3型3株、羊种1型3株。
2.2PFGE分型结果 菌株经XhoI酶切后用Bio Numerics软件分析,结果显示:63株菌株间相似性系数介于72.1%~100%,在94.4%相似性系数时可将菌株分为10个克隆复合群17种PFGE型别,可区分羊、猪、牛3个种,但不能有效区分同种异型;在100.0%相似性系数时可将菌株分为14个克隆复合群共29种PFGE型别。其中15株(23.8%)为单态群,另外48株(76.2%)分属于14种克隆复合群,各群分别含2~8 株菌。见图 1。

图1 63株布鲁氏菌 PFGE 分型聚类图
Fig.1ClustermapforPFGEtypein63strainsofBrucella
2.3MLVA 分型结果 经Bio Numerics软件分析聚类后,63 株菌株间相似性系数介于 16.9%~100%,在 52.3% 相似性系数时可将菌株分为 3 个克隆复合群 5 种 MLVA 型别,也可区分羊、猪、牛3个种的菌株,也不能有效区分同种异型;在 100.0% 相似性系数时可将菌株分为 8 个克隆复合群共 47 种 MLVA 型别,表现出较大的遗传多样性。其中 39 株(61.9%)为单态群,另外 24 株(38.1%)分属于 8 个克隆复合群,各群分别含 2~5 株菌。结果见图 2。
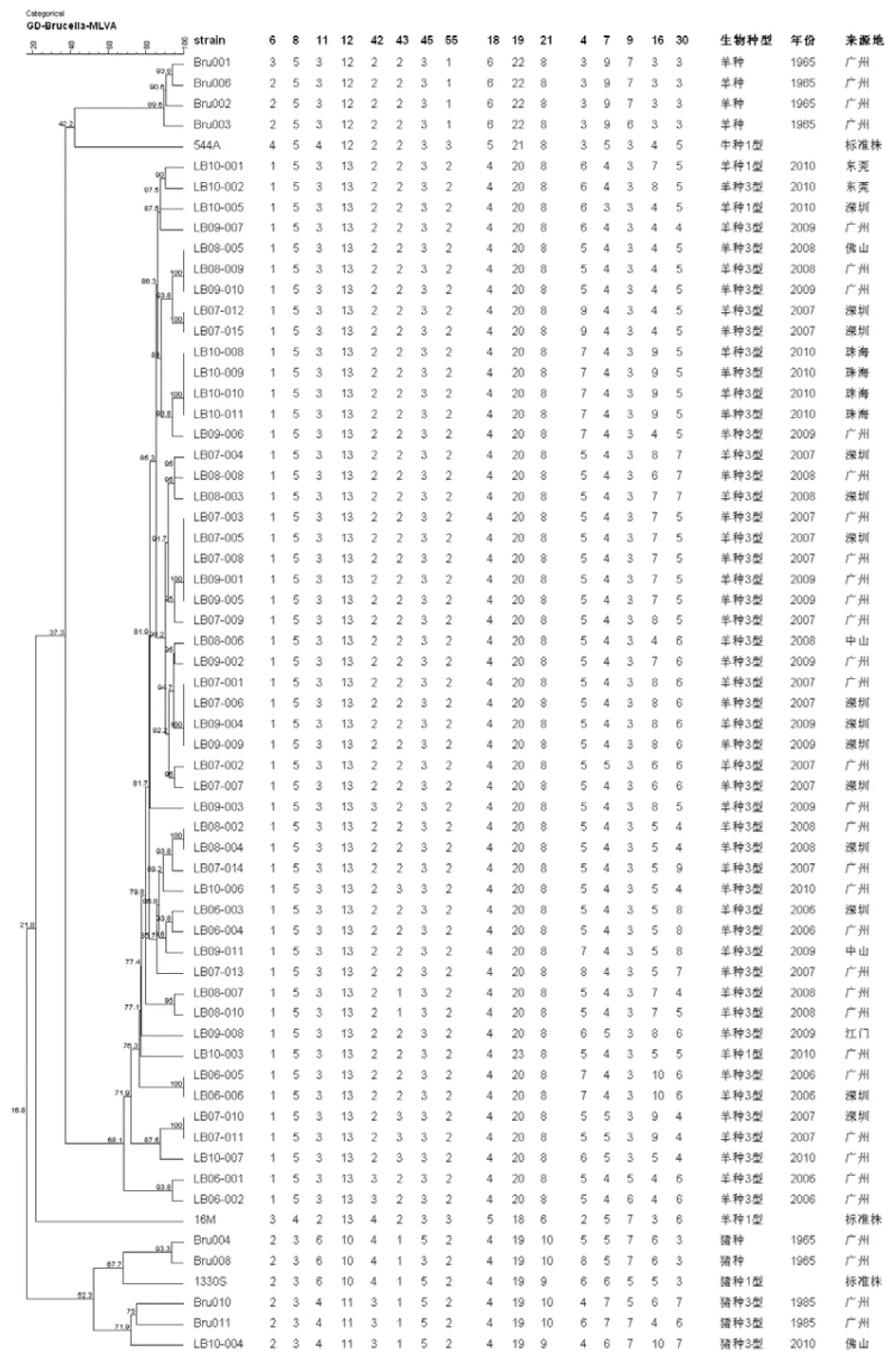
图2 63株布鲁氏菌MLVA分型聚类图
Fig.2ClustermapforMLVAtypein63strainsofBrucella
2.4PFGE和MLVA分型方法的比较和相关性 用DI值作为分型系统分辨力的指标,PFGE分型在100.0%相似性系数时可将菌株分为14个克隆复合群和15个单态群,共29种型别,DI为0.957 5;MLVA分型在100.0%相似性系数时可将菌株分为8个克隆复合群和39个单态群,共47种型别,DI为0.985 2。结合菌株的背景资料分析,存在同一暴发事件(2010年珠海的4株菌株)的菌株有相同的MLVA型别和相近的PFGE型别;也存在部分菌株有相同的PFGE型别,却分为不同MLVA型别或相同的MLVA型别,却分为不同PFGE型别的情况;还存在相同或相近年份的同种菌株的PFGE和MLVA型别都有较高的同源性特点。
3 讨 论
自1887年Bruce分离到羊种布鲁氏菌以来,该菌的分种分型一直存在争议。鉴于传统的分型方法周期较长且操作繁琐以及生物安全的风险,且近年来,国内出现了10%~30%的非典型菌株使用传统方法无法分型的情况[12],这些菌株应用传统的分型方法不能在分类表上找到合适的位置,说明传统的分型方法存在一定的缺陷,更重要的是严重影响对布病疫区分类的判定,从而不能采取合理有效的预防控制措施。
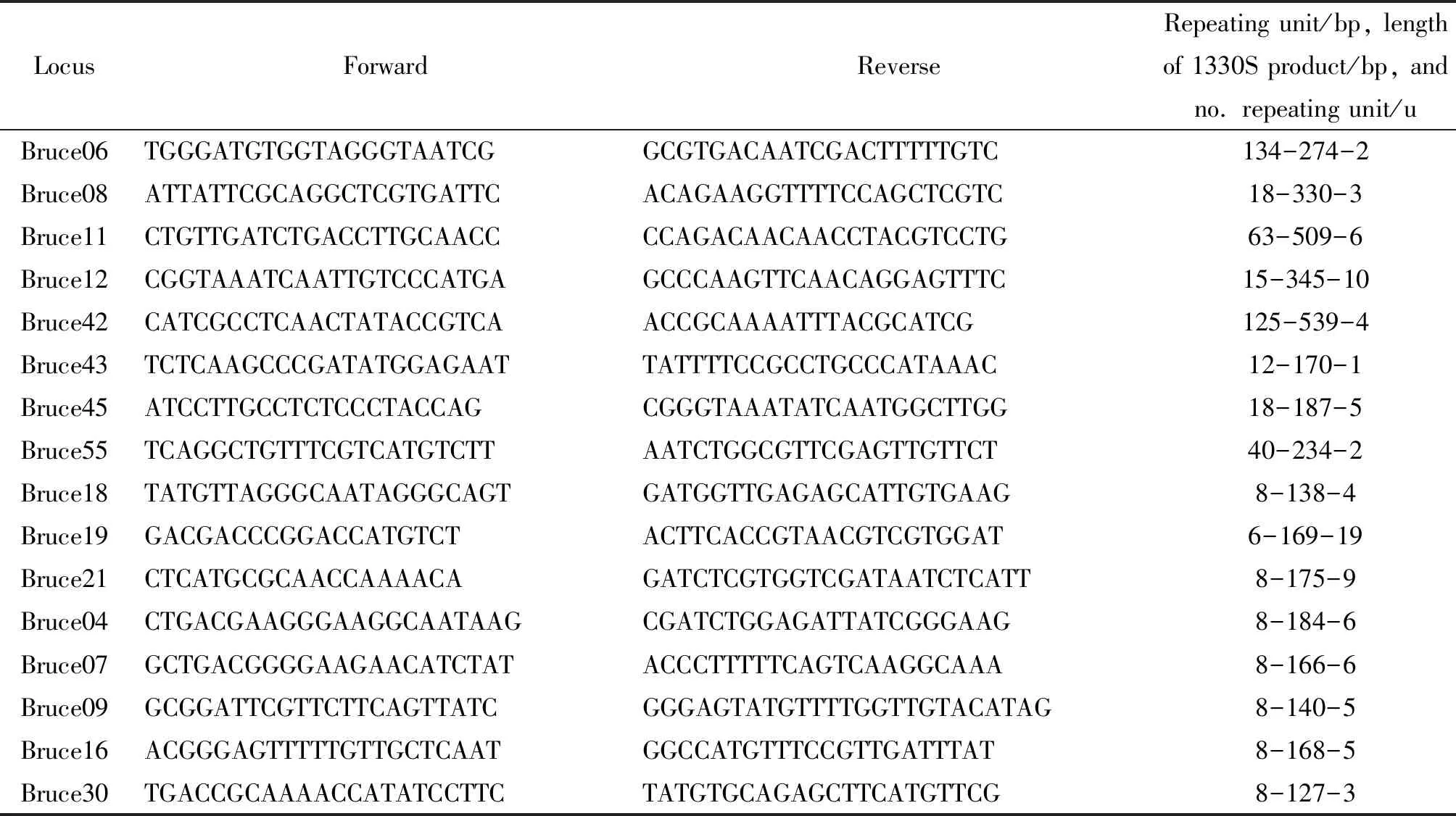
表1 MLVA-16引物
随着传统分型方法在实际工作中的受限和分子生物学基因分型技术的迅速发展,从基因水平对布鲁氏菌菌株进行研究的方法越来越多[1-6]。基因组研究显示布鲁氏菌属的羊、猪、牛种等大多数种均来源于同一克隆,种间同源性极高,高频率内切酶在鉴定分型方面存在局限性;又因其DNA发生重排,如插入、丢失,导致酶切位点的移动,或酶切位点发生点突变,但发现各种间都有保守的独特指纹谱带[7],因此有学者采用稀有位点内切酶对染色体进行消化[13]。根据酶切片段电泳图谱的不同,揭示基因组间差异,可以将布鲁氏菌在种的水平上区别开,因而引入脉冲场凝胶电泳(PFGE)技术作为传统生物分型的补充手段,对布鲁氏菌属进行分子分型。PFGE是用稀有位点的限制性核酸内切酶对细菌的染色体DNA进行消化酶切,得到较大的片段[14],然后在交变脉冲电场的作用下进行电泳,可将大小各异的DNA片段分开。由于该法使用细菌的原位酶切可反映整个细菌的基因情况,而不是某个片段,因而可显示基因组的微小变化,如酶切位点的突变、基因序列的插入或删除造成的条带的变大、变小或消失。因此,该方法可用于整个染色体基因组分析,对了解细菌的遗传特征有着重要意义[15]。作为病原菌分子分型的“金标准”,已广泛用于大肠埃希菌、沙门菌、金黄色葡萄球菌和布鲁氏菌等的分型研究[16]。Jensen等[17]应用PFGE方法对布鲁氏菌DNA核酸限制内切酶的图谱进行了分析鉴定,证实RB51菌株与其他牛种布鲁氏菌菌株不同。Ridler等[18]用限制性内切酶SwaⅠ消化12株绵羊附睾种布鲁氏菌后做脉冲场凝胶电泳,发现绵羊附睾种存在变种。崔步云等[19]对布鲁氏菌的19株标准菌株进行多次试验分析,证明PFGE应用于布鲁氏菌的可重复性和特异性较好。
研究也发现布鲁氏菌属的基因组存在大量重复序列,扩增细菌基因组中广泛分布短重复序列间片段,在各型别中拷贝数有所不同,从而对布鲁氏菌属进行分型[11]。MLVA是一种基于PCR技术的基因分型方法,该法根据散在于菌株基因组中不同独立位点可变数目串联重复序列(VNTR)的拷贝数多少来进行基因分型[20]。对研究病原菌的暴发感染和溯源起到重要作用,为确定菌株之间的亲缘关系提供了可靠的技术手段[21]。因具有操作简便、快速稳定、分辨率高和可重复性强[8]等优点,可广泛用于一些具有遗传同源性的病原菌如炭疽杆菌[22-23]、鼠疫耶尔森菌[24-25]、金黄色葡萄球菌[26]、结核菌属[27-28]和布鲁氏菌[8,29]的分型研究。该技术用于研究布鲁氏菌的基因分型,不仅可以用于布病暴发流行的传染源追溯和流行趋势的分析,还可用于探索国内外、同一地区的布病各菌株间的相关性、地理分布及传播过程等分子流行病学的研究。Bricker等[30]对美国97份畜牧业分离菌株和实验室6株牛种布鲁氏菌进行分析,结果显示分型的重复性很好,8个位点的等位基因多样性和频率都有很大差别;Le Fléche等[8]采用MLVA-15将来自不同地区的257株布鲁氏菌分为204个基因型,结果表明该法的分析结果与表型鉴定结果一致,而且将某地区的分离菌株与世界各国不同阶段的分离菌株进行遗传进化分析,可示踪布鲁氏菌在某一地区的流行趋势,查明该地区布鲁氏菌流行株传入途径。
本次研究利用PFGE、MLVA技术分析了不同年份、地域的不同种型布鲁氏菌的地方株和标准株,PFGE聚类分析可见:63株菌株间相似性系数介于72.1%~100%,在94.4%相似性系数时可将菌株分为10个克隆复合群17种PFGE型别;在100.0%相似性系数时可将菌株分为14个克隆复合群共29种PFGE型别。MLVA聚类分析可见:63株菌株间相似性系数介于16.9%~100%,在52.3%相似性系数时可将菌株分为3个克隆复合群5种MLVA型别;在100.0%相似性系数时可将菌株分为8个克隆复合群共47种MLVA型别,表现出较大的遗传多样性。两种方法聚类显示均可以在一定的相似系数从种的水平区分羊、猪、牛种的布鲁氏菌,但在种的基础上不能有效区分同种异型,该结论与骆利敏等的研究结果一致[4]。
本次研究的菌株病例个案流调资料显示除了珠海的4株菌为同一暴发事件所收集外,其它菌株均来自散发病例,部分病例都有加工、食用过羊胎盘或从事羊、猪、牛养殖、屠宰及售卖等接触史,但具体的羊、猪、牛来源调查由于涉及跨部门的协调问题局限了调查的深入进行,故未能深入追踪,掌握羊、牛的具体来源。但两种方法的聚类均提示多地的散发菌株呈现100%同源,提示这些菌株分别来自同一克隆,这可能由于来自同一疫区的染疫牲畜进入广东后分散到各地市再感染人所致,从而表现为各地表面上的散发。
聚类分析还显示:两种方法中的1965年的羊种与近几年(2005-2010年)的羊种同源性只有75.5%(PFGE)和37.3%(MLVA),均存在较大变异,各自独立成簇,表现出年度之间的差异,但因1965年的菌株发生变异不能确定其型别,但聚类看出1965年的羊种与近几年的羊种菌株分别来自不同的克隆;而牛种菌544A标准株与1965年的羊种菌反而有较高的相似性:PFGE(94.4%)和MLVA(42.2%),特别是PFGE方法显示出非常接近的遗传背景。
本研究选择了羊、猪、牛3个种的不同型别的63株布鲁氏菌菌株,结合菌株背景资料将PFGE(XhoI)与MLVA 基因分型方法同时进行分辨能力的比较。当辛普森指数DI值大于0.9即可认为该分型方法可信和具有较好的分辨能力[10]。结果显示两种分子分型方法都具有良好的分辨能力,但MLVA分辨力稍高于PFGE。PFGE 能把相同来源的菌株聚类成簇,MLVA把菌株从亲缘关系上细分,MLVA方法在分析菌株的亲缘进化关系方面具有一定的优势。PFGE、MLVA基因分型数据可在各实验室之间传输,可得到更多关于致病性基因和亲缘进化方面的研究资料,在建立全球数据库实现资源长期共享中具有较大的潜力。
致谢: 中国疾病预防控制中心传染病预防控制所布病室的全体专家对相关菌株的鉴定分析提供了帮助,谨致诚挚感谢。
参考文献:
[1]Al Dahouk S, Tomaso H, Prenger-Berninghoff E, et al. Identification ofBrucellaspecies and biotypes using polymerase chain reaction-restriction fragment length polymorphism (PCR-RFLP)[J]. Crit Rev Microbiol, 2005, 31: 191-196.DOI: 10.1080/10408410500304041
[2]Jiang H, Cui BY, Zhao HY, et al. Use of AMOS-PCR assay for the species identification ofBrucella[J].Chin J Zoonoses, 2009, 25(2): 107-109. (in Chinese)
姜海,崔步云,赵鸿雁,等.AMOS-PCR 对布鲁氏菌种型鉴定的应用[J].中国人兽共患病学报,2009,25(2):107-109.
[3]Marianelli C, Ciuchini F, Tarantino M, et al. Molecular characterization of the rpoB gene inBrucellaspecies: new potential molecular makers for genotyping[J]. Microbes Infect, 2006, 8(3): 860-865.
[4]Luo LM, Cui BY, Zhao HY, et al. Analysis ofBrucellareference strain s with pulsed field gel electrophoresis[J]. Lab Med, 2006, 21(5): 509-512. (in Chinese)
骆利敏,崔步云,赵鸿雁,等.脉冲场电泳分析布鲁菌标准菌株[J].检验医学,2006,21(5):509-512.
[5]Hanage WP, Feil EJ, Brueggemann AB, et al. Multilocus sequence typing: strain characterization, population biology, and patterns of evolutionary descent[M]. In Persing DH, Tenover FC, Versalovic J, et al (Eds). Molecular Microbiology: Diagnostic Principles and Practice. Washington DC: American Society Press, 2004: 235-243.
[6]Jiang H, Fan MG, Chen JD, et al. MLVA genotyping of Chinese humanBrucellamelitensisbiovar 1, 2 and 3 isolates[J]. BMC Microbiol, 2011, 11: 256-262. DOI: 10.1186/1471-2180-11-256
[7]Disease Prevention and Control Bureau, the Ministry of Health of People’s Republic China. Brucellosis prevention manual[M]. Beijing: People's Medical Publishing House, 2008. (in Chinese)
卫生部疾病预防控制局.布鲁氏菌病防治手册[M].人民卫生出版社 ,2008.
[8]Yang J, Zhao SL, Cui BY, et al. Establishment of standard operation procedures for multiple locus variable number tandem repeats analysis on typing ofBrucella[J]. Dis Surveill, 2012, 27(2): 137-140. (in Chinese)
杨杰,赵素莲,崔步云,等.多位点可变数目串联重复序列分型方法检测布鲁氏菌分型标准化操作方法的建立和应用[J].疾病监测,2012,27(2):137-140.
[9]Le Fleche P, Jacques I, Grayon M, et al. Evaluation and selection of tandem repeat loci for aBrucellaMLVA typing assay[J]. BMC Microbiol, 2006, 6: 9-11. DOI: 10.1186/1471-2180-6-9
[10]Al Dahouk S, Fleche PL, Nockler K, et al. Evaluation ofBrucellaMLVA typing for human brucellosis[J]. J Microbiol Methods, 2007, 69(1): 137-145. DOI: 10.1016/j.mimet.2006.12.015
[11]Hunter PR, Gaston MA. Numerical index of the discriminatory ability of typing systems: an application of Simpson′s index of diversity[J]. J Clinl Microbiol, 1988, 26(11): 2465-2466.
[12]Cui BY, Yin JM, Li LY, et al. Typing ofBrucellaisolates by repetitive element sequence-based polymerase chain reaction[J]. Dis Surveill, 2005, 20(8): 397-400. (in Chinese)
崔步云,尹继明,李兰玉,等.布鲁氏菌的 Rep-PCR 分型研究[J].疾病监测,2005,20(8):397-400.
[13]Shang DQ, Li LY. Study on molecular biology ofBrucellabacteria[J]. Chin J Ctrl Endem Dis, 2005, 20(5): 272-276. (in Chinese)
尚德秋,李兰玉.布氏菌分子生物学研究现况[J].中国地方病防治杂志,2005,20(5):272-276.
[14]Schwartz DC, Cantor CR. Separation of yeast chromosome-sized DNAs by pulsed field gradient gel electrophoresis[J]. Cell, 1984, 37(1): 67-75.
[15]Annick AS, Gisele B, Michel R, et al. DNA polymorphismin strains of the genusBrucella[J].J Bacteriol,1988,10: 4603-4607.
[16]Gerner-Smidt P, Hise K, Kincaid J, et al. PulseNet USA: a five-year update[J]. Foodborne Pathog Dis, 2006, 3(1): 9-19.
[17]Jensen AE, Cheville NF, Ewalt DR, et al. Application of pulsed-field gel electrophoresis for differentiation of vaccine strain RB51 from field isolates ofBrucellaabortusfrom cattle, bison, and elk[J]. American J Vet Res, 1995, 56(3): 308-312.
[18]Ridler AL, Leyland MJ, Fenwick SG, et al. Demonstration of polymorphism amongBrucellabovisfield isolates by pulsed-field gel electrophoresis[J]. Vet Microbiol, 2005, 108(1-2): 69-74. DOI: 10.1016/j.vetmic.2005.02.004
[19]Cui BY. Typing ofBrucellapulse field gel electrophoresis[D]. Taiyuan: Shanxi Medical University, 2006. (in Chinese)
崔步云.布鲁菌脉冲场凝胶电泳分型研究[D].太原:山西医科大学, 2006.
[20]Her M, Kang SI, Kim JY, et al. A genetic comparison ofBrucellaabortusisolates from animal sand human by using the MLVA assay[J]. J Microbiol Biotechnol, 2010, 20(12): 1750-1755. DOI: 10.4014/jmb.1005.05039
[21]Lukinmaa S, Takkunen E, Siitonen A. Molecular epidemiology ofClostridiumperfringensrelated to food borne outbreaks of disease in Finland from 1984 to 1999[J]. Appl Environ Microbiol, 2002, 68(8): 3744-3749.
[22]Keim P, Price LB, Klevytska AM, et al. Multiple-locus variable-number tandem repeat analysis reveals genetic relationships withinBacillusanthracis[J]. J Bacteriol, 2000, 182(10): 2928-2936.
[23]Zuo TT, Duan Q. MLVA and SNP analysis in the application ofBacillusanthracisgenotyping[J]. J Mil Med Sci Acad PLA, 2010, 34(3): 290-292. (in Chinese)
左庭婷,端青.MLVA和SNP分析在炭疽芽孢杆菌基因分型中的应用[J].军事医学科学院院刊,2010,34(3):290-292.
[24]Klevytska AM, Price LB, Schupp JM, et al. Identification and characterization of variable number tandem repeats in theYersiniapestisgenome[J]. J Clin Microbiol, 2001, 39(9): 3179-3185.
[25]Fu XP, Yu DZ, Hai R, et al. Application of tandem repeat sequence and genotyping ofYersiniapestis[J].Chin J Epidemiol, 2004, 25(3): 269-271. (in Chinese)
付秀萍,俞东征,海荣,等.串联重复序列及其在鼠疫菌基因分型中的应用[J].中华流行病学杂志,2004,25(3):269-271.
[26]Sabat A, Krzyszton-Russjan J, Strzalka W, et al. New method for typingStaphylococcusaureusstrains: multiple locus variable number tandem repeat analysis of polymorphism and genetic relationships of clinical isolates[J]. Clin Microbiol, 2003, 41(4): 1801-1804.
[27]Supply P, Mazars E, Lesjean S, et al. Variable human minisatellite-like regions in theMycobacteriumtuberculosisgenome[J]. Mol Microbiol, 2000, 36(3): 762-771.
[28]Skuce RA, McCorry TP, McCarroll JF, et al. Discrimination ofMycobacteriumtuberculosiscomplex bacteria using novel VNTR-PCR targets[J]. Microbiology, 2002, 148(Pt2): 519-528.
[29]Deng YQ, Wang JX, Lin DH, et al. Molecular identification of theBrucellastrains isolated in Fujian province[J]. Chin J Zoonoses, 2009, 25(7): 636-639. (in Chinese)
邓艳琴,王加熊,林代华,等.福建省布鲁氏菌分离株的分子生物学鉴定[J].中国人兽共患病学报,2009,25(7):636-639.
[30]Bricker BJ, Ewalt DR. Evaluation of the HOOF-Print assay for typingBrucellaabortusstrains isolated from cattle in the United States: results with four performance criteria[J]. BMC Microbiol, 2005, 5: 37.

