兰州鲇线粒体Cytb基因的克隆与序列分析
吴旭东肖 伟连总强侯玉霞
(1. 甘肃农业大学动物科学技术学院, 兰州 730070; 2. 宁夏回族自治区水产研究所, 银川 750001; 3. 中国农业大学理学院, 北京 100193)
WU Xu-Dong1,2, XIAO Wei2, LIAN Zong-Qiang1,2and HOU Yu-Xia3
(1. College of Animal Science and Technology, Gansu Agricultural University, Lanzhou 730070, China; 2. Ningxia Fisheries Research Institute, Yinchuan 750001, China; 3. College of Science, China Agricultural University, Beijing 100193, China)
兰州鲇线粒体Cytb基因的克隆与序列分析
吴旭东1,2肖 伟2连总强1,2侯玉霞3
(1. 甘肃农业大学动物科学技术学院, 兰州 730070; 2. 宁夏回族自治区水产研究所, 银川 750001; 3. 中国农业大学理学院, 北京 100193)
为克隆兰州鲇(Silurus lanzhouensis)线粒体Cytb基因全序列, 根据欧洲鲇(Silurus glanis)线粒体基因全序列设计特异引物进行兰州鲇线粒体Cytb基因的PCR扩增, 得到1138 bp兰州鲇线粒体Cytb基因序列。对兰州鲇和其他13种鱼的线粒体Cytb基因核苷酸和氨基酸序列之间进行同源性比较, 结果显示具有较高的同源性, 核苷酸同源性介于61.38%—91.12%, 氨基酸同源性介于76.62%—95.52%。对兰州鲇、欧洲鲇、大口鲇(Silurus meridionalis)、鲇(Silurus asotus)、越南鲇(Silurus cochinchinensis)的Cytb基因之间进行碱基替代分析, 结果显示兰州鲇Cytb基因与鲇之间替换率最低, 值为8.87%, 转换/颠换值为3.21; 与越南鲇之间替换率最高, 值为14.41%, 转换/颠换值为1.83。对本文克隆的兰州鲇Cytb基因与王庆容等测定的兰州鲇线粒体Cytb序列进行序列差异分析, 结果显示两者之间替换率为 11.16%, 存在 127个变异位点, 转换/颠换比为4.08, 遗传距离为0.1230。由NJ法基于兰州鲇和其他13种鱼的Cytb基因序列构建的系统进化树, 结果显示与传统的分类地位基本吻合。
兰州鲇; 线粒体Cytb; 序列分析
兰州鲇(Silurus lanzhouensis)又名黄河鲇, 隶属于鲇形目(Siluriformes)鲇科(Siluridea)鲇属(Silurus),是黄河中上游特有的大型经济鱼类。但近年来, 由于过度捕捞, 水域环境恶化及繁殖场所破坏, 兰州鲇种群数量急剧下降, 已被列为《中国物种红色名录》(第二卷)中的濒危物种[1]。目前, 有关兰州鲇的研究主要集中在兰州鲇分类学、形态学、营养学、繁殖学和遗传结构等方面。陈湘遴首次定名鲇属新种兰州鲇[2]; 吴旭东等于黄河宁夏段首次发现了宁夏鲇属一新记录种—兰州鲇, 对其形态学进行了较详细的研究, 随后对兰州鲇毒理学、繁殖学、育种学和遗传结构等方面进行了系统研究[3—6]; 邱小琮等对兰州鲇肌肉营养成分、消化酶活性和饲料学等方面进行了研究[7,8]; 王庆容等应用 PCR直接测序法测定了兰州鲇线粒体基因全序列[9]。
线粒体(Mitochondria)是真核细胞中小分子量的复制单位, 由于其具有拷贝数多、分子量小、编码效率高、母系遗传和进化速度较核基因组快等特点[10], 近年来越来越多的被作为分子标记, 广泛应用到动物进化遗传学、分子生态学、遗传多样性、物种及品系鉴定等方面[11]。线粒体细胞色素b(Mitochondria Cytochrome b)位于线粒体内膜磷脂双分子层中, 是参与氧化磷酸化合成ATP过程电子传递链中的重要物质, 也是线粒体自身编码的为数不多的蛋白质之一[12]。Meyer的研究表明, 对于初步进行某些物种分子系统发育研究是可以首先采用线粒体细胞色素b基因, 不仅它是13个蛋白质编码基因中了解的最为清楚的基因, 也被认为是最可信的分子标记之一[13], 同时用保守引物扩增线粒体Cytb基因较其他线粒体基因要容易的多, 因此研究较为广泛[14]。已有研究表明线粒体Cytb基因在研究亲缘关系较近的物种分类阶元系统关系和种系地理学方面非常有用[15]。本研究对兰州鲇线粒体 Cytb基因全序列进行克隆和序列分析, 并与GenBank中多个目的鱼类Cytb基因进行同源性比较分析, 旨在为兰州鲇种质资源保护、研究和利用提供基础研究数据。
1 材料与方法
1.1 材料
60尾兰州鲇样本[体长为(10±1.5) cm, 体重(120±8) g]采集于宁夏回族自治区水产研究所国家级兰州鲇原种场建设基地。EcoRⅠ、SalⅠ限制性内切酶、T4 DNA连接酶、Taq DNA聚合酶等购自大连宝生物公司; IPTG、X-gal、氨苄青霉素等生化试剂购自 Sigma公司; 大肠杆菌(Escherichia Coli)JM109,为本实验室保存; pMD18-T载体购自Promga公司。
1.2 兰州鲇基因组DNA的提取
取兰州鲇尾鳍0.5 g, 提取方法参考吴旭东等[16]的方法。经琼脂糖凝胶电泳检测其完整性后用Spectrophotometer测定DNA浓度及纯度, 按照测定的浓度值加适量超纯水将终浓度调整为50 ng/µL左右。
1.3 兰州鲇线粒体Cytb基因的克隆与筛选
引物设计 根据欧洲鲇线粒体全序列(GenBank登录号: AM398435.2)设计可扩增出兰州鲇线粒体Cytb基因全序列的扩增引物: HD: 5′-CCA AAGCGGCAAAATAAG-3′; LD: 5′-GGAGTTAGGG GCGGGAGT-3′
PCR反应体系及条件 PCR反应体系总体积为25 µL, 其中10×buffer(含Mg2+)3 µL, dNTP 2 µL,引物0.5 µL, Taq酶0.5 µL(2.5 U/µL), 加ddH2O至25 µL。PCR扩增程序为: 94℃预变性5min; 94℃变性30s, 54℃退火30s, 72℃延伸30s, 30个循环; 72℃延伸5min。PCR产物上样至1%琼脂糖凝胶电泳, 用紫外凝胶成像系统检测产物片段大小。
扩增产物的回收及阳性克隆的筛选 扩增产物经纯化回收后, 与 pMD18-T载体在 16℃下连接反应过夜, 转化大肠杆菌 JM109感受态细胞, 在含氨苄青霉素的LB培养基平板上37℃培养16h左右后,进行蓝白斑筛选, 挑取阳性克隆进行质粒提取[17]。
1.4 序列测定和系统发育分析
用Sal I酶和EcoR I酶对质粒做酶切鉴定插入片段的大小后, 选取鉴定正确的每个样品对应的菌液(2份)送至北京诺赛基因公司进行测序。测序结果用 DNAClub软件、Clustal x软件进行处理; 用Mega4.0[18]软件将兰州鲇线粒体Cytb基因翻译成氨基酸序列, 同时对兰州鲇线粒体Cytb核苷酸和氨基酸序列与其他鱼类进行同源性比较, 以及核苷酸替代率、颠换/转换计算; 用 Mega4.0中 Kimura’s 2—Parameter模型计算遗传距离, 由NJ法构建基于线粒体Cytb基因的系统进化树。
2 结果
2.1 兰州鲇线粒体Cytb基因全序列PCR扩增和酶切结果
PCR扩增产物经琼脂糖凝胶电泳显示有一条清晰明亮的区带, 分子大小约在1100 bp左右(图1A)。此扩增产物经纯化回收后与 T载体连接, 转化大肠杆菌JM109感受态细胞, 经质粒重组、筛选、酶切鉴定, 结果显示除大于2000 bp的载体外, 还有一条约1100 bp左右的基因片段(图1B)。上述结果证明,所挑选的质粒有目的片段的插入。

图1 兰州鲇线粒体Cytb基因全序列PCR扩增和酶切结果Fig. 1 The whole mitochondria Cytb PCR amplification product and enzyme digestion of S. lanzhouensis
2.2 兰州鲇线粒体Cytb全序列
测序结果用DNAClub软件打开, 剪切掉载体后,经过Clustal x软件处理后, 得到1138 bp基因序列,用Mega4.0软件将兰州鲇线粒体Cytb基因翻译成氨基酸序列。结果显示, 兰州鲇线粒体Cytb基因全序列由 1138个碱基组成, 共编码 379个氨基酸, 以ATG为起始密码子, T为不完全终止子。本文所克隆的基因序列已经录入 GenBank数据库, 序列号为HQ890503。
2.3 兰州鲇与13种鱼类Cytb基因的同源性比较
硬骨鱼亚纲 13种鱼类线粒体 Cytb基因由1137—1141个碱基组成, 编码 378—380个氨基酸(表 1), 其中除形目和鳗鲡目以 TAA为终止密码子外, 其余都以 T为不完全终止子, 这种现象与Perdices, et al.对中美洲Rhamdia属斑马鸭嘴鱼的进化史研究中的结果相符合[19]。鱼类Cytb基因碱基置换多数是沉默的, 很大程度上倾向于转换或颠换,密码子第三位点进化最快, 第二位点最保守, 且在一定的进化尺度内不受饱和效应的影响, 所以线粒体 Cytb基因显示的遗传变异信息和遗传分化水平在不同的科、属、种显示了良好的系统关系, 用线粒体 Cytb基因进行兰州鲇及其他硬骨鱼类的进化分析是适宜的[20]。

表1 兰州鲇与其他13种鱼类线粒体Cytb基因碱基组成及密码子比较Tab. 1 Comparison of base composition and codons of mitochondria Cytb genes between S. lanzhouensis and other 13 species
运用Mega4.0软件将兰州鲇线粒体Cytb的序列及其氨基酸序列, 与其他13种鱼的线粒体Cytb基因核苷酸序列和氨基酸序列进行同源性比较(表 2),结果显示具有较高的同源性, 其中兰州鲇与鲇的线粒体Cytb基因同源性最高, 核苷酸和氨基酸同源性分别为91.12%和95.52%。
应用Mega4.0中Kimura’s 2—Parameter模型计算遗传距离, 基于NJ法构建兰州鲇等5种鲇形目鱼类与其他九种鱼类线粒体Cytb的系统进化树, 枝上的数值为bootstrap1000次后的置信值 (图2)。
2.4 兰州鲇与几种鲇属鱼类 Cytb基因的碱基替代分析
运用Mega4.0软件对本文克隆的兰州鲇线粒体Cytb基因, 与欧洲鲇、鲇、大口鲇、越南鲇的线粒体 Cytb基因之间进行碱基替代分析(表 3), 结果显示本文克隆的兰州鲇线粒体 Cytb基因与鲇之间替换率最低, 值为 8.87%, 转换/颠换值为 3.21; 与越南鲇之间替换率最高, 值为14.41%, 转换/颠换值为1.83。
2.5 兰州鲇线粒体 Cytb不同测序结果之间的差异分析
王庆容等通过PCR扩增产物直接测序和引物行走法, 测定了兰州鲇的线粒体基因全序列[9](Gen-Bank序列号: JF895472)。运用Mega4.0软件对王庆容等测定的线粒体Cytb基因, 与欧洲鲇、鲇、大口鲇、越南鲇以及本文克隆的兰州鲇线粒体Cytb基因之间进行碱基替代分析(表4)。结果显示王庆容等测定的线粒体Cytb基因与欧洲鲇之间替换率最低, 值为 2.63%, 转换/颠换值为 4.00; 与越南鲇之间替换率最高为18.45%, 转换/颠换值为2.00; 与本文克隆的兰州鲇线粒体Cytb基因之间替换率为11.16%, 存在127个变异位点, 其中102个转换位点, 25个颠换位点, 转换/颠换值为4.08。
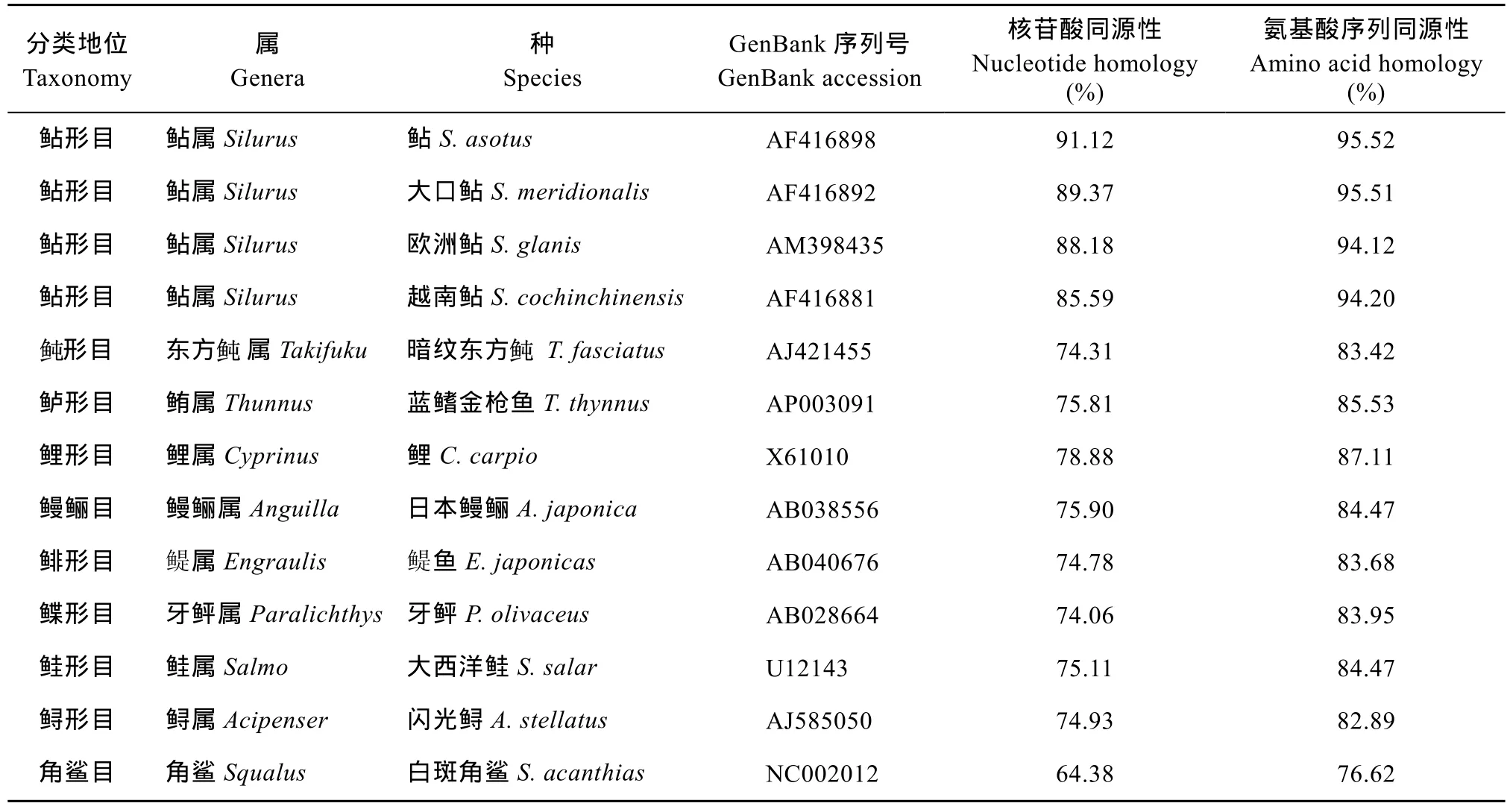
表2 兰州鲇与其他13种鱼类线粒体Cytb基因序列和氨基酸序列同源性比较Tab. 2 Comparoson of homology of nucleotide and amino acid of mitochondria Cytb between S. lanzhouensis and other 13 species

图2 用邻接法根据兰州鲇与其他13种鱼线粒体Cytb基因序列构建的分子系统树Fig. 2 Phylogenetic trees derived from mitochondria Cytb nucleotide sequences of S. lanzhouensis and other thirteen species of fishes based on Neighbor-Joining method
运用 Mega4.0软件基于 Kimura’s 2-Parameter模型计算本文克隆的兰州鲇线粒体Cytb基因与王庆容等测定的线粒体Cytb基因序列(在表5和图3中简称“W-fishes”)以及欧洲鲇、鲇、大口鲇、越南鲇这几种鲇属类Cytb基因之间的遗传距离(表 5)。结果显示本文克隆的兰州鲇线粒体Cytb基因与鲇之间的遗传距离最小, 与越南鲇之间的遗传距离最大; 而王庆容等测定的线粒体Cytb基因与欧洲鲇之间的遗传距离最小, 与越南鲇之间的遗传距离最大;本文克隆得到的兰州鲇线粒体Cytb基因与王庆容等测定的线粒体Cytb基因之间的遗传距离为0.1230。
基于表5中得到的遗传距离运用NJ法构建系统发育树(图3), 枝上的数值是bootstrap1000次后的置信值。从图中可以看出王庆容等的实验样本与欧洲鲇以 100%的置信值先聚为一支, 然后与南方鲇聚在一起, 越南鲇为单独的一支, 表明王庆容等采集的实验样本与欧洲鲇亲缘关系很近。

表3 兰州鲇与四种鲇属鱼类线粒体Cytb基因的碱基替换分析Tab. 3 Number and frequencies of nucleotides substitution of mitochondria Cytb genes between S. lanzhouensis and other four Silurus species
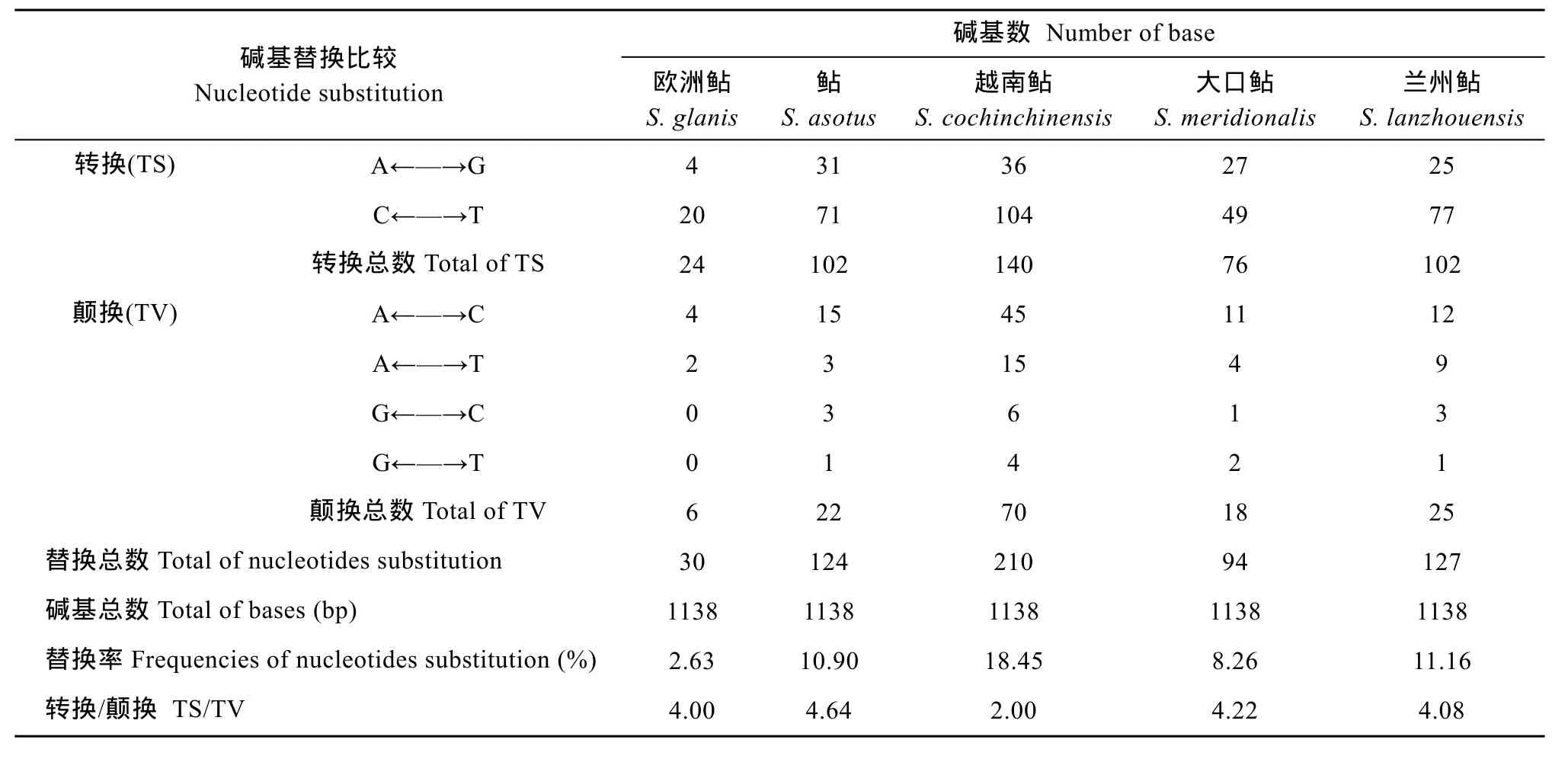
表4 王庆容等测定的线粒体Cytb基因与五种鲇属鱼类线粒体Cytb基因之间的碱基替代分析Tab. 4 Number and frequencies of nucleotides substitution of mitochondria Cytb genes between which was measured by Wang Qing-Rong and other five Silurus species

表5 兰州鲇等几种鲇形目鱼类基于线粒体Cytb基因的遗传距离Tab. 5 Genetic distance of S. lanzhouensis and other Siluriformes species based on mitochondria Cytb
3 讨论
造成种内同一基因差异的原因有很多, 隔离、漂变、杂交等都可能造成种内同一基因的变异。Avise认为同种的个体间同一基因存在0.1%—5%的差异[21], 而对于其他一些动物的Cytb基因序列的研究分析表明, 种内个体间的序列差异一般在 0—4.06%, 差异超过 6%的个体间已有明显的亚种和种的分化[22,23]。Robert, et al.等指出, 基于 Kimura’s 2-Parameter模型的同种内平均遗传距离为0.0039[24]; Kartavtsev, et al.等认为线粒体Cytb基因平均遗传距离在同种群间为(1.55±0.56)%, 同属之间为(5.52± 1.34)%, 在同科不同属之间为(10.69±1.34)%[25]。本文克隆的兰州鲇线粒体 Cytb全序列与王庆容测定的 Cytb全序列之间核苷酸差异率为 11.66%, 基于Kimura’s 2-Parameter模型的遗传距离为0.1230, 按照Avise和Kartavtsev的理论, 两者已经达到种间差异的水平。王庆容等测定的线粒体Cytb序列与欧洲鲇的序列差异为2.63%, 基于Kimura’s 2-Parameter模型的遗传距离为 0.0269, 表明王庆容等所采集的实验样本与欧洲鲇之间亲缘关系非常近。本文中兰州鲇采集于宁夏回族自治区水产研究所兰州鲇原种场, 该场是 2013年农业部命名的第一批“全国现代渔业种业示范场”, 养殖的兰州鲇种质纯正; 而王庆容等的实验样本采集于北京西门农贸市场(来源于山东养殖场)[9]。
褚新洛等在中国动物志•硬骨鱼纲•鲇形目的分类中关于兰州鲇的种级分布位于华西区陇西亚区,主要分布于黄河水系中上游, 具体分布位于黄河水系的兰州至内蒙古托克托县和巴彦淖尔盟四分滩[26];其次, 本文克隆的兰州鲇线粒体Cytb全序列与王庆容等测定的线粒体 Cytb基因全序列之间核苷酸差异率和遗传距离很大,已经达到不同种的水平; 王庆容等测定的线粒体 Cytb基因与欧洲鲇线粒体Cytb基因差异率很小, 亲缘关系很近, 推断王庆容等采集的样本应该不是兰州鲇, 而是欧洲鲇或其近缘种。

图3 基于邻接法根据本文克隆的兰州鲇线粒体 Cytb序列、王庆容等测定兰州鲇线粒体的Cytb序列及其他四种属鱼类的线粒体Cytb基因序列构建的系统进化树Fig. 3 Phylogenetic trees derived from mitochondria Cytb gene of S. lanzhouensis cloned by this paper and measured by Wang Qing-Rong et al. and other four species of Silurus based on Neighbor-Joining method
从图2可以看出五种鲇属鱼类聚在一起, 其中兰州鲇和鲇先聚为一支,再与大口鲇聚在一起; 构建的系统进化树将除软骨鱼亚纲角鲨目外的硬骨鱼亚纲九个目分成两组: 鲟形目和鳗鲡目聚为一组; 鲇形目、鲤形目、鲑形目、鲱形目、鲽形目、形目、鲈形目这 7个目为另一组; 其中后一组中鲇形目、鲤形目聚为一组, 鲑形目、鲱形目、鲽形目、形目和鲈形目聚为另一组。由此可见根据线粒体Cytb序列获得的分类结果, 与孟庆闻等采用Nelson的系统[27]提出的分类系统结果、系统发育演化基本吻合。
[1] Wang S, Xie Y. China Species Red List (Volume II) [M]. Beijing: Higher Education Press. 2009, 294—295 [汪松, 解焱.中国物种红色名录(第二卷). 北京: 高等教育出版社. 2009, 294—295]
[2] Cheng X L. Division of catfish fish overview of China [J]. Acta Hydrobiologica Sinica, 1977, 6(2): 197—216 [陈湘遴.我国鲇科鱼类的总述. 水生生物学集刊, 1977, 6(2): 197—216]
[3] Wu X D, Zhang Q, Zhao H X, et al. A new species of catfish in Ningxia—Silurus lanzhouensis and it’s intensive morphological description [J]. Freshwater Fisheries, 2006, 36(3): 26—29 [吴旭东, 张奇, 赵红雪, 等. 宁夏鲇属鱼类一新纪录种—兰州鲇形态学特征描述. 淡水渔业, 2006, 36(3): 26—29]
[4] Shi L N, Zhang Q, Wu X D, et al. Individual fertility of the Silurus lanzhouensis [J]. Journal of Gansu Agricultural University, 2008, 43(1): 67—70 [史丽娜, 张奇, 吴旭东,等. 兰州鲇个体繁殖力的研究. 甘肃农业大学学报, 2008, 43(1): 67—70]
[5] Wu X D, Lian Z Q, Hou Y X, et al. Application analysis of microsatellite markers from Silurus meriaionalis on three species of siluriformes [J]. Acta Hydrobiologica Sinica, 2011, 35(4): 638—644 [吴旭东, 连总强, 侯玉霞, 等. 大口鲇微卫星标记在三个鲇形目鱼类种群间适用性研究. 水生生物学报, 2011, 35(4): 638—644]
[6] Wu X D, Lian Z Q, Hou Y X, et al. Genetic structureanalyses between wild population and artificial breeding ones of Silurus lanzhouensis [J]. Freshwater Fisheries, 2011, 41(3): 34—38 [吴旭东, 侯玉霞, 连总强, 等. 兰州鲇野生群体和人工繁育群体遗传结构的比较研究. 淡水渔业, 2011, 41(3): 34—38]
[7] Qiu X C, Zhao H X, Wang Y J, et al. Nutrient analysis and nutritive evaluation in muscle of catfish Silurus lanzhouensis [J]. Fisheries Science, 2008, 27(8): 407—410 [邱小琮, 赵红雪, 王远吉, 等. 兰州鲇肌肉营养成分分析及营养价值评价. 水产科学, 2008, 27(8): 407—410]
[8] Qiu X C, Zhao H X, Wang Y J, et al. In vitro digestibility coefficient and enzymolysis rate of Silurus lanzhouensis to four common feed ingredients [J]. Freshwater Fisheries, 2008, 38(8): 31—35 [邱小琮, 赵红雪, 王远吉, 等. 兰州鲇对四种饲料原料的离体消化率和酶解能力. 淡水渔业, 2008, 38(2): 31—35]
[9] Wang Q R, Xu C, Xu C R, et al. Mitochondrial genome structure of Yellow River catfish (Silurus lanzhouensis) and phylogenetic analysis [J]. Acta Scientiarum Naturalium Universitatis Pekinensis, 2012, 48(3): 376—380 [王庆容,许昌, 许崇任, 等. 兰州鲇线粒体基因组序列结构及系统发育分析. 北京大学学报, 2012, 48(3): 376—380]
[10] Gray M W. Origin and evolution of mitochondrial DNA [J]. Annual Review of Cell Biology, 1989, 5: 25—50
[11] Xiao W H, Zhang Y P. Genetics and evolution of mitocchondrial DNA in fish [J]. Acta Hydrobiologica Sinica, 2000, 24(4): 384—391 [肖武汉, 张亚平. 鱼类线粒体DNA的遗传与进化. 水生生物学报, 2000, 24(4): 384—391]
[12] Schoepp B, Breton J, Parot P, et al. Relative orientation of the hemes of the cytochrome bc1complexes from Rhodobacter Sphaeroides,Rhodospirillum rubrum,and beef heart mitochondria, A linear dichroism study [J]. Biological Chemistry, 2000, 275(8): 5284—5290
[13] Meyer A. Shortcoming of the cytochrome b gene as a molecular marker [J]. Trends in Ecology and Evolution, 1994, 9: 278—289
[14] Katriina L I, Erie B T. Molecular resolution of the systematics of a problematic group of fishes (Teleostei: Osmeridae) and evidence for morphological homoplasy [J]. Molecular Phylogenetics and Evolution, 2009, 50(1): 163—175
[15] Song C B, Near T J, Page L M. Phylogenetic relations among percid fishes as inferred from mitochondrial cytochrome b DNA sequence data [J]. Molecular Phylogenetics and Evolution, 1998, 10(3): 343—353
[16] Wu X D, Zhang Q, Zhao H X, et al. The method of extracting and purifing genome DNA of a catfish in the Yellow River [J]. Freshwater Fisheries, 2006, 36(1): 19—21 [吴旭东, 张奇, 侯玉霞, 等. 黄河鲶基因组 DNA提取与纯化方法的研究. 淡水渔业, 2006, 36(1): 19—21]
[17] Sambrook J, Russell D W. Molecular Clonging: a Laboratory Manual [M]. New York: Cold Spring Harbor Laboratory Press. 2001, 611—627
[18] Tamura K, Dudley J, Nei M, et al. Mega4: Molecular evolutionary genetics analysis (MEGA) software version 4.0 [J]. Molecular Biology and Evolution, 2007, (10): 1092—1093
[19] Perdices A, Bermingham E, Montilla A, et al. Evolutionary history of the genus Rhamdia (Teleostei: Pimelodidae) in central America [J]. Molecular Phylogenetics and Evolution, 2002, 25: 172—189
[20] Peng Z, He S, Zhang Y. Phylogenetic relationships of glyptostemoid fishes(Siluriformes: Sisoridae) inferred from mitochondrial cytochrome b gene sequence [J]. Molecular Phylogenetics and Evolution, 2005, 31(3): 979—987
[21] Avise J C. Molecular Markers, Natural History and Evolution [M]. New York: Chapman and Hall. 1994, 120—144
[22] Cheng Q Q, Ma C Y, Zhuang P, et al. Genetic structure and evolution characters in three populations of Coilia mystus based on cytochrome b gene segment sequence of mitochondrial DNA [J]. Journal of Fisheries of China, 2008, 32(1): 2—6 [程起群, 马春艳, 庄平, 等. 基于线粒体细胞色素b标记探讨凤鲚3群体遗传结构和进化特征. 水产学报, 2008, 32(1): 2—6]
[23] Yang X G, Wang Y Q, Zhou K Y, et al. The authentication of Oviductus ranae and their original animals by using molecular marker [J]. Biopharm Bull, 2002, 25(8): 1035—1039
[24] Ward R D,Tyler S Z, Bronwyn H I, et al. DNA barcoding Australia’s fish species [J]. Philosophical Transactions Royal Society B, 2005, 360: 1847–1857
[25] Kartavtsev Y P, Lee J S. Analysis of nucleotide diversity at the cytochrome b and cytochrome oxidase 1 genes at the population, species, and genus levels [J]. Russian Journal of Genetics, 2006, 42(4): 437–461
[26] Chu X L, Zheng B S, Dai D Y, et al. Fauna Sinica-Osteich-thyes Siluriformes [M]. Beijing: Science Press. 1999, 87—88 [褚新洛, 郑葆珊, 戴定远, 等. 中国动物志·硬骨鱼纲·鲇形目. 北京: 科学出版社. 1999, 87—88]
[27] Meng Q W, Su J X, Miao X Z. Taxonomy of Fishes [M]. Beijing: China Agriculture Press. 1995, 995—998 [孟庆闻,苏锦祥, 缪学祖. 鱼类分类学. 北京: 中国农业出版社. 1995, 995—998]
THE CLONING AND SEQUENCE ANALYSIS OF THE MITOCHONDRIAL CYTOCHROME b GENE OF SILURUS LANZHOUENSIS
To clone the sequence of the mitochondrial cytochrome b (Cytb) gene of Silurus lanzhouensis, we designed a specific pair of primers from the mitochondrial genome of S. glanis. The 1138 bp S. lanzhouensis Cytb gene was obtained after PCR. Then we compared the homology of the mitochondrial Cytb gene between S. lanzhouensis and 13 other species. The results showed that these species have high homology in Cytb gene. The homology value of the nucleotides was between 61.38% and 91.12%, and the value of the amino acids was between 76.62% and 95.52%. We also analyzed the nucleotides substitution of the mitochondrial Cytb gene among S. lanzhouensis, S. glanis, S. meridionalis, S. asotus, and S. cochinchinensis, and found that the lowest frequency of nucleotides substitution occurred between S. lanzhouensis and S. asotus with the value of 8.87%, and TS/TV was 3.21. The highest frequency was between S. lanzhouensis and S. cochinchinensis with the value of 14.41%, and TS/TV was 1.83. Qing-Rong Wang, et al. previously reported the sequence of the mitochondrial Cytb gene of S. lanzhouensis. Here we conducted the sequence difference analysis between our results and theirs. The results showed that the frequency of nucleotides substitution was 11.16%, there were 127 variable sites, TS/TV was 4.08, genetic distance was 0.1230. The phylogenetic tree of S. lanzhouensis and 13 other species constructed by NJ method was consistent with the traditional taxonomic results.
Silurus lanzhouensis; Mitochondria Cytochrome b; Sequence analysis
WU Xu-Dong1,2, XIAO Wei2, LIAN Zong-Qiang1,2and HOU Yu-Xia3
(1. College of Animal Science and Technology, Gansu Agricultural University, Lanzhou 730070, China; 2. Ningxia Fisheries Research Institute, Yinchuan 750001, China; 3. College of Science, China Agricultural University, Beijing 100193, China)
S917
A
1000-3207(2014)04-0772-08
10.7541/2014.108
2013-07-05;
2014-02-13
国家自然科学基金项目(30760191)资助
吴旭东(1967—), 男, 博士, 研究员; 研究方向为水生动物分子生物学及种质资源保护与增养殖。E-mail: amy95@163.com

