基于线粒体COⅠ基因的沙鳅亚科鱼类DNA条形码及其分子系统发育研究
毛云涛甘小妮王绪祯
(1. 中国科学院水生生物研究所, 水生生物多样性与保护重点实验室, 武汉 430072; 2. 中国科学院大学, 北京 100049)
基于线粒体COⅠ基因的沙鳅亚科鱼类DNA条形码及其分子系统发育研究
毛云涛1,2甘小妮1王绪祯1
(1. 中国科学院水生生物研究所, 水生生物多样性与保护重点实验室, 武汉 430072; 2. 中国科学院大学, 北京 100049)
选择线粒体 COⅠ基因作为分子标记, 进行沙鳅亚科鱼类(Botiinae)DNA条形码及其分子系统发育研究。研究获得了沙鳅亚科7属19种共131个个体的COⅠ基因序列, 利用MEGA5.0软件分析了沙鳅亚科鱼类COⅠ基因的序列特征, 计算了种内及种间遗传距离。沙鳅亚科鱼类的分子系统发育关系的重建分别采用NJ法和Bayesian法。研究发现, 沙鳅亚科COⅠ基因的碱基组成为: A 24.4%、T 29.5%、G 18.0%、C 28.1%。沙鳅亚科鱼类种内平均遗传距离为0.002±0.000, 种间平均遗传距离为0.148±0.008。DNA条形码研究结果显示, 所分析的19种沙鳅鱼类各自分别聚成单系分支, 表明COⅠ基因在本研究中具有100%的物种鉴别率。同时, 系统发育分析支持各属的单系性, 并且结果显示沙鳅亚科鱼类聚为两个分支, 其中一支由薄鳅属和副沙鳅属构成, 另一分支则包括: (沙鳅属、色鳅属)和 [中华沙鳅属、(缨须鳅属、安彦鳅属)]。因此, COⅠ基因可以作为有效的分子标记对沙鳅亚科进行DNA条形码研究以及分子系统发育研究。
DNA条形码; COⅠ基因; 沙鳅亚科; 物种鉴定
沙鳅亚科(Botiinae)是鲤形目的一群中小型鱼类,包括7个属[1], 近60种。沙鳅亚科鱼类分布范围北至中国黑龙江和日本, 西至巴基斯坦, 南至印度尼西亚的爪哇岛, 但集中分布于中国长江以南、泰国、缅甸、老挝及印度东北部等地区[2]。根据颊部有无鳞片和眼下刺是否分叉, 朱松泉[3]认为沙鳅亚科由薄鳅属、沙鳅属和副沙鳅属组成。Nalban, et al.[4]和Kottelat, et al.[5]将沙鳅亚科划分为薄鳅族与沙鳅族,并对原有沙鳅属进行分类整理, 将其亚属提升到属的水平, 认为沙鳅亚科包括7个属。
随着环境的污染, 生态系统的破坏, 鱼类物种多样性呈现丧失的现象[6]。因此, 对鱼类物种的保护工作显得格外重要, 而进行鱼类物种多样性保护工作的前提是对物种进行正确的鉴定和分类[7]。基于形态学特征的传统分类研究对构建沙鳅亚科的分类系统起到了重要的作用。然而, 在实践过程中, 由于会遇到标本的个体较小及保存不完整等现实问题,这些都给传统的物种鉴定工作带来了巨大困难。尤其对一些不具备分类学背景的研究者而言, 依据形态学特征鉴定物种将是一个重大的挑战。随着分子分类学的发展, 寻求分子生物学的辅助手段对沙鳅亚科鱼类进行准确的鉴定, 已经成为鱼类学研究中的一个迫切需要解决的问题。
DNA条形码(DNA barcode)作为对物种进行识别和鉴定的一项新技术, 正受到国内外研究者的广泛关注[8—11]。DNA条形码是指生物体内能够代表该物种的、标准的、有足够变异的、易扩增且相对较短(约650 bp)的DNA片段[12,13]。在线粒体DNA中, COⅠ基因拥有长度适宜、进化速率适中及富含系统发育信号等特点[14]。已有文献证明, 在实验中, 能够较好地通过通用引物对大多数动物的 COⅠ基因进行有效扩增, 因此, 在动物界物种鉴定中, 通常选择 COⅠ基因作为条形码[15—17]。目前, 世界各地已经开展了从湖泊[18]到海洋[19,20]多个地理区域的鱼类DNA条形码研究计划, 在物种保护[2]和鉴定[22,23]方面做了诸多探索。
近年来, 研究者开始利用分子系统发育的理论和方法开展沙鳅亚科鱼类的分类学研究, 通常选择线粒体的细胞色素b (Cytb)和控制区(Control region)的 DNA 序列作为分子标记[22,23]。本研究选择了DNA条形码标准基因——线粒体COⅠ基因作为标记, 进行沙鳅亚科鱼类 DNA条形码及其分子系统发育研究。本研究的目的就是通过沙鳅鱼类COⅠ基因序列的比对和分析, 探讨COⅠ基因作为DNA条形码在沙鳅亚科鱼类种类鉴定中的可行性, 通过重建沙鳅亚科鱼类的系统发育关系来探讨其内部种级和属级分类阶元的演化关系。
1 材料与方法
1.1 实验材料
本研究包含了沙鳅亚科鱼类的 7属 19种, 共131个个体, 其中从 GenBank下载 COⅠ基因序列44条。本研究所采集的标本、物种的形态学特征鉴定主要依据《中国淡水鱼类检索》[3]和《薄鳅属一新种——小眼薄鳅》[24]进行。鲤形目包括鳅科、平鳍鳅科、鲤科、双孔鱼科和胭脂鱼科, 基于鲤形目是单系类群这一普遍认同观点[25], 我们选择了除鳅科和平鳍鳅科以外的6种鲤形目鱼类作为外类群。它们分别来自鲤科中鲢(Hypophthalmichthys molitrix)、三角鲂(Megalobrama terminalis)、鲤(Cyprinus carpio)、斑马鱼(Danio rerio); 双孔鱼科的双孔鱼(Gyrinocheilus aymonieri)和胭脂鱼科中胭脂鱼(Myxocyprinus asiaticus)。研究样本和外类群的样本信息见表1。采集标本的鳍条或肌肉组织保存在95%的酒精中, 标本收藏于中国科学院水生生物研究所。
1.2 DNA提取、PCR扩增和测序
在本研究中采用了略作改动的酚–氯仿抽提法[26]和高盐浓度抽提法提取总DNA, 提取好的DNA置于–20℃冰箱保存。
使用特异性引物COⅠ-F1 (5′-TCAACCAACCA CAAAGACATTGGCAC-3′)和COⅠ-R1 (5′-TAGACT TCTGGGTGGCCAAAGAATCA-3′)[16]扩增线粒体COⅠ基因序列。PCR反应体系为 30 μL, 其中含ddH2O 20.625 μL, 10×PCR buffer 3 μL(1 mol/L), dNTP 1.5 μL(2.5 mmol/L), Taq酶0.375 μL(5 U/μL),正反向引物各1.5 μL(10 pmol/L), 总的基因组DNA模板约为100 ng。PCR反应条件为: 95℃预变性5min; 95℃变性30s, 50℃退火45s, 72℃延伸45s, 共31个循环; 最后再72℃延伸10min。PCR产物经1.2%琼脂糖凝胶电泳检测后送北京天一辉远测序公司进行正反向测序。10×PCR buffer、dNTP和Taq酶均购自Takara公司。
1.3 数据分析
用DNAstar软件中SeqMan程序对序列进行拼接, 运用 BioEdit软件查看序列结果并辅助测序峰图进行人工校正, 排除可能引入的错误信息。序列比对使用的是Clustalw2 (Gap Open Penalty缺省值设置为10), 用MEGA5.0计算序列的碱基组成、序列间的碱基变异频率和转换颠换频率及其比率, 基于Kimura’s 2-parameter[27]模型计算各物种的遗传距离。
在本研究中, 分子系统树的构建采用邻接法(Neighbor-joining, NJ)和贝叶斯法(Bayesian)。使用modeltest3.7软件选择最佳替代模型, 基于AIC标准选择了TrN+I+G模型作为核苷酸的最佳模型, 用于邻接法分析和贝叶斯分析。邻接树(NJ)的构建基于MEGA5.0软件, 并进行 1000次自展分支检验检验分支树的可靠性。贝叶斯分析运用了 MrBayes3.1.2软件, 运算中马尔科夫蒙特卡洛(MCMC)参数设置如下: ngen = 1000000, nchains = 4, samplefreq = 100, temp = 0.02, startingtree = random。运用Trace 1.5软件检查MCMC结果的收敛性, 结果表明链收敛于前200000代之内, 因此burnin设置为2000。
2 结果
2.1 COⅠ基因序列特征
本研究共获得19种沙鳅亚科鱼类131个个体的线粒体COⅠ基因的DNA片段, 长684 bp。沙鳅鱼类COⅠ基因序列的碱基组成为: A 24.4%、T 29.5%、C 28.1%、G 18.0%。在648 bp排列位点中, 变异位点数目为237个, 转换位点和颠换位点分别为58个和24个。在COⅠ基因的3个密码子位点中, 第2密码子位点没有变异, 转换位点和颠换位点均为 0;而第 3密码子位点的转换位点和颠换位点数最多,分别为51个和23个。沙鳅鱼类COⅠ基因的转换位点与颠换位点比值范围为0.21—6.45。
沙鳅鱼类的 COⅠ基因序列平均 G+C含量为42%—49.4%, 平均值为 46%。其中, 含量最低的是皇冠沙鳅(42%), 最高的是白氏薄鳅(49.4%)。这种差异主要是由第 3密码子位点 G+C含量的差异引起的。对于沙鳅鱼类而言, 除白氏薄鳅外, 其余各物种的平均G+C含量在第1密码子、第2密码子、第3密码子中依次递减。第1 密码子的 G+C含量贡献值最大, 变化范围为54%—60%; 第2密码子位点的平均G+C含量变化范围最小, 为43%—44%; 第3密码子的G+C含量变化范围为29%—45%。
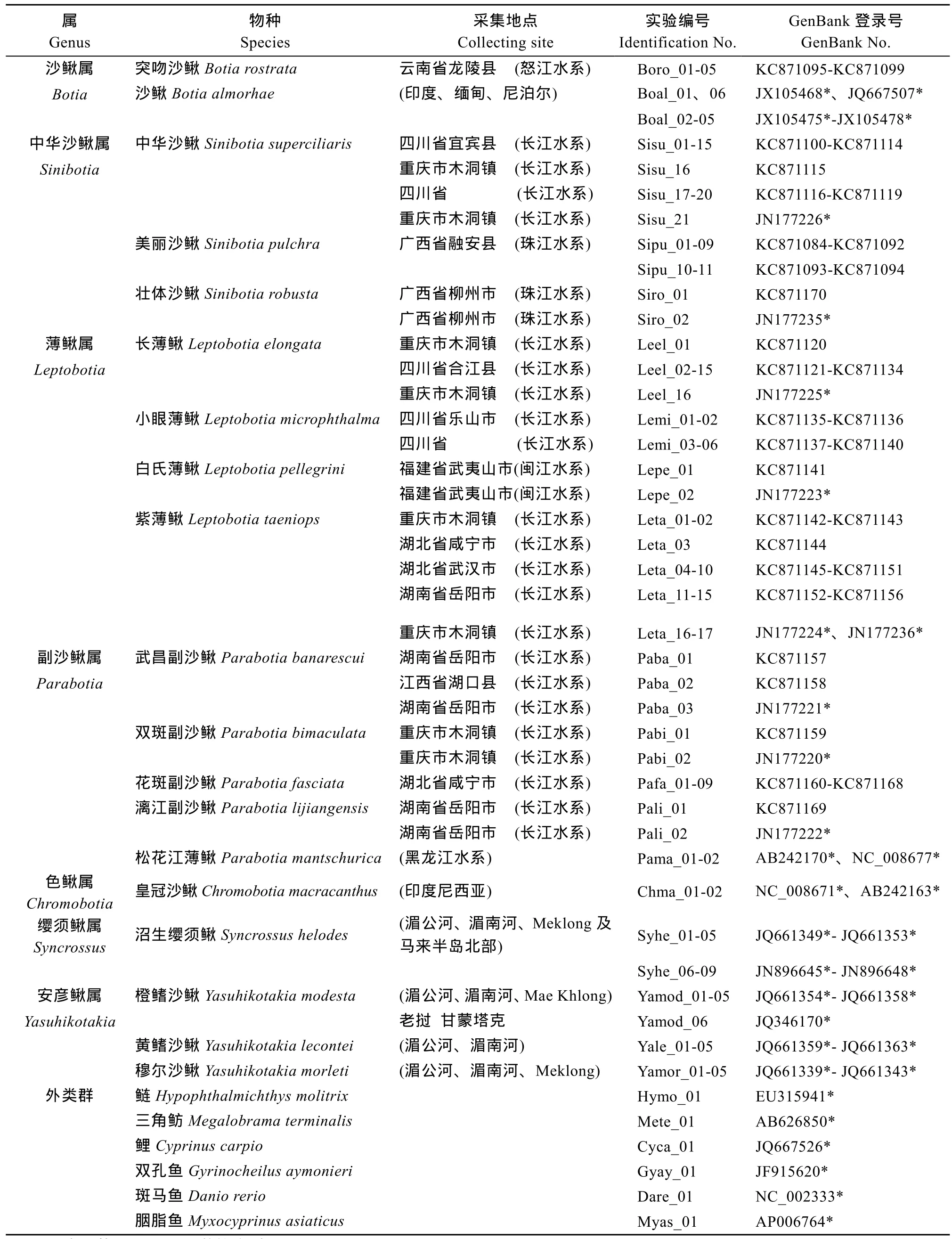
表1 研究所用样本名录及相关信息Tab. 1 Sampling information and GenBank accession numbers of COⅠ genes
2.2 种内及种间的遗传距离
在本研究中, 沙鳅亚科鱼类的种内遗传距离为0—0.009, 其中, 橙鳍沙鳅的种内遗传距离最大为0.009, 其余18种的种内遗传距离小于或等于0.003,种内平均遗传距离为0.002±0.000。沙鳅亚科鱼类的种间遗传距离为0.019—0.233(表2), 种间遗传距离平均为0.148±0.008。
2.3 分子系统树
贝叶斯法构建的分子系统树的拓扑结构与 NJ法一致, 仅部分节点的支持率存在差异。采用NJ法构建的沙鳅亚科分子系统树如图 1所示, 其中加入了贝叶斯法得到的节点支持率。在沙鳅亚科中, 每种鱼类的不同个体均各自聚成单系类群, 且具有较高的节点支持率, 其中NJ树中节点的自展支持率均为100%, 而Bayesian树中, 除花斑副沙鳅 (Parabotia fasciata) 外, 其余节点的后验概率均为1。另外, 我们的系统发育分析结果也支持沙鳅亚科的7个属各自构成单系。
3 讨论
Saccone, et al.[28]对9种硬骨鱼和3种软骨鱼的线粒体基因组全序列进行分析, 结果表明硬骨鱼类的平均G+C含量高于软骨鱼类。Ward, et al.[16]分析了207种澳大利亚鱼类的COⅠ基因序列, 结果同样支持硬骨鱼类的平均G+C含量显著高于软骨鱼类。本研究中沙鳅鱼类的 COⅠ基因的平均 G+C含量(46.5%)高于Saccone, et al.和Ward, et al.得出的软骨鱼类的平均G+C含量, 与Saccone, et al.和Ward, et al.的研究结果一致。在DNA进化过程中, 转换发生的频率要比颠换高, R值(si/sv)的大小标志着物种亲缘关系的远近, 在沙鳅亚科中, 密码子的 R值分别为2.45, 大于2.0, 表明基因序列的突变未达到饱和[29],适用于系统发育分析。
利用 DNA条形码对物种进行正确分类取决于种内遗传距离和种间遗传距离之间的差异大小, 即条形码缺口(barcoding gap)。Hebert, et al.[12,14]指出,有效利用 COⅠ基因序列鉴别物种的关键是种间遗传距离必须大于种内遗传距离, 并且距离差异大约10倍。在本研究中, 沙鳅鱼类的种间平均遗传距离是 0.148±0.008, 种内平均遗传距离是 0.002±0.000,两者相差74倍, 这表明COⅠ基因序列在沙鳅亚科鱼类中的遗传差异为有效区分该类群中的各个物种奠定了良好的基础。薄鳅属内的种间遗传距离相对较小, 其中, 松花江薄鳅(Parabotia mantschurica)与花斑副沙鳅(Parabotia fasciata)的种间遗传距离为0.019。而松花江薄鳅分布于黑龙江水系, 花斑副沙鳅则采自于长江流域的湖北省咸宁市, 二者间较小的遗传差异并不是由于种间杂交造成, 可能与谱系分化时间较短有关。总体而言, 本研究所分析的 19种沙鳅鱼类每种各自聚成单系分支, 表明 COⅠ基因在沙鳅亚科物种中具有100%的物种鉴别率。因此,对沙鳅亚科而言, COⅠ基因可以作为一个有效的DNA条形码标准基因。
本研究分子系统发育分析显示, 沙鳅亚科鱼类的不同个体在种与属水平上均分别聚成单系分支,为形态学分类中属的单系性提供了分子生物学证据。就属级相互关系而言, 沙鳅亚科鱼类聚为两个分支, 其中一支由薄鳅属和副沙鳅属构成, 二者互为姐妹群。薄鳅属和副沙鳅属主要分布于青藏高原以东的东亚地区, 为我国特有属。沙鳅亚科中另一分支包括沙鳅属、色鳅属、中华沙鳅属、缨须鳅属和安彦鳅属。沙鳅属与色鳅属为姐妹群, 就地理分布而言, 这个姐妹群主要分布于青藏高原以南的南亚及东南亚的主要水系中。缨须鳅属和安彦鳅属主要分布于东南亚地区的湄公河流域, 其姐妹群关系与其地理分布上的密切联系一致。缨须鳅属和安彦鳅属的姐妹类群中华沙鳅属则主要分布于长江以南流域, 向南至湄公河流域的老挝和泰国等地。总体而言, 本文系统发育研究也揭示了沙鳅鱼类的三个单系类群的谱系进化关系与其地理分布之间的密切相关性。

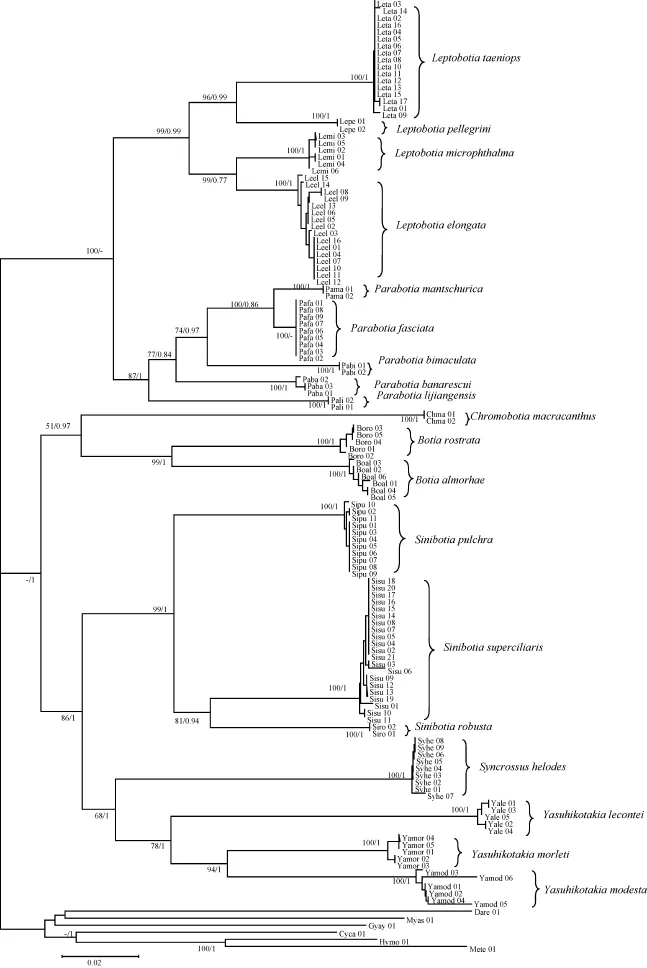
图1 利用COⅠ基因序列构建的NJ树(节点支持率中, 前者为自展支持率, 后者为后验概率)Fig. 1 NJ tree based on the COⅠ gene sequences (Numbers among the Botiinae of the nodes represent bootstrap values, The former are based on NJ method while the latter are based on Bayesian method)
本研究系统发育分析还揭示了沙鳅亚科中属级阶元的单系性。唐琼英等[1]基于细胞色素 b基因的沙鳅亚科分子系统发育研究支持除安彦鳅属外的其余 6属的单系性, 该研究认为安彦鳅属为复系类群, 其中黑线沙鳅(Yasuhikotakia nigrolineata)及小沙鳅(Yasuhikotakia sidthimunki)和安彦鳅属其他物种没有聚为一支。而黑线沙鳅和小沙鳅被认为属于Ambastaia属[30], 这种分类订正也进一步明确了安彦鳅属的单系性。因此, 本研究系统发育分析结果在沙鳅亚科属级阶元的单系性及其相互关系上与以前的分子系统发育研究结果基本一致。线粒体 COⅠ基因可以作为沙鳅亚科分子系统发育研究中的有效的分子标记。
[1] Tang Q Y, Yu D, Liu H Z. Leptobotia zebra should be revised as Sinibotia zebra (Cypriniformes: Botiidae) [J]. Zoological Research, 2008, 29(1): 1—9 [唐琼英, 俞丹, 刘焕章. 斑纹薄鳅(Leptobotia zebra)应该为斑纹沙鳅(Sinibotia zebra). 动物学研究, 2008, 29(1): 1—9]
[2] Chen J X. A study on the classification of the Botoid fishes of China [J]. Zoological Research, 1980, 1(1): 3—26 [陈景星. 中国沙鳅亚科鱼类系统分类的研究. 动物学研究, 1980, 1(1): 3—26]
[3] Zhu S Q. Synopsis of Freshwater Fishes of China [M]. Nanjing: Jiangsu Science and Technology Publishing House. 1995, 1—549 [朱松泉. 中国淡水鱼类检索. 南京: 江苏科学技术出版社. 1995, 1—549]
[4] Nalbant T T, Nalbant T T. Sixty million years of evolution. Part one: family Botiidae (Pisces: Ostariophysi: Cobitidae) [J]. Travaux Du Muséum D'histoire Naturelle “Grigore ntipa”, 2002, 44: 343—379
[5] Kottelat M. Botia kubotai, a new species of loach (Teleostei : Cobitidae) from the Ataran River basin (Myanmar), with comments on botiine nomenclature and diagnosis of a new genus [J]. Zootaxa, 2004, 40(1): 1—18
[6] Zhang C G, Zhao Y H, Xing Y C, et al. Fish species diversity and conservation in Beijing and adjacent areas [J]. Biodiversity Science, 2011, 19(5): 597—604 [张春光, 赵亚辉, 邢迎春, 等. 北京及其邻近地区野生鱼类物种多样性及其资源保育. 生物多样性, 2011, 19(5): 597—604]
[7] Frezal L, Leblois R. Four years of DNA barcoding: Current advances and prospects [J]. Infection Genetics and Evolution, 2008, 8(5): 727—736
[8] Bhattacharjee M J, Laskar B A, Dhar B, et al. Identification and re-evaluation of freshwater catfishes through DNA barcoding [J]. PLoS One, 2012, 7(11): e49950. doi:10.1371/ journal.pone.0049950
[9] Kochzius M, Seidel C, Antoniou A, et al. Identifying fishes through DNA barcodes and microarrays [J]. PLoS One, 2010, 5(9): e12620. doi:10.1371/journal.pone.0012620
[10] Peng J L, Wang X Z, He S P. The progress and application of DNA barcoding [J]. Acta Hydrobiologica Sinica, 2008, 32(6): 916—919 [彭居俐, 王绪祯, 何舜平. DNA条形码技术的研究进展及其应用. 水生生物学报, 2008, 32(6): 916—919]
[11] Wang X, Huang B. Advancement of DNA barcoding in animal taxonomy [J]. Biotechnology Bulletin, 2006, 4(4): 67—72 [王鑫, 黄兵. DNA条形编码技术在动物分类中的研究进展. 生物技术通报, 2006, 4(4): 67—72]
[12] Hebert PDN, Cywinska A, Ball S L, et al. Biological identifications through DNA barcodes [J]. Proceedings of the Royal Society B-Biological Sciences, 2003, 270(1512): 313—321
[13] Tavares E S, Baker A J. Single mitochondrial gene barcodes reliably identify sister-species in diverse clades of birds [J]. BMC Evolutionary Biology, 2008, 8(81) doi:10.1186/1471-2148-8-81
[14] Hebert PDN, Ratnasingham S, deWaard J R. Barcoding animal life: cytochrome c oxidase subunit 1 divergences among closely related species [J]. Proceedings of the Royal Society B-Biological Sciences, 2003, 270(Supplement 1): S96—S99
[15] Vences M, Thomas M, Bonett R M, et al. Deciphering amphibian diversity through DNA barcoding: chances and challenges [J]. Philosophical Transactions of the Royal Society B-Biological Sciences, 2005, 360(1462): 1859—1868
[16] Ward R D, Zemlak T S, Innes B H, et al. DNA barcoding Australia's fish species [J]. Philosophical Transactions of the Royal Society B-Biological Sciences, 2005, 360(1462): 1847—1857
[17] Witt JDS, Threloff D L, Hebert PDN. DNA barcoding reveals extraordinary cryptic diversity in an amphipod genus: implications for desert spring conservation [J]. Molecular Ecology, 2006, 15(10): 3073—3082
[18] Aquilino S V, Tango J M, Fontanilla I K, et al. DNA barcoding of the ichthyofauna of Taal Lake, Philippines [J]. Molecular Ecology Resources, 2011, 11(4): 612—619
[19] Lakra W S, Verma M S, Goswami M, et al. DNA barcoding Indian marine fishes [J]. Molecular Ecology Resources, 2011, 11(1): 60—71
[20] Mecklenburg C W, Møller P R, Dirk S. Biodiversity of arctic marine fishes: taxonomy and zoogeography [J]. Marine Biodiversity, 2011, 41(1): 109—140
[21] Ardura A, Linde A R, Moreira J C, et al. DNA barcoding for conservation and management of Amazonian commercial fish [J]. Biological Conservation, 2010, 143(6): 1438—1443
[22] Cawthorn D M, Steinman H A, Witthuhn R C. Establishment of a mitochondrial DNA sequence database for the identification of fish species commercially available in South Africa [J]. Molecular Ecology Resources, 2011, 11(6): 979—991
[23] Wong L L, Peatman E, Lu J, et al. DNA barcoding of catfish: species authentication and phylogenetic assessment [J]. PLoS One, 2011, 6(3), e17812. doi:10.1371/journal. pone.0017812
[24] Fu T Y, Ye M R. On a new species of the family CobitidaeLeptobotia microphthalma, SP. NOV [J]. Zoological Research, 1983, 4(2): 121—124 [傅天佑, 叶妙荣. 薄鳅属一新种——小眼薄鳅. 动物学研究, 1983, 4(2): 121—124]
[25] Fink S V, Fink W L. Interrelationship of ostariophysan fishes [J]. Zoological Journal of Linnean Society, 1981, 72(4): 297—353
[26] Kocher T D, Thomas W K, Meyer A, et al. Dynamics of mitochondrial-DNA evolution in animals - amplification and sequencing with conserved primers [J]. Proceedings of the National Academy Sciences of the U S A, 1989, 86(16): 6196—6200
[27] Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide-sequences [J]. Journal of Molecular Evolution, 1980, 16(2): 111—120
[28] Saccone C, De Giorgi C, Gissi C, et al. Evolutionary genomics in Metazoa: the mitochondrial DNA as a model system [J]. Gene, 1999, 238(1): 195—209
[29] Kumar S. Molecular clocks: four decades of evolution [J]. Nature Reviews Genetics, 2005, 6(8): 654—662
[30] Kottelat M. Conspectus Cobitidum: An inventory of the loaches of the world (Teleostei: Cypriniformes: Cobitoidei) [J]. Raffles Bulletin of Zoology, 2012, Supplement (26): 1—175
DNA BARCODES AND MOLECULAR PHYLOGENY OF BOTIINAE FISHES BASED ON THE MITOCHONDRIAL COⅠ GENE
MAO Yun-Tao1,2, GAN Xiao-Ni1and WANG Xu-Zhen1
(1. The Key Laboratory of Aquatic Biodiversity and Conservation of Chinese Academy, Institute of Hydrobiology, Chinese Academy of Sciences, Wuhan 430072, China; 2.University of Chinese Academy of Sciences, Beijing 100049, China)
To analyze the characteristics of the sequences and to calculate the genetic distance within and between species, the 648 bp mitochondrial COⅠ gene was used as DNA barcode to analyze phylogenetic relationships of Botiinae fishes in 131 sequences from 19 species representing 7 genera of Botiinae. MEGA5.0 software was used in the current study. Phylogenetic analyses were carried out using neighbor-joining and Bayesian methods. Our result revealed that the nucleotide compositions were 24.4% A, 29.5% T, 18.0% G, and 28.1% C. The mean inter- and intra-specific genetic distances were 0.148±0.008 and 0.002±0.000, respectively. Individuals of each species were clustered into monophyletic groups based on the molecular phylogenetic tree, indicating that the mitochondria COⅠ gene is suitable for species discrimination. Moreover, the phylogenetic analyses supported monophyly of genera within Botiinae. Two clades were recovered within the subfamily Botiinae: one clade includes the sister-group of Leptobotia and Parabotia and the other contains Botia + Chromobotia and Sinibotia + (Syncrossus, Yasuhikotakia). This study indicated that the mitochondria COⅠ gene is a potentially effective marker for DNA barcoding and phylogenetic studies of Botiinae fishes.
DNA barcoding; COⅠ gene; Botiinae; Species identification
Q949+.1
A
1000-3207(2014)04-0737-08
10.7541/2014.104
2013-05-28;
2013-11-23
国家自然科学基金项目(31272290)资助
毛云涛(1988—), 男, 湖北仙桃人; 硕士; 主要从事鱼类学研究。E-mail: maoyuntao1988@163.com
王绪祯, E-mail: wangxzh@ihb.ac.cn

