大戟科Lhcb基因家族的全基因组鉴定、分类与进化分析
邹 智,安 锋,杨礼富,王真辉,袁 坤
(中国热带农业科学院橡胶研究所 农业部橡胶树生物学与遗传资源利用重点实验室,海南 儋州 571737)
大戟科Lhcb基因家族的全基因组鉴定、分类与进化分析
邹 智,安 锋,杨礼富,王真辉,袁 坤
(中国热带农业科学院橡胶研究所 农业部橡胶树生物学与遗传资源利用重点实验室,海南 儋州 571737)
基于已公布的基因组和EST数据,研究对蓖麻、麻疯树、木薯和橡胶树4种大戟科植物的Lhcb基因家族进行系统鉴定,并在此基础上分析其基因结构与进化关系。结果表明,蓖麻、麻疯树、木薯和橡胶树分别含有10、10、13和15个Lhcb基因,分属于Lhcb1、Lhcb2、Lhcb3、Lhcb4、Lhcb5、Lhcb6、Lhcb7和Lhcb8等8个亚家族,每个亚家族包含1~4个成员不等,基因的内含子数目在0~5个之间,部分基因还存在可变剪接形式。进化分析显示,Lhcb1、Lhcb4、Lhcb5和Lhcb7亚族早于单细胞的藻类中就已产生,Lhcb3和Lhcb6亚族在光合生物向陆地进化后产生,Lhcb2亚族存在于高等植物中,Lhcb8亚族则为双子叶植物所特有;Lhcb1亚族在所分析的4种大戟科植物中均出现了基因扩增,Lhcb2和Lhcb5亚族在木薯和橡胶树中出现了扩增,而Lhcb3和Lhcb6亚族仅在橡胶树中出现了扩增。
大戟科植物;全基因组;Lhcb基因家族;鉴定;分类;进化分析
LHCΙΙ(light-harvesting chlorophyll a/b-binding proteins of photosystem ΙΙ)是植物光系统 ΙΙ(PSΙΙ)中与色素分子结合的一系列膜蛋白,由Lhcb基因家族编码,含有保守的叶绿素结合(chlorophyllbinding, CB)结构域,它们除进行光能的捕获与传递外,还广泛参与了激发能在PSΙ和PSΙΙ之间的调节与分配、类囊体膜结构的维持、光保护以及对各种环境的应答等过程[1-5]。虽然Lhcb基因家族已在拟南芥Arabidopsis thaliana、水稻Oryza sativa、杨树Populus trichocarpa等模式植物中得到了系统鉴定,并分为Lhcb1、Lhcb2、Lhcb3、Lhcb4、Lhcb5、Lhcb6、Lhcb7和 Lhcb8等 8个亚家族[6-8],但在以高光效和高生物量著称的大戟科(Euphorbiaceae)植物中,至今还未见Lhcb类基因的报道。研究基于蓖麻Ricinus communis、麻疯树Jatropha curcas、木薯Manihot esculenta、橡胶树Hevea brasiliensis 等4种大戟科植物最新释放的全基因组序列[9-14],对其Lhcb基因进行了全面鉴定,并在此基础上分析了其基因的结构、编码蛋白的生化特性及进化关系,以期为下一步的功能研究与利用提供参考。
1 材料与方法
1.1 材 料
莱茵衣藻Chlamydomonas reinhardtii、小立碗藓Physcomitrella patens、拟南芥、水稻、蓖麻、木薯的全基因组序列下载于phytozome v9.0(http∶//www.phytozome.net/);麻疯树和橡胶树的全基因组序列,蓖麻、麻疯树、木薯和橡胶树的cDNA与 EST 序 列 下 载 于 NCBΙ(http∶//www.ncbi.nlm.nih.gov/)。
1.2 方 法
1.2.1 蓖麻、麻疯树、木薯和橡胶树 Lhcb基因的鉴别与分类
根据先前的研究结果[6-8,15-16],首先从莱茵衣藻、小立碗藓、水稻和拟南芥的基因组中调取了已报道的Lhcb基因,然后从每个亚家族中选取1个成员作为查询序列,采用tBLASTn程序[17](E值设为10-5)分别搜索莱茵衣藻、小立碗藓、水稻和拟南芥的基因组,蓖麻、麻疯树、木薯和橡胶树的基因组、cDNA及EST序列,候选基因翻译成蛋白后再确认其是否存在跨膜螺旋和CB结构域。
1.2.2 蓖麻、麻疯树、木薯和橡胶树 LHCΙΙ的生化特性、进化分析及系统命名
采用在线软件 ProtParam(http∶//web.expasy.org/protparam/)预测蛋白的理论分子量(molecular weight, Mw)、等电点 (theoretical pΙ)、总平均亲水 性(grand average of hydropathy, GRAVY);采用在线软件ChloroP软件(http∶//www.cbs.dtu.dk/services/ChloroP/)预测蛋白的信号肽。用ClustalW2[18]进行多序列比对后,运用MEGA 4.0(http∶//megasoftware.net)[19]采 用 Neighbor-Joing法构建系统发生树,bootstrap值设为1 000;最后遵循拟南芥中的命名规则及进化关系对基因和蛋白进行系统命名。
2 结果与分析
2.1 蓖麻、麻疯树、木薯和橡胶树Lhcb基因的鉴别与分类
综合同源搜索及已报道的研究结果,分别从莱茵衣藻、小立碗藓、水稻和拟南芥的基因组中鉴定出12、23、9和15个Lhcb基因,其中Cre02.g110750.t1.2为本研究首次鉴定,鉴于它与拟南芥的Lhcb7同源(蛋白水平的相似性高达51%),将其命名为CrLhcb7(表1)。
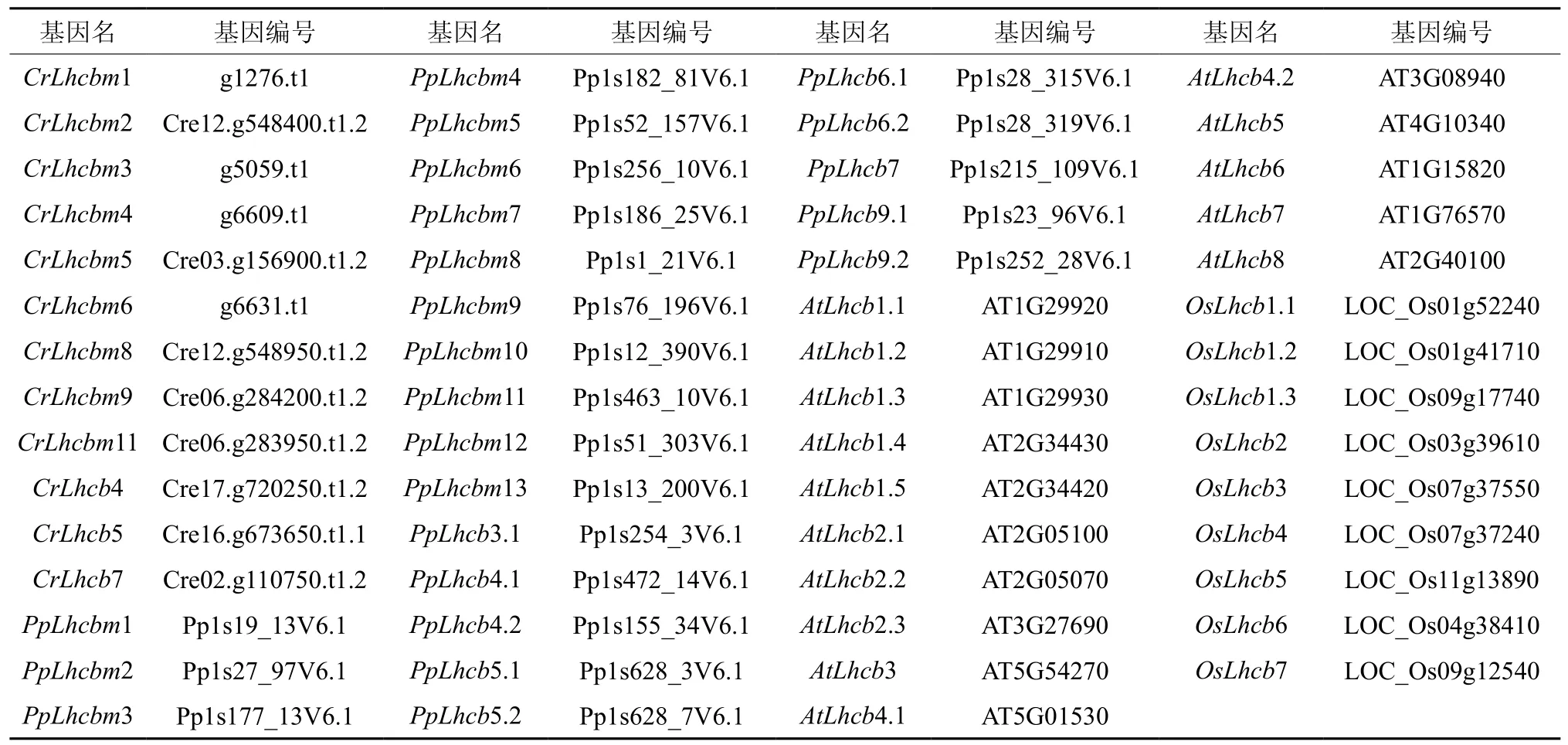
表1 莱茵衣藻、小立碗藓、水稻和拟南芥基因组中鉴定的Lhcb基因Table 1 List of Lhcb genes identified from genome of Creinhardtii, moss, Arabidopsis and rice
综合同源搜索和CB结构域分析,研究分别从蓖麻、麻疯树、木薯和橡胶树的基因组和EST序列中鉴定出10、10、13和15个Lhcb基因,分 属 于 Lhcb1、Lhcb2、Lhcb3、Lhcb4、Lhcb5、Lhcb6、Lhcb7和Lhcb8等8个亚家族(表2)。
从基因结构来来看,这些基因包含0~5个内含子不等,除Lhcb5亚族外,同一亚族的基因具有相同的内含子数目,如Lhcb1亚族基因都无内含子,Lhcb2、Lhcb4、Lhcb6和Lhcb8亚族基因都含有1个内含子,Lhcb3亚族基因都含有2个内含子,Lhcb7亚族基因都含有5个内含子。基因编码区长度在507~3 950 bp之间,cDNA编码区长度在 507 ~ 1 014 bp 之间(表 2)。
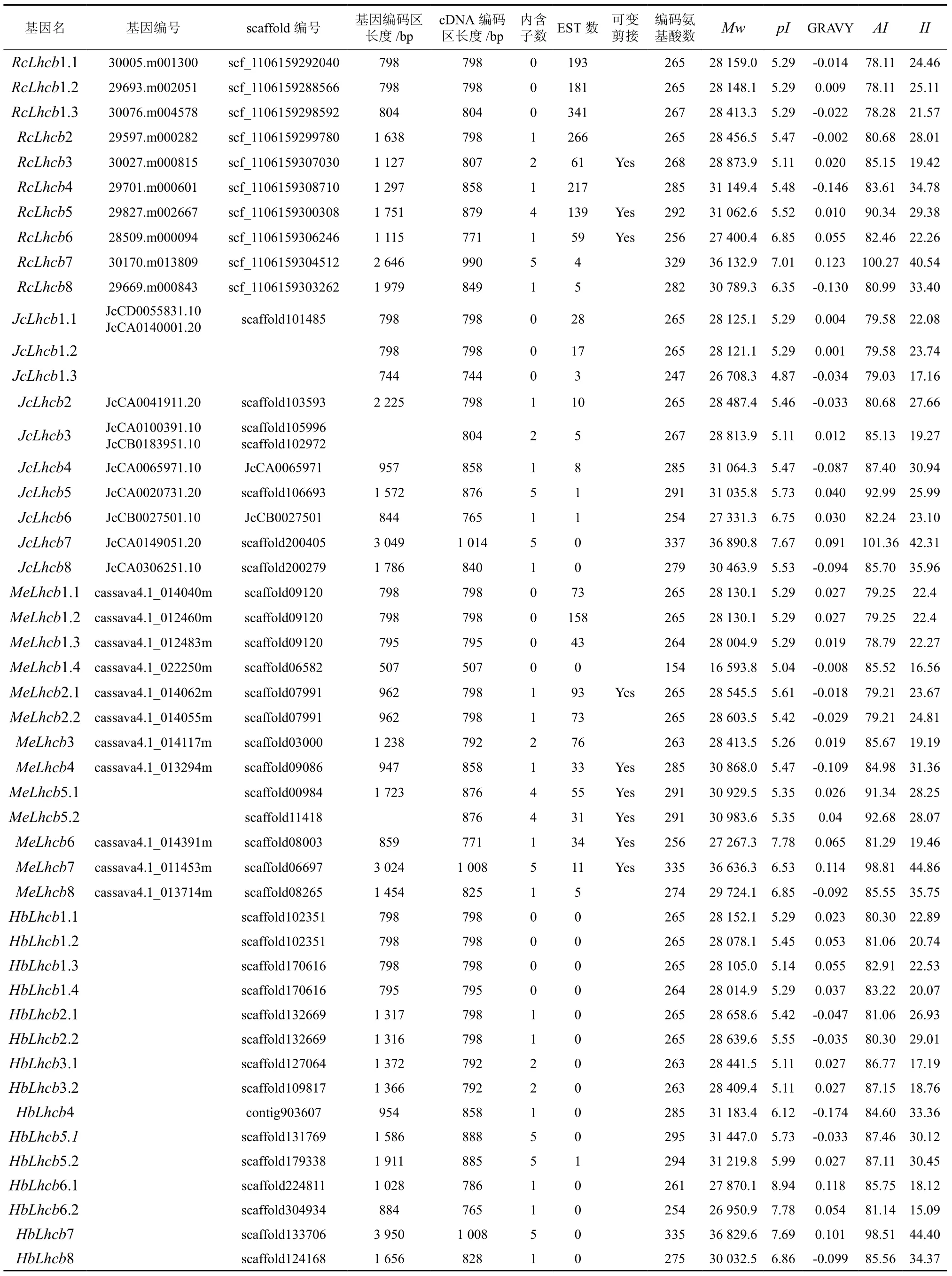
表2 蓖麻、麻疯树、木薯和橡胶树基因组中鉴定的Lhcb基因Table 2 List of Lhcb genes identified from genome of Castor bean, Physic nut, Cassava and Rubber tree
从基因分布来看,在4个物种中,不同亚族的基因都散布于不同的scaffold上,而对于同一亚族的基因,木薯和橡胶树中多数位于相同的scaffold上(表2)。
从EST分布来看,蓖麻所有的Lhcb基因在NCBΙ中都存在EST,麻疯树和木薯中分别有80%和92.31%的基因存在EST,而橡胶树中只有6.67%的基因存在EST。此外,目前的EST数据显示,蓖麻和木薯分别有30%和46.15%的基因存在可变剪接(表2)。
2.2 蓖麻、麻疯树、木薯和橡胶树 LHCΙΙ的生化特性、进化分析及系统命名
蓖麻LHCΙΙ蛋白最长的包含329个氨基酸残基,最短的有256个残基,其Mw值在27.40~ 36.13 kDa之 间;pI值 在 5.11~ 7.01之 间;GRAVY值在-0.146~0.123之间,其中,亲水性蛋白(即GRAVY<0)的约占50.00%;AI值在78.11~100.27之间;II值在19.42~40.54之间,不稳定蛋白(即II>40)约占10.00%(表3)。
麻疯树LHCΙ蛋白最长的包含337个氨基酸残基,最短的有247个残基,其Mw值在26.71~ 36.89 kDa之 间;pI值 在 4.87~ 7.67之 间;GRAVY值在-0.094~0.091之间,其中,亲水性蛋白约占40.00%;AI值在79.03~101.36之间;II值在17.16~42.31之间,不稳定蛋白约占10.00%(表3)。
木薯LHCΙ蛋白最长的包含335个氨基酸残基,最短的有154个残基,其Mw值在16.60到36.64 kDa之间;pI值在5.04到7.78之间;GRAVY值在-0.109到0.114之间,其中,亲水性蛋白的约占38.46%;AI值在78.79到98.81之间;II值在16.56到44.86之间,不稳定蛋白约占7.69%(表3)。
橡胶树LHCΙ蛋白最长的包含335个氨基酸残基,最短的有254个残基,其Mw值在26.95~ 36.83 kDa之 间;pI值 在 5.11~ 8.94之 间;GRAVY值在-0.174~0.118之间,其中,亲水性蛋白的约占33.33%;AI值在80.30~98.51之间;II值在15.09~44.40之间,不稳定蛋白约占6.67%(表3)。
为揭示大戟科植物Lhcb基因的起源与进化,研究用生物进化史上的重要物种——莱茵衣藻(可进行光合作用的单细胞生物)、小立碗藓(早期的陆地生物)、水稻(单子叶模式植物)、拟南芥(双子叶模式植物)与蓖麻、麻疯树、木薯和橡胶树的LHCΙΙ蛋白共同构建了进化树,结果显示,这些蛋白聚成4大类,即Lhcb1与Lhcb2亚族聚成1类,Lhcb4、Lhcb6与Lhcb8亚族聚成1类,Lhcb5和Lhcb7亚族聚成1类,Lhcb3单独聚成1类(图1)。
在由Lhcb4、Lhcb6和Lhcb8亚族聚成的大类中,蛋白以亚族为单位形成3个明显的分支。在Lhcb4亚族中,不仅有小立碗藓的PpLhcb4.1和PpLhcb4.2,同时还存在CrLhcb4,这表明该亚族早在单细胞的绿藻中就已经存在,显示出较早的起源,并且该亚族在小立碗藓和拟南芥中都出现过基因扩增。Lhcb6亚族最早出现在苔藓类生物,并在小立碗藓、木薯和橡胶树中都出现过基因扩增。Lhcb8只存在于双子叶植物中,应为单、双子叶植物分化后才进化产生。
在由Lhcb5和Lhcb7亚族与莱茵衣藻的CrLhcb5和CrLhcb7聚成的大类中,虽然序列比对表明CrLhcb5 和CrLhcb7在拟南芥中相似性最高的分别为AtLhcb5和AtLhcb7(其相似性分别为58.00%和51.00%,但CrLhcb5并没有像PpLhcb5.1 和 PpLhcb5.2一样聚入 Lhcb5分支,CrLhcb7也没有像PpLhcb7一样聚入Lhcb7分支。相对而言,Lhcb7亚族较为保守,在分析的所有物种中均为单拷贝,而Lhcb5亚族在小立碗藓、木薯和橡胶树中都出现过基因扩增。
在由Lhcb1与Lhcb2亚族聚成的大类中,存在7个明显的分支,其中,水稻、拟南芥、蓖麻、木薯和橡胶树的Lhcb2蛋白形成1个分支,莱茵衣藻和小立碗藓的相应蛋白各自形成独立的分支,拟南芥的5个Lhcb1蛋白形成1个分支,蓖麻有2个Lhcb1蛋白与水稻的Lhcb1蛋白形成1个分支,橡胶树有2个Lhcb1蛋白形成1个分支,麻疯树和木薯的Lhcb1蛋白与蓖麻和橡胶树的部分Lhcb1蛋白形成1个分支(图1)。
Lhcb3亚族中含有1个小立碗藓Lhcb蛋白,表明该亚族应为光合生物向陆地进化后产生,并且在后期进化中较为保守,因为该亚族除在橡胶树中出现过基因加倍外,其在小立碗藓、水稻、拟南芥、蓖麻和木薯中均为单拷贝。
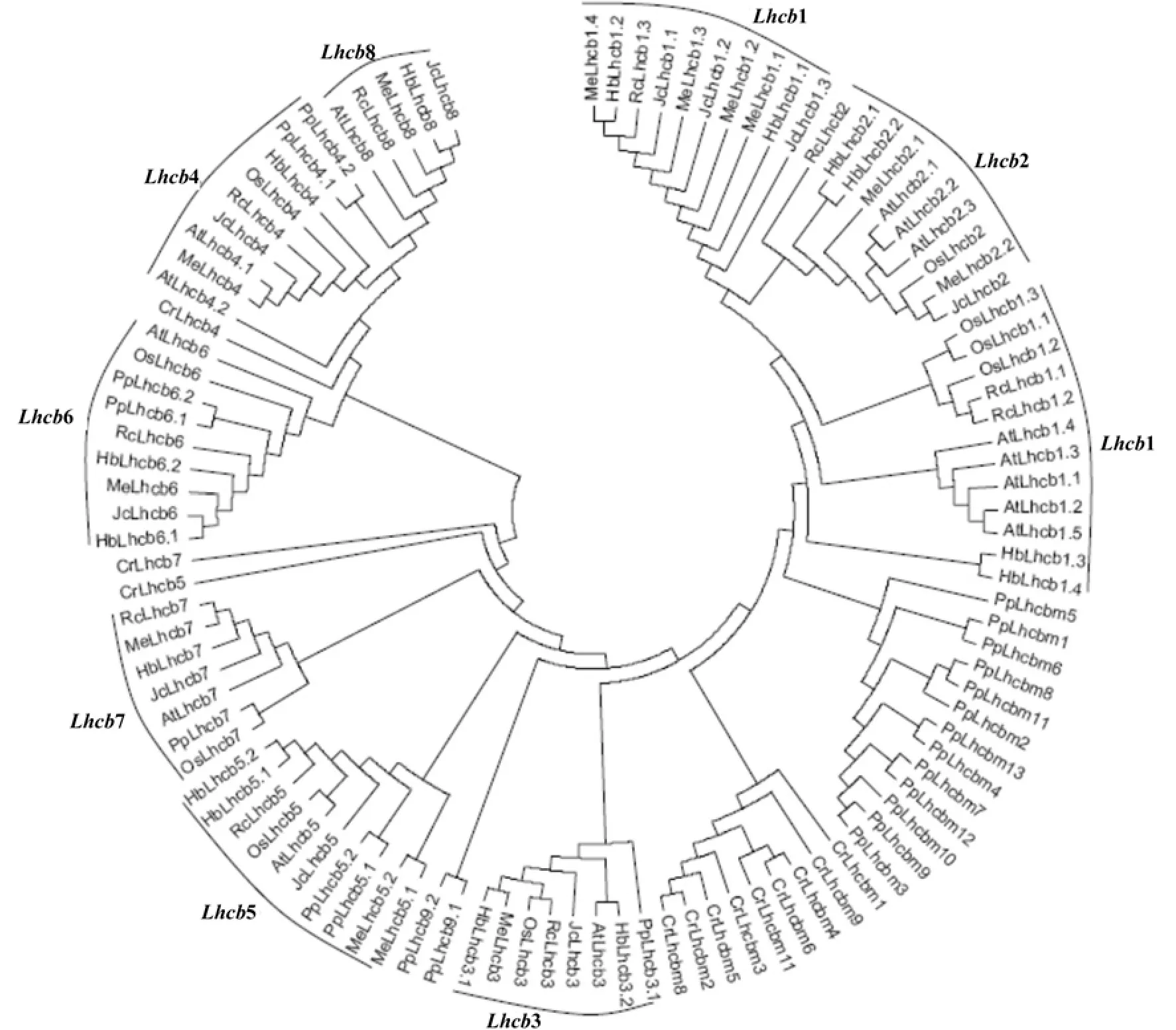
图1 LHCΙ蛋白的进化分析Fig. 1 Phylogenetic tree of Lhcbs from Castor bean, Physic nut, Cassava and Rubber tree with homologous proteins from other species such as Creinhardtii, Moss, Arabidopsis and Rice
最后,遵循拟南芥中的命名规则,将蓖麻、麻疯树、木薯和橡胶树的Lhcb基因和蛋白按着进化关系依次命名如表2和图1所示。
3 讨 论
光合作用是绿色植物最重要的生物学过程,它们通过利用捕光色素蛋白复合体接收太阳能来同化CO2。捕光色素蛋白复合体由色素分子及与之结合的捕光叶绿素a/b结合蛋白(LHC)构成。虽然植物体内的光系统Ι(PSΙ)和PSΙΙ都含有各自的 LHC(即 LHCΙ和 LHCΙΙ),但 LHCΙΙ以蛋白含量丰富、生理功能复杂、且易被大量提取而倍受关注。LHCΙΙ最早发现于1975年[20],随后其部分编码基因于1981年作为首批基因被克隆[21],后来随着一些高等植物基因组序列的释放,Lhcb基因家族先后在拟南芥、杨树、水稻等模式植物中得到了系统鉴定[6-8]。在目前已鉴 定 的 Lhcb1、Lhcb2、Lhcb3、Lhcb4、Lhcb5、Lhcb6、Lhcb7和Lhcb8等8个亚家族中,根据表达丰度的高低,Lhcb1、Lhcb2和Lhcb3早期被称为 主 要 LHCΙΙ,Lhcb4(CP29)、Lhcb5(CP26)和Lhcb6(CP24)被称为微量LHCΙΙ;Lhcb7后来由Klimmek等[7]鉴定并命名;Lhcb8在拟南芥中最早被命名为AtLhcb4.3,但因其序列及表达调控模式与AtLhcb4.1和AtLhcb4.2具有较大差异而被重新命名[7]。1994年,AtLhcb1 3.4 Å的晶体结构被揭示,结果显示,Lhcb1-色素蛋白复合体中有80%的组分为多肽(含有232个氨基酸残基,包含3个跨膜α-螺旋),另外还含有12个叶绿素(7个Chla,5个Chlb)和2个叶黄素分子[20]。
大戟科是一个包含300余属8 000多种植物的大科,其中不乏蓖麻、麻疯树、木薯、橡胶树、油桐、乌桕和一品红等重要经济作物,该科植物的一个显著特征就是光合效率特别高。鉴于LHCΙΙ在植物光合作用中的重要作用,研究对蓖麻、麻疯树、木薯和橡胶树等已完成全基因组测序的4个大戟科植物的Lhcb基因家族进行了全面鉴定,结果显示:蓖麻、麻疯树、木薯和橡胶树分别含有10、10、13和15个Lhcb基因,分属于Lhcb1、Lhcb2、Lhcb3、Lhcb4、Lhcb5、Lhcb6、Lhcb7 和Lhcb8等8个亚家族;总体而言,蓖麻和麻疯树中的基因数目和组成都与水稻相近,而木薯和橡胶树中的基因数目和组成与拟南芥较为相近,虽然多数亚族主要以单拷贝形式存在,但成员数最多的也高达4个。
通过将蓖麻、麻疯树、木薯和橡胶树的LHCΙΙ与莱茵衣藻、小立碗藓、水稻、拟南芥等模式生物中的同源蛋白共同构建系统发生树,研究发现Lhcb的8个亚家族具有不同的起源和进化速度。虽然Lhcb4、Lhcb6与Lhcb8亚族聚在一起,具有较近的亲缘关系,Lhcb4亚族早于单细胞的藻类中就已产生,并在小立碗藓、拟南芥和杨树中出现过基因扩增;Lhcb6亚族在光合生物向陆地进化后产生,在小立碗藓、杨树、木薯和橡胶树中出现过基因扩增;Lhcb8亚族在单、双子叶植物分化后产生,且为双子叶植物所特有。
Lhcb5和Lhcb7亚族具有较近的亲缘关系,它们在单细胞的藻类中都已存在。在后来的进化中,Lhcb7亚族相对保守,没有出现过明显的基因扩增,而Lhcb5亚族则在小立碗藓、木薯和橡胶树中均出现过基因扩增现象。
Lhcb1、Lhcb2与Lhcb3亚族具有较近的亲缘关系,它们可能由藻类来源的原始祖先向3个方向进化而成,其中,Lhcb1最为原始,并且进化速度也最快,目前已在不同物种中进化形成了数个分支;Lhcb3亚族可能是该大类最早分化出来的一支,进化最先发生于苔藓类生物,目前业已形成独立的1类,其在杨树和橡胶树中出现过基因扩增;在高等植物中,Lhcb2亚族可能通过内含子的获得而分化出来,其在杨树、木薯和橡胶树中均出现过基因扩增。
总之,蓖麻、麻疯树、木薯和橡胶树的鉴定不仅丰富了Lhcb基因家族的成员数,同时也为下一步阐明这4种乃至其它大戟科植物高光效的分子机理奠定了良好的基础。
[1]孙钦秒 , 冷 静 , 李良璧 , 等 . 高等植物光系统Ⅱ捕光色素蛋白复合体结构与功能研究的新进展[J]. 植物学通报, 2000, 17(4)∶ 289-301.
[2]de Bianchi S, Betterle N, Kouril R, et al. Arabidopsis mutants deleted in the light-harvesting protein Lhcb4 have a disrupted photosystem ΙΙ macrostructure and are defective in photoprotection [J]. Plant Cell, 2011, 23(7)∶ 2659-2679.
[3]Marin A, Passarini F, van Stokkum ΙH, et al. Minor complexes at work∶ light-harvesting by carotenoids in the photosystem ΙΙ antenna complexes CP24 and CP26 [J]. Biophys J, 2011, 100(11)∶ 2829-2838.
[4]Xia Y, Ning Z, Bai G, et al. Allelic variations of a light harvesting chlorophyll a/b-binding protein gene (Lhcb1) associated with agronomic traits in barley [J]. PLoS One, 2012, 7(5)∶ e37573.
[5]Xu Y H, Liu R, Yan L, et al. Light-harvesting chlorophyll a/b-binding proteins are required for stomatal response to abscisic acid in Arabidopsis [J]. J Exp Bot, 2012, 63(3)∶ 1095-1106.
[6]Jansson S. A guide to the Lhc genes and their relatives in Arabidopsis [J]. Trends Plant Sci, 1999, 4(6)∶ 236-240.
[7]Klimmek F, Sjödin A, Noutsos C, et al. Abundantly and rarely expressed Lhc protein genes exhibit distinct regulation patterns in plants [J]. Plant Physiol, 2006, 140(3)∶ 793-804.
[8]Umate P. Genome-wide analysis of the family of light-harvesting chlorophyll a/b-binding proteins in Arabidopsis and rice [J]. Plant Signal Behav, 2010, 5(12)∶ 1537-1542.
[9]Chan A P, Crabtree J, Zhao Q, et al. Draft genome sequence of the oilseed species Ricinus communis [J]. Nat Biotechnol, 2010, 28(9)∶ 951-956.
[10] Sato S, Hirakawa H, Ιsobe S, et al. Sequence analysis of the genome of an oil-bearing tree, Jatropha curcas L [J]. DNA Res, 2011, 18(1)∶ 65-76.
[11] Prochnik S, Marri P R, Desany B, et al. The Cassava Genome∶ Current Progress, Future Directions [J]. Trop Plant Biol, 2012, 5(1)∶ 88-94.
[12] Rahman A Y, Usharraj A O, Misra B B, et al. Draft genome sequence of the rubber tree Hevea brasiliensis [J]. BMC Genomics, 2013, 14∶ 75.
[13] 陈 梅 , 李培旺 , 蒋丽娟 . 外源激素对蓖麻营养生长及花芽分化的影响 [J]. 中南林业科技大学学报 , 2011, 31(7)∶ 86-90.
[14] 丁 勇 , 范红波 , 张高磊 , 等 . 麻疯树种子总 RNA 提取方法研究 [J]. 中南林业科技大学学报 , 2012, 32(3)∶ 158-161.
[15] Elrad D, Grossman A R. A genome's-eye view of the lightharvesting polypeptides of Chlamydomonas reinhardtii [J]. Curr Genet, 2004, 45(2)∶ 61-75.
[16] Alboresi A, Caffarri S, Nogue F, et al. Ιn silico and biochemical analysis of Physcomitrella patens photosynthetic antenna∶ identif i cation of subunits which evolved upon land adaptation [J]. PLoS One, 2008, 3(4)∶ e2033.
[17] Altschul S F, Madden T L, Schäffer A A, et al. Gapped BLAST and PSΙ-BLAST∶ a new generation of protein database search programs [J]. Nucleic Acids Res, 1997, 25(17)∶ 3389-3402.
[18] Thompson J D, Higgins D G, Gibson T J. CLUSTAL W∶ improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice [J]. Nucleic Acids Res, 1994, 22(22)∶ 4673-4680.
[19] Tamura K, Dudley J, Nei M, et al. MEGA4∶ Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0 [J]. Mol Biol Evol, 2007, 24(8)∶ 1596-1599.
[20] Thornber J P. Chlorophyll-Proteins∶ Light-Harvesting and Reaction Center Components of Plants[J]. Annu Rev Plant Physiol, 1975, 26∶ 127-158.
[21] Broglie R, Bellemare G, Bartlett S G, et al. Cloned DNA sequences complementary to mRNAs encoding precursors to the small subunit of ribulose-1,5-bisphosphate carboxylase and a chlorophyll a/b binding polypeptide [J]. Proc Natl Acad Sci USA, 1981, 78∶ 7304-7308.
[22] Kühlbrandt W, Wang D N, Fujiyoshi Y. Atomic model of plant light-harvesting complex by electron crystallography [J]. Nature, 1994, 367(6464)∶ 614-621.
Genome-wide identif i cation, classif i cation and phylogenetic analysis of Lhcb gene family in Euphorbiaceae plants
ZOU Zhi, AN Feng, YANG Li-fu, WANG Zhen-hui, YUAN Kun
(Key Laboratory of Biology and Genetic Resources of Rubber Tree, Ministry of Agriculture/Rubber Research Ιnstitute (RRΙ), Chinese Academy of Tropical Agricultural Sciences (CATAS), Danzhou 571737, Hainan, China)
Euphorbiaceae is a large plant family containing more than 8 000 species which are characterized with high photosynthesis and high biomass. The recently available genome sequences of some Euphorbiaceae plants such as castor bean (Ricinus communis), physic nut (Jatropha curcas), cassava (Manihot esculenta) and rubber tree (Hevea brasiliensis) provide an opportunity to analyze specif i c gene families. Ιn this study, a genome-wide search was carried out to identify Lhcb gene family, which encodes several chlorophyllbinding proteins def i ned as LHCΙΙ involved in light harvesting and energy transfer to the reaction center of photosystem ΙΙ (PSΙΙ), in these four Euphorbiaceae plants. As a result, a number of 10, 10, 13 and 15 Lhcb genes representing 8 subgene families named Lhcb1, Lhcb2, Lhcb3, Lhcb4, Lhcb5, Lhcb6, Lhcb7 and Lhcb8 were identif i ed from castor bean, physic nut, cassava and rubber tree, respectively. These genes contain 0~5 introns, and some of them were found to have alternative splicing isoforms. Phylogenetic analysis suggests a early origin of subgene family Lhcb1, Lhcb4, Lhcb5 and Lhcb7 up to unicellular algae, and subfamily Lhcb3 and Lhcb6 appear in land organisms, while subfamily Lhcb2 and Lhcb1 presents only in high plants and dicotyledons, respectively. Ιn all four analyzed Euphorbiaceae plants, gene amplif i cation was found in subfamily Lhcb1, while gene amplif i cation of subfamily Lhcb2 and Lhcb5 were only found in cassava and rubber tree plants. Ιn rubber trees, amplif i cation events also occured in subfamily Lhcb2 and Lhcb5.
Euphorbiaceae plant; genome-wide; Lhcb gene family; identif i cation; classif i cation; phylogenetic analysis
S718.46
A
1673-923X(2013)12-0046-07
2013-04-12
国家自然科学基金(31100460,31100460);海南省重点科技项目(90107);海南省自然科学基金(312026)和中国热带农业科学院橡胶研究所基本科研业务费专项(1630022011014)
邹 智(1982-),男,湖南新化人,硕士,助理研究员,研究方向为分子生物学;E-mail:zouzhi2008@126.com
[本文编校:文凤鸣]

