基于核密度估计的动物生境适宜度制图方法
张桂铭,朱阿兴 ,杨胜天,秦承志,肖 文,Steve K.Windels
(1.北京师范大学地理学与遥感科学学院,北京 100875;2.中国科学院地理科学与资源研究所资源与环境信息系统国家重点实验室,北京 100101;3.美国威斯康星大学麦迪逊分校地理系,麦迪逊 WI53706;4.大理学院东喜玛拉雅资源与环境研究所,大理 671003;5.美国Voyageurs国家公园,国际瀑布城 MN56649)
动物生境是动物生存和繁衍的场所,为动物提供资源、庇护所、筑巢位置和交配场所[1]。自然界的动物经过长期的生境选择都形成了自己特定的生境,生境质量的优劣,对于物种的延续和繁衍非常重要[2]。生境适宜度是衡量生境质量的重要指标,生境适宜度制图能提供野生动物适宜生境的空间分布信息,从而为野生动物种群管理及保护地规划提供决策依据[3]。
生境适宜度制图是通过构建生境适宜度模型(HSM)来实现的[4]。生境适宜度模型表达生境适宜度与环境因子之间的关系,可以简单地写成HS=f(E),式中HS为生境适宜度;E为环境因子的集合;f为生境适宜度和环境因子之间的数量关系,一般通过对动物生境利用数据(例如出现位置数据)的分析来建立。随着地理信息系统(GIS)和遥感技术的发展,快速获取和分析大量环境因子数据以刻画动物生存的环境特征已经成为可能,它们目前已经在种群动态的时空分析与模拟、野生动物生境等方面得到越来越多的应用[5-8]。根据建模所需动物出现位置数据的不同,HSM可以分为两类:第一类只需要动物的出现位置数据来构建的模型,第二类同时需要动物的出现位置和不出现位置数据来构建的模型。虽然有研究表明第二类模型的精度要高于第一类模型[9],但是由于动物不出现位置的数据通常难以获取并且精度难以保证[10],因此只需要动物出现位置数据进行建模的第一类HSM的应用更加广泛[11]。目前,收集动物出现位置数据的手段包括野外直接跟踪观测、痕迹法、无线电遥测定位、热红外相机自动感应照相以及GPS追踪定位技术等[12-15]。运用这些方法与技术,研究者能够获得丰富的动物出现位置数据,但如何利用这些动物出现位置数据并结合环境因子数据提取准确的生境适宜度与环境因子之间的数量关系f仍然是一个值得研究的问题。
生境适宜度和环境因子之间的数量关系f可以通过分析动物出现与环境因子之间的关系来建立[16]。现有的只基于动物出现位置数据分析f的方法主要包括早期发展起来的生物气候分析预测系统(BIOCLIM)[17]和环境距离模型DOMAIN[18],以及当前国内外应用最为广泛的生态位因子分析法(ENFA)[19]、规则集生成遗传算法(GARP)[20]和最大熵法(MaxEnt)[21]。生境适宜度与环境因子之间的关系可能相当复杂,这体现在生境适宜度与各个环境因子之间的数量关系往往为非线性且不符合固定的函数形式[22],而基于环境包络线的BIOCLIM和基于环境距离的DOMAIN等简单方法尚不能表达这种复杂的非线性关系。基于最大熵理论的MaxEnt和基于遗传算法的GARP都是机器学习方法,虽然它们通常能够很好地拟合数据并表达生境适宜度与环境因子之间复杂的非线性数量关系,但是通常这种数量关系及其生态学意义都是隐含的。基于生态位理论的ENFA首先将环境因子进行主成分变换得到几个相互独立的生态位因子,然后在这些生态位因子的基础上计算生境适宜度值,虽然这个计算过程有一定的生态学依据,但是由于该方法依赖于将环境因子进行主成分变换后得到的生态位因子,所以也不能直接表达生境适宜度与各个环境因子之间的数量关系。综上所述,现有的只基于动物出现位置数据分析f的方法还不能很好地直接表达动物生境适宜度和各个环境因子之间具有生态学意义的数量关系,也就不能很好地体现各个环境因子对动物生境利用的生态学作用,从而导致模型的可解释性较差。
本文提出了一种基于核密度估计构建HSM进行动物生境适宜度制图的方法。该方法首先运用核密度估计对动物出现位置数据进行分析,估计出动物出现对各个环境因子的概率密度函数来直接表达动物生境适宜度与各个环境因子之间具有生态学意义的数量关系;然后对生境适宜度与多个环境因子之间的数量关系进行综合构建HSM;最后在GIS支持下基于构建的 HSM进行生境适宜度制图。本文运用该方法对美国Voyageures国家公园的白尾鹿(Odocoileus virginianus)进行了生境适宜度制图的案例研究以验证方法的有效性。
1 理论基础
自然界的动物具有可移动性特征,能够通过生境选择来调整其与环境之间的相互关系,使其处于最佳状态[23]。可以假定动物在某种环境条件下出现的概率大小直接指示了它在这种环境条件下的生境适宜度高低[24]。因此可以用动物出现对某一环境因子的概率密度函数(PDF)来表达动物生境适宜度与该环境因子之间具有生态学意义的数量关系并体现该环境因子对动物生境利用的生态学作用。
在自然界中,环境因子对生物不会单独地、孤立地起作用,各个环境因子对于生物是共同起作用的,即因子的综合作用[25]。动物生境是各种环境因子的组合,因此需要按照一定的原则对动物生境适宜度与各个环境因子之间的数量关系进行综合,才能得到动物在某种环境特征条件(由多个环境因子刻画)下的生境适宜度。
2 研究方法
2.1 基于核密度估计的HSM构建方法
2.1.1 生境适宜度与单个环境因子之间数量关系的建立
动物出现位置数据中包含了动物分布与环境因子之间关系的信息,可以通过对它们的分析来估计动物出现对各个环境因子的PDF。如引言部分所述,动物出现位置数据可以通过多种手段获得,然而无论运用哪种手段,往往都是按照一定的采样方法进行的,由此得到的动物出现位置数据是这种动物分布数据总体中的样本。
核密度估计(KDE)是一种能从样本估计总体的PDF的方法。一方面,应用KDE估计总体PDF不需要引入对数据分布的先验假设,只从样本本身出发获取数据分布特征,因此可以用来估计任意形状的PDF;另一方面,相比于直方图等其他密度估计方法,通过KDE得到的PDF连续性更好,并且不会依赖于选取的区间长度[26-27]。KDE的基本表达式为:

式中,f(x)为根据环境因子x在n个出现位置处的值x1,x2,…,xn估计得到的动物出现对环境因子x的PDF;K为核函数;h为带宽,其取值影响PDF曲线的形状和平滑程度[28]。本文选用高斯核作为核函数,并根据经验法则计算带宽 h[27,29]:
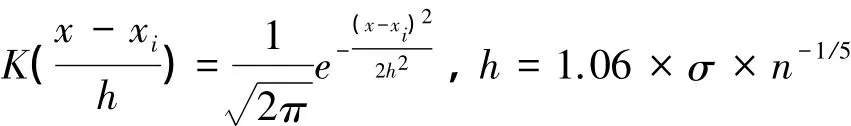
式中,σ为样本标准差,n为出现位置个数。
利用动物出现位置数据和环境因子数据,运用核密度估计方法得到动物出现对每一个环境因子的PDF(对于类别型环境因子数据,如地表覆被类型,直接用频率分布统计来估计PDF),对这些PDF进行归一化处理,作为动物生境适宜度和每一个环境因子之间具有生态学意义的数量关系:
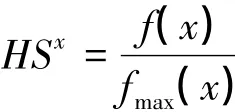
式中,f(x)是动物出现对环境因子x的PDF;fmax(x)是概率密度函数f(x)(也即PDF)的最大值;HSx是将该PDF归一化后得到的结果。
2.1.2 生境适宜度与多个环境因子之间数量关系的综合
各个环境因子对动物生境适宜度的影响是综合起作用的,生境适宜度建模方法体现这种因子综合作用通常采用“加权平均”或“最小限制因子”的处理方式。采用“加权平均”的处理方式需要依据专家知识赋予各个环境因子不同权重来表达它们对动物生境适宜度的不同贡献,然后加权平均计算生境适宜度值[30-31]。因此,采用“加权平均”的处理方式要求对各个环境因子影响动物生境适宜度的相对重要性有深入的认识并且能够给出权重将这种知识定量化。然而,这方面的专家知识通常难以获得而且往往具有较强的主观性[30]。“最小限制因子”处理方式基于利比希的“最小因子定律”[25],具有一定的生态学理论依据。采用“最小限制因子”的处理方式只需要根据少量定性专家知识挑选出适当的影响动物生境适宜度的环境因子,并不要求将各个环境因子的重要性进行定量化,可以避免引入过多的主观性。因此,在对于环境因子影响动物生境适宜度的知识不够全面或不够准确的情况下,“最小限制因子”的处理方式不失为一种简单而行之有效的方法[32-33]。所以,本文按照“最小限制因子”对生境适宜度与各个环境因子之间的关系进行综合,以对各个环境因子生境适宜度值中的最小值作为最终的动物生境适宜度值:

式中,HSj为位置j处的动物生境适宜度值;HSkj是位置j处动物对第k个环境因子的生境适宜度值;m为环境因子个数。
2.2 基于HSM的生境适宜度制图
基于以上构建HSM的思路,可分三步计算研究区内每一个栅格处动物的生境适宜度值:首先基于动物出现位置数据结合环境因子数据,运用核密度估计建立动物生境适宜度和单个环境因子之间的数量关系;然后根据这些数量关系以及每个栅格处的环境因子值计算动物对各个环境因子的生境适宜度值;最后以对各个环境因子生境适宜度值中的最小值作为该栅格的生境适宜度值。
2.3 生境适宜度图验证
本文运用Hirzel等提出的连续Boyce指数[34]对生境适宜度制图结果进行定量评价。该指数的计算只需要以动物出现位置作为验证数据,它常被用来评价只基于动物出现位置数据构建的HSM及生境适宜度制图结果。
连续Boyce指数是对Boyce指数[35]的改进。计算Boyce指数时首先将生境适宜度分成r个等级(如r=10),然后依次计算验证数据中所在位置生境适宜度值落入第l(l=1,2,…,r)级生境适宜度范围的动物出现位置点频率Pl和期望频率El的比值Fl:

其中
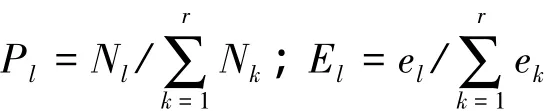
式中,Pl为验证数据中值落入第l级生境适宜度范围的Nl个动物出现位置点个数占验证数据的动物出现位置点总数的比例;El为生境适度图上值落入第l级生境适宜度范围的栅格数el占生境适宜度图栅格总数的比例。如果HSM能正确地预测出适宜生境分布,则Fl的值应该随着生境适宜度等级l的上升而单调增加。Boyce指数Br即用于度量这种单调增加的趋势,它是Fl和l之间的Spearman等级相关系数,取值范围在-1.0到1.0之间。Br>0且越接近1.0表示基于生境适宜度模型的生境适宜度制图结果与验证数据中的动物出现位置数据分布符合越好(即验证数据中越多的出现位置落在了预测的生境适宜度高的区域),模型预测能力越强;Br<0表示模型预测的生境适宜度图与验证数据分布相反;Br=0表示模型的预测能力和随机模型相当。
Boyce指数的一个不足之处是它对生境适宜度的分级数目r和边界很敏感,为此Hirzel等[34]提出连续Boyce指数Bcont(w)作为对它的改进。Hirzel等[34]在宽度为w(如w=0.1)的滑动窗口内计算Fl:第1次计算的生境适宜度等级区间为[0,w];第2次计算的生境适宜度等级区间为[Δx,Δx+w](Δx为窗口滑动距离,本文取 Δx=0.01;w 为窗口宽度,本文取 w=0.1),…,第 l次计算的生境适宜度等级区间为[(l-1)Δx,(l-1)Δx+w],…;直到(l-1)Δx≥1.0时计算结束。由此计算Fl和l之间的Spearman等级相关系数就得到Bcont(w),它的取值范围及对模型预测能力的指示意义均和Boyce指数相同。
3 应用案例
3.1 研究区
Voyageurs国家公园(48°18'N—48°38'N,92°27'W—93°11.5'W)位于美国明尼苏达州北部美国和加拿大交界处。公园总面积约为828 km2,其中湖泊和其他水域面积约占总面积的40%;公园内地势平缓,最大地形起伏为80—90 m[36]。公园内分布的主要哺乳动物除了白尾鹿外,还有驼鹿(Alces alces)、灰狼(Canis lupus)、黑熊(Ursus americanus)和山猫(Felis rufus)。灰狼是白尾鹿冬季主要的捕食者,黑熊和山猫只会在春季和夏季捕食白尾鹿的幼崽。白尾鹿和驼鹿是公园内仅有的两种有蹄类动物,但是它们之间并不存在对食物和生存空间的竞争关系[37]。公园内白尾鹿当前的种群数量大约为2000—3000只。
3.2 出现位置数据
公园管理部门于2009、2010和2011连续3a冬季(具体时间:2009年1月16、24—25、27日;2010年1月11—13日;2011年1月18—20日)以空中调查[38]的方式对公园内的驼鹿和白尾鹿的种群数量和分布状况进行了调查[39]。调查将公园全区分为33个调查单元(面积为9.8—27.0 km2)[36]并对所有调查单元都进行了调查。对每个调查单元进行野外调查时,搭载着观察员的小型飞机沿着事先设计好的西—东向平行样线飞行(样线间距约550 m,飞行速度125—145 km/h,飞行高度150—215 m,采样强度约1.5 min/km2),观察员沿途记录观察到的驼鹿或白尾鹿的性别、年龄、种群数量和位置等信息。其中观察到的驼鹿或白尾鹿所在的位置通过参照高分辨率航空摄影影像图和大比例尺地形图上的山峰、沟谷、湖泊、湿地和道路等特征来确定,以点的形式标注在影像图或地形图上。后续室内处理时将野外调查记录的原始数据进行了数字化,得到公园内白尾鹿出现位置点总共391个(图1)。
391个白尾鹿出现位置点中有26个点落入了离湖边不远的湖泊内。调查在冬季(1月)进行,此时公园内湖面已经冻结,由于湖边冰面上的积雪较浅,白尾鹿在上面行走相对容易,这些落入湖泊的点是白尾鹿在冰面上行走时被记录的点。考虑到白尾鹿出现在冰面上仅仅是为了行走方便而不会在冰面上停留进行取食等其他活动,这些点对于白尾鹿的生境适宜度并无指示意义,因此本文剔除这26个点,只基于剩余的365个白尾鹿出现位置点进行分析。
3.3 环境特征的刻画
选择环境因子刻画白尾鹿生活的环境特征遵循两个原则:一是要依据已有知识选择影响其生境利用的主要环境因子,二是用于刻画环境因子的数据可以获得。已有对白尾鹿冬季生境选择和利用的研究[40-42]表明:白尾鹿冬季生境选择和利用一般会尽量避开积雪较深的地方,地表覆被类型会影响白尾鹿对冬季庇护所的选择,白尾鹿在冬季倾向于到森林边界处觅食。此外,研究区内地形起伏不大(最大地形起伏为80—90 m),海拔高度变化对白尾鹿的生境利用的影响较小,但局部地区陡峭的地形仍然会限制白尾鹿的生境利用。除了这些影响白尾鹿冬季庇护所选择、取食条件和活动通达性的环境因子外,人为干扰因素对白尾鹿的生境选择和利用的影响很小(Voyageurs国家公园属于自然保护区);白尾鹿与驼鹿之间不存在对食物和生存空间的竞争[37];白尾鹿冬季的生境选择和利用会受捕食者灰狼的影响[43],但目前无法获得相关数据来刻画这一捕食者因素。因此本文选择1月平均积雪深度、地表覆被类型、半径150 m圆形区域内的森林边界线长度(后文称为森林边界长度)、坡度等4个环境因子来刻画冬季白尾鹿生活的环境特征(表1)。
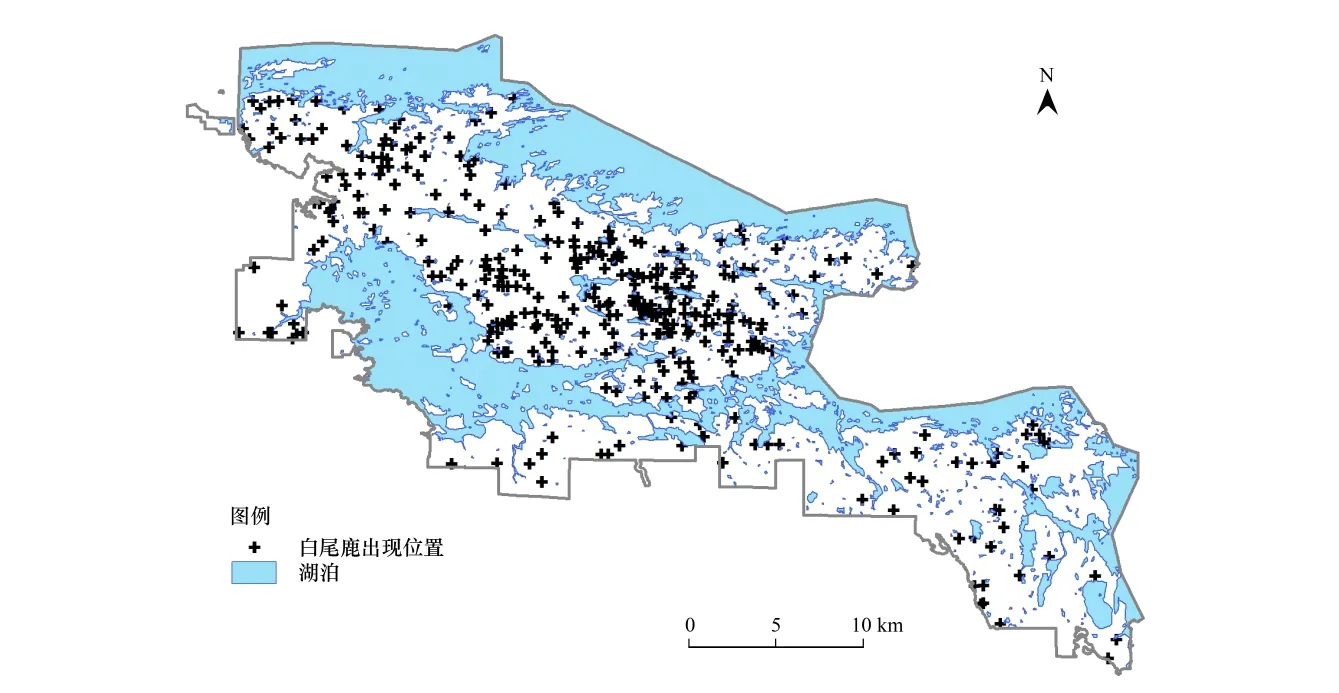
图1 白尾鹿出现位置Fig.1 White-tailed deer occurrence data in study area
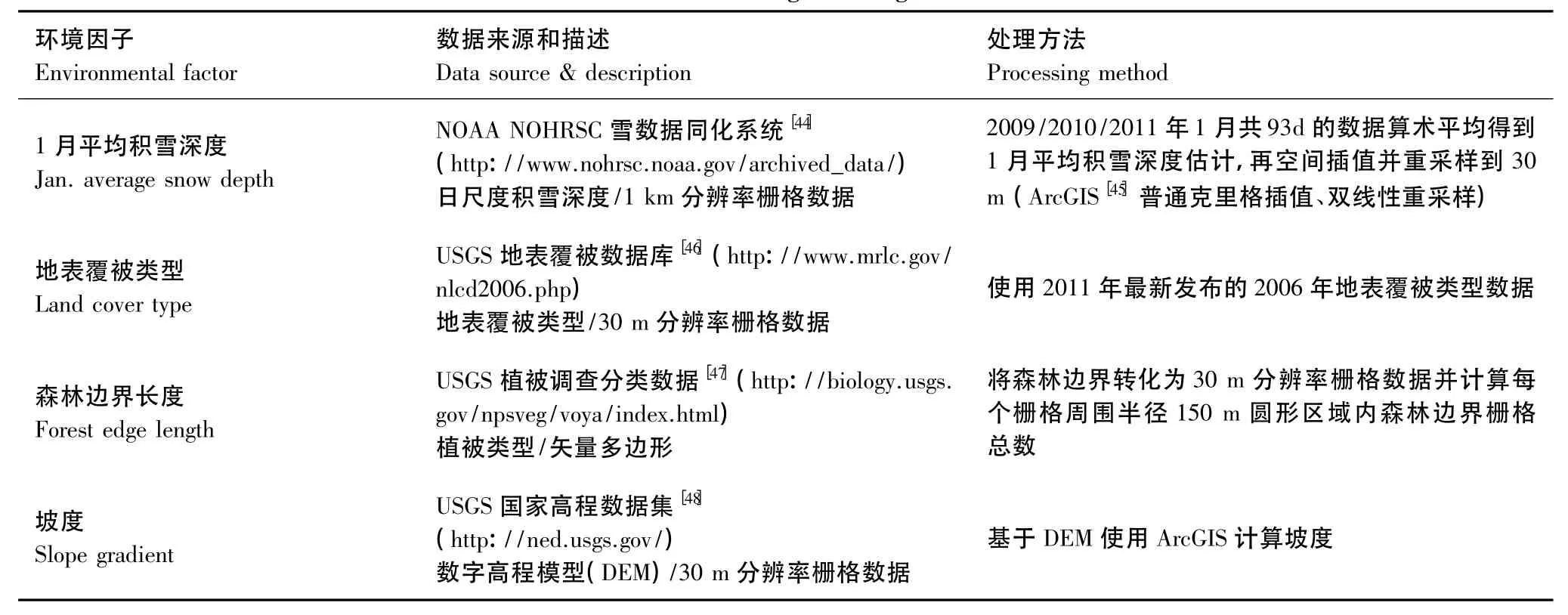
表1 刻画白尾鹿生活环境特征的环境因子Table 1 Environmental factors characterizing the living environment of White-tailed deer
3.4 生境适宜度图
基于365个出现位置结合环境因子数据运用核密度估计方法建立了白尾鹿生境适宜度与各个环境因子之间的数量关系(图2),在此基础上构建HSM进行生境适宜度制图得到了研究区内白尾鹿的生境适宜度图(图3)。由图2可知,公园内白尾鹿生境适宜度最高的区域1月平均积雪深度在0.42 m左右、地表覆被类型为混合林或落叶林、150 m圆形区域内处于森林边界总长度在1050—1500 m(即35—50个栅格)之间、坡度约在3°左右。生境适宜度图上几处范围较大的低值区主要由积雪深度这一环境因子决定:这些区域的1月平均积雪较深,不宜于白尾鹿的生境利用;生境适宜度图上的高值区分布呈现较破碎的斑块状,一方面是由于众多湖泊和河流水域的分割,另一方面是环境因子(坡度、地表覆被类型、森林边界长度)空间异质性较强造成的。
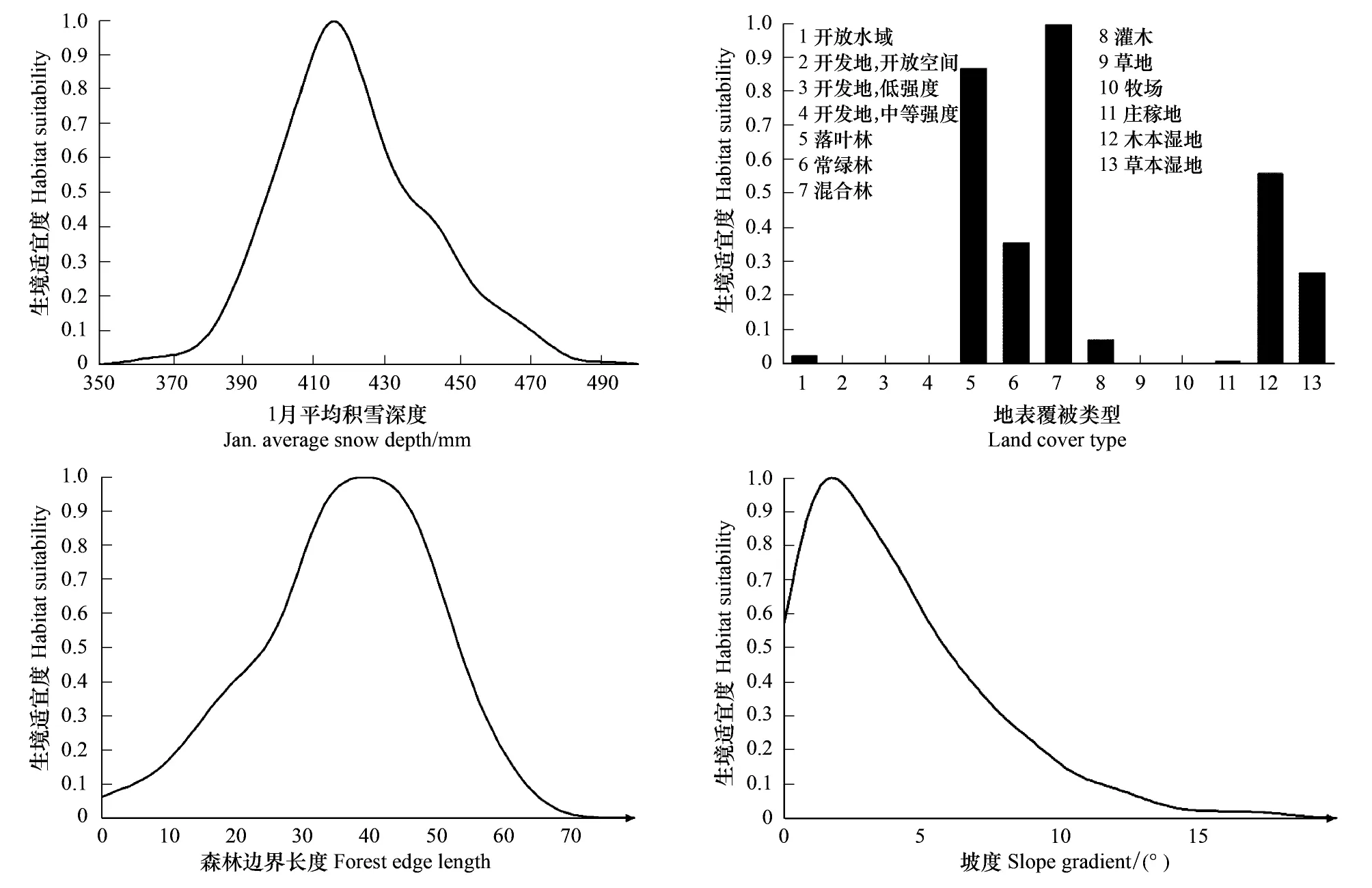
图2 白尾鹿生境适宜度与各个环境因子的数量关系Fig.2 The quantitative relationship between White-tailed deer habitat suitability and environmental factors
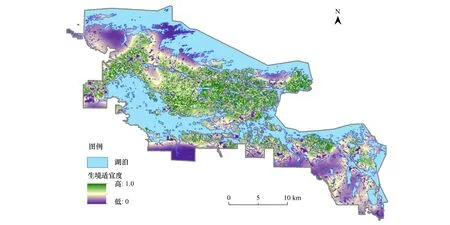
图3 白尾鹿生境适宜度图Fig.3 White-tailed deer habitat suitability map
3.5 验证
由于缺少独立获取的出现位置作为验证数据,本文以交叉验证[49]的方式计算连续Boyce指数对生境适宜度制图结果进行定量评价,检验基于核密度估计进行生境适宜度制图方法的有效性。每次将365个白尾鹿出现位置点随机平均分为两组[50],其中一组用于构建生境适宜度模型进行生境适宜度制图,另一组作为验证数据计算连续Boyce指数Bcont(0.1)。该过程重复10次,计算得到10个相应的Bcont(0.1)值(表2)。10次交叉验证Bcont(0.1)的平均值到达了0.75(标准差为0.11),表明生境适宜度制图结果达到了较高精度,按照本文方法构建的生境适宜度模型预测能力强(Bcont(0.1)达到0.7以上即可视为表现优秀[34])。

表2 10次交叉验证计算的连续Boyce指数B cont(0.1)Table 2 Continuous Boyce Indexes calculated from 10 repetitions of 2 folds cross validation
4 结论与讨论
4.1 本文方法的优点
本文基于代表性动物出现位置结合环境因子数据,运用核密度估计得到动物出现对各个环境因子的PDF来表达动物生境适宜度与各个环境因子之间的数量关系,然后对这些关系进行综合构建HSM进行生境适宜度制图,验证表明生境适宜度制图结果达到了较高精度,按照本文方法建立的HSM预测能力强。此外,该方法能建立并直接表达生境适宜度和环境因子之间具有生态学意义的数量关系(图2),以此体现环境因子对动物生境利用的生态学作用,就模型的可解释性而言,本文方法要优于MaxEnt、GARP、ENFA等其他构建HSM的方法,有助于增进对动物生境选择和生境利用的认识。
目前,研究者或野生动物保护部门可以利用多种技术和手段方便地收集动物出现位置数据,GIS和遥感技术的发展也使得快速获取各种环境因子数据成为可能。本文方法只需要动物出现位置数据和相应环境因子数据即可进行动物生境适宜度制图服务于野生动物保护工作,因此,与那些同时需要动物出现位置和不出现位置进行HSM建模的方法相比,本文方法在保护工作中也具有更大的实际应用价值。
4.2 应中用需要注意的问题
4.2.1 对动物出现位置数据的要求
基于核密度估计进行动物生境适宜度制图方法的关键是根据有限的动物出现位置数据样本估计出动物出现对各个环境因子的PDF,以此表达动物生境适宜度与环境因子之间的数量关系。为了使得到的动物生境适宜度与环境因子之间的数量关系能代表实际的动物生境适宜度与环境因子之间的关系,要求动物出现位置数据样本具有良好的代表性[51],即能代表动物出现的各种典型环境条件。如果收集动物出现位置数据时运用的采样方法没有偏差(如随机采样、规则采样),则收集的动物出现位置数据样本应该能代表动物出现的各种典型环境条件(文中使用的白尾鹿出现位置数据即是通过规则采样方式收集的代表性出现位置数据);反之,如果收集的出现位置数据样本代表性差,那么在此基础上运用核密度估计方法构建的HSM可能就无法正确预测该动物在某些环境条件下的生境适宜度。
此外,动物出现位置样本数量也可能影响基于本文方法构建HSM进行生境适宜度制图的精度。一般而言,用于HSM建模的动物出现位置样本数量越多越好。至于本文方法最少需要多少动物出现位置样本才能保证生境适宜度制图精度,本文没有设计专门的试验对该问题进行讨论,在后续研究中需要对该问题进行深入研究。
4.2.2 环境因子的选择
选择环境因子刻画动物生存的环境特征需要了解动物的生态学特征,基于动物生境选择和利用的知识选取那些对目标动物生境利用产生影响的主要环境因子。一般可以从生境的基本要素例如地形条件、庇护所、食物、水源、与其他物种之间的相互作用(竞争、捕食)等出发,选择相应的环境因子来刻画这些要素。本文应用案例选择环境因子时就是根据已有知识选择了1月平均积雪深度、地表覆被类型、森林边界长度和坡度四个影响白尾鹿冬季庇护所选择、取食条件和活动通达性环境因子来刻画白尾鹿生存的环境特征。此外,选择环境因子还会受到数据源的限制,虽然目前已经有越来越多的遥感和GIS数据可供选择,但有些用于刻画动物生境利用影响因素的数据仍然难以获得。在本文应用案例中,捕食者是白尾鹿冬季生境选择和利用的重要影响因素之一,但由于无法获得相应数据来刻画这一因素,在进行白尾鹿生境适宜度制图时并没有考虑其冬季主要捕食者灰狼对白尾鹿生境适宜度的影响。
4.2.3 对动物生境适宜度与多个环境因子之间的数量关系的综合
本文依据“最小限制因子”对动物生境适宜度与各个环境因子之间的数量关系进行综合,其中隐含假设了各个环境因子对动物生境适宜度的影响同等重要且各因子间不能相互补偿,其生态学理论依据是利比希的“最小因子定律”[25]。“加权平均”处理方式基于的假设是在影响动物生境适宜度时各个环境因子可以相互补偿[25],采用这种处理方式要求能够获得客观准确的关于环境因子之间的补偿作用机制、相对重要性的知识。然而,环境因子之间的补偿作用机制可能相当复杂而难以认知,实际工作中也难以获取全面准确的专家知识来确定各个环境因子的重要性权重并避免主观性[30]。因此,在选择环境因子时充分考虑动物的生态学特征只选择那些对其生境适宜度会产生显著影响的环境因子的前提条件下,依据“最小限制因子”对动物生境适宜度与各个环境因子之间的关系进行综合是一种简单可行的解决办法[32-33]。但是,无论采用哪一种处理方式来对动物生境适宜度与各个环境因子之间的数量关系进行综合,对所采用的处理方式的前提假设有清楚的认识将有助于正确理解由此产生的结果。
[1]Reunanen P,Monkkonen M,Nikula A.Habitat requirements of the Siberian flying squirrel in northern Finland:comparing field survey and remote sensing data.Annales Zoologici Fennici,2002,39(1):7-20.
[2]Dai Q,Gu H J,Wang Y Z.Theories and models for habitat selection.Zoological Research,2007,28(6):681-688.
[3]Jin L R,Sun K P,He H S,Zhou Y F.Research advances in habitat suitability index model.Chinese Journal of Ecology,2008,27(5):841-846.
[4]Guisan A,Zimmermann N E.Predictive habitat distribution models in ecology.Ecological Modelling,2000,135(2/3):147-186.
[5]Sanchez-Hernandez C,Boyd D S,Foody GM.Mapping specific habitats from remotely sensed imagery:support vector machine and support vector data description based classification of coastal saltmarsh habitats.Ecological Informatics,2007,2(2):83-88.
[6]Wang J L,Chen Y.Applications of 3Stechnology in wildlife habitat researches.Geography and Geo-Information Science,2004,20(6):44-47.
[7]Wang Z J,Zhang A B,Cheng JA,Li DM.The development of spatiotemporal analysis and simulation of population dynamics based on GIS.Acta Ecologica Sinica,2002,22(1):104-110.
[8]Tian B,Zhou Y X,Zhang L Q,Ma Z J,Yang B,Tang C D.A GIS and remote sensing-based analysis of migratory bird habitat suitability for Chongming Dongtan Nature Reserve,Shanghai.Acta Ecologica Sinica,2008,28(7):3049-3059.
[9]Václavík T,Meentemeyer R K.Invasive species distribution modeling(iSDM):are absence data and dispersal constraints needed to predict actual distributions?Ecological Modelling,2009,220(23):3248-3258.
[10]Gu W D,Swihart R K.Absent or undetected?Effects of non-detection of species occurrence on wildlife-habitat models.Biological Conservation,2004,116(2):195-203.
[11]Remm K.Case-based predictions for species and habitat mapping.Ecological Modelling,2004,177(3/4):259-281.
[12]Guan H L,Tong K G F.Review on satellite tracking of migratory birds and its prospect.Zoological Research,2000,21(5):412-415.
[13]Liu B W,Jiang Z G.Feces analysis and its role in wildlife research.Zoological Research,2002,23(1):71-76.
[14]Ma M,Xu F,Chundawat R S,Jumabay K,Wu Y Q,Ai Z Z,Zhu M H.Camera trapping of snow leopards for the photo capture rate and population size in the Muzat Valley of Tianshan Mountains.Acta Zoologica Sinica,2006,52(4):788-793.
[15]Harris S,Cresswell W J,Forde P G,Trewhella WJ,Woollard T,Wray S.Home-range analysis using radio-tracking data-a review of problems and techniques particularly as applied to the study of mammals.Mammal Review,1990,20(2/3):97-123.
[16]Ortigosa G R,de Leo G A,Gatto M.VVF:integrating modelling and GIS in a software tool for habitat suitability assessment.Environmental Modelling and Software,2000,15(1):1-12.
[17]Busby J R.BIOCLIM:a bioclimatic analysis and prediction system//Margules C R,Austin M P,eds.Nature Conservation:Cost Effective Biological Surveys and Data Analysis.East Melbourne,Australia:CSIRO,1991:64-68.
[18]Carpenter G,Gillison A N,Winter J.DOMAIN:a flexible modelling procedure for mapping potential distributions of plants and animals.Biodiversity and Conservation,1993,2(6):667-680.
[19]Hirzel A H,Hausser J,Chessel D,Perrin N.Ecological-niche factor analysis:how to compute habitat-suitability maps without absence data?Ecology,2002,83(7):2027-2036.
[20]Stockwell D.The GARP modelling system:problems and solutions to automated spatial prediction.International Journal of Geographical Information Science,1999,13(2):143-158.
[21]Phillips SJ,Anderson R P,Schapire R E.Maximum entropy modeling of species geographic distributions.Ecological Modelling,2006,190(3/4):231-259.
[22]Lek S,Delacoste M,Baran P,Dimopoulos I,Lauga J,Aulagnier S.Application of neural networks to modelling nonlinear relationships in ecology.Ecological Modelling,1996,90(1):39-52.
[23]Yan Z C,Chen Y L.Habitat selection in animals.Chinese Journal of Ecology,1998,17(2):43-49.
[24]Braunisch V,Bollmann K,Graf R F,Hirzel A H.Living on the edge-Modelling habitat suitability for species at the edge of their fundamental niche.Ecological Modelling,2008,214(2/4):153-167.
[25]Sun R Y.Principles of Animal Ecology.Beijing:Beijing Normal University Press,2001:19-29.
[26]Sheather SJ.Density estimation.Statistical Science,2004,19(4):588-597.
[27]Silverman B W.Density Estimation for Statistics and Data Analysis.London:Chapman and Hall,1986:34-72.
[28]Wand M P,Jones M C.Kernel Smoothing.London:Chapman and Hall,1995.
[29]Sheather SJ,Jones M C.A reliable data-based bandwidth selection method for kernel density estimation.Journal of the Royal Statistical Society.Series B(Methodological),1991,53(3):683-690.
[30]Robinson V B.A Perspective on the fundamentals of fuzzy sets and their use in geographic information systems.Transactions in GIS,2003,7(1):3-30.
[31]Bolívar-CiméB,Gallina S.An optimal habitat model for the White-tailed deer(Odocoileus Virginianus)in central Veracruz,Mexico.Animal Production Science,2012,52(8):707-713.
[32]Zhu A X,Band L E.A knowledge-based approach to data integration for soil mapping.Canadian Journal of Remote Sensing,1994,20(4):208-218.
[33]Zhu A X.Detail Digital Soil Survey:Models and Methods.Beijing:Science Press,2008:103-106.
[34]Hirzel A H,Le Lay G,Helfer V,Randin C,Guisan A.Evaluating the ability of habitat suitability models to predict species presences.Ecological Modelling,2006,199(2):142-152.
[35]Boyce M S,Vernier P R,Nielsen SE,Schmiegelow FK A.Evaluating resource selection functions.Ecological Modelling,2002,157(2/3):281-300.
[36]Gogan P JP,Kozie K D,Olexa E M,Duncan N S.Ecological status of moose and white-tailed deer at Voyageurs National Park,Minnesota.Alces,1997,33:187-201.
[37]Cobb M A,Gogan P JP,Kozie K D,Olexa E M,Lawrence R L,Route W T.Relative spatial distributions and habitat use patterns of sympatric moose and white-tailed deer in Voyageurs National Park,Minnesota.Alces,2004,40:169-191.
[38]Gasaway W C,Dubois S D,Reed D J,Harbo S J.Estimating moose population parameters from aerial surveys.University of Alaska Biological Papers,1986,22:1-108.
[39]Windels SK.2009 aerial moose survey protocol for Voyageurs National Park.Unpublished report,Voyageurs National Park,Minnesota,2009.
[40]Ozoga J J,Gysel L W.Response of White-tailed deer to winter weather.The Journal of Wildlife Management,1972,36(3):892-896.
[41]Blouch R I.Northern Great Lakes states and Ontario forests//Halls LK,ed.White-Tailed Deer Ecology and Management.Harrisburg:Stackpole Books,1984.
[42]Kie JG,Bowyer R T,Nicholson M C,Boroski B B,Loft E R.Landscape heterogeneity at differing scales:effects on spatial distribution of mule deer.Ecology,2002,83(2):530-544.
[43]National Park Service,U.S.Department of the Interior.Gray wolves in and adjacent to Voyageurs National Park,Minnesota:research and synthesis 1987-1991.[2012-04-12].http://www.nrmsc.usgs.gov/files/norock/products/VOYA_wolves_1987-1991.pdf.
[44]National Operational Hydrologic Remote Sensing Center.Snow Data Assimilation System(SNODAS)data products at NSIDC.[2012-03-20].http://www.nohrsc.noaa.gov/archived_data/.
[45]Environmental Systems Resource Institute.ArcMap version 10.0.[2012-05-15].http://resources.arcgis.com/content/arcgisdesktop/10.0/about.
[46]Fry JA,Xian G,Jin S,Dewitz JA,Homer CG,Yang L,Barnes C A,Herold N D,Wickham JD.Completion of the 2006 National Land Cover Database for the Conterminous United States.Photogrammetric Engineering and Remote Sensing,2011,77(9):858-864.
[47]U.S.Geological Survey.USGS-NPSVegetation Mapping Program.[2012-04-13].http://biology.usgs.gov/npsveg/voya/index.html.
[48]Gesch D,Oimoen M,Greenlee S,Nelson C,Steuck M,Tyler D.The national elevation dataset.Photogrammetric Engineering and Remote Sensing,2002,68(1):5-11.
[49]Hastie T,Tibshirani R,Friedman J H.The Elements of Statistical Learning:Data Mining,Inference,and Prediction.New York:Springer-Verlag,2001
[50]Beyer H L.Geospatial Modelling Environment.[2012-06-23].http://www.spatialecology.com/gme/.
[51]Phillips SJ,Dudík M,Elith J,Graham CH,Lehmann A,Leathwick J,Ferrier S.Sample selection bias and presence-only distribution models:implications for background and pseudo-absence data.Ecological Applications,2009,19(1):181-197.
参考文献:
[2]戴强,顾海军,王跃招.栖息地选择的理论与模型.动物学研究,2007,28(6):681-688.
[3]金龙如,孙克萍,贺红士,周宇飞.生境适宜度指数模型研究进展.生态学杂志,2008,27(5):841-846.
[6]王金亮,陈姚.3S技术在野生动物生境研究中的应用.地理与地理信息科学,2004,20(6):44-47.
[7]王正军,张爱兵,程家安,李典谟.基于GIS的种群动态的时空分析与模拟研究的方法进展.生态学报,2002,22(1):104-110.
[8]田波,周云轩,张利权,马志军,杨波,汤臣栋.遥感与GIS支持下的崇明东滩迁徙鸟类生境适宜性分析.生态学报,2008,28(7):3049-3059.
[12]关鸿亮,通口广芳.卫星跟踪技术在鸟类迁徙研究中的应用及展望.动物学研究,2000,21(5):412-415.
[13]刘丙万,蒋志刚.粪样在野生动物研究中的作用.动物学研究,2002,23(1):71-76.
[14]马鸣,徐峰,Chundawat R S,Jumabay K,吴逸群,艾则孜,朱玛洪.利用自动照相术获得天山雪豹拍摄率与个体数量.动物学报,2006,52(4):788-793.
[23]颜忠诚,陈永林.动物的生境选择.生态学杂志,1998,17(2):43-49.
[25]孙儒泳:动物生态学原理.北京:北京师范大学出版社,2001:19-29.
[33]朱阿兴.精细数字土壤普查模型与方法.北京:科学出版社,2008:103-106.

