基于28S, COI和Cytb基因序列的薜荔和爱玉子传粉小蜂分子遗传关系研究
吴文珊,陈友铃,孙伶俐,毛建萍,杨问新,王爱芳
(福建师范大学生命科学学院,福建省发育与神经生物学重点实验室,福州 350117)
基于28S,COI和Cytb基因序列的薜荔和爱玉子传粉小蜂分子遗传关系研究
吴文珊,陈友铃*,孙伶俐,毛建萍,杨问新,王爱芳
(福建师范大学生命科学学院,福建省发育与神经生物学重点实验室,福州 350117)
薜荔和爱玉子均属于桑科榕属植物,二者为同一物种的原变种与变种的关系,早期研究认为这两种榕树与同一种传粉榕小蜂(Wiebesiapumilae(Hill))建立了稳定的互利共生关系,但近期在形态学、生态学、传粉生物学等方面对二者的研究结果表明,薜荔传粉小蜂和爱玉子传粉小蜂之间可能发生了遗传分化。实验用核糖体28SrDNAD1-D3区、线粒体Cytb及COI基因部分序列,对采自福建3个不同样地的薜荔传粉小蜂和3个不同品系的栽培爱玉子的传粉小蜂进行分析,结果表明:(1)薜荔传粉小蜂和爱玉子传粉小蜂的核糖体28S序列的碱基组成中A,T,G,C 4种含量较平均,C+G的平均含量(56%)稍高于A+T的含量(44%)。线粒体Cytb序列中A+T的含量(76.1%)明显高于C+G的含量(23.9%),COI序列中A+T的含量(71.9%)也明显高于G+C的含量(28.1%),这是膜翅目昆虫线粒体基因的普遍特征。在薜荔和爱玉子传粉小蜂的线粒体Cytb及COI基因中,密码子第三位点A+T的含量最高。(2)比较薜荔和爱玉子传粉小蜂的3种分子标记的变异范围显示,28S进化速度较Cytb及COI序列慢,比较保守,更适合科、亚科等较高分类单元的研究。薜荔传粉小蜂与爱玉子传粉小蜂之间的亲缘关系较近,采用Cytb与COI序列进行分析更为精确。(3)用Cytb及COI序列对薜荔传粉小蜂与爱玉子传粉小蜂之间的遗传距离进行分析显示,薜荔传粉榕小蜂个体间Cytb序列平均遗传距离为0.0054,爱玉子传粉小蜂个体间的Cytb遗传距离为0.0164;薜荔传粉小蜂与爱玉子传粉小蜂群体之间的Cytb序列平均遗传距离为0.1385;COI序列的薜荔传粉榕小蜂个体间遗传距离为0.0048,爱玉子传粉小蜂各样本间平均遗传距离为0.0102;薜荔传粉小蜂与爱玉子传粉小蜂群体间COI序列平均遗传距离为0.1896,两群体间的遗传距离(差异大于10%以上)明显大于群体内各样本之间的遗传距离,表明薜荔传粉小蜂与爱玉子传粉小蜂之间已经发生了很大的遗传分化,其变异水平达到了种间分化水平,即薜荔传粉小蜂与爱玉子传粉小蜂为两个不同的种。
薜荔; 爱玉子; 榕小蜂; COI;Cytb;28S
薜荔(Ficuspumilavar.pumila)隶属桑科榕属,为攀援或匍匐灌木,主要分布于福建、江西、浙江、安徽、江苏、台湾、湖南、广东、广西、贵州、云南东南部、四川及陕西等地,被广泛用于园林的绿化[1- 2],爱玉子(Ficuspumilavar.awkeotsang)为薜荔的变种,是我国特有的植物,原产自台湾,在福建、浙江等地少量的野生分布,目前在台湾和大陆长江以南有广泛栽培[1,3]。
薜荔和爱玉子在植株性状、茎叶形态、花与花序的形态结构等方面均十分相似,二者究竟是原变种与变种的关系,拟或为不同的两个种,长期以来争论不休。李和惠用trnT-trnL序列和核内NIA-i3和ITS2序列对薜荔和爱玉子植株进行研究发现,核内NIA-i3和ITS2序列两者之间不存在差异,trnT-trnL序列上存在微小差异,认为两者为同一个种[4]。而林贊標的研究表明,爱玉子瘦果中的果胶甲酯酶的活性(95.8活性单位)比薜荔(4.8活性单位)高很多,且爱玉子果胶甲酯酶有两个亚型,在薜荔果胶甲酯酶只有一个亚型[5],暗示薜荔和爱玉子可能是亲缘关系很近的两个不同的种。
长期以来,人们一直认为薜荔与爱玉子的传粉小蜂为同一种传粉榕小蜂(Wiebesiapumilae(Hill))[6- 7]。1991年何坤耀从台湾各地采集的爱玉子及薜荔的传粉小蜂进行研究,也认为台湾地区的薜荔与爱玉子共享一种传粉小蜂,并且与香港的模式标本无形态差异,同时,还观察到在台湾存在薜荔的雄株的地方,栽培爱玉子均能成功繁殖[8]。但近年来,越来越多的证据表明爱玉子传粉小蜂与薜荔传粉小蜂之间可能已经发生了变异,例如,Chen 等发现福建爱玉子栽培园内即使种植薜荔雄株(有传粉小蜂),却不能使爱玉子结实,同时,薜荔、爱玉子繁殖生物学的调查表明,二者之间已经存在生殖隔离[9]。江少华在台湾高雄地区开展的薜荔和爱玉子之间的人工辅助杂交实验结果表明:1)薜荔的传粉小蜂可以进入爱玉子的雌花期花序内授粉并能结实,但无法进入爱玉子雄花序;2)爱玉子传粉小蜂可以进入薜荔的雌花期花序内授粉并能结实,也可以进入雄花序产卵,但幼虫的死亡率高,小蜂发育与花序发育异步,导致羽化的小蜂无法出飞[10]。随着分子生物学的发展,有关榕小蜂的分子生物学研究越来越多,李和惠对两个来自台湾不同地区的薜荔传粉小蜂和爱玉子传粉小蜂进行COI序列研究发现,两者的遗传距离为11.7%,认为两种小蜂已经发生了很大的变异[4]。同时,陈艳对我国东南沿海及岛屿不同地理分布的薜荔榕小蜂进行研究发现,薜荔榕小蜂存在Sp.A、Sp.B、Sp.C3个隐存种[11]。以上证据表明,亲缘关系密切的薜荔传粉小蜂和爱玉子传粉小蜂之间可能发生了遗传分化。
由于薜荔传粉小蜂与爱玉子传粉小蜂之间的形态差异极其细微,仅依赖传统的形态标记分类方法,难以将它们区别开来。薜荔传粉小蜂和爱玉子传粉小蜂究竟是同种?还是两个不同的种?或是互为隐种关系?这一直是悬而未决的问题。分子标记技术的快速发展,给这一难题的解决带来希望。本文用核糖体28SrDNAD1—D3,以及线粒体COI和 Cytb三种分子标记对分类地位模糊的薜荔和爱玉子传粉小蜂进行鉴定,明确薜荔传粉小蜂与爱玉子传粉小蜂之间的分子遗传关系,以期在爱玉子的经济栽培中起到指导性作用。
1 材料和方法
1.1 样本的采集及保存
从2010年3月至2012年12月间于福建省福州市、莆田市等地采集实验小蜂,样本及采集地点(表1)。将采集来的榕小蜂存放入含有无水乙醇保存液的1.5 mL的离心管中,使小蜂迅速死亡,置于-20 ℃保存。
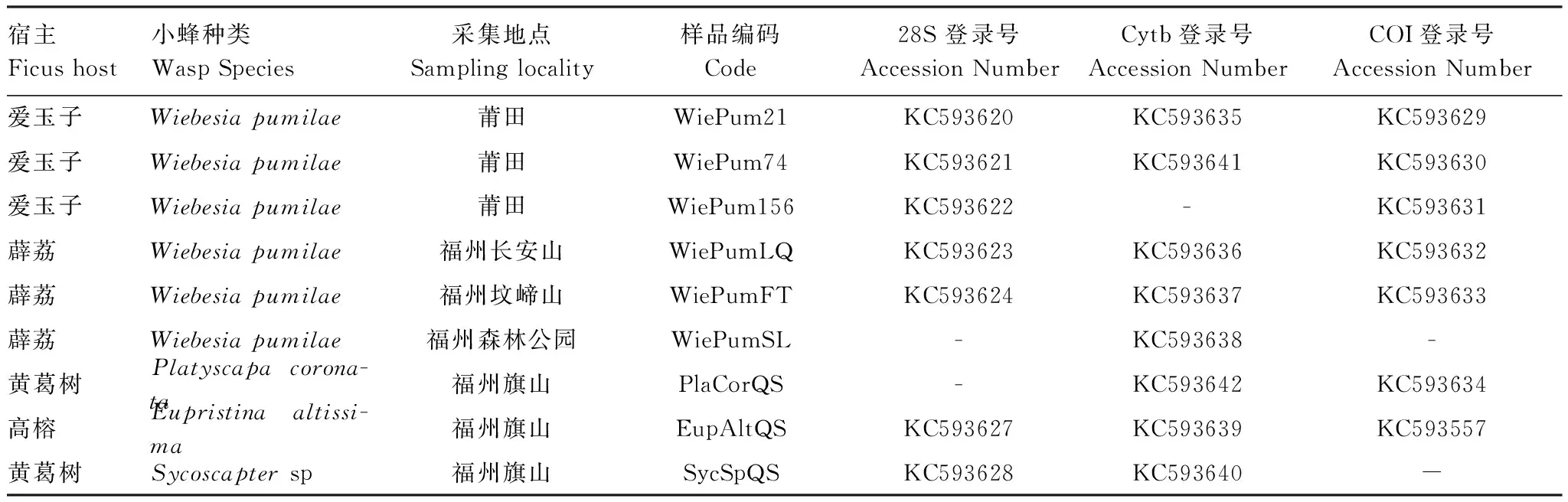
表1 实验材料及采集地及其登陆号
爱玉子:F.pumilavar.awkeotsang;薜荔:F.pumilavar.pumila;黄葛树:F.virens;高榕:F.altissima; “-”表示无此样本
1.2 基因组DNA的提取,PCR及测序
取95%的酒精-20 ℃下储存的榕小蜂样品,用酚氯仿抽提法进行基因组DNA提取。用提取的基因组DNA进行目的片段PCR扩增。
28S序列上游引物 28S-F:5′-ACCCGCTGAATTTAAGCATAT-3′
28S序列下游引物 28S-R:5′-TAGTTCACCATCTTTCGGGTC-3′
28S序列扩增条件为94 ℃预变性3 min,94 ℃变性60 s,60 ℃退火50 s,72 ℃延伸70 s,34个循环后72℃延伸7 min结束,4 ℃保温。
COI序列上游引物 COI-F:5′CAACATTTATTTTGATTTTTTGG3′
COI序列下游引物 COI-R:5′TCCAATGCACTAATCTGCCATATTA3′
COI序列PCR扩增条件为:94℃预变性3 min,94 ℃变性60 s,54 ℃退火40 s,72 ℃延伸80 s,34个循环后72 ℃延伸7 min结束,4 ℃保温。
Cytb序列上游引物 Cytb-F:5′TATGTACTACCATGAG GACAAATATC3′
Cytb序列下游引物 Cytb-R:5′ATTACACCTCCTAATTTATTAGGAAT3′
Cytb序列扩增条件为:94 ℃预变性4 min,94 ℃变性30 s,57 ℃退火60 s,72 ℃延伸60 s,34个循环后72 ℃延伸5 min结束,4 ℃保温。
对PCR扩增的产物直接进行测序,所有PCR产物均送至生工生物公司进行测序。将测序所得序列上传GenBank上。
1.3 数据处理
得到测序序列后,在NCBI上用BLAST进行相似性检索,确定所得的序列确实为目的片段。将这些片段用CLUSTAL X 1.83进行序列比对, BioEdit 3.3校正比对结果,截取相同长度的序列进行分析,运用MEGA 4.0软件计算不同序列的碱基组成及碱基替换,用邻接法(Neighbor-Joining)构建系统进化树并进行系统进化分析,计算K2P遗传距离。
2 结果
2.1 榕小蜂核糖体28S序列分析
2.1.1 榕小蜂28S序列组成分析
对榕小蜂28S序列,用CLUSTAL X 1.83软件进行序列比对,截取对准的941 pb序列进行分析,用Mega4.0软件中的Data explore软件进行各小蜂的碱基组成分析。在28S序列中,不变位点702个,变异位点233个,简约信息位点91个,自裔位点140个,变异性为24.8%。各样品的碱基组成分析见表2,由表2可知,所有的小蜂样本碱基组成都比较平均,C+G的含量在55.7%—58.4%,C+G的含量略大于A+T的含量。
2.1.2 榕小蜂核糖体28S序列的遗传距离分析
用Mega4.0软件中的K2P法计算遗传距离(表3)。所有样品间的遗传距离在0.0000—0.1503之间,爱玉子传粉小蜂样品间的遗传距离为0.0000;薜荔传粉小蜂样品间遗传距离为0.0017;两群体间的平均遗传距离为0.0043。

表2 榕小蜂28S序列的碱基组成
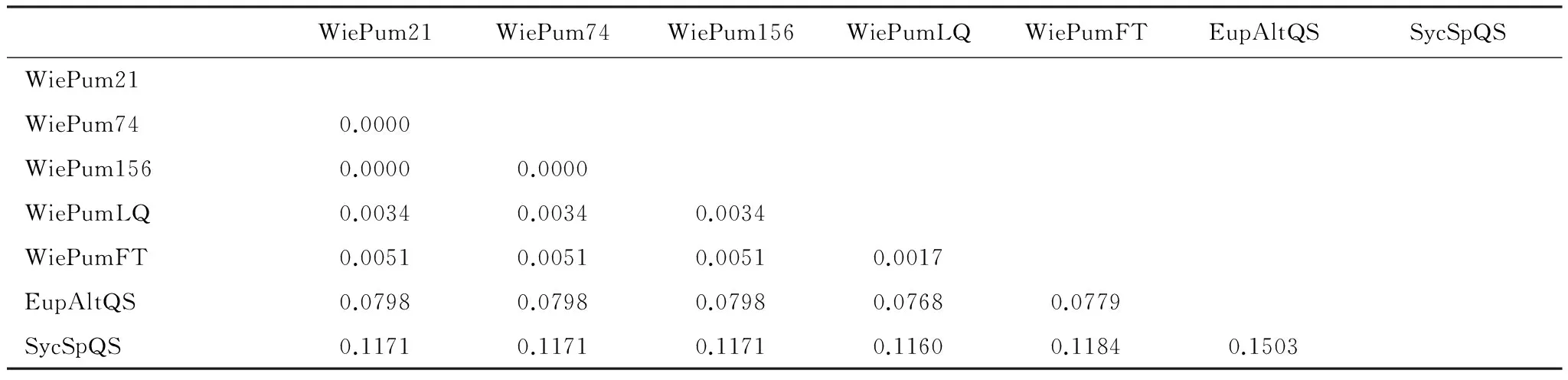
表3 基于Kimura双参数模型榕小蜂28S序列的遗传距离
2.1.3 榕小蜂核糖体28S序列系统发育树的构建
在NCBI网站上对实验中的28S序列进行同源性检索,下载4种前人研究过的榕小蜂28S序列(表4)进行分析。

表4 GenBank上下载的榕小蜂28S序列
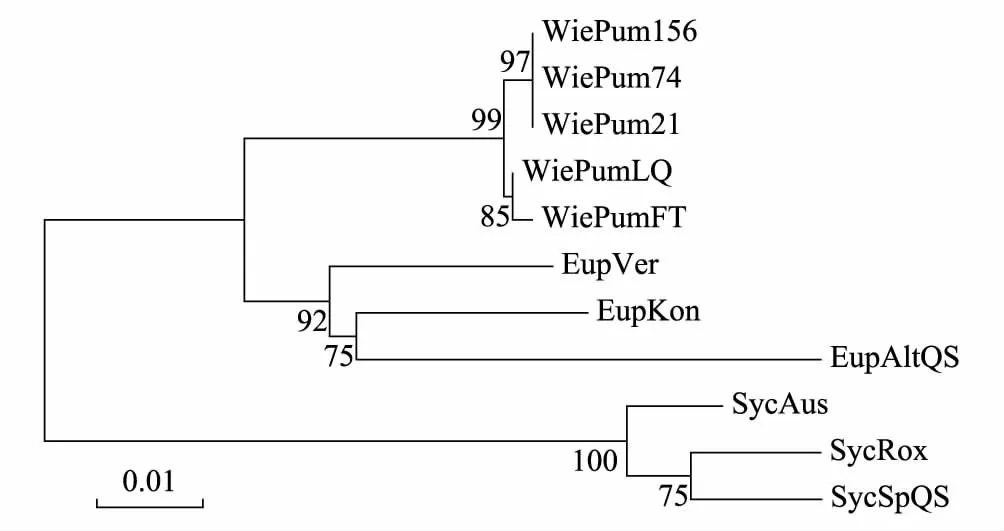
图1 邻接法构建的榕小蜂28S序列系统发育树 Fig.1 Phylogenetic tree reconstructed using NJ based on 28S sequence of fig wasps
用Mega4.0软件对实验所得与GenBank中下载的榕小蜂28S序列用NJ(Neighbor-joining)法,同时用重复抽样1000次检验分子系统树各分支的置信值,建立系统发育树见图1。图中显示,相同属的小蜂汇聚到一起,显示出较近的亲缘关系,爱玉子传粉小蜂3个样本和薜荔传粉小蜂两个样本各自先汇聚成一支,再汇聚到一起,表明薜荔传粉小蜂与爱玉子传粉小蜂群体之间已经产生了一定的变异。图中显示每个属的小蜂都能够明显的区分开来,这说明28S序列可以做为小蜂分子分类的鉴定依据。
2.2 榕小蜂线粒体Cytb及COI序列分析
2.2.1 Cytb与COI序列碱基组成及变异分析
(1) Cytb序列组成及变异分析
对榕小蜂Cytb序列,用CLUSTAL X 1.83软件进行序列比对,截取对准的376pb序列进行分析,用Mega 4.0软件中的Data explore软件进行各小蜂的碱基组成分析。在Cytb序列中,不变位点243个,变异位点132个,简约信息位点73个,自裔位点59个,变异性为35.1%。A,T,G,C四种碱基的平均含量为32.9%,43.2%,9.7%,14.1%,A+T的平均含量为76.1%,显著高于G+C的含量,符合膜翅目昆虫线粒体基因序列的共同特点。
Cytb密码子不同位点的核苷酸频率及碱基替换统计见表5,表中可知,Cytb序列密码子的各个位点上A+T含量均偏高,第三位点A+T的含量高达92.3%,A+T偏向尤为明显,其中T的使用最为频繁为54.3%。说明Cytb基因在密码子使用上具有偏向性。实验样本中Cytb序列上核苷酸颠换数大于转换数。密码子第三位点的转换(si)和颠换(sv)发生的频率明显高于第一、二位点。
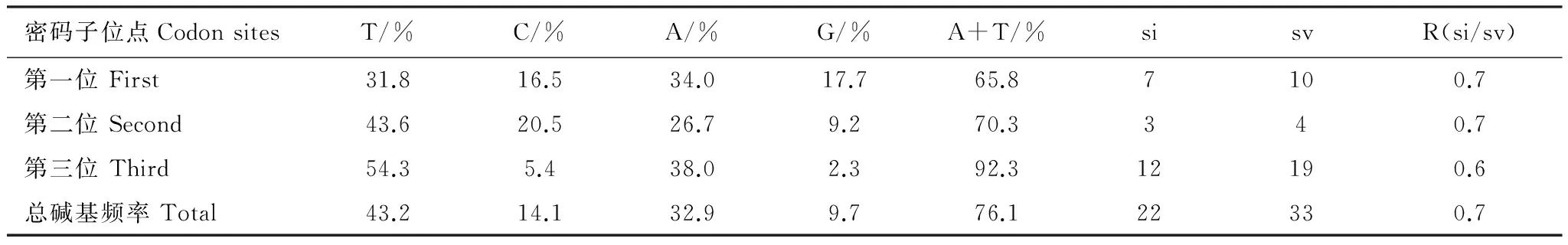
表5 Cytb密码子不同位点的碱基频率及碱基替换
(2) COI序列组成及变异分析
对榕小蜂COI序列,用CLUSTAL X 1.83软件进行序列比对,截取对准的931pb序列进行分析,用Mega4.0软件中的Data explore软件进行各小蜂的碱基组成分析,在COI序列中,有不变位点641个,变异位点285个,简约信息位点99个,自裔位点186个,总体变异性为14.2%,较Cytb序列总体变异性低。A,T,G,C四种碱基的平均含量为32.5%,39.4%,16.8%,11.2%,A+T的平均含量为71.9%,显著高于G+C的含量,与Cytb序列一致符合膜翅目昆虫线粒体基因序列的共同特点。
COI密码子不同位点的核苷酸频率及碱基替换统计见表6,表中可知,与Cytb序列相似COI序列密码子的各个位点上A+T含量均偏高,第三位点A+T的含量为86.2%,A+T偏向最为明显。说明COI基因在密码子使用上也具有偏向性。
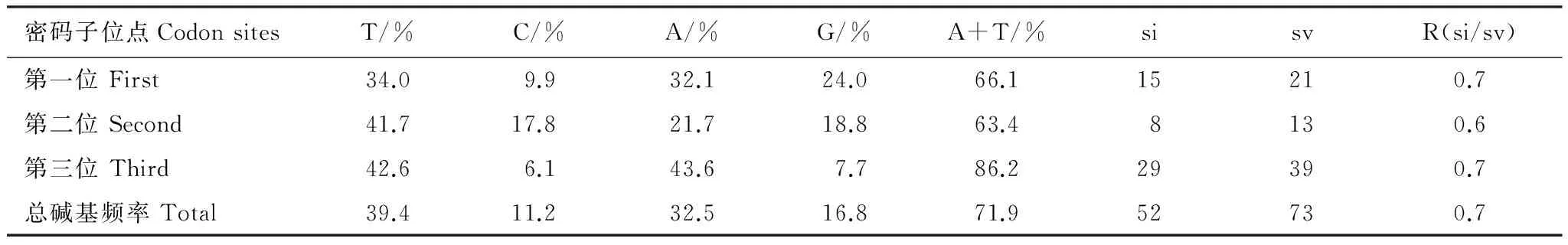
表6 COI密码子不同位点的碱基频率及碱基替换
2.2.2 Cytb与COI序列遗传距离分析
(1) Cytb序列的遗传距离分析
运用Mega 4.0软件,基于K2p模型计算两两序列间的遗传距离,对各样品间遗传距离的统计见表7。试验小蜂群体核苷酸遗传距离在0—0.3322之间,爱玉子传粉小蜂个体间的遗传距离为0.0164,薜荔传粉榕小蜂个体间遗传距离为0—0.0081,平均遗传距离为0.0054。爱玉子与薜荔传粉小蜂之间的遗传距离为0.1369—0.1416,平均遗传距离为0.1385。
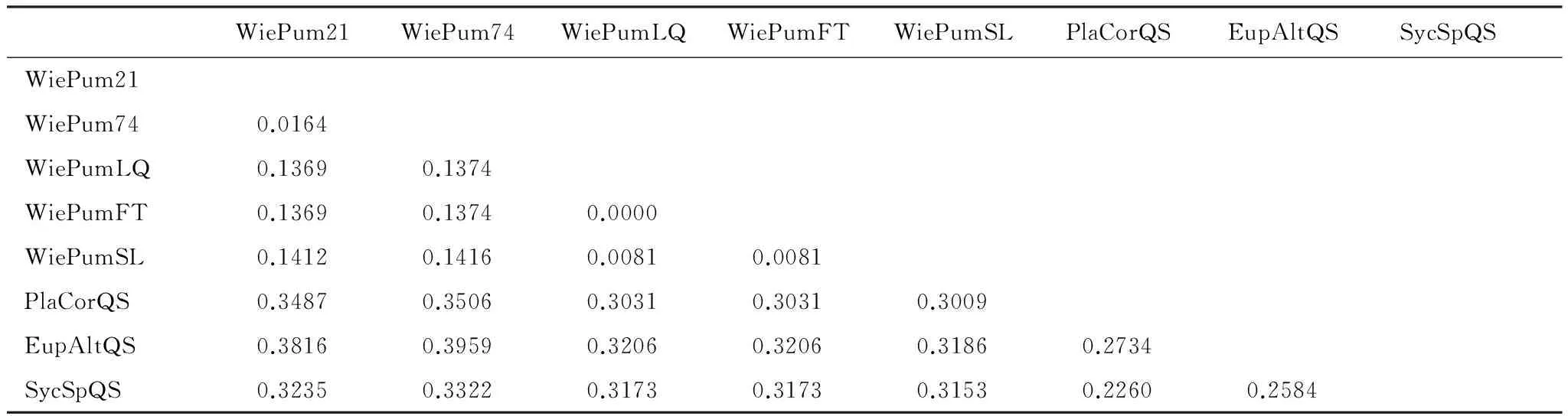
表7 基于Kimura双参数模型榕小蜂Cytb序列的遗传距离
(2) COI序列的遗传距离分析
运用Mega 4.0软件,基于K2P模型计算两两序列间的遗传距离,对各样品间遗传距离的统计见表8。试验小蜂群体COI序列核苷酸遗传距离0.0038—0.8707在之间,爱玉子传粉小蜂个体间的遗传距离在0.0038—0.0153,平均遗传距离为0.0102。薜荔传粉榕小蜂各样本间遗传距离为0.0048。爱玉子与薜荔传粉小蜂之间的遗传距离为0.1854—0.1938,平均遗传距离为0.1896。
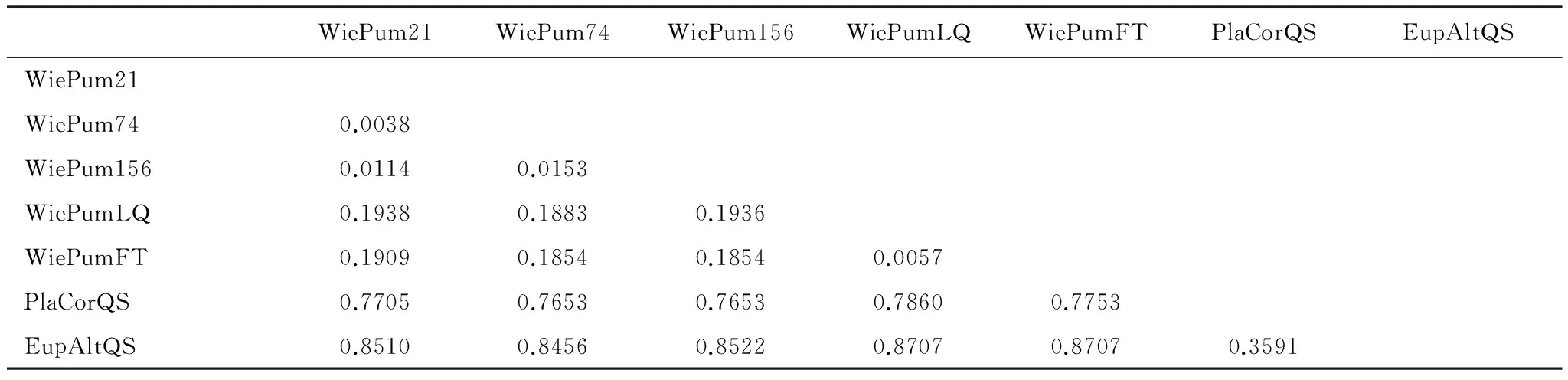
表8 基于Kimura双参数模型榕小蜂COI序列的遗传距离
2.2.3 Cytb及COI序列系统发育树的建立
实验所得序列在NCBI网站上进行Blast相似性检索,下载得到8种有榕小蜂的Cytb序列5种COI序列(见表9)与实验所得序列一起建立系统发育树(图2,图3),来分析小蜂之间的系统发育情况。
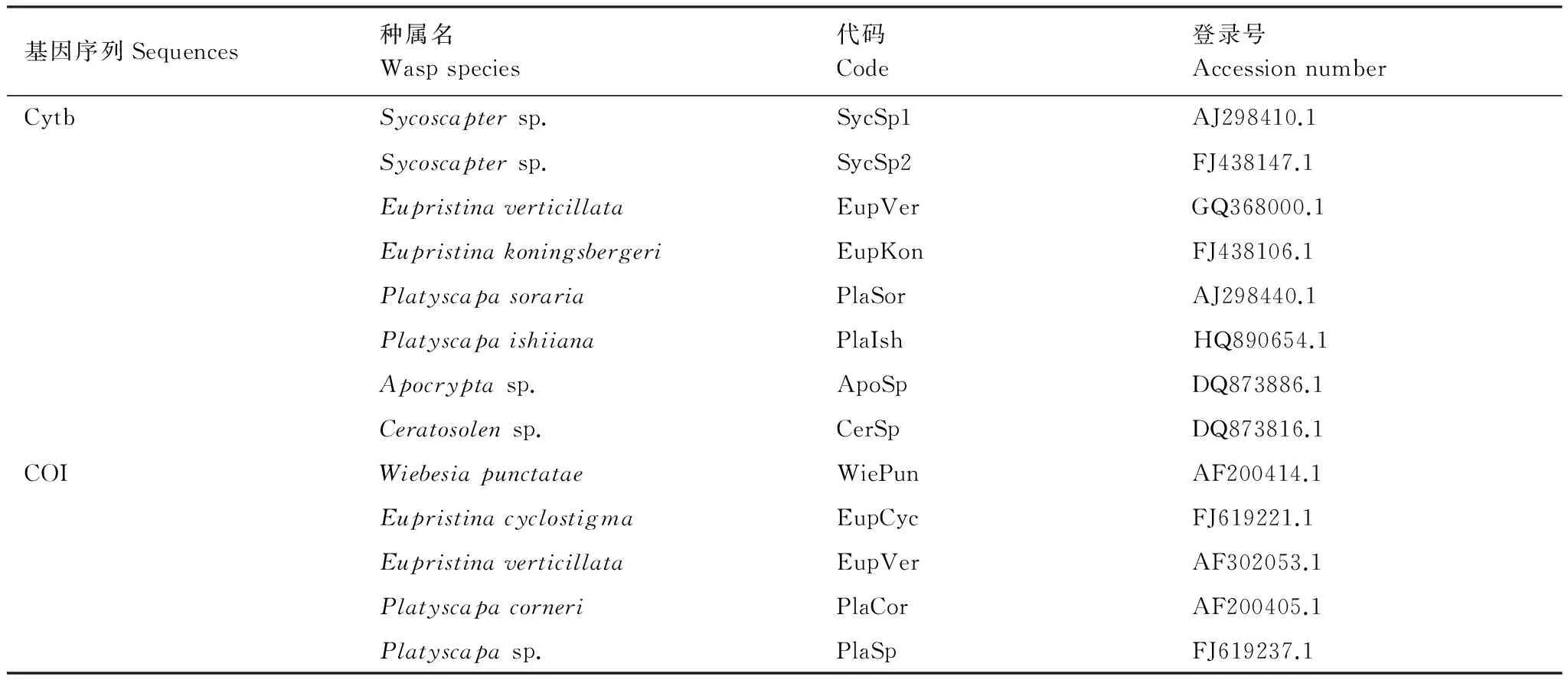
表9 GenBank上下载的榕小蜂Cytb与COI序列
用Mega4.0软件对实验所得与GenBank中下载的榕小蜂Cytb与COI序列用NJ(Neighbor-joining)法,同时用重复抽样1000次检验分子系统树各分支的置信值,建立系统发育树见图2、图3。图中显示,样本中属于同一个属的不同种的样本汇聚到一起,显示出较近的亲缘关系。基于两种序列建立的系统发育树在薜荔与爱玉子小蜂上显示出相似的拓扑结构,爱玉子传粉小蜂与薜荔传粉小蜂个体先分别汇聚成一支然后两群体再汇聚到一起,这显示出两群体之间亲缘关系相对较近,同时产生了一定的变异。每个属的小蜂都能够明显的区分开来,这说明Cytb与COI基因为榕小蜂鉴别的良好的分子标记。
3 讨论
3.1 3种分子标记特征分析
很多学者认为,如果用分子的手段进行种类鉴定时,不能只用某一个基因,因为一个基因所含有的基因数量有限,只用一个基因来进行种类鉴定存在很大的风险。与用传统的形态学方法来进行鉴定物种一样,很少只用某一个特征来鉴定物种,因为这样往往使鉴定的结果不够准确[12]。本研究用核糖体28S,线粒体Cytb及COI三个片段共同进行研究,这样就避免了一个基因片段含有的信息量有限而给研究带来的误差。对3种遗传标记序列分析结果表明,28S序列中4种碱基的含量较为平均,G+C碱基的平均含量为56%,比A+T的含量稍高,符合Campbell小蜂总科28SD2序列对小蜂总科及各科间的系统发育关系进行研究时得出的28SD2序列G+C碱基的含量偏高的结论[13]。而Cytb与COI基因片段的碱基组成都一致的显示出A+T含量明显较高的特点,有研究显示榕小蜂线粒体基因有A+T含量偏高的特点[11, 14]。并且Cytb及COI基因在密码子的不同位点上显示出了对碱基的不同偏好,特别是第三位点,Cytb基因密码子的第三位点上A+T的含量高达92.7%,COI基因该位点A+T的平均含量为86.2%。这种偏好性产生的原因可能是因为密码子第三位点产生的突变多为同义突变,发生该突变不会影响基因编码的蛋白质序列,对榕小蜂的影响较小,受到自然选择的压力较小,突变容易被保留下来[15- 16]。
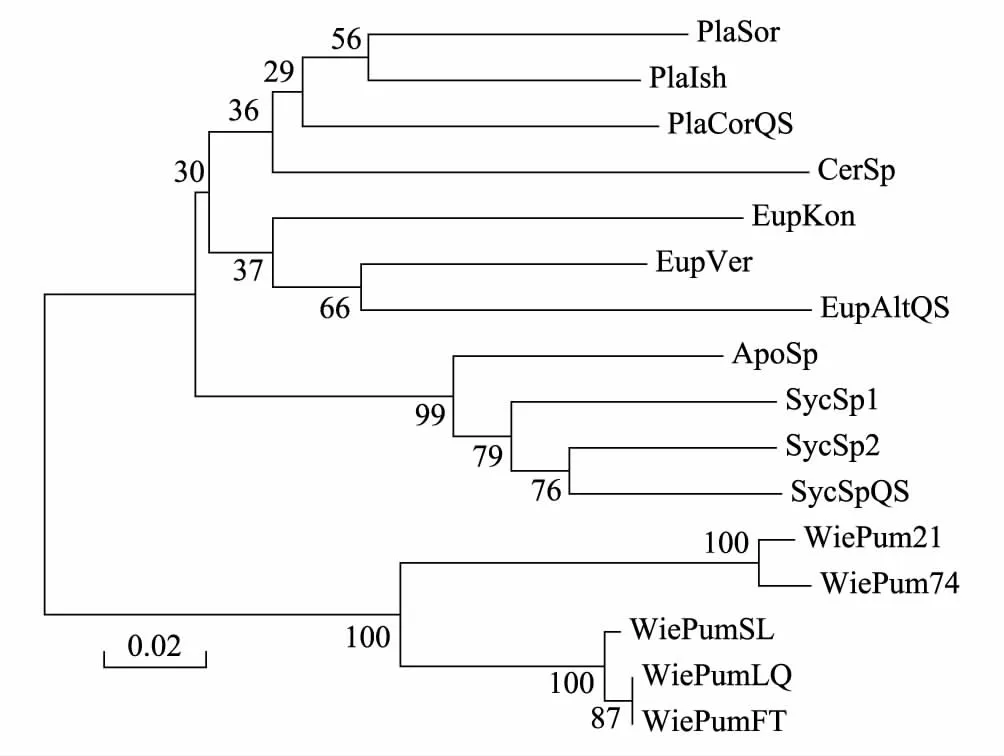
图2 邻接法构建的榕小蜂Cytb序列系统发育树 Fig.2 Phylogenetic tree reconstructed using NJ based on Cytb sequence of fig wasps
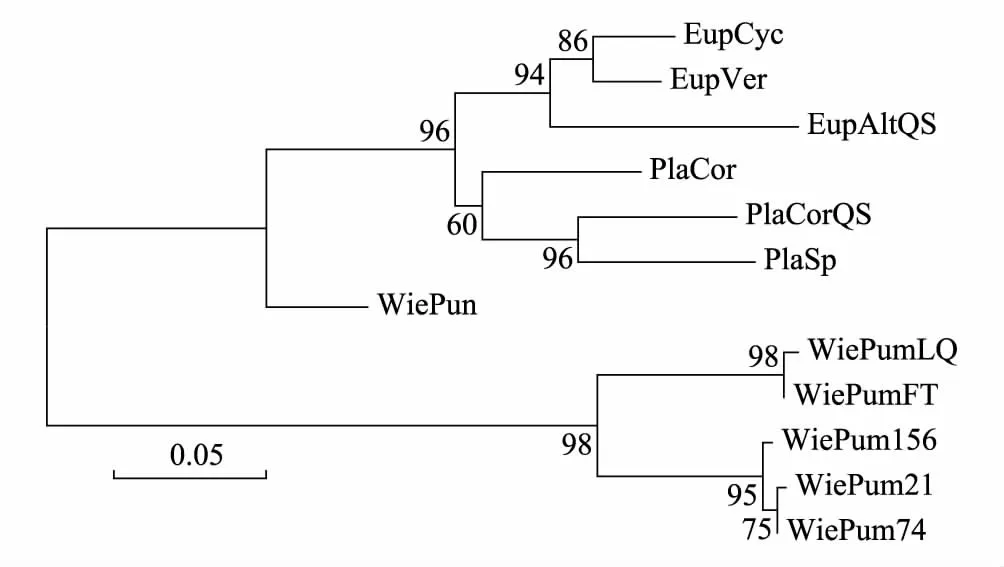
图3 邻接法构建的榕小蜂COI序列系统发育树 Fig.3 Phylogenetic tree reconstructed using NJ based on COI sequence of fig wasps
3.2 3种分子标记适用性分析
选择合适的分子标记,是用DNA序列进行准确分析的前提,不同的DNA序列在生物体中行使不同的功能,所承受的环境的选择压力不同,因而在序列的进化速度上存在差异。对生物不同分类阶元进行鉴定时,对所选择的分子标记的变异速度有不同的要求。在对亚种、种、属等较低的分类阶元进行研究时,要求所选择的分析标记要有较快的变异速度,这样才能确保在亲缘关系较近的不同个体之间存在足够的变异能够将它们区别开来。但是当研究样本之间是科、目等较高的分类群体时,由于较高的分类阶元之间产生分化的时间比较久远,在不同的群体之间不仅在形态上往往存在较大的差异外,在分子水平上也已经形成了稳定而显著的差异,因此在分析时要选择进化速度较慢,相对保守的分子标记进行分析。对实验中3种序列进行K2P遗传距离分析显示,28S序列小蜂群体的遗传距离为0—0.1503,Cytb序列小蜂遗传距离为0—0.3322,COI序列分析小蜂遗传距离为0.0038—0.8707,这说明Cytb与COI基因进化速度较快,而28S序列相对比较保守。有研究认为28S片段非常保守,更适合高级阶元(属、亚科)系统发育的研究[13, 17]。薜荔传粉榕小蜂与爱玉子传粉榕小蜂之间的亲缘关系很近,甚至被认为是同一个种,所以用进化速度相对较快的Cytb及COI序列对两者进行系统发育分析较为妥当。
3.3 薜荔传粉小蜂与爱玉子传粉小蜂分子遗传关系确定
对一些动物的 Cytb基因序列分析表明,种内个体间的差异一般在0%—4.06%,当个体差异超过6%时就说明个体间已有亚种或种的分化[18- 19]。Xiao等研究显示榕小蜂Cytb基因序列种间差异一般在11%—22%之间[20],本实验中爱玉子传粉小蜂个体间的Cytb遗传距离为0.0164,薜荔传粉榕小蜂个体间Cytb序列平均遗传距离为0.0054;爱玉子传粉小蜂与薜荔传粉小蜂群体之间的Cytb序列遗传距离为0.1385,两群体间的遗传距离明显大于群体内部各样本之间的遗传距离,表明薜荔传粉小蜂与爱玉子传粉小蜂之间的变异已达到了种间分化的水平。
Herbert利用COI序列对鳞翅目分属5个科的200个近缘种昆虫进行K2P遗传距离的研究显示,其种内平均差异仅为0.25%,属内种间差异在5.8%—9.1%之间,种间平均差异为6.8%[21]。目前关于榕小蜂种间、种内差异的标准还比较少,姜自锋(2005)利用COI基因开展榕小蜂分子系统学研究时,将遗传距离3%的差异水平作为区分种间和种内关系的界限[22]。李艳伟对189种榕小蜂进行COI序列分析显示,约123个种(65%)的种内差异小于2%,32个种(17%)的种内差异在2%—5%之间,即共有155个种(82%)的种内差异小于5%;约132个种(70%)的种间差异在10%—15%之间,有32个种(17%)的种间差异大于20%[12]。Zhou等在对Philotrypesis小蜂进行分子鉴定时,显示榕小蜂的COI序列差异达到5.8%—6.0%时可以鉴定成不同的种[23]。本实验中薜荔传粉榕小蜂个体间COI序列遗传距离为0.0048,爱玉子传粉小蜂各样本间COI序列平均遗传距离为0.0102;爱玉子传粉小蜂与薜荔传粉小蜂群体间COI序列平均遗传距离为0.1896,两榕小蜂群体间的遗传距离明显大于群体内部各样本之间的遗传距离,实验中所研究的爱玉子传粉小蜂与薜荔传粉小蜂之间已经发生了很大的分化,其变异水平已经达到了种的分异水平。
综上所述,基于28S,Cytb及COI序列所得的结果显示,福建地区的薜荔传粉小蜂和爱玉子传粉小蜂各自群体内部的遗传距离都很小,没有隐存种的分化,Cytb与COI序列分析结果一致表明二者群体之间的遗传距离达到了种间分异的水平,加之Chen等人研究显示福建地区的薜荔传粉小蜂与爱玉子传粉小蜂之间已存在生殖隔离[9],因此薜荔传粉小蜂与爱玉子传粉小蜂可以被认为是两个不同的种。
致谢:感谢福建师范大学南方生物医学研究中心陈骐教授对写作的帮助。
[1] Zhang X S, Wu Z Y, Cao Z Y. Flora of China. Vol. 23, No. l. Beijing: Science Press, 1998: 205- 206.
[2] Wu W S, Chen Y L. Comparison of reproduction ecology of differentFicusawkeotsangstrains. Acta Ecologica Sinica, 2008, 28(10): 4692- 4702.
[3] Chen Y L, Wu W S. Volatile compounds from the syconia ofFicusawkeotsangMakino and their attractiveness to pollinator wasps. Acta Ecologica Sinica, 2010, 30(8): 2212- 2219.
[4] Lee H H. Genetic Differentiation betweenFicuspumilavar.pumilaandFicuspumilavar.awkeotsangand Their Pollinators [D]. Taibei: Department of Entomology College of Bioressources and Agriculture National Taiwan University, 2009.
[5] Lin T-P, Liu C-C, Yang C-Y, Huang R-S, Lee Y-S, Chang S-Y. Morphological and biochemical comparison of syconium ofFicusawkeotsangandFicuspumila. Research Report Quarterly of Taiwan Forestry Research Institute, 1990, 5(1): 37- 43.
[6] Chen Y, Li H Q, Ma W L. The reproductive character ofFicuspumilavar.pumila,F.pumilavar.awkeotsangand their pollinators. Acta Phytoecologica Sinica, 2002, 26(1): 58- 63.
[7] Chen Y, Li H Q, Ma W L. Pollination ecology of cultivatedFicuspumilavar.awkeotsang. Chinese Journal of Applied Ecology, 2006, 17(12): 2403- 2407.
[8] He K Y. The Pollinators ofFicuspumilavar.Awkeotsang// Lin T P, ed.Ficuspumilavar.awkeotsangMonograph. Taibei: Taiwan Forestry Research Institute, 1991: 83- 96.
[9] Chen Y, Liu H Q, Ruan S J, Ma W L. Pollination of a cultivated fig,Ficuspumilavar.awkeotsang, in South China. Symbiosis, 2008, 45(1/3): 33- 36.
[10] Jiang S H. Morphological Differences between Pollinating Fig Wasps ofFicuspumilaL. var.pumilaandFicuspumilavar.Awkeotsang(Makino) Corner and Their Asymmetric Host Specificity [D]. Taibei: Department of Entomology College of Bioressources and Agriculture National Taiwan University, 2011.
[11] Chen Y. The Pollinating Fig Wasps ofFicuspumila[D]. Shanghai: East China Normal University, 2009.
[12] Li Y W. DNA Barcoding in Chinese Fig Wasps [D]. Shandong: College of Plant Protection Shandong Agricultural University, 2009.
[13] Campbell B C, Steffen-Campbell J D, Werren J H. Phylogeny of the Nasonia species complex (Hymenoptera: Pteromalidae) inferred from an internal transcribed spacer (ITS2) and 28S rDNA sequences. Insect Molecular Biology, 1994, 2(4): 225- 237.
[14] Lopez-Vaamonde C, Rasplus J Y, Weiblen G D, Cook J M. Molecular phylogenies of fig wasps: partial cocladogenesis of pollinators and parasites. Molecular Phylogenetics and Evolution, 2001, 21(1): 55- 71.
[15] Gao J B, Zhang X W, Zhou G N, Liu J X. Genetic structure ofPinecaterpillars(Dendrolimus) populations based on the analysis of Cyt b gene sequences. Acta Ecologica Sinica, 2011, 31(6): 1727- 1734.
[16] Zhou J L, Zhang Y P, Huang M H, Chen Y J, Chen X Q, Yao G D. Phylogenetic relationships among Crotalinae based on mitochondrial cytochrome B gene sequence variations. Acta Zoologica Sinica, 2001, 47(4): 361- 366.
[17] Dowton M, Austin A D. Simultaneous analysis of 16S, 28S, COI and morphology in the Hymenoptera: Apocrita-evolutionary transitions among parasitic wasps. Biological Journal of the Linnean Society, 2001, 74(1): 87- 111.
[18] Yang X G, Wang Y Q, Zhou K Y, Liu Z Q. Phylogenetic relationships of Chinese brown frogs (Rana) based on sequence of mitochondrial cytochrome b gene. Zoological Research, 2001, 22(5): 345- 350.
[19] Yang X G, Wang Y Q, Zhou K Y, Liu Z Q. Authentication of oviductus ranae and its original animals using molecular marker. Biological and Pharmaceutical Bulletin, 2002, 25(8): 1035- 1039.
[20] Xiao J H, Wang N X, Li Y W, Murphy R W, Wan D G, Niu L M, Hu H Y, Fu Y G, Huang D W. Molecular approaches to identify cryptic species and polymorphic species within a complex community of fig wasps. PLoS One, 2010, 5(11): e15067.
[21] Hebert P D, Penton E H, Burns J M, Janzen D H, Hallwachs W. Ten species in one: DNA barcoding reveals cryptic species in the neotropical skipper butterflyAstraptesfulgerator. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(41): 14812- 14817.
[22] Jiang Z F. Molecular Systematics of Fig Wasps [D]. Beijing: Chinese Academy of Sciences, 2005.
[23] Zhou M J, Xiao J H, Bian S N, Li Y W, Niu L M, Hu H Y, Wu W S, Murphy R W, Huang D W. Molecular approaches identify known species, reveal cryptic species and verify host specificity of ChinesePhilotrypesis(Hymenoptera: Pteromalidae). Molecular Ecology Resources, 2012, 12(4): 598- 606.
参考文献:
[1] 张秀实, 吴征镒, 曹子余. 中国植物志 (第二十三卷, 第一分册). 北京: 科学出版社, 1998: 205- 206.
[2] 吴文珊, 陈友铃. 爱玉子 (Ficusawkeotsang) 不同品系的繁殖生态学比较. 生态学报, 2008, 28(10): 4692- 4702.
[3] 陈友铃, 吴文珊. 爱玉子花序挥发物成分以及对其传粉小蜂的吸引作用. 生态学报, 2010, 30(8): 2212- 2219.
[4] 李和惠. 薜荔和爱玉子及其授粉小蜂之遗传分化 [D]. 台北: 台湾大学昆虫学研究所, 2009.
[5] 林赞标, 刘哲政, 杨居源, 黄瑞祥, 李永生, 张森永. 爱玉与薜荔隐花果形态与其生化特性比较. 林业试验所研究报告季刊, 1990, 5(1): 37- 43.
[6] 陈勇, 李宏庆, 马炜梁. 薜荔和爱玉及其传粉昆虫繁殖特性. 植物生态学报, 2002, 26(1): 58- 63.
[7] 陈勇, 李宏庆, 马炜梁. 栽培爱玉的传粉生态. 应用生态学报, 2006, 17(12): 2403- 2407.
[8] 何坤耀. 爱玉子授粉小蜂 // 林赞标. 爱玉子专论. 台北: 台湾林业实验所, 1991: 83- 96.
[10] 江少华. 薜荔及爱玉子授粉榕小蜂之形态差异及其寄主专一性的不对称性 [D]. 台北: 台湾大学昆虫学研究所, 2011.
[11] 陈艳. 薜荔之传粉小蜂 [D]. 上海: 华东师范大学, 2009.
[12] 李艳伟. DNA条形码在中国榕小蜂中的应用研究 [D]. 山东: 山东农业大学农业昆虫与害虫防治专业, 2009.
[15] 高宝嘉, 张学卫, 周国娜, 刘军侠. 基于Cyt b基因序列分析的松毛虫种群遗传结构研究. 生态学报, 2011, 31(6): 1727- 1734.
[16] 周继亮, 张亚平, 黄美华, 陈永久, 陈小青, 姚耿东. 蝮亚科蛇线粒体细胞色素b基因序列分析及其系统发育. 动物学报, 2001, 47(4): 361- 366.
[18] 杨学干, 王义权, 周开亚, 刘中权. 从细胞色素b基因序列探讨我国林蛙属动物的系统发生关系. 动物学研究, 2001, 22(5): 345- 350.
[22] 姜自锋. 榕小蜂分子系统学研究 [D]. 北京: 中国科学院研究生院, 2005.
ThemoleculargeneticrelationshipbetweenthepollinatorsofFicuspumilavar.pumilaandFicuspumilavar.awkeotsang
WU Wenshan, CHEN Youling*, SUN Lingli, MAO Jianping, YANG Wenxin, WANG Aifang
ProvincialKeyLaboratoryforDevelopmentalBiologyandNeurosciences,CollegeofLifeScience,FujianNormalUniversity,Fuzhou350117,China
Ficuspumilavar.pumilaandFicuspumilavar.awkeotsangare two varieties of the same species (Moraceae); one is the original variant while the other is a later variant and, they have long been considered to interact with the same species of pollinator. However, the recent studies in morphology, ecology and pollination biology suggest that a differentiation between the pollinators ofFicuspumilavar.pumilaandFicuspumilavar.awkeotsangwasps may have occurred during the evolution. In the present studies, we used three molecular markers including 28SrDNA D1—D3, the mitochondrial Cytb and COI genes to investigate the genetic relationship of these two pollinators. The pollinator samples were collected fromFicuspumilavar.pumilaat three different locations as well as three cultivars ofFicuspumilavar.awkeotsang. The data are summarized as follows: (1) The distribution of the four bases including A, T, G, C in the 28SrDNA sequence from the pollinators ofFicuspumilavar.pumilaandFicuspumilavar.awkeotsangis relatively akin. The average content of C + G (56%) is slightly higher than the average content of A+T (44%). The average content of A+T in the mitochondrial Cytb sequence is 76.1%, significantly higher than the average content of C+G (23.9%). The COI sequence analysis shows a similar result, with the A+T content (71.9%), higher than the G+C content (28.1%). This base composition pattern is consistent cross along hymenopteran mitochondrial sequences. Furthermore, in the Cytb and COI sequences, it appears to have the highest content of A/T base at the third position of a codon. (2) The 28SrDNA sequence evolution appears to be slower than the Cytb and COI gene sequences, is relatively conservative, and maybe more suitable for senior-order (family or subfamily) phylogenetic studies. In contrast, the Cytb and COI molecular markers may be more suitable for the genetic relationship analyses between the pollinators ofFicuspumilavar.pumilaandFicuspumilavar.awkeotsangdue to their close ties. (3) The results derived from genetic distance analyses show that the average distance of the Cytb sequence of each sample withinFicuspumilaL.pumilapollinator or withinFicuspumilavar.awkeotsangpollinator is 0.0054 or 0.0164. The average genetic distance of the Cytb sequence between the two pollinator groups is 0.1385. The average genetic distance of the COI sequence of each sample withinFicuspumilavar.pumilaorFicuspumilavar.awkeotsangis 0.0048 or 0.0102, while the average genetic distance between the two wasp populations is 0.1896. Since the genetic distance between the two wasp groups are significantly greater than the genetic distance within the respective group, these data suggest that the two groups of fig wasps have differentiated during the evolution and reached to the level of species identification, namely the pollinators ofFicuspumilavar.pumilaandFicuspumilavar.awkeotsangare the two different wasp species.
Ficuspumilavar.pumila;Ficuspumilavar.awkeotsang; fig wasps; COI; Cytb; 28S
国家自然科学基金资助项目(31270440);福建省科技厅重点资助项目(2011N0014);福建省高等学校学科带头人培养计划资助项目;福建师范大学生命科学学院生物学拔尖生培养计划资助项目
2013- 06- 09;
2013- 07- 25
*通讯作者Corresponding author.E-mail: chenyouling2000@126.com
10.5846/stxb201306091500
吴文珊,陈友铃,孙伶俐,毛建萍,杨问新,王爱芳.基于28S, COI和Cytb基因序列的薜荔和爱玉子传粉小蜂分子遗传关系研究.生态学报,2013,33(19):6049- 6057.
Wu W S, Chen Y L, Sun L L, Mao J P, Yang W X, Wang A F.The molecular genetic relationship between the pollinators ofFicuspumilavar.pumilaandFicuspumilavar.awkeotsang.Acta Ecologica Sinica,2013,33(19):6049- 6057.

