基于神经氨酸酶结构的流感病毒抑制剂的计算机辅助药物设计
刘 磊,马 英,王润玲,王树青,徐为人
(1.天津医科大学药学院,天津市临床药物关键技术重点实验室,天津 300070;2.天津药物研究院,天津市新药设计与发现重点实验室,天津 300193)
流感病毒是一种严重影响人类正常生活的流行性疾病,始终处于不间断的变异中[1-3]。流感病毒表面蛋白神经氨酸酶(NA)在流感病毒生命过程中起着至关重要的作用,是抗流感病毒药物设计的重要靶点。神经氨酸酶抑制剂类抗流感病毒药物可同时抑制A、B 型流感病毒,不易引起抗药性且耐受性好,神经氨酸酶抑制剂的发现是抗流感化学治疗药物研究中取得的重大突破性进展[4-6]。2006年Skehel 等对H5N1 流感病毒的N1 型神经氨酸酶三维结构解析成功并发现其与先前药物设计的靶点N2和N9 型结构有很大的区别,其潜在的新配体结合空腔(150-cavity)为新型流感病毒药物的设计提供更多的机会[7-9]。本研究旨在找到基于流感病毒表面蛋白NA 结构的新型抑制剂化合物,不仅靶向原来达菲占据的靶点,而且向新发现的150-cavity 伸展,使新型抑制剂更有利于抑制NA 的活性。
1 材料与方法
1.1 软件及数据库 DELL T5500 计算工作站,涉及软件主要有Schrodinger 2009,Desmond2.4,在ChemBioOffice2010 上绘图。受体来自PDB-Bank 数据库,分子碎片库是本实验室手绘库及已有碎片库。
1.2 方法 虚拟药物筛选也称计算机筛选,即在进行生物活性筛选之前,在计算机上对化合物分子进行预筛选,以降低实际筛选化合物数目,同时提高先导化合物发现效率。
1.2.1 靶点选择及活性位点分析 本文所用受体晶体结构是从PDB Bank 下载的,PDB ID为2HU0[10]。遵循以下条件:所选靶点晶体结构为人源的;晶体结构中氨基酸残基没有突变;晶体结构分辨率小于2.6魡。
1.2.2 靶点蛋白的优化 蛋白结构处理由Schrodinger Suite2009 软件中的Protein preparation wizard 模块完成。将下载的NA 靶点晶体结构(2HU0)导入Schrodinger Suite2009,首先对蛋白质结构进行修正,参数设定为Assign bond orders(修正化学键);Add hydrogens(加氢);Treat metals(处理金属离子);Cap termini by adding ACE&NMA residues(修正N 端和C 端的残基结构错误),经处理后除去蛋白质晶体中的水分子和杂原子,只保留蛋白分子与原始配体。再对蛋白质结构进行优化,氢键优化参数为Sample water orientations,优化立场选择OPLS_2005[11],以RMSD0.5魡为收敛标准。
1.2.3 对接方法的验证 在对小分子库进行筛选之前,应验证对接程序及所选参数是否适用于本研究。首先将原始配体OSE(达菲)从2HU0 靶点蛋白晶体复合物中剥离出来,再与原受体活性位点进行对接,如果对接后的配体结构能够与原晶体中配体结构较好吻合,则表明该对接程序及所选参数适用于本研究的筛选。
1.2.4 配体准备 对Leadnow 分子库中的分子进行配体优化处理,此步在LigPre 模块中完成。小分子优化的力场为OPLS_2005,产生pH 值7.0±2.0 下的离子化状态,去盐,产生各种可能的互变异构体。
1.2.5 受体盒子生成 此步在Glid 模块中完成,选择Receptor Grid Generation。Site,按需要选择,大小选择10魡。
1.2.6 分子对接 分子对接利用Schrodinger Suite 2009 软件中的Glide[12]模块进行,以原始配体小分子为中心,自动生成对接盒子文件。对接精度为SP(标准精度),采用柔性对接,柔性对接时要求考虑各种环的柔性情况,要求对接函数在打分时对非平面型的酰胺键构象进行罚分,其它参数为默认值。虚拟筛选中对接精度为HTVS(高通量筛选)。
1.2.7 Core-Hopping Core-Hopping 模块具有替换片段和分子对接两大功能。在“Core Hopping”过程中,第一步是定义模板化合物。首先定义需要替换的片段,这一任务由Define Combinations step 完成。第二步需要定义受体盒子,这部分已在Receptor Grid Generation 中介绍。第三部分准备分子片段,分子片段来自于drug-like 数据库和ZINC[13]数据库中的片段数据库。第四步把小分子片段连接到母核上,得到的结构对接到受体里。最后,依据对接得分进行筛选,得分的绝对值越大,亲和力越强,结合力越好。
1.2.8 分子动力学 分子动力学模拟由Schrodinger Suite 2009 软件中的Desmond2.4 模块完成。使用SPC 水模型模拟人体内环境。为了使系统的密度维持恒定,采用周边性边界条件。为了得到电中性的环境,在蛋白质中加入0.15 mol/L 钠离子和氯离子,离子在模型中自动排布。用Nose-Hoover 定温计算法,调整平衡体系的温度到300 K。用NPT 系综保持体系温度、压强和原子数量不变,压强为1 atm。最后进行系统能量最小化,用relax 去掉局部高能量,防止原子碰撞。3 ns 之后系统达到平衡,然后进行了10 ns 的模拟。
2 结果
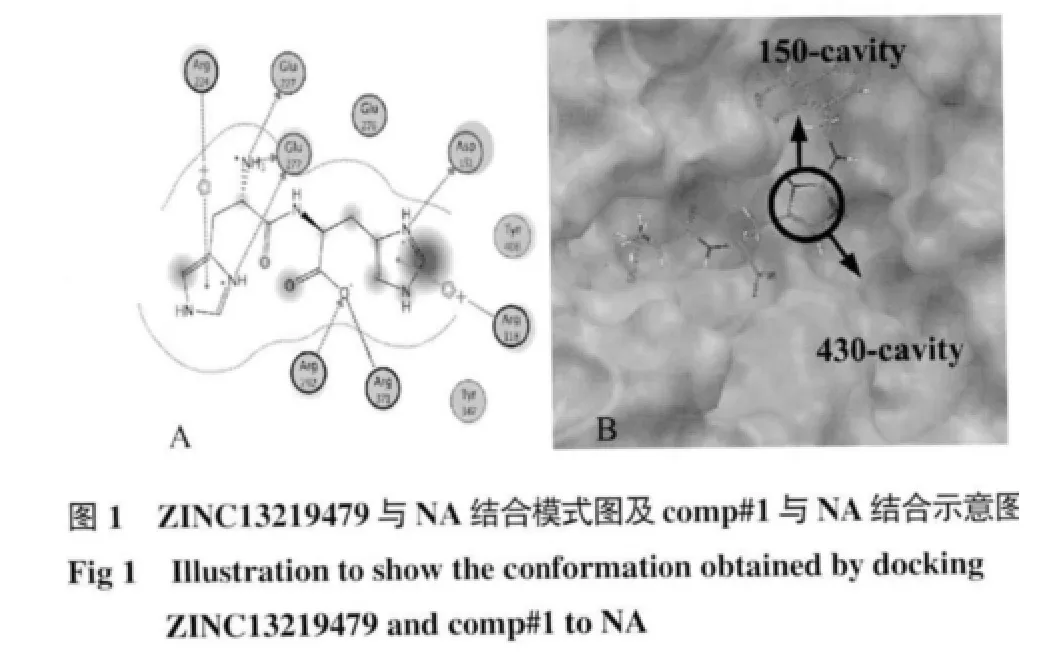
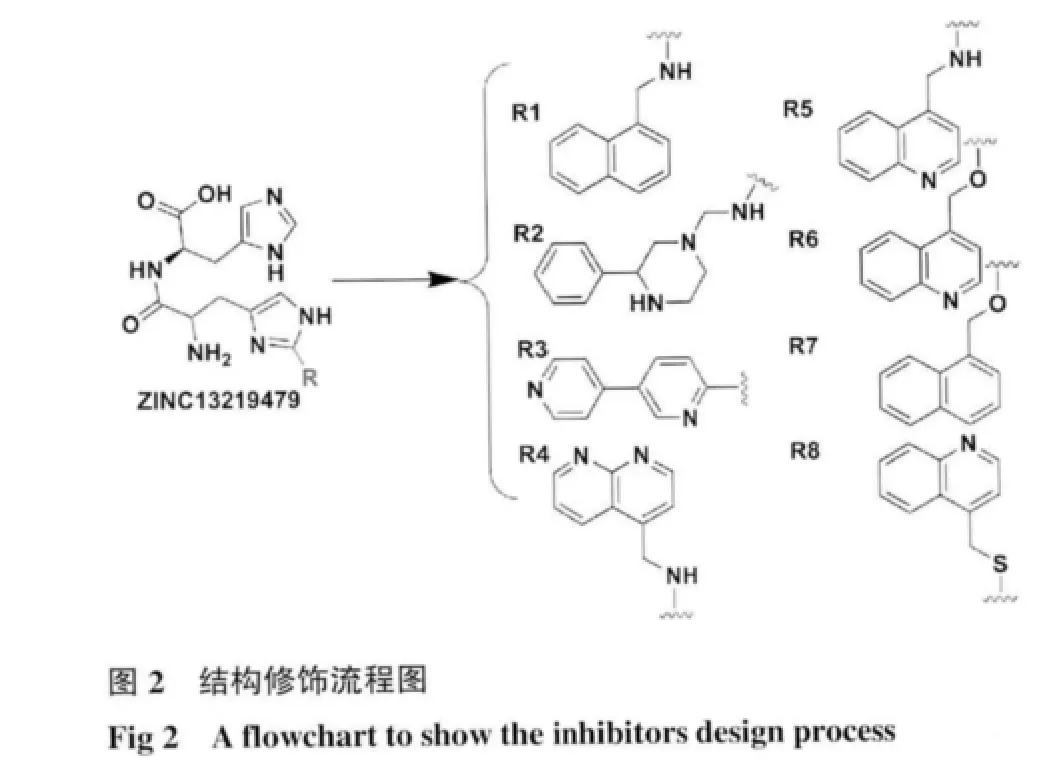
2.1 高通量虚拟筛选,设计能够进入150-cavity 的抑制剂 对Leadnow 数据库中10 000个化合物进行筛选,选出和达菲与靶点结合力相似的1个化合物ZINC13219479。对此结构应用“Core Hopping”进行结构改造得出5个较好的结构,见表1。对接结果显示:ZINC13219479 对接得分明显高出Oseltamivir。从图1(A)中可以看出ZINC13219479 不但与s 空腔结合良好,还与Arg118 形成p-π 共轭。由此,选ZINC13219479 作为进一步修改的候选化合物。开放式的150-loop 拥有较低的能量,所以可以尝试设计出能够占据150-cavity 的高效抑制剂。具体修饰过程如图2 所示:在环上添加疏水分子片段以提高与受体的疏水臂的匹配性,找出8个优选片段R1、R2、R3、R4、R5、R6、R7、R8。对接结果显示:分子结构与NA 具有较好的匹配性,羧酸头上的羰基氧原子能与Arg118、Arg371 形成氢键,起到与Oseltamivir 的1-位羧酸类似的作用,还有咪唑环能与Arg118 形成p-π 共轭;通过-NHCO-伸入150-loop,使刚性环占据150-cavity 提高活性。图1为ZINC13219479、comp#1 结合模式图。选出得分最高的结构comp#1 进行动力学模拟,对对接结果做进一步验证。
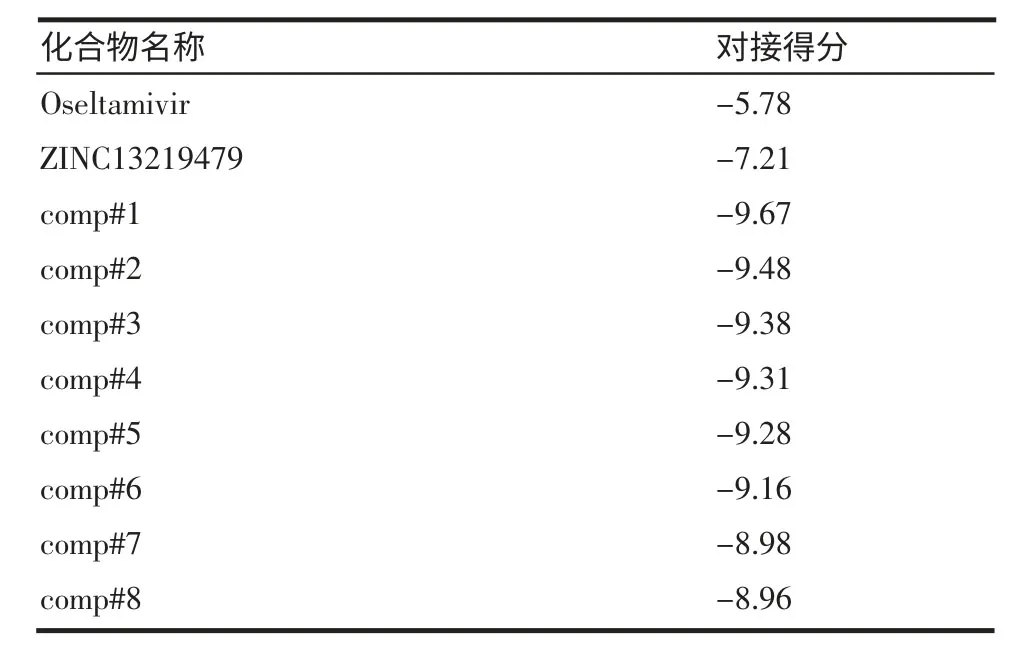
表1 对ZINC13219479 修饰结果Tab 1 The results of docking by Glide
2.2 动力学模拟结果 图3 是NA 的空载蛋白及靶点与配体小分子复合物comp#1、Oseltamivir 的骨架原子RMSD 值随时间变化图。可以看出,空载蛋白在2 ns 左右达到了平衡,对于NA 与配体复合物体系在1 ns 左右达到了平衡。在达到平衡状态后,NA 与配体复合物体系RMSD 较空蛋白时低,说明与配体结合后,NA 的柔性降低了,配体占住了靶点。
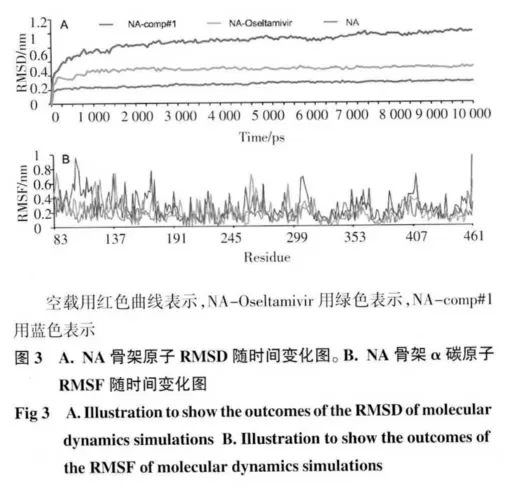
如图3 所示,NA 的活性中心波动减少,说明设计的抑制剂与NA 的匹配程度很高。而新设计的抑制剂使150-cavity 区的波动减少,说明设计的抑制剂能抑制住150-cavity。
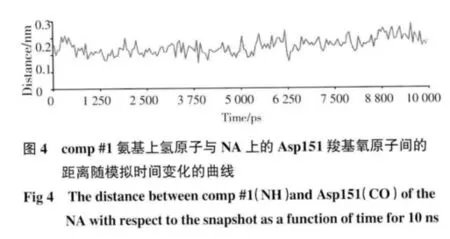
功能性保守残基Asp151,其构象不会从“开放式”的形式变化为“封闭”的形式。因此,设计的化合物与Asp151 所及氧原子形成的氢键相互作用是一直很稳定的。为揭示这种机制,绘制了关键原子对的距离随模拟时间变化的曲线。由图4 可知,NAcomp#1 氨基上氢原子与Asp151 羧基氧原子间的距离均大约在0.15~0.25 nm 范围内波动。说明稳定的氢键已经存在于配体和蛋白之间。
3 讨论
3.1 NA 受体抑制剂的设计 S1为由3个精氨酸残基组成的带正电荷区域,能与抑制剂带负电的取代基(如羧基)形成盐桥。S2为由Glu227 和Glu119形成的带负电的区域,能与抑制剂分子中的羧基、氨基或胍基以氢键的形式相互作用。S3为包括有Trp178 和Ile222 的侧链形成的疏水性区域和与之相邻的由Arg152 的侧链形成的极性区域。抑制剂的羰基氧与Arg152 形成氢键,而甲基等疏水性基团与Trp178 和Ile222 形成的疏水区口袋作用。S4为由Ile222、Ala246 的侧链和Arg224 的疏水面形成的较大的疏水口袋。S5为由Glu276 的羧基和Ala246 的甲基构成混合极性的区域,其中Glu276能与抑制剂侧链的羟基形成氢键[14]。本文首先论证了所选择NA(PDB ID∶2HU0)用作虚拟筛选的可行性。接着对Leadnow 数据库的筛选得出优于达菲的小化合物ZINC13219479,对其改造找出能伸入150-cavity 的结构。分子结构与NA 具有较好的匹配性,羧酸头上的羰基氧原子能与Arg118、Arg371 形成氢键,起到与达菲相似的作用;刚性环能与150-cavity空腔匹配,提高抑制作用。筛选得到化合物均能与NA 受体有较好的相互作用,具有潜在的抑制作用,因此可以作为针对NA 的抗病毒化合物。
3.2 NA 受体抑制剂分子动力学研究 结果显示模拟过程中NA 与配体复合物体系是稳定的,结合位点是正确的。作为抑制剂,comp#1 类小分子配体与NA 的活性区域残基形成稳定的氢键作用(如Arg118,292,371),牢牢占据NA 的催化位点,同时使S4、S5 区的残基波动减小,从而抑制酶的活性;还可以伸入150-cavity 与150-loop 上的Asp151 和Arg118 形成氢键,阻止150-cavity 闭合。动力学模拟结果与分子对接结果一致。希望此设计理念和方法能为今后的研究奠定基础。
[1]Chen Z,Kim L,Subbarao K,et al.The 2009 pandemic H1N1 virus induces anti-neuraminidase(NA)antibodies that cross-react with the NA of H5N1 viruses in ferrets[J].Vaccine,2012,30(15):2516
[2]Tao P,Luo M,Zhu D,et al.Virus-like particle vaccine comprised of the HA,NA,and M1 proteins of an avian isolated H5N1 influenza virus induces protective immunity against homologous and heterologous strains in mice[J].Viral Immunol,2009,22(4):273
[3]Chen S J,Sun L,Liu W J,et al.Recombinant fowlpox virus coexpressing HA and NA gene from subtype H5N1 of avian influenza virus and its protective efficacy[J].Wei Sheng Wu Xue Bao,2006,46(1):111
[4]Russell R J,Haire L F,Stevens D J,et al.The structure of H5N1 avian influenza neuraminidase suggests new opportunities for drug design[J].Nature,2006,443(7107):45
[5]Du Q S,Wang S Q,Chou K C.Analogue inhibitors by modifying oseltamivir based on the crystal neuraminidase structure for treating drug-resistant H5N1 virus[J].Biochem Biophys Res Commun,2007,362(2):525
[6]An J,Lee D C,Law A H,et al.A novel small-molecule inhibitor of the avian influenza H5N1 virus determined through computational screening against the neuraminidase[J].J Med Chem,2009,52(9):2667
[7]D’Souza C,Kanyalkar M,Joshi M,et al.Probing molecular level interaction of oseltamivir with H5N1-NA and model membranes by molecular docking,multinuclear NMR and DSC methods [J].Biochim Biophys Acta,2009,1788(2):484
[8]Durrant J D,McCammon J A.Potential drug-like inhibitors of Group 1 influenza neuraminidase identified through computeraided drug design[J].Comput Biol Chem,2010,34(2):97
[9]Gong K,Li L,Wang J F,et al.Binding mechanism of H5N1 influenza virus neuraminidase with ligands and its implication for drug design[J].Med Chem,2009,5(3):242
[10]Mitrasinovic P M.On the structure-based design of novel inhibitors of H5N1 influenza A virus neuraminidase(NA)[J].Biophys Chem,2009,140(1):35
[11]Kaminski G A,Friesner R A,Tirado-Rives J,et al.Evaluation and reparametrization of the OPLS-AA force field for proteins via comparison with accurate quantum chemical calculations on peptides[J].J Physic Chem B,2001,105:6474
[12]Halgren T A,Murphy R B,Friesner R A,et al.Glide:a new approach for rapid,accurate docking and scoring.2.Enrichment factors in database screening[J].J Med Chem,2004,47(7):1750
[13]Irwin J J,Shoichet B K.ZINC--a free database of commercially available compounds for virtual screening[J].J Chem Inf Model,2005,45(1):177
[14]Li Y,Zhou B,Wang R.Rational design of Tamiflu derivatives targeting at the open conformation of neuraminidase subtype 1[J].J Mol Graph Model,2009,28(3):203

