从生命科学领域解读《Science》2012年度十大突破
张 婷 王晓民
(首都医科大学基础医学院神经生物学系,北京100069)
《Science》2012年度十大科学突破:
1)希格斯玻色子的发现
2)古人类DNA序列的完美呈现
3)基因组的巡航导弹
4)中微子“第三种震荡”
5)超越基因的基因组
6)“好奇号”的火星着陆系统
7)X射线激光器测定蛋白质结构
8)脑机接口的成功应用
9)马约拉纳费米子确实存在
10)干细胞诱导成为卵细胞
生命科学领域中的突破:
1 古人类DNA序列的完美呈现[1]
目前,人们探索古人类的主要手段是通过各种化石等遗物。近年来,古遗传学家对尼安德特人及丹尼索瓦人基因组全序列的揭示为该领域研究带来了新的方式[2]。然而,与目前广泛应用的活组织DNA测序相比,古人类DNA测序结果还不够精确,因为大部分从化石中提取的DNA已降解成单链,自动测序仪无法识别。今年,德国马普进化人类学研究所的博士后Meyer建立了一种新型扩增单链DNA的技术,并带领其研究小组对丹尼索瓦人基因组重新测序,其结果与当今活组织DNA测序结果的精确度非常接近。
Meyer建立的单链DNA文库制备方法为(图1):①将古人类DNA去磷酸化,加热变性;②与生物素标记的寡核苷酸接头相连接,通过streptavidin包被的磁珠后,标记的DNA被吸附下来;③加入与寡核苷酸接头互补的引物;④通过碱基互补配对原则复制单链DNA;⑤新合成的DNA单链与另一个双链接头的一条链连接;⑥加热磁珠,释放DNA。这一方法避免了DNA纯化步骤,减少了样本的消耗[3]。
通过此技术对居住在西伯利亚丹尼索瓦人洞穴中的女孩进行研究,只需要6毫克小拇指指骨样本,一次至少能拷贝99.9%的基因组,同时对其中92%的基因组可重复拷贝20次(这是确定核苷酸位置可靠性的指标)[3]。这是研究者第一次应用基因组信息对古人类进行研究。此研究的意义在于,丹尼索瓦人化石迄今仅存一小块指骨和两颗磨牙,却得到了完整的DNA序列,而尼安德特人尽管已有上百块化石,但基因组序列却并不完整。该方法将进一步应用在更多化石标本上,尼安德特人基因组序列有望在2013年得到完整揭示。
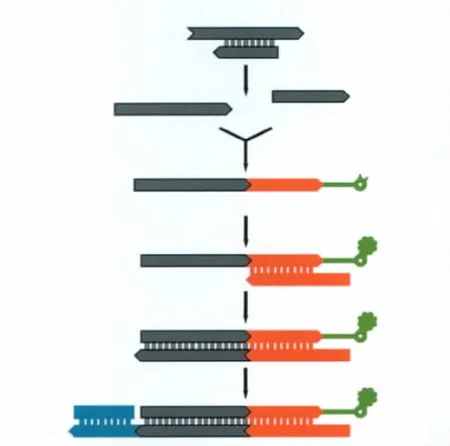
图1 单链DNA文库制备方法[3]
2 基因组的巡航导弹[1]
几年前,成功的进行基因敲入或敲除还是偶然现象,而且,人们也无法控制外源基因在宿主基因组中的插入位置,或者基因敲除实验中敲除的是哪个DNA。这种情况下,明确某一特定基因的作用或者纠正某个患病基因就非常困难。今年,研究人员得到了一些DNA修饰的有利工具,其中之一,被称为转录激活子样效应因子核酸酶(transcription activator– like effectors nucleases,TALENs)。正如巡航导弹击中军事目标一样,TALEN可特异识别DNA序列并进行切割,从而进行基因修饰或敲除[4]。
2009年,《Science》同时发表两篇文章,报道在植物病原菌黄单胞菌(Xanthomonas)上发现的转录激活子样效应因子(transcription activator– like effectors,TALEs)可特异性地结合到DNA上,在该病原菌感染过程中对植物基因进行调控[5-6]。TALE蛋白的DNA结合域由一串连续排列、数目不同(12~30个不等)、序列高度同源的蛋白结构域模块构成,每个模块大小为33~35个氨基酸。模块的第12位和13位的氨基酸,对应识别DNA分子中四个碱基中的一个(图2)。TALEN是由TALEs DNA结合域和核酸内切酶FokⅠ的切割域融合而成。其中,TALEs的DNA结合域能够识别特异的DNA序列,FokⅠ可通过二聚体产生核酸内切酶活性,在特异的靶DNA序列上切断双链[7]。
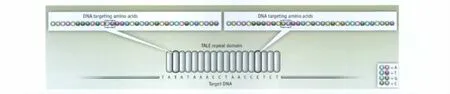
图2 TALE识别特异DNA序列[4]
发现TALEN时间非常短暂,却已在各种模型中证实有效。Jaenisch等[8]在人胚胎干细胞(embryonic stem cell,ES)及诱导的多潜能干细胞(pluripotent stem cell,iPS)中的五个不同的基因位点应用TALEN,此前这五个基因位点曾使用锌指核酸酶(zinc-finger nucleases,ZFNs)研究过,结果证实TALEN在位点特异性DNA修饰方面与ZFN具有相似的准确性和有效性;Carlson[9]及同事应用TALEN建立了小型猪单等位基因或双等位基因敲除低密度脂蛋白受体基因的家族性高胆固醇血症疾病模型;Ekker等[10]在斑马鱼中应用TALEN进行基因修饰的同时,引入了一个定制的EcoRⅤ位点和一个修饰的loxP(mloxP)位点,后者可条件性开启或关闭基因,用于观察该基因在发育过程中的作用;Zhao等[11]应用TALEN在人源细胞上对镰刀型红细胞贫血症相关基因进行纠正,证实了TALEN可用于基因治疗。
基因编辑(gene editing)是研究生物学过程的有利手段。基因编辑的工具目前主要包括核酸内切酶(endonucleases),ZFNs和 TALENs[12]。其中,ZFN 的效果最引人注目,并已进入临床实验。但其局限性是制备困难,而且关键技术均被一家公司申请了专利。而TALEN与ZFN相比同样有效,但制备更加简单便宜。目前,一些研究者甚至认为TALENs会成为所有分子生物学实验室的必备工具。
3 超越基因的基因组[1]
人类基因组计划(HGP)研究已测定出人类基因组DNA的30亿个碱基对序列,其中编码蛋白的碱基对只有2%或更少。继HGP之后又一大型国际合作项目“DNA元件百科全书”(the Encyclopedia of DNA Elements,ENCODE)对各基因之间的DNA序列进行研究,发现这些一度被认为“荒漠”的基因组序列发挥了非常重要的作用,可开启或关闭基因表达。ENCODE计划旨在完成HGP遗留的任务,为潜藏在“荒漠”中的功能性DNA序列编制目录。
2003年,ENCODE开展前期调研,对基因组中1%的部分进行深入研究。结果提示,只有少数DNA被转录翻译成蛋白,许多基因组被“转录”成非编码的RNA分子。该发现公布不久后,ENCODE开展了全面研究(图3),对至少147种细胞类型进行了1 648项实验[13]。通过这些实验,研究者精确定位了每个基因的起始位点、终止位点及编码区,发现有一些DNA编码RNA后并不翻译成蛋白,这些RNA存在于不同的细胞器[14]。因此,有人认为基因组或遗传学的最基本单位不是基因,而是DNA编码的RNA。
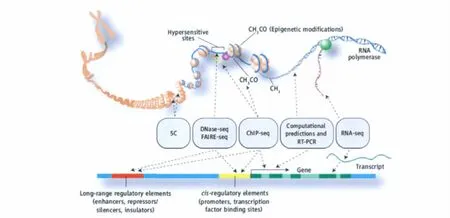
图3 ENCODE对DNA功能的全面研究[13]
其中一项对DNA的功能性研究评价了其在不同物种之间,以及同一物种不同个体之间的保守性。研究显示在人类和哺乳动物之间并不保守的序列在人群不同个体之间呈现保守性,如与神经生长及视锥细胞发育基因相邻的区域,于是构成了人类独有的特征性表现;同时,在人类和哺乳动物之间保守的序列在人群中却有差异,提示这些区域可能不再具有功能[15]。
另外一项研究是针对发挥基因调控功能的DNA序列,它们通过与转录因子或其他蛋白结合而发挥作用。其中,DNase-seq和 FAIRE-seq研究提示,42%的基因组可结合转录因子或蛋白,而这些区域可能与不同疾病的发病风险相关[16]。另外,该研究组[17]还发现此研究可预测参与疾病发病的细胞类型,例如,有两种类型的T细胞参与了克罗恩病的发病。另外,ChIP-seq研究还对组蛋白与DNA的结合进行了揭示[18]。另一项5C的技术对一个染色体上距离较远的DNA,甚至是不同染色体上的DNA相互作用进行了研究[19]。
4 X射线激光器测定蛋白质结构[1]
X射线晶体衍射法(X-ray diffraction)是目前测定蛋白质三维结构的最主要和最精确的方法。X射线射至蛋白质晶体上,可产生不同方向的衍射,X线片则接受衍射光束,形成衍射图。然后借助计算机绘制出三维空间的电子密度图。该方法要求首先将蛋白质制备成晶体,但一些膜蛋白无法形成晶体。今年,研究人员用一种比传统的同步加速辐射源亮10亿倍的X射线激光测定了一种蛋白结构(图4),有可能对传统X射线无法测定的蛋白质进行解码[20]。

图4 X射线激光解密蛋白质结构[20]
非洲昏睡病每年要夺走3万人的生命。该疾病由单细胞寄生虫布氏锥虫引起,半胱氨酸蛋白酶组织蛋白酶 B(TbCatB)是其存活的关键酶。Chapman等[20]在细胞中过度表达该酶,得到毫米水平的晶体。应用X射线激光器,研究人员测定了TbCatB的无活性前体结构,发现其活性区域覆盖着一种分子安全帽—前肽—而使其失活。该发现将有助于人们找到与这种前肽结构相似的分子,从而抑制TbCatB的活性,杀死布氏锥虫。
今年年初,日本研究人员也开启了X射线激光器研究。欧洲研究人员正在构建此研究项目,估计2015年可以开启。这一应用的宏伟目标是将X射线衍射推向极限,通过X射线激光器解密蛋白结构。
5 脑机接口的成功应用[1]
Collinger等[20]报道,一位由于遗传性神经退变导致颈部以下瘫痪的53岁女性患者,学会了用思想控制机械臂,完成一系列日常活动。
研究者将两个4×4毫米的96通道微电极植入患者大脑采集信号,植入区域为参与手部运动计划的脑区。电脑将这些信号转换成机械臂的运动指令,使该机械臂具备与人手臂运动相同的能力(图5)。本研究持续了13周,研究者对患者进行训练,目标是能控制假肢进行7个自由度的活动,包括三维转译,三维定向以及一维抓取。患者控制假肢的能力通过上肢功能评分来判断。通过这一系统,患者能够控制机械臂完成一系列日常活动。训练进行的第二天,患者就能够在三维的空间内控制机械臂。13周后,患者已经可以常规完成七维的运动。任务型抓取的成功率为91.6%,完成时间不断缩短(从最初的148秒降至112秒)。患者能够控制假肢进行技巧性和协调性的抓取和抓握运动,上肢功能评分有显著的进步,没有不良反应发生[21]。
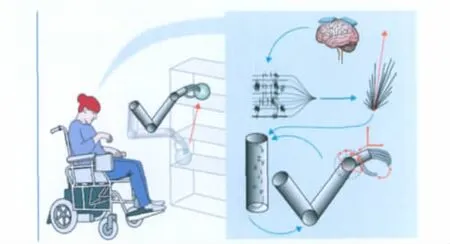
图5 瘫痪患者应用脑机接口完成日常生活[23]
该研究呈现了瘫痪患者应用脑机接口(brain-machine interface,BMI)能完成的最复杂运动。2012年5月,Hochberg等[21]报道了第一例瘫痪患者应用BMI完成复杂的三维运动的研究。在此基础上,本研究实现了更流畅更自然的运动。这一发现对于瘫痪患者来说是非常鼓舞人心的,然而真正的临床应用仍需要时间:机械臂仍处在试验阶段,价格昂贵,其使用尚需技术人员的辅助;与未损伤肢体相比,BMI控制运动不够迅速和流畅。当然,这些可以通过变换算法模块的改进,使大脑信号解码更准确,对电脑及机械臂的指令更明确实现。这一技术为中风、脊髓损伤或其他原因导致的瘫痪患者带来了新的希望。
6 干细胞诱导成为卵细胞[1]
2011年,日本京都研究组[24]报道了干细胞形成可繁殖精子的成果,2012年,该实验室[25]报道了形成卵子的类似过程。上述两项研究所使用的干细胞都是胚胎干细胞(ES)和诱导多潜能干细胞(iPS)。从理论上讲,两者都能够形成所有类型的体细胞,但大多数研究人员一直无法把它们变成生殖细胞,即精子和卵子的前体细胞。由干细胞生物学家Mitinori Saitou率领的研究小组找到了一个可行的方法。他们将ES和iPS细胞置于各种生长因子及蛋白混合的“鸡尾酒”中进行孵育,将其诱导成为原生殖细胞(primordial germ cell,PGC)样细胞。PGC为产生生殖细胞的早期细胞,既可分化为精原细胞,又可分化为卵原细胞[26]。为了得到卵母细胞,研究人员[25]随后将这些PGC与胎鼠的卵巢细胞混合,并最终将其移植到活体小鼠的正常卵巢中。4周零4天后,PGC发育成为卵母细胞。研究人员去除卵巢,得到卵母细胞,并且对其进行体外受精,然后再将得到的受精卵移植到雌鼠体内。大约3周后,正常的小鼠诞生了(图6)。

图6 iPS细胞诱导成为卵细胞,体外受精后诞生正常小鼠[25]
这一发现对于研究生殖细胞成熟过程中的分子机制非常重要。同时,研究人员仍在进一步探索完全体外诱导成熟卵细胞的方法,此方法将会对治疗不孕不育症提供新的途径。如果干细胞能够体外诱导成为生殖细胞,那么完全可以通过不育症妇女的体细胞先诱导成为iPS细胞,再体外诱导iPS细胞形成卵细胞继而体外受精。当然,此过程中的技术难题以及伦理问题仍有待解决。
致谢:感谢宫晓丽在本文撰写过程中对参考资料的搜集。
[1]Breakthrough of the year.The runners-up[J].Science,2012,338(6114):1525-1532.
[2]Green R E,Krause J,Briggs A W,et al.A draft sequence of the Neandertal genome[J]. Science,2010,328(5979):710-722.
[3]Meyer M,Kircher M,Gansauge M T,et al.A high-coverage genome sequence from an archaic Denisovan individual[J].Science,2012,338(6104):222-226.
[4]Pennisi E.The tale of the TALEs[J].Science,2012,338(6113):1408-1411.
[5]Boch J,Scholze H,Schornack S,et al.Breaking the code of DNA binding specificity of TAL-type III effectors[J].Science,2009,326(5959):1509-1512.
[6]Moscou M J,Bogdanove A J.A simple cipher governs DNA recognition by TAL effectors[J].Science,2009,326(5959):1501.
[7]Joung J K,Sander J D.TALENs:a widely applicable technology for targeted genome editing[J].Nat Rev Mol Cell Biol,2012,14(1):49-55.
[8]Hockemeyer D,Wang H,Kiani S,et al.Genetic engineering of human pluripotent cells using TALE nucleases[J].Nat Biotechnol,2011,29(8):731-734.
[9]Carlson D F,Tan W,Lillico S G,et al.Efficient TALEN-mediated gene knockout in livestock[J].Proc Natl Acad Sci U S A,2012,109(43):17382-17387.
[10]Bedell V M,Wang Y,Campbell J M,et al.In vivo genome editing using a high-efficiency TALEN system[J].Nature,2012,491(7422):114-118.
[11]Sun N,Liang J,Abil Z,et al.Optimized TAL effector nucleases(TALENs)for use in treatment of sickle cell disease[J].Mol Biosyst,2012,8(4):1255-1263.
[12]McMahon M A,Rahdar M,Porteus M.Gene editing:not just for translation anymore[J].Nat Methods,2011,9(1):28-31.
[13]Pennisi E.Genomics.ENCODE project writes eulogy for junk DNA[J].Science,2012,337(6099):1159,1161.
[14]Dunham I,Kundaje A,Aldred S F,et al.An integrated encyclopedia of DNA elements in the human genome[J].Nature,2012,489(7414):57-74.
[15]Ward L D,Kellis M.Evidence of abundant purifying selection in humans for recently acquired regulatory functions[J].Science,2012,337(6102):1675-1678.
[16]Pennisi E.The Biology of Genomes.Disease risk links to gene regulation[J].Science,2011,332(6033):1031.
[17]Maurano M T,Humbert R,Rynes E,et al.Systematic localization of common disease-associated variation in regulatory DNA[J]. Science,2012,337(6099):1190-1195.
[18]Landt S G,Marinov G K,Kundaje A,et al.ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia[J].Genome Res,2012,22(9):1813-1831.
[19]Sanyal A,Lajoie B R,Jain G,et al.The long-range interaction landscape of gene promoters[J].Nature,2012,489(7414):109-113.
[20]Redecke L, Nass K, Deponte D P, et al. Natively Inhibited Trypanosoma brucei Cathepsin B Structure Determined by Using an X-ray Laser[J].Science,2012,[Epub ahead of print].
[21]Collinger J L,Wodlinger B,Downey J E,et al.High-performance neuroprosthetic control by an individual with tetraplegia[J].Lancet,2012,[Epub ahead of print].
[22]Hochberg L R,Bacher D,Jarosiewicz B,et al.Reach and grasp by people with tetraplegia using a neurally controlled robotic arm[J].Nature,2012,485(7398):372-375.
[23]Courtine G,Micera S,Digiovanna J,et al.Brain-machine interface:closer to therapeutic reality[J]Lancet,2012,[Epub ahead of print].
[24]Hayashi K,Ohta H,Kurimoto K,et al.Reconstitution of the mouse germ cell specification pathway in culture by pluripotent stem cells[J].Cell,2011,146(4):519-532.
[25]Hayashi K,Ogushi S,Kurimoto K,et al.Offspring from oocytes derived from in vitro primordial germ cell-like cells in mice[J].Science,2012,338(6109):971-975.
[26]Saitou M,Yamaji M.Primordial germ cells in mice[J].Cold Spring Harb Perspect Biol,2012,4(11):a008375.

