三重实时荧光PCR检测纯培养物和环境水体中霍乱弧菌方法的建立及应用*
麻丽丹,王殿夫,王多春,曹际娟
(1.丹东出入境检验检疫局,辽宁 丹东,118000;2.辽东学院畜牧兽医系,辽宁 丹东,118000;3.国家疾控中心腹泻重点实验室,北京,102206;4.辽宁出入境检验检疫局,辽宁 大连,116001)
霍乱是一种急性肠道传染病,对一些亚洲和非洲国家来说目前仍是严重的公共卫生问题,霍乱弧菌主要通过受污染的水和食物传播,与不严格的环境管理密切相关。因此,加强霍乱弧菌环境水体监测,可提供环境污染危险性的评价及对传播来源的分析,为制定霍乱防控对策提供科学依据,是霍乱防治工作的重要组成部分,同时,对环境水体的霍乱弧菌的监测可作为霍乱流行的预警指标[1-4]。水体中的霍乱弧菌检测通常是采用传统的浓缩碱性蛋白胨增菌法。国家卫生部编制的《霍乱防治手册》及GB15984-1995《霍乱诊断标准及处理原则》首选方法也是此法(以下简称常规法)。但在霍乱的非流行期,霍乱弧菌在外环境水体中自然生存状态下菌量较少,同时由于外环境水体中存在大量其它杂菌,会对霍乱弧菌的分离鉴定产生干扰;另外,霍乱弧菌可以以活的非可培养及生物膜状态存在,常规培养无法检测到,对监测技术研发提出了更高的要求。并且研究显示,非O1群和非O139群霍乱弧菌也能引起霍乱的爆发,造成在疫情监测和调查中产生误差,不适应烈性传染病的监测需要[5-12]。
针对这些不足,为了提高外环境水体中霍乱弧菌的检出率,减少漏检的可能性,在确保标本采集和增菌的技术质量前提下,作者将实时荧光PCR(real time fluorecent PCR)运用于对霍乱弧菌的快速筛选。实时荧光PCR检测是近几年发展起来的PCR技术,具有快速、特异、敏感的特点,可对样品进行精确定量。本研究以霍乱弧菌(O1群和O139群)O抗原编码基因(rfb)和特异性的溶血素基因(hlyAA)序列为靶序列[13-14],建立三重实时荧光 PCR的实验方法,为水体标本霍乱弧菌的初筛与纯培养物鉴定提供了一条可行的途径。
1 材料与方法
1.1 实验菌株
霍乱弧菌国际标准菌株N16961(O1群)、MO45(O139群)、O1群 El Tor型霍乱弧菌30株(2002—2008年分离),O139群霍乱弧菌5株(2003—2006年分离),非O1/非O139群霍乱弧菌25株(2007—2008年分离)以及O22和N53、以及拟态弧菌、副溶血弧菌、溶藻弧菌、麦氏弧菌、河弧菌、创伤弧菌、弗尼斯弧菌、嗜水气单胞菌等8种环境水体中能分离到的弧菌。实验菌株均经过血清或生化鉴定,本室保存。
1.2 引物和探针的设计
根据O1群和O139群霍乱弧菌O抗原编码基因rfb序列和霍乱弧菌特异性的溶血素基因(hlyA)序列,使用Oligo 6.0软件设计并合成针对O1群和O139群特异性rfb和霍乱弧菌特异性的溶血素基因(hlyA)的PCR引物和探针(见表1)。

表1 实时荧光PCR所用引物和探针Table 1 Primers and probes for real-time fluorecent PCR assay
1.3 灵敏度实验
将N16961、O22和MO45标准菌株于选择性培养基中培养一定时间,测OD600nm值估计细菌数,将菌液倍比稀释到10-7,取10-4、10-5、10-6、10-7菌液进行平板计数,各稀释度重复3个,按照相应的方法培养后进行菌落计数取平均值确定其真实的菌密度。同时每个稀释液各取1mL菌液于2mL离心管中并作3管重复,用于提取DNA测其OD260nm/OD280nm值并进行实时荧光PCR检测,确定反应体系的灵敏度。
1.4 单重实时荧光PCR反应条件
实时荧光PCR采用20μL反应体系,每个反应中含10μL通用 PCR 反应预混液(TaqMan Gene Expression Master Mix(2×),ABI 产 品);引 物 对(10μmol/L)各0.4μL、探针(10μmol/L)0.2μL、灭菌蒸馏水7μL、模板2μL。反应条件为第一步:预变性,进行1个循环:95℃10min;第二步:PCR反应 共40个循环,95℃15s;60℃1min。
1.5 三重实时荧光PCR反应条件
实时荧光PCR采用20μL反应体系,每个反应中含10μL 通用 PCR 反应预混液(TaqMan Gene Expression Master Mix(2×),ABI产 品)、O1 引 物 对(20μmol/L)各 0.25 μL、O1 探 针 (20 μmol/L)0.125μL、O139引物对(20μmol/L)各1μL、O139探针(20μmol/L)0.5μL 、hlyA 引物对(20μmol/L)各0.5μL、hlyA 探针(20μmol/L)0.4μL、灭菌蒸馏水3.15μL、模板2μL。反应条件为第一步:预变性,进行1个循环:95℃10min;第二步:PCR反应 共40个循环,95℃15s;60℃1min。
1.6 河口水标本的检测和霍乱弧菌的分离
2007年3月~2008年6月间,对丹东市区的鸭绿江河段及其入海口处,采集水体标本共计252份。每份采集450mL,回实验室后加入50mL 10倍浓缩的碱性蛋白胨增菌液,置37℃增菌6h,取其中1mL提取细菌DNA(ABI公司)做模板,按实时荧光PCR反应条件检测O1、O139、非O1和非O139群霍乱弧菌,另分别取10μL涂TCBS平板分离霍乱弧菌,对生长的可疑菌落进行O1群及O139群霍乱诊断血清玻片凝集试验和霍乱弧菌生化试验。
2 结果与分析
2.1 灵敏度分析
采用 N16961(O1 群;1.9×107cfu/mL)、MO45(O139群;2.2×107cfu/mL)、O22(非O1/O139群;1.7×107cfu/mL)进行灵敏度和定量实验研究,实时荧光PCR可检测出至10-6,对应的菌落数分别为19cfu/mL、22cfu/mL和 17cfu/mL。定量关系式分别为:y=-3.122 9 x+37.814,R2=0.998 8;y=-3.206 6 x+41.526,R2=0.995;y= -3.657 7 x+40.882,R2=0.996 6。式中x分别表示O1群霍乱弧菌起始拷贝数以10为底的对数、O139群霍乱弧菌起始拷贝数以10为底的对数和O22霍乱弧菌起始拷贝数以10为底的对数,y表示CT值。灵敏度实验结果和定量实验结果见图1~3。

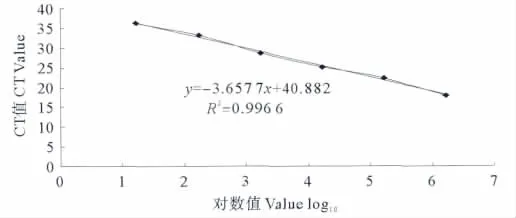
图3 O22群霍乱弧菌定量实验结果Fig.3 Quantity amplified curve for Vibrio cholerae O22serogroups assay
2.2 特异性分析
用建立的实时荧光PCR分别检测不同年代分离、不同型别的霍乱弧菌DNA(31株O1群、27株非O1/O139群和5株O139群)以及其它水体中能分离出的8种常见弧菌。分别进行单重实时荧光PCR反应检测O1群、O139群霍乱弧菌和所有血清型的霍乱弧菌,均表现出了100%的特异性。当模板中同时存在O1群、非O1/O139群和O139群DNA时,采用三重实时荧光PCR法时3种目的片段均获得扩增(见图4),并且结果互不干扰,并且能够与其它弧菌和细菌相区别。说明O1群、O139群特异性rfb基因和霍乱弧菌特异性的溶血素基因(hlyA)共同检测时特异性高,没有交叉反应和干扰现象。

图4 霍乱弧菌三重实时荧光PCR反应特异性实验扩增曲线Fig.4 Specific amplified curve for Vibrio cholerae three multiplex Real-Time Fluorescent PCR
2.3 水体标本的检测
本研究在丹东市鸭绿江水体的市区河段以及入海口处采集了水体进行霍乱弧菌的监测,对252份水体样本进行霍乱弧菌的增菌后分离,同时用建立的O1群、非O1/O139群和O139群霍乱弧菌三重实时荧光PCR检测增菌液DNA。常规分离菌株份数为14份O1群、149份非O1/O139群和2份O139群,而利用实时荧光PCR检测,阳性份数为29份O1群、165份非O1/O139群和6份O139群。其中,所有常规分离方法阳性的标本,其实时PCR检测也均为阳性,同时血清群检测结果也一致。

表2 三重实时荧光PCR对水体标本中霍乱弧菌的检测Table 2 Vibrio cholerae detected by three multiplex Real-Time Fluorescent PCR in Water samples
3 讨论
荧光定量PCR已被应用于检测水体环境中的霍乱弧 菌 等 病 原 菌[15-17],Lyon 等[14]采 用 溶 血 素 基 因(hlyA)做探针,使用TaqMan PCR法检测O1群、O139群以及非O1群和非O139群霍乱弧菌,获得较高的灵敏度。本研究建立了基于荧光探针的实时荧光PCR法,用于水体增菌标本中快速检测O1群、O139群、非O1和非O139群霍乱弧菌的方法,检测的目标基因是O1群和O139群霍乱弧菌的O抗原特异的rfb基因片段和霍乱弧菌特异性的溶血素基因(hlyA)为靶序列,如果扩增出hlyA基因片段而未检出O1群和O139群霍乱弧菌,则可认为检出非O1和非O139群霍乱弧菌。实时荧光PCR法具有高特异性和高灵敏度并且快速可用于水体中霍乱弧菌的筛选。
本研究显示,对标本第一次增菌液使用商品化试剂盒提取DNA标本,对于一份样品,从DNA提取到实时荧光PCR检测时间在2h以内,由于不需要对扩增产物的凝胶电泳分离,因此缩短了对样品的检测周期。另外实时荧光PCR比分离培养有明显的高灵敏性,因此在处理标本时,可以先进行实时荧光PCR检测,然后仅对阳性标本的增菌液进行后续分离鉴定,这样能够明显增加监测工作中处理的标本量,从而明显提高监测工作的灵敏性和工作效率。需注意的是水体中常能检测到霍乱弧菌,但在非流行期,环境中常能检到非产毒的霍乱弧菌,这类菌株有报道能引起轻微的腹泻。
[1] Lipp E K,I N Rivera,A I Gil,et al.Direct detection of Vibrio cholerae and ctxA in Peruvian coastal water and plankton by PCR[J].Appl Environ Microbiol,2003,69(6):3676-3680.
[2] Theron,J J,Cilliers,M,Du Preez,et al.Detection of toxigenic Vibrio cholerae from environmental water samples by an enrichment broth cultivation-pit-stop seminested PCR procedure[J].Appl Environ Microbiol,2000,89(3):539-546.
[3] Tauxe R L,Seminario R,Tapia M,et al.The Latin American epidemic,In I.K.Wachsmuth,F.A.Blake,and O.Olsvik(ed.),Vibrio cholerae and cholera:molecular to global perspectives[M].Washington:ASM Press,1994:321-344.
[4] WHO.Health topics:cholera[EB/OL].[2011-11-23].http://www.who.int/topics/cholera/en/.
[5] 陈德云,张慧红,胡伟宁,等.沉淀缓冲增菌法提高水中El Tor霍乱弧菌阳性检出率的研究和应用[J].中国卫生检验杂志,2004,14(6):693-695.
[6] Kaper J B,Nataro J P,Roberts N C,et al.Molecular epidemiology of non-O1 Vibrio cholerae and Vibrio mimicus in the U.S.Gulf Coast region[J].J Clin Microbiol,1986,23(3):652-654.
[7] Morris J G.Non-O group 1 Vibrio cholerae:a look at the epidemi-ology of an occasional pathogen.Epidemiol[J].Epidemiologic Reviews,1990,12(1):179-191.
[8] Mukhopadhyay A K,Saha P K,Garg S K,et al.Distribution and virulence of Vibrio cholerae belonging to serogroups other than O1 and O139:a nationwide survey[J].Epidemiol Infect,1995,114(1):65-70.
[9] 杨辉,龚成,吴丽娟,等.产毒型O1群霍乱弧菌特异性基因序列实时荧光双重TaqMan PCR快速检测体系的建立[J].中国医药生物技术,2009,4(1):9-13.
[10] 梁晅,李柏生,牟成惠,等.实时荧光PCR技术检测珠江水霍乱弧菌[J].南方医科大学学报,2010,30(8):2000-2001.
[11] 郝民,阚飙.霍乱弧菌的环境生存及其监测[J].疾病监测,2010,25(6):485-489.
[12] 金玉娟,甘莉萍,陈应坚,等.深圳市龙岗区水产品和外环境水体中致病性弧菌污染调查[J].中国卫生检疫杂志,2011,21(6):1512-1514.
[13] 王晓梅,王多春,谭海玲,等.实时PCR检测O1群和O139群霍乱弧菌方法的建立及应用[J].中华流行病学杂志,2007,28(8):768-771.
[14] Lyon W J.TaqMan PCR for detection of Vibrio cholerae O1,O139,non-O1,and non-O139in pure cultures,raw oysters,and synthetic seawater[J].Appl Environ Microbiol,2001,67(10):4685-4693.
[15] Gubala A J.Multiplex real-time PCR detection of Vibrio cholerae[J].Microbiol Methods,2005,65(2):278-293.
[16] Blackstone G M,Nordstrom J L,Vickery M C L,et al.Detection of pathogenic Vibrio parahaemolyticus in oyster enrichments by real time PCR[J].Microbiol Methods,2003,53(2):149-155.
[17] Rivera I N,Lipp E K,Gil A,et al.Method of DNA extraction and application of multiplex polymerase chain reaction to detect toxigenic Vibrio cholerae O1and O139from aquatic ecosystems[J].Environ Microbiol,2003,5(7):599-606.

