一种来源于毕赤酵母的高对映选择性羰基还原酶的性质及底物谱分析
田来强,刘卫东,陈曦,冯进辉,杨洪江,吴洽庆,朱敦明,马延和
1 天津科技大学生物工程学院,天津 3004572 中国科学院天津工业生物技术研究所 工业酶国家工程实验室,天津 300308
一种来源于毕赤酵母的高对映选择性羰基还原酶的性质及底物谱分析
田来强1,2,刘卫东2,陈曦2,冯进辉2,杨洪江1,吴洽庆2,朱敦明2,马延和2
1 天津科技大学生物工程学院,天津 300457
2 中国科学院天津工业生物技术研究所 工业酶国家工程实验室,天津 300308
田来强, 刘卫东, 陈曦, 等. 一种来源于毕赤酵母的高对映选择性羰基还原酶的性质及底物谱分析. 生物工程学报,2013, 29(2): 169−179.
Tian LQ, Liu WD, Chen X, et al. Biochemical characterization and substrate profile of a highly enantioselective carbonyl reductase fromPichia pastoris. Chin J Biotech, 2013, 29(2): 169−179.
手性醇是一类非常重要的化合物,羰基还原酶催化酮的不对称还原生成对应的手性醇。从毕赤酵母Pichia pastorisGS115基因组数据中找到一个潜在的 NADPH依赖的羰基还原酶,研究毕赤酵母P. pastorisGS115中的羰基还原酶。根据其核酸序列设计引物,从P. pastorisGS115基因组中扩增到目的基因ppcr,大肠杆菌BL21 (DE3) 中表达,Ni-NTA纯化,对酶的性质和底物谱进行了研究。PPCR的最适反应温度为35 ℃,最适反应pH为6.0,低于45 ℃时有很好的稳定性。对3-甲基-2-羰基丁酸乙酯的Km和kcat分别为9.48 mmol/L和0.12 s-1。PPCR表现出广泛的底物谱和很高的对映选择性,对醛、α-酮酯、芳香族β-酮酯及芳香族酮都表现出了很好的活性,在测定的底物中,除极少数底物外,ee值均达到 97%以上。因此,PPCR具有较好的应用前景。
羰基还原酶,不对称还原,手性醇,毕赤酵母
2006年美国FDA批准的小分子药物中,80%是手性化合物,75%是手性化合物的单构型立体异构体[1]。手性醇是一类非常重要的化合物,在医药、农药、液晶材料等方面广泛应用,可以通过有机合成和生物转化两种方式获得,生物转化法因其具有环境友好及高立体选择性的特点而被人们认可[2-4],用生物转化生产光学纯的手性醇可以用酶催化法[5-10]或者全细胞催化法[11-14]进行,羰基还原酶是生物转化法中一类非常重要的酶。
羰基还原酶的分类有很多,按催化功能及一级结构可以分为3大类:醛还原酶 (AKR)、短链脱氢酶 (SDR) 及锌依赖的醇脱氢酶。其中SDR是非常庞大的酶家族,该家族中不同酶之间的氨基酸序列同源性较低,仅有几个非常保守的氨基酸残基,但它们都有N端的Rossmann折叠结构、NAD/NADP-结合位点区域以及 C端的催化区域[15-16]。Classical SDRs和 Extended SDRs是SDRs家族中最重要的两个亚族,Classical SDRs家族的酶有共同的辅酶结合区域及结合序列:TGXXX[AG]XG (X代表任意的一种氨基酸);它们亦有保守的催化序列:YXXXK。酪氨酸在催化过程中发挥着重要作用,是催化中心的关键氨基酸,而除了酪氨酸和催化序列中的赖氨酸外,催化序列上游经常有丝氨酸和天冬酰胺对酶的催化起到辅助作用,C端通常在序列上变化较大,可能与酶的底物特异性密切相关[17-18]。
通过基因组数据库挖掘,从P. pastorisGS115中找到一个潜在羰基还原酶基因ppcr,成功克隆并构建到载体PET28a (+) 上,在大肠杆菌Escherichia coliBL21中表达,Ni-NTA纯化,并对 PPCR的酶学性质和生物转化进行了研究。PPCR表现出广泛的底物谱和很好的对映选择性,对醛、α-酮酯、β-酮酯及芳香族的酮表现出了很高的活性和对映选择性 (ee值),是一种新的有应用潜力的羰基还原酶。
1 材料与方法
1.1 材料
P. pastorisGS115为本实验室保存,pET28a (+)载体和E. coliBL21 (DE3) 购自Novagen公司;PCR相关酶购自TaKaRa (大连)公司;限制性内切酶NcoⅠ和XhoⅠ以及 T4 DNA连接酶购自NEB公司;基因组提取、质粒提取纯化试剂盒购自Tiangen公司;Ni-NTA填料购自GE Health Care公司;BCA试剂盒购自CWBIO公司;考马斯亮蓝 R250购自 Solarbio公司;实验中所使用的羰基化合物均来自 Alfa Aesar或Sigma Aldrich公司;其他试剂均为分析纯或色谱纯。
1.2 PPCR的克隆与表达
用酵母基因组试剂盒提取P. pastorisGS115基因组:细胞用 200 μL 裂解液 (2%Triton X-100,1% SDS,100 mmol/L NaCl,10 mmol/L Tris-HCl (pH 8.0),1 mmol/L EDTA)重悬,于-80 ℃冷冻20 min,95 ℃水浴1 min,重复上述过程3次,涡旋振荡30 s,用试剂盒提取基因组。
以潜在羰基还原酶基因 (GenBank Accession No. XM_002491410) 为模板设计扩增引物,正向引物:5'-CATGCCATGG GTACTG TCATTATTTTAACT-3';反向引物:5'-CCG CTCGAG GGTTTGGTACTGTTCCAATA-3',下划线分别为NcoⅠ和XhoⅠ酶切位点。以提取的酵母基因组为模板扩增目的基因,PCR条件:94 ℃预变性 5 min;94 ℃变性 30 s,56.5 ℃退火30 s,72 ℃延伸50 s,30个循环;72 ℃延伸15 min。PCR产物双酶切 (NcoⅠ和XhoⅠ) 后构建到pET28a (+) 上,转化E. coliBL21 (DE3),提取质粒测序并双酶切验证。成功表达的目的蛋白C端有6×His标签。培养基为LB培养基:1%蛋白胨,0.5%酵母粉,1% NaCl。培养时加入50 mg/L的卡那霉素,菌体在37 ℃、200 r/min生长至OD600为0.6~1.0时加入0.5 mmol/L的异丙基-β-D 硫代半乳糖苷 (Isopropyl-β-D-thiogal acto-pyranoside;IPTG),20 ℃诱导 10 h,4 ℃、6 500 r/min离心15 min,收集菌体。
1.3 目的蛋白的纯化
收集菌体用缓冲液A (20 mmol/L Tris-HCl,pH 8.0,5%甘油,500 mmol/L NaCl,5 mmol/L咪唑) 重悬,4 ℃高压匀浆破碎。12 000 r/min离心30 min,弃细胞碎片,上清用0.45 μm的滤膜过滤,Ni-NTA柱纯化,先用A液洗涤,再用B液 (20 mmol/L Tris-HCl,pH 8.0,5%甘油,500 mmol/L NaCl,500 mmol/L 咪唑) 进行0~250 mmol/L咪唑线性洗脱。所得纯酶用Amicon Ultra-15超滤管 (截留分子量:10 kDa)浓缩,缓冲液C (20 mmol/L Tris-HCl,pH 8.0,100 mmol/L NaCl) 脱盐后,用于后续的酶学性质检测和催化反应。蛋白质浓度用BCA试剂盒检测,使用牛血清白蛋白BSA作为标准蛋白。
1.4 酶学性质测定
PPCR在溶液中的聚集状态通过SDS-PAGE及分子筛 (Superdex 200 10/300 GL) 来确定。分子筛所用流动相为20 mmol/L磷酸缓冲液 (pH 7.2),150 mmol/L NaCl。标准蛋白为:Ovalbumin(43.0 kDa),Conalbumin (75.0 kDa),Conalbumin(158.0 kDa),Ferritin (440.0 kDa),Thyroglobulin(669.0 kDa)。根据标准蛋白与目的蛋白的保留体积计算PPCR的相对分子质量。
PPCR活性测定:使用微板光谱仪连续监测340 nm辅酶NADPH的降低 (ε=6 200 L/(mol·cm))来进行。测活反应总体积为200 μL,缓冲液为100 mmol/L的磷酸钠缓冲液 (pH 6.0),底物3-甲基-2-羰基丁酸乙酯终浓度为 6.5 mmol/L,NADPH终浓度为 0.25 mmol/L,PPCR终浓度0.005 g/L。一个酶活性单位定义为每分钟消耗1 μmol NADPH所需要的酶量。
最适pH:测定纯酶在不同pH缓冲液中的酶活来确定。所用缓冲液分别为:100 mmol/L醋酸-醋酸钠缓冲液 (pH 4.0~6.0),100 mmol/L磷酸氢二钠-磷酸二氢钠缓冲液 (pH 6.0~8.0),所用底物为3-甲基-2-羰基丁酸乙酯。
最适反应温度:测定底物 3-甲基-2-羰基丁酸乙酯在磷酸缓冲液 (100 mmol/L,pH 6.0)中不同温度 (25 ℃~60 ℃)下的酶活来确定。酶热稳定性是将酶放在含有 100 mmol/L NaCl的Tris-HCl (20 mmol/L,pH 8.0) 缓冲液中,在不同温度下分别孵育不同时间,然后检测残余酶活力。
动力学参数Km和Vmax:在最适温度、pH条件下,测定不同底物浓度时酶的活力。底物为3-甲基-2-羰基丁酸乙酯,辅酶 NADPH的终浓度为0.25 mmol/L,动力学参数Km和Vmax通过非线性拟合方法进行计算。
1.5 底物谱测定
以不同类型的羰基化合物测定 PPCR活性,对有活性的底物均建立催化反应体系以检测反应产物构型。反应体系 (1 mL) 包含:100 mmol/L的磷酸二氢钠-磷酸氢二钠缓冲液(pH 6.0),葡萄糖18 mg,NADP+0.5 mg,葡萄糖脱氢酶 2~3个酶活单位,0.02 mmol底物(1%的DMSO助溶),PPCR (约2个活力单位)。30 ℃下200 r/min反应24 h后,结束反应。
加入相同体积的甲基叔丁基醚萃取,使用高效液相 (HPLC) 或气相 (GC) 检测产物的产率以及ee值。HPLC检测中将萃取液在通风橱中晾干,用体积比为9∶1的异丙醇和正己烷溶解,用0.22 μm的滤膜过滤后,滤液用于HPLC分析,手性柱为IB、OD-H或AD-H。GC检测中,萃取液用0.22 μm滤膜过滤后,进行GC分析,手性柱为 Gamma DEXTM 225 Capillary Column (30 m × 0.25 mm × 0.25 mm,SUPELCO,Japan)。
2 结果与分析
2.1 羰基还原酶PPCR序列比对
羰基还原酶 PPCR与其他几个来自酵母的羰基还原酶比对结果如图 1所示。PPCR与Classical SDRs家族的酶 (图1中的PSCR、S1、KACR) 有共同的辅酶结合序列和催化序列,即位于N端的辅酶结合序列TGXXX[A/G]XG (图中黑框标出的第52~59位氨基酸) 及位于C端的催化序列YXXXK (图中标出的第208~212位氨基酸)。在催化序列的上游有两个对催化起到辅助作用的氨基酸即:丝氨酸 (图1 PPCR序列中黑框标出的第 192位氨基酸) 和天冬酰胺(图1 PPCR序列中黑框标出的第155位氨基酸)。PPCR与来自毕赤酵母的 PSCR (GenBank Accession No. XP_001387287)[19]、木兰假丝酵母的S1 (GenBank Accession No. BAB21578)[20]、克鲁维酵母属的KACR (GenBank Accession No.BAD01116)[21]和假丝酵母的 CPCR1 (GenBank Accession No. AFD29185)[22]的同源性分别是27%、31%、30%和37%。如图1所示,CPCR1没有Classical SDRs家族的酶的保守序列,因而不属于同一亚族。PPCR与PSCR、S1及KACR相比,C端的同源性较低,可能具有较为独特的底物谱。
2.2 羰基还原酶PPCR的克隆表达与纯化
以P. pastorisGS115基因组为模板,PCR扩增得到与目的片段大小一致 (759 bp) 的基因,测序验证,目的基因电泳图谱如图2所示。构建重组质粒 pET28a(+)-ppcr,在大肠杆菌BL21 (DE3) 中表达,Ni-NTA纯化得到高纯度目的蛋白。表达及纯化图谱见图 3,纯化参数如表1所示。
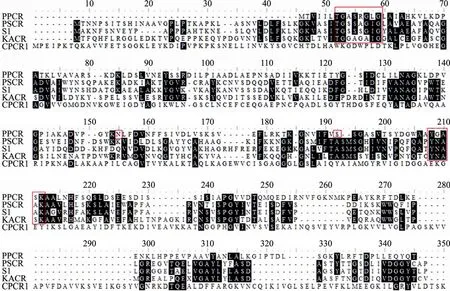
图1 PPCR与其他酵母来源的还原酶序列比对结果Fig. 1 Sequence alignment of PPCR to other carbonyl reductase. Sequences PSCR from Pichia stipotis, S1 from Candida magnolia, KACR from Kluyveromyces aestuarii, CPCR1 from Candida parapsilosis, PPCR of this study from P. pastoris GS115.

图2 PCR产物电泳Fig. 2 Analysis of the PCR product. 1: marker; 2:genomic DNA of P. pastoris GS115; 3: PCR product of PPCR.
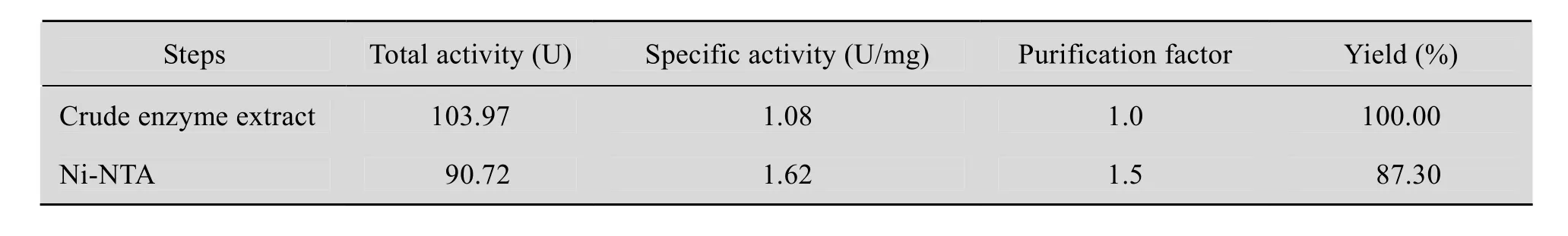
表1 PPCR纯化表Table 1 Purification of PPCR

图3 蛋白表达纯化图谱Fig. 3 SDS-PAGE analysis of expression and purification of PPCR. 1: supernatant of cell-free extract; 2: precipitation of cell-free extract; 3: Ni-NTA flow through; 4: purified PPCR; 5: low MW protein marker.
2.3 酶学性质测定
PPCR单体理论分子量为27.3 kDa,分子筛结果测得其分子量约为60 kDa,因此,PPCR聚集状态为二聚体。
PPCR对 NADPH有较高的偏好性,但对NADH也显示出微弱活性。以 4-氯乙酰乙酸乙酯为底物时,其对NADPH以及NADH的比活分别为0.50 U/mg和0.05 U/mg。
以 3-甲基-2-羰基丁酸乙酯为底物进行最适pH测定,结果如图 4所示,从图中可以看出PPCR的最适pH为6.0左右,磷酸缓冲液与来源于毕赤酵母的PSCR和假丝酵母的CPCR1相似,而来源于木兰假丝酵母的羰基还原酶S1和克鲁维酵母属的KACR的最适pH较低,分别是5.5和5[19,21-23]。当pH在7.5以上时,酶活骤然下降。在磷酸缓冲液里比其他的缓冲液酶活更高,说明PPCR更适合在磷酸缓冲液中反应。
最适温度研究结果表明:羰基还原酶PPCR在35 ℃下表现出了最高活性,而到60 ℃时基本没有活性 (图5),可能在该温度下大部分酶已失活。温度稳定性测定结果 (图 6) 表明:35 ℃时PPCR有很好的稳定性,45 ℃和55 ℃稳定性下降,尤其55 ℃,稳定性下降非常明显。在45 ℃时PPCR的半衰期约40 min,而在55 ℃的半衰期只有9 min左右。
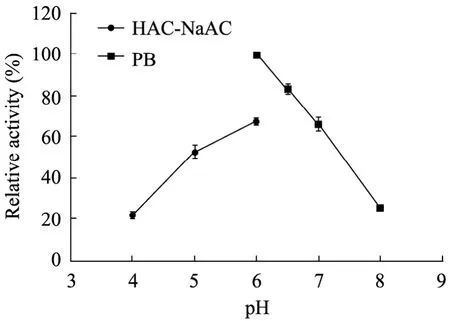
图4 pH对酶活的影响Fig. 4 Effect of pH on enzyme activity. acetic acid /sodium acetate buffer (pH 4.0 to 6.0), sodium phosphate buffer (pH 6.0 to 8.0). The activity of enzyme at optimal buffer and pH (PB buffer, pH 6.0) was taken as 100%.

图5 反应温度对酶活的影响Fig. 5 Effect of temperature on enzyme activity. The activity of enzyme at optimal temperature (35 °C) was taken as 100%.
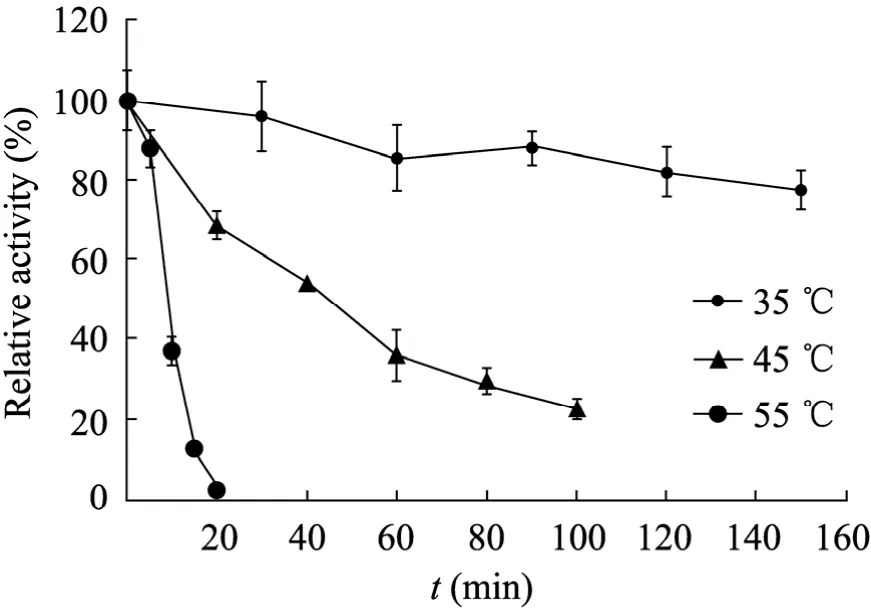
图6 温度对酶稳定性的影响Fig. 6 Effect of temperature on enzyme stability. The activity of enzyme without pre-incubation was taken as 100%.
2.4 动力学参数测定
以 3-甲基-2-羰基丁酸乙酯为底物测定动力学参数Km和kcat,测定条件如1.4所述,反应在最适pH和最适温度下进行,PPCR对底物3-甲基-2-羰基丁酸乙酯的Km为 9.48 mmol/L,kcat为 0.12 s-1。
2.5 底物谱测定
PPCR底物谱的测定结果表明,PPCR有广泛的底物谱,包括醛类、α-酮酯、β-酮酯以及芳香族的酮等。测定结果如表2~5所示。
如表2所示,PPCR能还原对位取代的苯甲醛类底物,对位的取代基类型会影响到羰基还原酶PPCR对醛基还原的活性,它们的顺序是F、Cl、NO2依次增强。
从表 3可以看出,PPCR对 3-甲基-2-羰基丁酸乙酯的活性是 3-苯基-2-羰基丁酸乙酯活性的 6倍以上,引起这种现象的原因可能是苯环与甲基相比位阻比较大,对位阻较小的甲基容易反应,而对位阻较大的苯环反应比较困难。但 PPCR对 α-酮酯都有较好活性,能够非常有效地转化 α-酮酯生成对应的 (R)-a-羟基羧酸酯,而且产物构型单一,ee都大于99%,说明PPCR对α-酮酯的对映选择性很好,在α-酮酯类底物上有很好的应用潜力。

表2 PPCR对醛的活性Table 2 Reduction of various aldehydes by PPCR
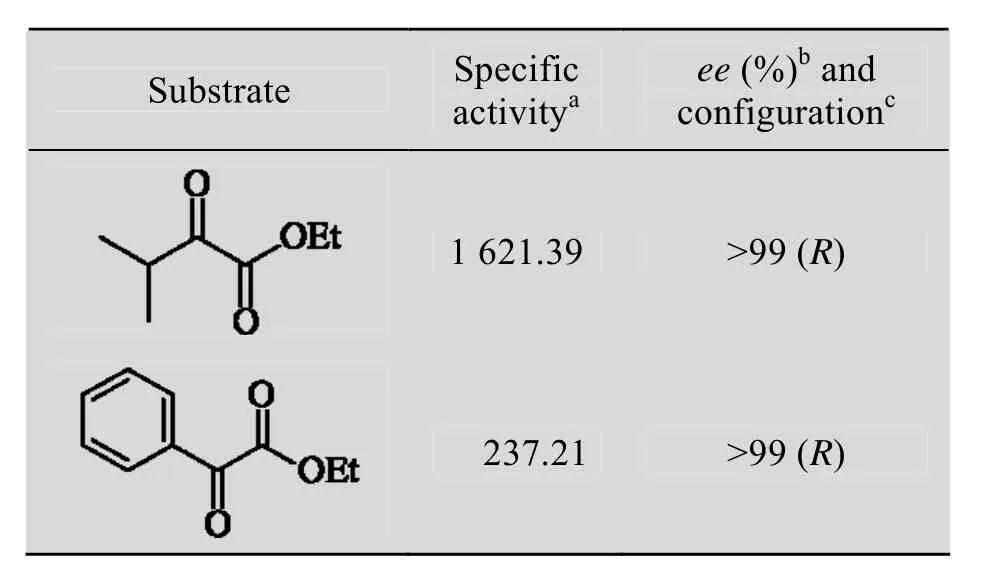
表3 PPCR对α-酮酯的活性Table 3 Reduction of α-ketoesters by PPCR
表4表明,PPCR对β-酮酯表现出较高的活性,并且有很高的立体选择性,取代基 R基团对 PPCR催化 β-酮酯的活性有较大的影响,而对产物ee值的影响很小。从表中可以看出当取代基 R为-CH2CH3时,活性明显好于-CH3,说明底物碳链的长度会影响酶对底物的活力。当取代基为-CH2Cl时,活力明显好于-CH3,说明取代基上的Cl促进了酶与底物的相互作用。对双甲基取代的底物活性降低,可能是双甲基的位阻比单甲基位阻大引起的。PPCR对β-酮酯还原产物ee均在99%以上,说明PPCR对β-酮酯类底物也有很好的应用前景。
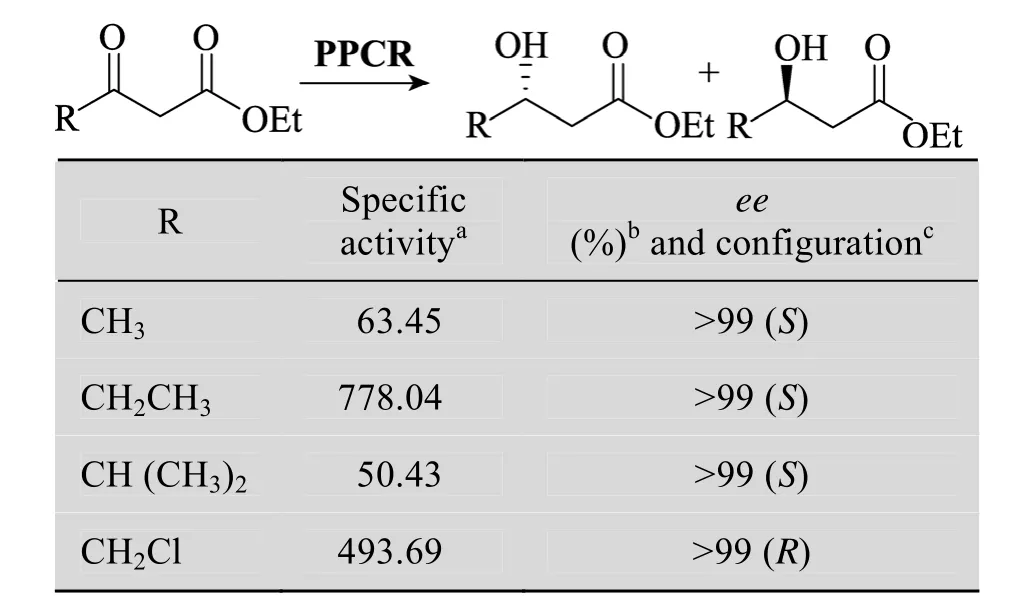
表4 PPCR对β-酮酯的活性Table 4 Reduction of β-ketoesters by PPCR
表5表明,羰基还原酶 PPCR可以催化很多芳香族酮的不对称还原生成相应的手性醇。a-位的取代基对酶的活性和立体选择性都有较大的影响,如a位被溴或羟基取代时,产物的ee值明显降低,而a-位是其他取代基时,催化活性相对较低。对于苯环取代的苯乙酮类底物,苯环上的取代基团对 PPCR的活性有一定的影响,而对产物的ee值的影响较小。
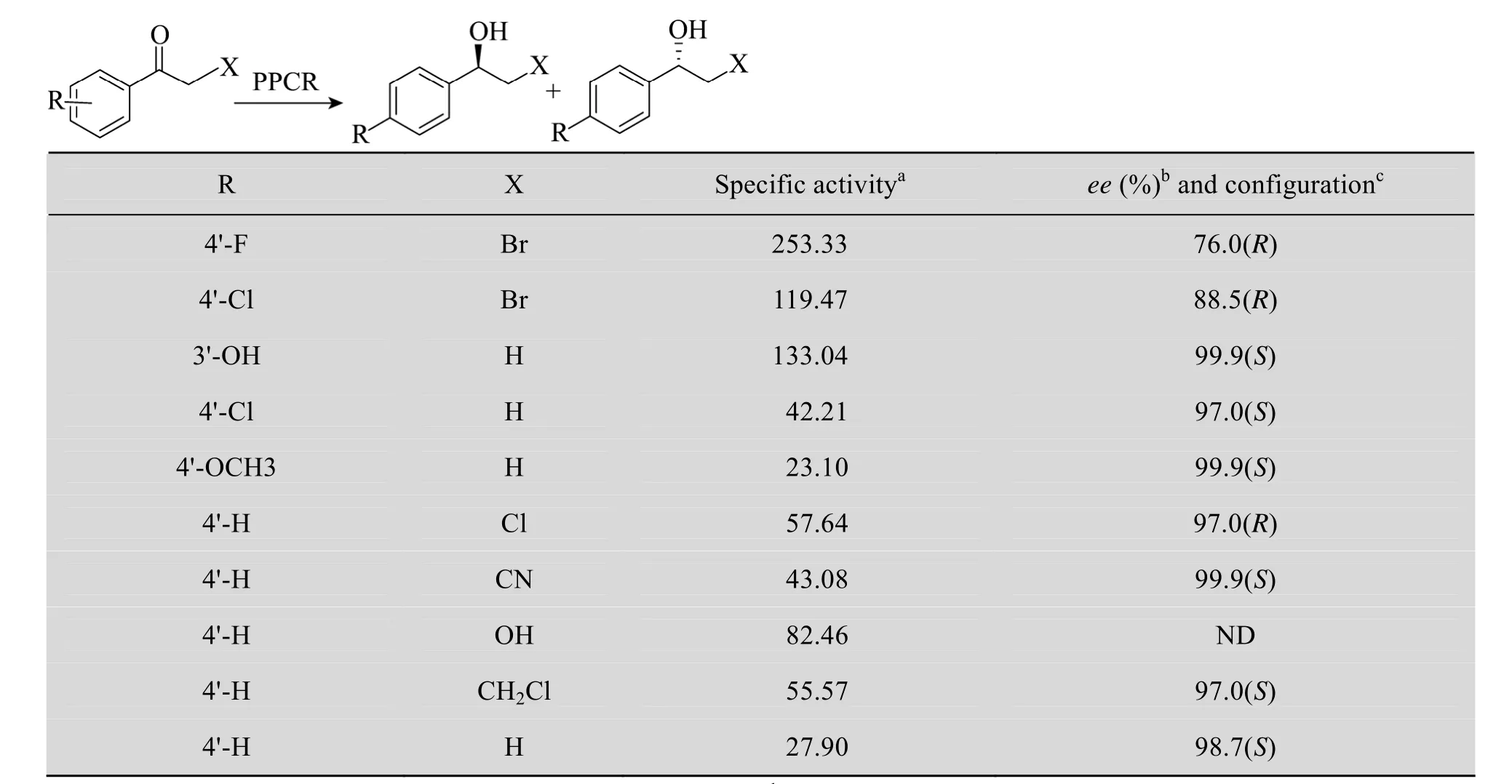
表5 PPCR对芳香族酮的活性Table 5 Reduction of various aryl ketones by PPCR
3 总 结
成功从P. pastorisGS115中克隆出一个新的羰基还原酶,在大肠杆菌中表达,Ni柱纯化得到纯酶,并对其酶学性质进行了研究。该酶以NADPH为辅酶,最适反应温度为35 ℃,最适反应pH为6.0,在45 ℃以下有良好的稳定性。该酶与一些已知的来源于酵母的羰基还原酶同源性较低 (30%左右),使得它们可能具有不同的催化性能。PPCR对醛类、α-酮酯、β-酮酯及芳香族的酮都表现出较高的活性,催化 α-酮酯及β-酮酯还原的产物ee值都达到99%以上,对苯乙酮类化合物的还原,除a溴代或羟基取代的底物外,产物的ee值也大于97%。虽然文献中对图 1中这些已知的来源于酵母的羰基还原酶底物谱有所研究,但只对少数底物如 4-氯乙酰乙酸乙酯的还原反应的立体选择性进行了报道。来源于克鲁维酵母的KACR、木兰假丝酵母的羰基还原酶S1和毕赤酵母的PSCR对β-酮酯和二酮类化合物表现出较好的催化活性,而对其他的酮如苯乙酮类底物,以及醛则没有活性或者没有报道[19-23]。与这些来源于酵母的 Classical SDRs家族的酶相比,PPCR具有一些独特的性质,如较广的底物谱和优良的立体选择性,这有可能与PPCR的C端与PSCR、S1及KACR的同源性较低有关。因此,PPCR在生物催化手性醇的合成中具有较好的应用前景。
[1]Zhu DM, Wu QQ. Engineering the enantioselectivity of biocatalysts. Chin J Biotech,2009, 25(12): 1770−1778 (in Chinese).
朱敦明, 吴洽庆. 生物催化剂立体选择性的基因工程改造. 生物工程学报, 2009, 25(12):1770−1778.
[2]Kaluzna IA, Matsuda T, Sewell AK, et al.Systematic investigation ofSaccharomyces cerevisiaeenzymes catalyzing carbonyl reductions.J Am Chem Soc, 2004, 126(40): 12827−12832.
[3]Patel RN. Biocatalytic synthesis of intermediates for the synthesis of chiral drug substances. Curr Opin Biotechnol, 2001, 12(6): 587−604.
[4]Gruber CC, Lavandera L, Faber K, et al. From a racemate to a single enantiomer: deracemization by stereoinversion. Adv Synth Catal, 2006, 348(14):1789−1805.
[5]Inoue K, Makino Y, Itoh N. Production of(R)-chiral alcohols by a hydrogen-transfer bioreduction with NADH-dependentLeifsoniaalcohol dehydrogenase (LSADH). Tetrahedron:Asymmetry, 2005, 16(15): 2539−2549.
[6]Ema T, Yagasaki H, Okita N, et al. Asymmetric reduction of a variety of ketones with a recombinant carbonyl reductase: identification of the gene encoding a versatile biocatalyst.Tetrahedron: Asymmetry, 2005, 16(6): 1075−1078.
[7]Hanson RL, Goldberg S, Goswami A, et al.Purification and cloning of a ketoreductase used for the preparation of chiral alcohols. Adv Synth Catal,2005, 347(7/8): 1073−1080.
[8]Kaluzna IA, Feske BD, Wittayanan W, et al.Stereoselective, biocatalytic reductions of α-chloro-β-keto esters. J Org Chem, 2005, 70(1):342−345.
[9]Gröger H, Hummel W, Rollmann C, et al.Preparative asymmetric reduction of ketones in a biphasic medium with an (S)-alcohol dehydrogenase under in situ-cofactor-recycling with a formate dehydrogenase. Tetrahedron, 2004,60(3): 633−640.
[10]Shorrock VJ, Chartrain M, Woodley JM. An alternative bioreactor concept for application of an isolated oxidoreductase for asymmetric ketone reduction. Tetrahedron, 2004, 60(3): 781−788.
[11]Zhang J, Witholt B, Li Z. Coupling of permeabilized microorganisms for efficient enantioselective reduction of ketone with cofactor recycling. Chem Commun, 2006(4): 398−400.
[12]Poessl TM, Kosjek B, Ellmer U, et al. Non-racemic halohydrinsviabiocatalytic hydrogen-transfer reduction of halo-ketones and one-pot cascade reaction to enantiopure epoxides. Adv Synth Catal,2005, 347(14): 1827−1834.
[13]Ernst M, Kaup B, Müller M, et al. Enantioselective reduction of carbonyl compounds by whole-cell biotransformation, combining a formate dehydrogenase and a (R)-specific alcohol dehydrogenase. Appl Microbiol Biotechnol, 2005,66(6): 629−634.
[14]Stampfer W, Edegger K, Kaup B, et al. Simple biocatalytic access to enantiopure (S)-1-heteroarylethanols employing a microbial hydrogen transfer reaction. Adv Synth Catal, 2004, 346(1):57−62.
[15]Filling C, Berndt KD, Benach J, et al. Critical residues for structure and catalysis in short-chain dehydrogenases/reductases. J Biol Chem, 2002,277(28): 25677−25684.
[16]Forrest GL, Gonzalea B. Carbonyl reductase. Chem Biol Interact, 2000, 129(1/2): 21−40.
[17]Joernvall H, Persson B, Krook M, et al. Short-chain dehydrogenases/reductases (SDR). Biochemistry,1995, 34(18): 6003−6013.
[18]Tanaka N, Nonaka T, Tanabe T, et al. Crystal structures of the binary and ternary complexes of 7α-hydroxysteroid dehydrogenase fromEscherichia coli. Biochemistry, 1996, 35(24):7715−7730.
[19]Ye Q, Yan M, Yao Z, et al. A new member of the short-chain dehydrogenases/reductases superfamily:purification, characterization and substrate specificity of a recombinant carbonyl reductase fromPichia stipitis.Bioresour Technol, 2009,100(23): 6022−6027.
[20]Yasohara Y, Kizaki N, Hasegawa J, et al.Molecular cloning and overexpression of the gene encoding an nadph-dependent carbonyl reductase fromCandida magnoliae, involved in stereoselective reduction of ethyl 4-chloro-3-oxobutanoate. Biosci Biotechnol Biochem, 2000,64(7): 1430−1436.
[21]Yamamoto H, Mitsuhashi K, Kimoto N, et al. A novel NADH-dependent carbonyl reductase fromKluyveromyces aestuariiand comparison of NADH-regeneration system for the synthesis of ethyl (S)-4-chloro-3-hydroxybutanoate. Biosci Biotechnol Biochem, 2004, 68(3): 638−649.
[22]Jakoblinnert A, Bocola M, Bhattacharjee M, et al.Who's who? Allocation of carbonyl reductase isoenzymes fromCandida parapsilosisby combining bio-and computational chemistry.Chembiochem, 2012, 13(6): 803−809.
[23]Wada M, Kataoka M, Kawabata H, et al.Purification and characterization of NADPH-dependent carbonyl reductase, involved in stereoselective reduction of ethyl 4-chloro-3-oxobutanoate, fromCandida magnoliae. Biosci Biotechnol Biochem, 1998, 62(2): 280−285.
September 19, 2012; Accepted: November 30, 2012
Dunming Zhu. Tel: +86-22-84861962; Fax: +86-22-84861996; E-mail: zhu_dm@tib.cas.cn
中国科学院知识创新工程重点方向项目 (No. KSCX2-EW-G-14), 国家重点基础研究发展计划 (973计划) (No. 2011CB710801) 资助。
Biochemical characterization and substrate profile of a highly enantioselective carbonyl reductase fromPichia pastoris
Laiqiang Tian1,2, Weidong Liu2, Xi Chen2, Jinhui Feng2, Hongjiang Yang1,Qiaqing Wu2, Dunming Zhu2, and Yanhe Ma2
1College of Bioengineering,Tianjin University of Science and Technology,Tianjin300457,China
2National Engineering Laboratory for Industrial Enzymes,Tianjin Institute of Industrial Biotechnology,Chinese Academy of Sciences,Tianjin300308,China
Carbonyl reductases catalyze carbonyl compounds to chiral alcohols that are important building blocks in fine chemical industry. To study carbonyl reductase fromPichia pastorisGS115 (ppcr), we discovered a new gene (ppcr)encoding an NADPH-dependent carbonyl reductase by genomic data mining. It was amplified by PCR from the genomic DNA, and expressed inEscherichia coliBL21 (DE3). The recombinant protein was purified to homogeneity. The optimum temperature was 37○C and the optimum pH of PPCR was 6.0. PPCR was stable below 45○C. TheKmandkcatvalue of the enzyme for ethyl 3-methyl-2-oxobutanoate were 9.48 mmol/L and 0.12 s-1, respectively. The enzyme had broad substrate specificity and high enantioselectivity. It catalyzed the reduction of aldehydes, α-ketoesters, β-ketoesters and aryl ketones to give the corresponding alcohols with >97%eewith only a few exceptions, showing its application potential in the synthesis of chiral alcohols.
carbonyl reductase, asymmetric reduction, chiral alcohol,Pichia pastoris
book=178,ebook=129
Supported by: Knowledge Innovation Project of Chinese Academy of Sciences (No. KSCX2-EW-G-14), National Basic Research Program of China (973 Program) (No. 2011CB710801).
Qiaqing Wu. Tel: +86-22-84861963; Fax: +86-22-84861996; E-mail: wu_qq@tib.cas.cn
(本文责编 陈宏宇)

