草鱼Smad4基因的克隆、生物信息学分析及反义真核载体的构建
吕池波,郁二蒙,谢 骏,王广军,余德光,李志斐,王海英,龚望宝
(1 中国水产科学研究院 珠江水产研究所,农业部热带亚热带水产资源利用与养殖重点实验室,广东 广州510380;2 上海海洋大学 水产与生命学院,上海201306)
在Smads 蛋白家族中已发现9 个成员[1],根据结构和功能的差异可分为通用型、受体激活型和抑制型Smads.通用型Smads 的主要功能是与受体激活型Smads 结合形成异源寡聚体,将信号转至细胞核内,调节基因表达[2],目前仅发现Smad4 这1 种.Smad4 是TGF-β/smads 信号转导通路中的中心转导分子,它通过形成异源寡聚体,将信号转导至细胞核内,从而调控靶基因的转录并诱导下游基因的表达[3],因此它的表达情况与生长、发育等生命过程密切相关.在鱼类研究中,只有斑马鱼Danio rerio、鲤Cyprinus carpio、虹鳟Oncorhynchus mykiss、金鱼Carassius auratus 等少数物种的Smad4 基因被克隆.在鲤体内发现Smad4 基因有不同的组织表达模型,暗示着Smad4 通过改变组织表达水平来调控TGF-β 信号通路[4].在斑马鱼胚胎发育研究中发现Smad4 的时空分布广泛且表达量大,而与之相关的Smad3 基因呈现一种空间限制性表达模式[5].将金鱼的Smad4结构与Smad2、Smad3 及Smad7 比较分析发现,Smad4 编码的氨基酸序列中间连接区域少了42 个氨基酸,这可能就是它在LT-2 细胞中失活的原因[6].目前,有关草鱼Smad4 基因的研究鲜见报道.
研究基因功能常用的方法有基因转导技术、反义技术、基因敲除和敲入、基因诱捕技术等,其中反义技术利用DNA 或RNA 分子通过Watson-Crick 碱基配对原则与目的基因的mRNA 互补结合,通过各种机制使其降解或抑制其编码蛋白的翻译,从而抑制特定目的基因的表达[7].作为反义技术中的一种,反义转基因技术已成为下调生物体内源目的基因表达效果的一种有效手段.其中,具有延熟保鲜特性的转基因番茄Solanum lycopersicum 的培育是最经典的成功范例[8].采用反义转基因技术导入反义重组基因,以抑制机体内的基因表达,可阻断其生理功能.中国科学院水生生物研究所研究团队利用促性腺激素释放激素的反义基因抑制了转基因鲤鱼中促性腺激素的合成和性腺发育[9];反义桃拉综合病毒外衣蛋白基因转基因虾显著增强了病毒抵抗力[10].在有关Smad4 基因的研究中,通过构建反义Smad4 真核表达载体可以抑制Smad4 蛋白的表达,从而减少Ⅰ型胶原的表达量[11].为此,本研究拟通过PCR 方法和RACE 技术获得草鱼Smad4 cDNA 全序列,并将开放阅读框(Open reading frame,ORF)序列反向插入真核表达载体pcDNA3.1(+)构建Smad4 基因反义真核表达载体pcDNA3.1(+)-Anti-Smad4,期望为进一步研究Smad4 基因在草鱼胚胎发育过程中的生理机制打下基础.
1 材料与方法
1.1 试验材料
2010年5月在广东省中山食品水产集团有限公司南头养殖场采集3 尾体质健康的草鱼,体质量为(2.0 ±0.10)kg,去鳞解剖后,采集肌肉组织,并立即置于液氮中保存,运回实验室,置于-40 ℃冰箱冷冻保存备用.
1.2 主要试剂
总RNA 提取试剂盒(百泰克公司);DNA Marker DL2000、PrimeScriptTMRT-PCR Kit、pMD19-T 载体、DH5α、LA Taq DNA 聚合酶、EcoR Ⅰ、Xho Ⅰ和T4 DNAligase(大连TaKaRa 公司);SMARTTMRACE cDNA Amplification Kit(Clontech 生物工程公司);Plasmid Mini KitⅠ、Gel Extraction Kit(OMEGA 公司).
1.3 试验方法
1.3.1 总RNA 提取与cDNA 的合成 RNA 提取参照百泰克公司总RNA 提取试剂盒说明书进行,利用琼脂糖凝胶电泳检测RNA 纯度,然后使用微量分光光度计测总RNA 浓度.按照TaKaRa PrimeScriptTMRT-PCR Kit 说明书进行反转录合成cDNA:Oligo dT Primer 1 μL、总RNA 4 μL、dNTP Mixture 1 μL、RNase Free ddH2O 4 μL,反应程序为65 ℃5 min,4 ℃条件下保存;然后加入5×PrimeScript ⅡBuffer 4 μL、RNase inhibitor 0.5 μL、PrimeScript ⅠRTase 1 μL 和RNase Free ddH2O 4.5 μL,反应程序为42 ℃ 30 min,95 ℃5 min,4 ℃条件下保存.
1.3.2 草鱼Smad4 基因cDNA 全序列克隆 以反转录合成的cDNA 为模板,PCR 扩增草鱼Smad4 基因核心序列片段.PCR 反应体系25 μL:ddH2O 19.7 μL、PCR Buffer 2.5 μL、4×dNTP Mixture 0.5 μL、cDNA 1 μL、Smad4 F(5'-TGTGTGACCATACAGAGAAC-3')0.5 μL、Smad4 R(5'-TCCAGCAGGGCGTCTCTTTA-3')0.5 μL 、LA Taq 0.3 μL.PCR 反应条件:预变性94 ℃5 min;94℃1 min,65 ℃30 s,72 ℃90 s,30个循环;然后72 ℃延伸8 min.PCR 产物经10 g·L-1琼脂糖凝胶电泳检测后回收并克隆、测序,所得的cDNA 片段与GenBank 数据库中的已有物种的Smad4 cDNA 序列进行BLAST 相似性分析.
3'RACE 试验按照SMARTTMRACE cDNA Amplification 说明书进行.用上述的逆转录反应液、5×PCR Buffer 10 μL、ddH2O 28.75 μL、TaKaRa Ex Taq® HS 0.25 μL、Smad4-3' RACE(5'-CCTGCTATCAGTTTGTCT-3')0.5 μL、M13 Primer M4 0.5 μL 进行首次PCR 反应,反应条件:94 ℃预变性3 min;94 ℃30 s,55 ℃30 s,72 ℃1 min,共30 个循环;72 ℃延伸10 min.巢式PCR 使用的引物为M13 Primer M4 和Smad4-3' 2RACE(5'-ACCCACGGCAGAGCATTA-3').PCR 产物在琼脂糖凝胶中电泳检测后进行胶回收,并克隆测序.
根据前一步克隆的Smad4 基因的cDNA 序列,设计2 个下游引物Smad4-5'RACE(5'-GTAGGGGTTCACACAGAC-3')与Smad4-5'2RACE(5'-AGCGTTCTCTGTATGGTC-3'),合成5'RACE cDNA 方法参照试剂盒说明书进行.首次PCR 反应体系:10×Advantage 2 PCR Buffer 5 μL、dNTP Mix(10 mmol/L)1 μL、10×UPM 5 μL、Smad4-5' RACE 1 μL、5'-RACEReady cDNA 2.5 μL、50×Advantage 2 Polymerase Mix 1 μL、PCR-Grade water 34.5 μL.反应程序与3'RACE一致,将退火温度改为65 ℃,循环数为25.取首次PCR 产物5 μL,用Tricine-EDTA Buffer 稀释50 倍.然后取5 μL 作为模板,引物为Smad4-5' 2RACE 和NUP 各1 μL,进行巢式PCR 反应.RACE 试验产物进行胶回收并克隆测序.
1.3.3 草鱼Smad4 基因的生物信息学分析 Smad4基因的分析、Smad4 蛋白的疏水性分析及功能结构域分析在expasy 网站(http:∥www.expasy.org/)完成;蛋白功能位点及二级结构分析在https:∥www.predictprotein.org/完成;系统发育树使用MEGA4.0软件完成.
1.3.4 Smad4 基因ORF 片段的获得与鉴定 以反转录合成cDNA 为模板,设计引物P1(5'-ATGTCTATCACAAACACGCCCACC-3')、P2(5'-TCAGTCTAACGGCGTGGGGTC-3'),PCR 扩增Smad4 基因ORF片段,产物经凝胶电泳检测后回收并克隆测序.
1.3.5 带酶切位点的Smad4 基因ORF 片段的获得与鉴定 将上述进行过验证的菌液提取质粒pMD19-T-Anti-Smad4,并以此为模板,P3(5'-CCGCTCGAGATGTCTATCACAAACACG-3')、P4(5'-CGGAATTCTCAGTCTAACGGCGTGGG-3')(加粗的为保护碱基,带下划线为酶切位点)为引物进行PCR 扩增,反应体系及反应条件同1.3.2.获得两端含有XhoⅠ和EcoRⅠ2 个酶切位点的Smad4 ORF 片段.PCR 产物经凝胶电泳检测后回收并克隆测序.
1.3.6 反义真核表达载体的构建 如图1 所示,将Smad4 片段反向插入pcDNA3.1(+)载体中.同时用XhoⅠ和EcoRⅠ对质粒pcDNA3.1(+)进行双酶切,反应体系:Xho Ⅰ1 μL,EcoR Ⅰ1 μL,10×H Buffer 2 μL,质粒1 μg,加无菌水至20 μL.反应条件:37 ℃,2 h.利用相同的反应体系对pMD19-T-Anti-Smad4 进行双酶切.产物进行10 g·L-1琼脂糖凝胶电泳,按照OMEGA胶回收试剂盒说明书纯化回收目的片段.连接载体和Smad4 片段,反应体系:10×T4 DNA Ligase buffer 2 μL,Smad4 酶切片段15 μL,pcDNA3.1(+)酶切片段2 μL,T4 DNA Ligase 1 μL.16 ℃连接15 h,转化并将菌液送至上海英骏生物技术有限公司测序.
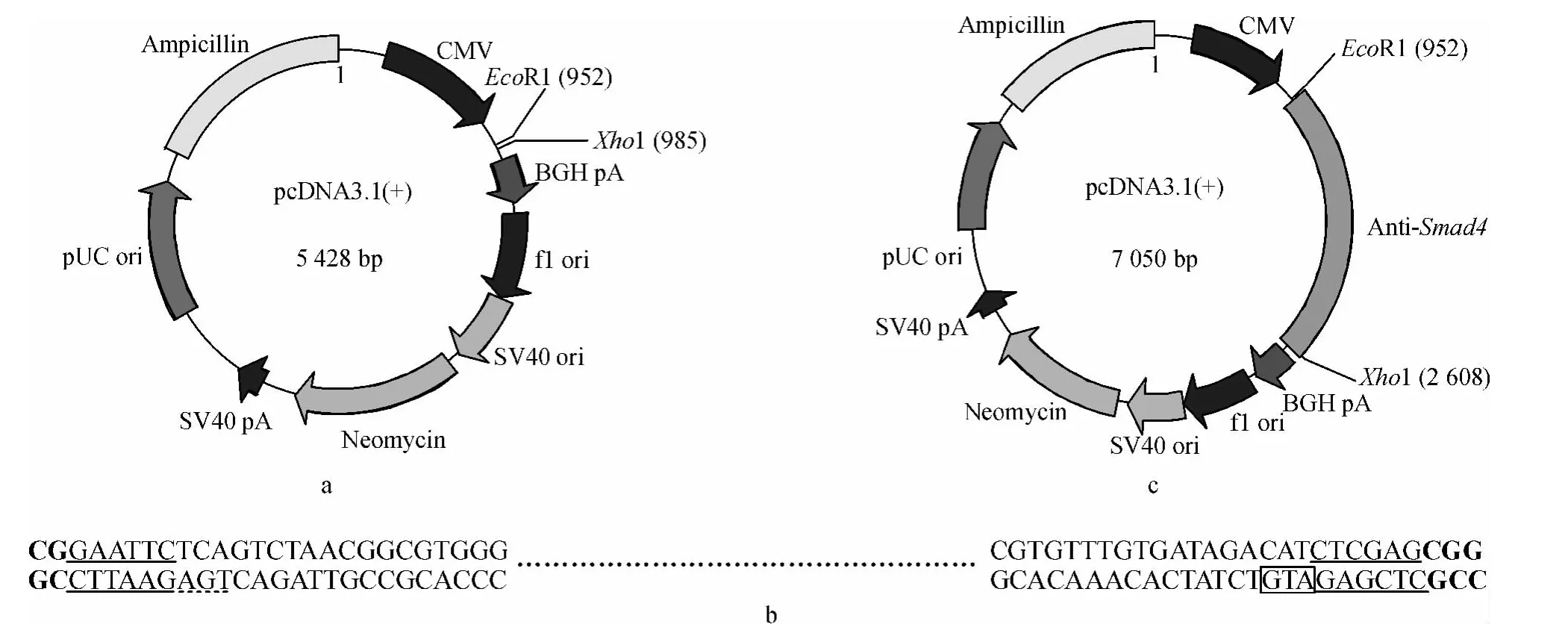
图1 反义Smad4 重组载体图谱Fig.1 Recombinant vector of antisense Smad4
1.3.7 重组载体的PCR 检测和酶切鉴定 提取重组载体pcDNA3.1(+)-Anti-Smad4,方法按照OMEGA 质粒提取试剂盒说明书进行.以P1、P2 为引物,抽提的质粒为模板进行PCR 检测,反应体系和条件同1.3.2.用XhoⅠ和EcoRⅠ双酶切重组载体pcDNA3.1(+)-Anti-Smad4,酶切产物进行10 g·L-1琼脂糖凝胶电泳分析.然后,参照测序结果筛选阳性的反义表达载体.
2 结果与分析
2.1 草鱼Smad4 基因cDNA 全序列及推导的氨基酸序列分析
测序结果显示:草鱼Smad4 cDNA 全长为2 974 bp,开放阅读框为1 644 bp(第367~2 010 bp),编码547 个氨基酸,其相对分子质量为59 690.3,分子式为C2632H4073N753O791S24,理论等电点为6.5,无信号肽和跨膜序列.草鱼Smad4 基因含有2 个N-糖基化位点,2 个蛋白激酶C 磷酸化位点,4 个酪蛋白激酶II 磷酸化位点,9 个N-豆蔻酰化位点和1 个酰胺化位点(图2).
2.2 草鱼与其他生物Smad4 蛋白氨基酸序列的相似性比较和进化树分析
草鱼Smad4 氨基酸序列与其他生物的相似性比较高,其全长序列与斑马鱼、鲤、非洲爪蟾Xenopus laevis、小鼠和人的相似性分别达到了95.61%、94.33%、84.63%、85.77%和85.95%(表1).

图2 草鱼Smad4 基因全长cDNA 序列推导的氨基酸序列Fig.2 Smad4 gene full-length cDNA sequence-deduced amino acid sequence from grass carp
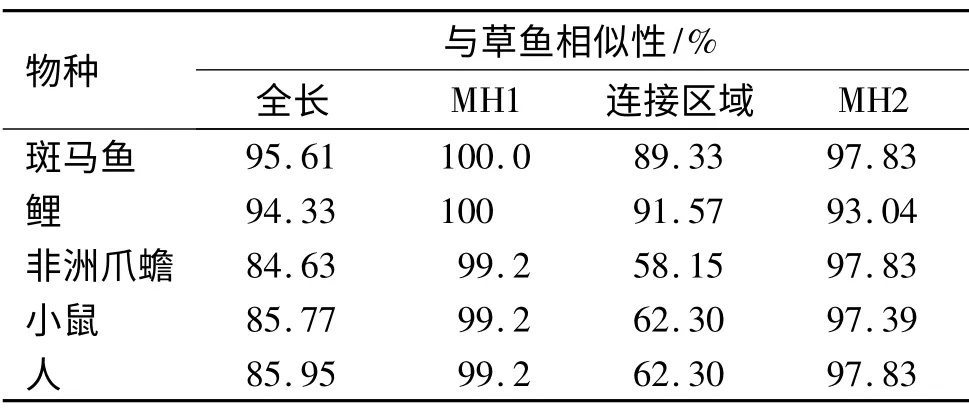
表1 草鱼Smad4 蛋白氨基酸序列相似性比较Tab.1 Homology comparison of Smad4 amino acid sequence from grass carp
用MEGA 软件构建了Smad4 蛋白氨基酸序列的系统进化树(图3).分析结果显示,草鱼和鲤鱼Smad4 蛋白的亲缘关系比较近.
2.3 Smad4 蛋白疏水性分析
Protscale 软件的疏水性预测结果如图4 所示,Smad4 蛋白存在6 个高分值(Scare>1.5)峰,分布在第16~18、473~474、477、480~481、483、497 氨基酸位点,属于高疏水性域;19 个低分值(Scare<-1.5)峰,分布在第26~28、45~50、100~101、103~104、106~107、158~164、179、243~245、247、250、255~258、265、267、270、277~285、314、414~415、436~444、510~513 氨基酸位点,属于高亲水性域.
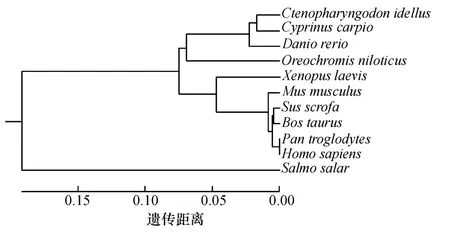
图3 草鱼Smad4 蛋白氨基酸序列系统进化树分析Fig.3 Phylogenetic analysis of amino acid sequence of Smad4 protein from grass carp
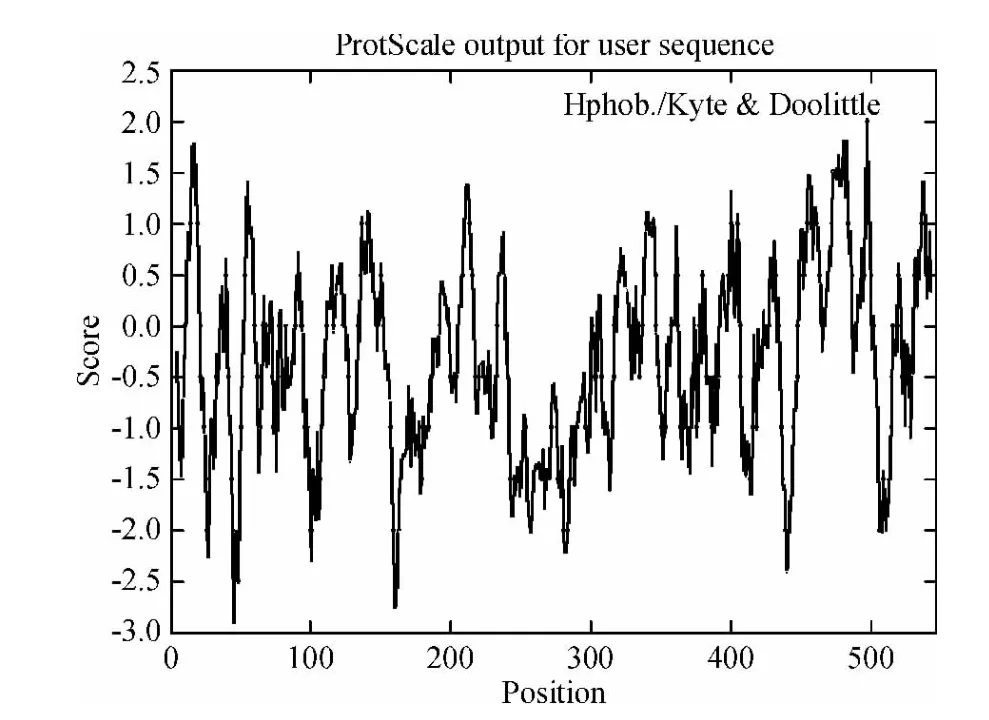
图4 Smad4 蛋白氨基酸序列的疏水性分析Fig.4 Hydrophobicity analysis of amino acid sequence of Smad4 protein
2.4 Smad4 蛋白功能结构域和二级结构特征预测
用ExPasy 服务器中的Prosite 工具对Smad4 蛋白进行预测,发现2 个结构域(图5a),第1 个为MH1 结构域,由第15~139 共125 个氨基酸残基组成;第2 个是MH2 结构域,由第318~547 共230 个氨基酸残基组成.
Predict protein 软件预测结果如图5b 所示,Smad4蛋白二级结构中有5 个α 螺旋结构域,分布在第12~21、30~60、368~378、435~461、525~537 氨基酸区域;有12 个β-折叠结构域,分布在第69~71、89~95、319~325、333~336、342~345、382~387、392~395、402~405、421~424、430~433、493~502、518~523 氨基酸区域,其余区域均为无规则卷曲域.
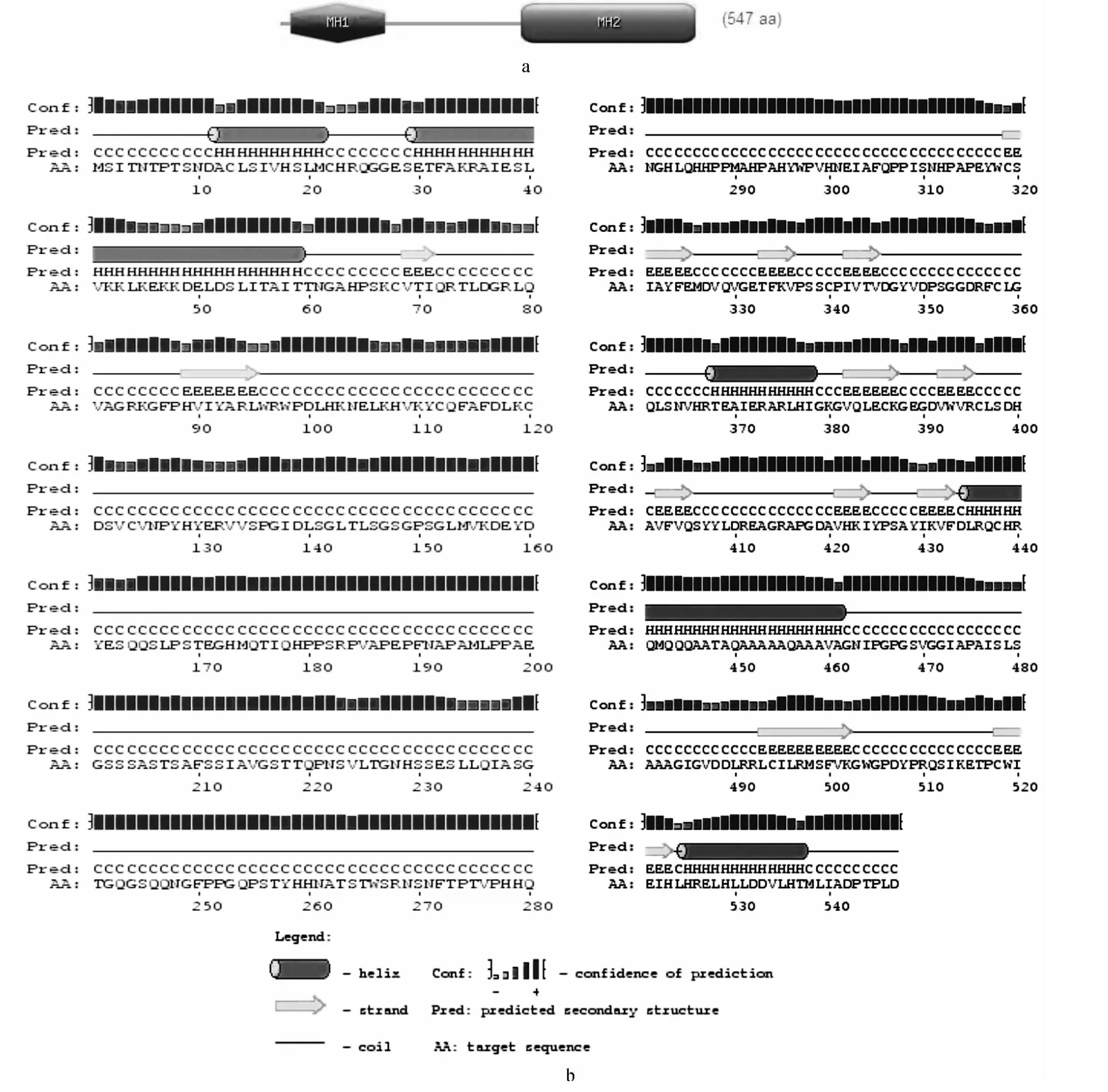
图5 草鱼Smad4 蛋白序列二级结构和功能结构域预测分析Fig.5 Prediction of domains and secondary structure of Smad4 protein from grass carp
2.5 重组质粒构建与PCR 检测和酶切鉴定
以pcDNA3.1(+)-Anti-Smad4 为模板的PCR扩增,可见1 条长度约1.6 kb 的目的条带.将测序结果与已知的草鱼Smad4 基因ORF 序列比对,序列一致,Smad4 片段已经成功地反向插入了pcDNA3.1(+)载体.如图6 所示,pcDNA3.1(+)共由5 428 bp 组成,EcoRⅠ和XhoⅠ分别位于952 和985 bp 处,经双酶切后除去33 bp,再加上新插入的片段1 655 bp,重组载体的长度为7 050 bp.重组表达载体pcDNA3.1(+)-Anti-Smad4 经XhoⅠ和EcoRⅠ双酶切,可见在1.6 kb 处有1 条目的条带.
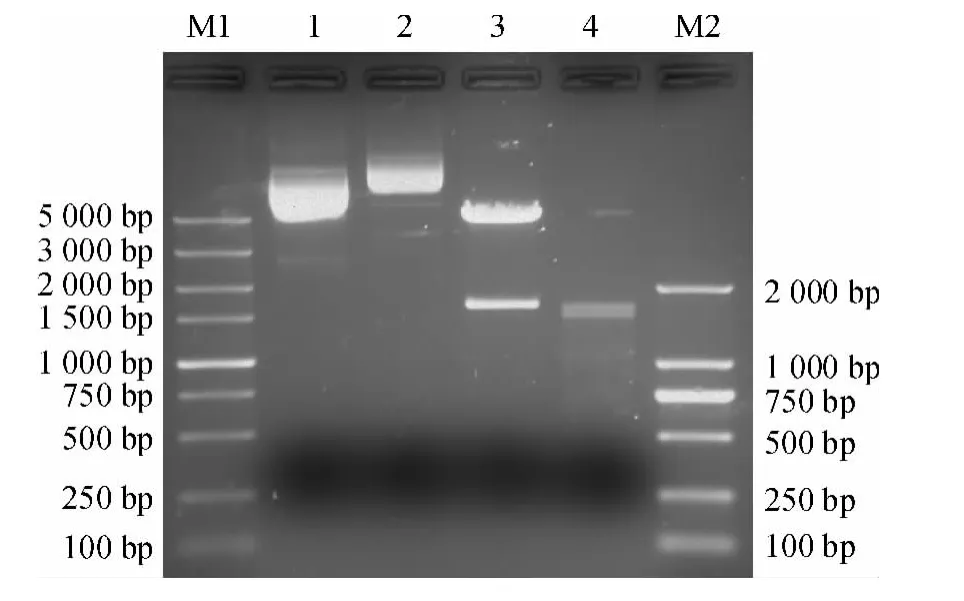
图6 草鱼Smad4 反义真核表达载体的PCR 检测和双酶切鉴定Fig.6 PCR and double enzyme identification of recombinant vector pcDNA3.1(+)-Anti-Smad4 from grass carp
3 讨论与结论
从系统进化树分析结果来看,Smad4 基因推导的氨基酸序列系统进化树主要分成2 大支:鱼类和哺乳类.系统进化树显示,草鱼与鲤、斑马鱼等鲤科鱼类具有较近的亲缘关系,其中与鲤的亲缘关系最近.进化树中各个物种Smad4 蛋白的亲缘远近与各自的分类地位相符合,说明Smad4 蛋白的氨基酸序列具有很高的保守性.相似性比较结果显示,草鱼Smad4 基因符合Smad4 基因高度保守的特征,与其他鱼类相比,氨基酸序列的相似性都在90%以上;与人和小鼠等其他物种相比,氨基酸序列的相似性则达到了85%左右.草鱼Smad4 基因推导的氨基酸序列系统进化树分析结果和相似性分析都与传统的鱼类分类地位相吻合.
Smads 蛋白非常保守,它们结构相似,均由保守的N 端MH1 结构域[12]、C 端MH2 结构域[13]和富含脯氨酸的连接区域构成.MH1 结构域是Smads 蛋白的1 个DNA 绑定区域,通过β-发夹结构与DNA 结合,MH2结构域位于Smads 蛋白的C 端,对与I 型受体结合、磷酸化触发、异源低聚物形成及转录激活等都有作用,对于Smad4 与其他Smad 分子形成低聚物及调节转录有十分重要的作用.结构域MH1 和MH2 可相互作用,MH2 结构域抑制MH1 结构域的DNA 结合功能,MH1结构域抑制MH2 结构域的转录激活功能[14].本试验克隆的草鱼Smad4 基因经软件分析发现,由它推导的氨基酸序列MH1 结构域位于第15~139 个氨基酸残基处,MH2 结构域位于318~547 个氨基酸残基处,中间为连接区域,是典型的Smads 蛋白结构.
已有研究发现Smad4 蛋白的2 个功能结构域(MH1、MH2)比较保守,而富含脯氨酸的连接区域是可以进行不同剪切的,不同的选择性剪切可以造成Smads 蛋白的连接区域长短不同.在非洲爪蟾体内发现了2 种Smad4 的剪切体,尽管二者在连接区域存在差异,但是两端的功能结构域都非常保守[15],而且2 个剪切体都具有相似的转录活性[16],在鲤中也发现了4 种不同的Smad4 剪切体[12],它们的MH1 结构域均含有141 个氨基酸残基,MH2 结构域氨基酸数量也相差不多.草鱼与其他几个物种的Smad4 氨基酸序列相似性比较显示,MH1 结构域的相似性极高,与斑马鱼和鲤达到了100%,MH2 结构域的相似性也很高,除了鲤以外,相似性均在97%以上,而中间连接区域的相似性则相对较低,除了与斑马鱼和鲤相比在90%以外,与其他几个物种相比均在60%左右.据此推测,Smad4 蛋白结构和功能的保守性可能与MH1 和MH2 这2 个功能结构域的高度保守有关.
Smad4 是TGF-β 信号传导的中心环节,它在分子伴侣TRAP1(TNFR associated protein1,肿瘤坏死因子受体相关蛋白1)[17]的作用下与受体激活型Smads 结合形成异源复合物,将信号传导至细胞核内,研究发现,受体激活型Smad 与Smad4 形成的异源复合物是由2 个受体激活型Smads 和Smad4 组成的异源三聚体[18],异源三聚体进入细胞核后通过3种方式作用于靶基因.第1 种是异源三聚体直接与DNA 结合,Smad4 通过识别靶基因上的5'-AGAC-3'序列,并通过N-端MH1 结构域利用β-发夹结构与之结合,从而调节靶基因的转录[19];第2 种是Smads蛋白通过与其他DNA 结合因子相互作用而与DNA结合[20];第3 种是Smads 蛋白复合物与非DNA 结合因子相互作用[16].根据软件预测结果,草鱼Smad4蛋白的MH1 结构域也存在2 个β-折叠,具备形成β-发夹结构的条件,暗示草鱼Smad4 蛋白可能与其他物种的Smad4 蛋白一样具有DNA 结合功能[19].
1984年,Lzant 等[21]就发现反义RNA 具有下调目的基因表达的功能.目前,反义基因技术已在鱼类研究中得到了应用,Uzbekova 等[22]研究发现带重组反义sGnRH(促性腺激素释放激素)载体的转基因大西洋鲑鱼Salmo salar 的sGnRH 表达量远远低于非转基因鱼.有关鱼类的Smad4 反义基因技术的研究鲜见报道,本试验构建了草鱼反义Smad4 基因真核表达载体,以探究草鱼Smad4 基因在其胚胎发育等过程中的作用.在构建反义真核表达载体的过程中,本文选择真核表达载体pcDNA3.1(+)[23],载体上人巨噬细胞介导的启动子(CMV)保证重组蛋白高效地表达[24],丰富的多克隆位点有助于目的片段的有效插入和扩增,牛生长激素poly A 尾巴(BGH pA)能高效地终止转录[25],载体上的pUC ori 则保证了载体在大肠埃希菌Escherichia coli 体内的增殖和合成.因此,本试验构建的草鱼反义Smad4 基因真核表达载体pcDNA3.1(+)-Anti-Smad4 可为进一步研究草鱼Smad4 基因的功能及在胚胎发育过程的作用机理奠定基础.
[1]GRONEBERG D A,WITT H,ADCOCK I M,et al.Smads as intracellular mediators of airway inflammation[J].Exp Lug Res,2004,30(3):223-250.
[2]CHEN Xin,WEISBERG E,FRIDMACHER V,et al.Smad4 and FAST-1 in the assembly of activin-nesponsive factor[J].Nature,1997,389(6646):85-89.
[3]CHEN X,RUBOCK M J,WHITMAN M.A transcriptional partner for MAD protein in TGF-beta signaling[J].Nature,1996,383(6602):691-696.
[4]WANG Zhiyong,FUTAMI K,NISHIHARA A,et al.Four types of Smad4 found in the common carp,Cyprinus carpio[J].J Exp Zool:Part B:Mol Dev Evol,2005,304(3):250-258.
[5]DICK A,MAYR T,BAUER H,et al.Cloning and characterization of zebrafish smad2,smad3 and smad4[J].Gene,2000,246(1/2):69-80.
[6]LAU M T,GE Wer.Cloning of Smad2,Smad3,Smad4,and Smad7 from the goldfish pituitary and evidence for their involvement in activin regulation of goldfish FSH promoter activity[J].Gen Comp Endocr,2005,141(1):22-38.
[7]CROOKE S T.Progress in antisense technology[J].Annu Rrv Med,2004(55):61-95.
[8]OELLER P W,LU M W,TANLOR P L,et al.Reversible inhibition of tomato fruit senescence by antisense RNA[J].Science,1991,254(5030):437-439.
[9]HU Wei,LI Shuangfei,TANG Bin,et al.Antisense for gonadotropin-releasing hormone reduces gonadotropin synthesis and gonadal development in transgenic common carp(Cyprinus carpio)[J].Aquaculture,2007,271(1/2/3/4):498-506.
[10]LU Yuanan,SUN P S.Viral resistance in shrimp that express an antisense Taura syndrome virus coat protein gene[J].Antiviral Res,2005,67(3):141-146.
[11]XU X B,HE Z P,LIANG Z Q,et al.Obstruction of TGF-beta1 signal transduction by anti-Smad4 gene can therapy experimental liver fibrosis in the rat[J].Chin J hepatol,2004,12(5):263-266.
[12]GRISHIN N V.MH1 domain of Smad is a degraded homing endonuclease[J].J Mol Biol,2001,307(1):31-37.
[13]SHI Yigong.Structural insights on Smad function in TGFβ function[J].Bioessays,2001,23(3):223-232.
[14]SHI Yigong,MASSAGUE J.Mechanisms of TGF-β signaling from cell membrane to the nucleus[J].Cell,2003,113(6):685-700.
[15]HOWELL M,ITOH F,PIERREUX C E,et al.Xenopus Smad4beta is the co-smad component of developmentally regulated transcription factor complexes responsible for induction of early mesodermal genes[J].Dev Biol,1999,214(2):354-369.
[16]MASUYAMA N,HANAFUSA H,KUSAKABE M,et al.Identification of two Smad4 proteins in Xenopus:Their common and distinct properties[J].J Biol Chem,1999,274(17):12163-12170.
[17]WURTHNER J U,FRANK D B,FELICI A,et al.Transforming growth factor-β receptor-associated protein 1 is a Smad4 chaperone[J].J Biol Chem,2001,276(22):19495-19502.
[18]CHACKO B M,QIN B Y,TIWARI A,et al.Structural basis of heteromeric smad protein assembly in TGF-beta signaling[J].Mol Cell,2004,15(5):813-823.
[19]DAI Jiale,BANSAL R K,KERN S E.G1cell cycle arrest and apoptosis induction by nuclear Smad4/Dpc4:Phenotypes reversed by a tumorigenic mutation[J].Cell Biol,1999,96(4):1427-1432.
[20]DERYNCK R,ZHANG Y E.Smad-depended and Smadindepended pathways in TGF-β family signaling[J].Nature,2003,425(6958):577-584.
[21]IZANT J G,WEINTRAUB H.Inhibition of thymidine kinase gene expression by anti-sense RNA:A molecular approach to genetic analysis[J].Cell,1984,36(4):1007-1015.
[22]UZBEKOVA S,CHYB J,FERRIERE F,et al.Transgenic rainbow trout expressed sGnRH-antisense RNA under the control of sGnRH promoter of Atlantic salmon[J].J Mol Enoocrinol,2000,25(3):337-350.
[23]CHEN Rongyi,CHEN Hongxiang,LIN Jiaxi,et al.In-vivo transfection of pcDNA3.1-IGFBP7 inhibits melanoma growth in mice through apoptosis induction and VEGF down expression[J].J Exp Clin Canc Res,2010,29(1):13-20.
[24]MICHAEL B,FRANK W,GERHARD J,et al.A very strong enhancer is located upstream of an immediate early gene of human cytomegalovirus[J].Cell,1985,41(2):521-530.
[25]GOODWIN E C,ROTTMAN F M,The 3'-flanking sequence of the bovine growth hormone gene contains novel elements required for efficient and accurate polyadenylation[J].J Biol Chem,1992,267(23):16330-16334.

