一株嗜盐古菌DSFD111的分类学鉴定
程露露,傅金辰,杨丹丹,张含薇,俞姗杉,陈 敏
(杭州师范大学生命与环境科学学院,浙江 杭州 310036)
一株嗜盐古菌DSFD111的分类学鉴定
程露露,傅金辰,杨丹丹,张含薇,俞姗杉,陈 敏
(杭州师范大学生命与环境科学学院,浙江 杭州 310036)
采用形态学和系统发育分析对一株嗜盐古菌DSFD111进行了分类学鉴定.分离到的DSFD111菌株为革兰氏阴性球菌,不产芽胞,细胞内含类脂粒.最适生长温度为37 ℃, 最适pH为7, 最适生长NaCl浓度为4.3 mol/L,表明该菌株属于极端嗜盐菌.根据16S rRNA基因序列的系统发育分析,DSFD111菌株归于盐盒菌属(Haloarcula),与最近缘的Haloarculaargentinensis的相似度为99%.
嗜盐古菌;菌种鉴定;16S rRNA
嗜盐菌(Halophiles)是指生长需要盐且在一定盐浓度的环境中生长最佳的微生物[1].长期的选择压力使嗜盐菌形成了对环境胁迫的适应机制,能够产生多种嗜(耐)盐功能酶及其他特殊性质的活性物质[2],成为一类新型的、极具应用前景和开发潜力的极端微生物资源.
我国的云南、新疆和内蒙古等地区存在着数量众多的盐湖或盐碱湖,对这类自然高盐环境中的嗜盐微生物已有较多的研究[3-6].而我国东部沿海地区拥有众多的盐田、盐场,是典型的人工高盐环境,对此类环境中的包括中度嗜盐菌在内的微生物的研究相对还较少[7-9].岱山是浙江省最大的产盐县,岛上有数万亩盐田,为研究盐田环境嗜盐菌资源提供了极好的条件.2010年,本实验室从岱山盐场采集土壤及卤水样品,利用选择性培养基共分离得到181株嗜盐菌菌株,其中DSFD111菌株经检测具有较高的淀粉酶和脂肪酶活性(结果已另文报道),本研究主要对DSFD111菌株进行分类学鉴定和系统发育分析,为进一步的开发应用提供理论基础.
1 材料和方法
1.1 菌株
DSFD111菌株:分离自岱山盐场卤水样品,本实验室保存.
1.2 培养基
嗜盐菌RM培养基:MgSO4·7H2O,20.0 g·L-1;柠檬酸钠,3.0 g·L-1;KCl,2.0 g·L-1;无水CaCl2,0.2 g·L-1;细菌蛋白胨(L 37),10.0 g·L-1;NaCl,250 g·L-1;琼脂,15.0 g·L-1;pH 7.0.
1.3 形态学鉴定
1.3.1 菌落形态:取试管斜面保藏的菌种在25% NaCl的RM固体平板培养基上划线,保鲜膜包覆,倒置在37 ℃光照培养箱中培养1周,观察平板上单菌落的颜色、大小、形状、边缘、表面突起及透明程度等.
1.3.2 菌体形态[10]:载玻片上滴1滴已灭菌的25% NaCl溶液,从平板上挑取新鲜培养的菌种,涂片风干后,以2% 的冰乙酸覆盖5 min固定脱盐,洗去多余的乙酸并晾干;用结晶紫覆盖1~2 min进行简单染色,蒸馏水洗去染液,晾干,普通光学显微镜观察细胞的形状,并测定其大小.
1.3.3 革兰氏染色:涂片方法同上.革兰氏染色用结晶紫初染90 s,水洗,滴加卢戈氏(Lugol’s)碘液冲去残水,覆盖1 min,水洗,95%的乙醇滴洗,直至流出的液体不呈紫色,约20~30 s,立即水洗,0.5%番红水溶液复染1~2 min,水洗,晾干后镜检.
1.3.4 芽孢染色:采用改良Schaeffer-Fulton染色法,取1~2滴无菌盐水于小试管中,用接种环挑取培养2周的菌体于该试管中,充分打散混匀,制成浓稠的菌悬液.加2~3滴5% 的孔雀绿染液于小试管中,轻轻摇晃,使染液与菌液充分混合.将小试管用沸水浴加热15~20 min.用接种环从试管底部挑取几环菌液于洁净的载玻片上,制成涂片,通过酒精灯火焰2~3次固定,晾干.水洗至流出的水无绿色止.加0.5% 番红水溶液复染 5 min,倾去染液,用吸水纸吸干.油镜下观察.
1.3.5 类脂粒(PHB)颗粒检测[11]:按1.3.2的方法制备涂片.苏丹黑染液染10 min,用水冲去染剂,再用二甲苯冲洗涂片至无色素洗脱.用0.5%番红复染1~2 min,水洗,干燥,镜检.类脂粒染成蓝黑色,菌体其他部分呈红色.
1.4 系统发育分析
1.4.1 菌体DNA的提取[12]:纯化后的单菌落接种至相应NaCl浓度的液体培养基中,37 ℃,180 r/min恒温光照摇床培养过夜.取1 mL培养好的菌悬液至1.5 mL离心管中,10 000 r/min高速离心5 min,弃去上清液,加无菌水1 mL冲洗,充分旋涡后离心去上清液,重复2次.加双蒸水100 μL,旋涡震荡器充分震荡悬浮,加保鲜膜封口,于沸水浴中煮沸10 min,取出后立即置于冰中.瞬时高速离心,直接取2 μL上清液作为PCR反应模板.
1.4.2 16S rRNA 扩增:采用古菌通用引物[13]22:5′-ATTCCGGTTGATCCTGC-3′和1540R:5 ′-AGGAGGTGATCCAGCCGCAG-3′, PCR反应条件:95 ℃ 5 min;95 ℃ 45 s,50 ℃ 45 s,72 ℃ 90 s,30个循环;72 ℃ 10 min.1.2%琼脂糖凝胶电泳检测,稳定电压90 V,电泳25 min.
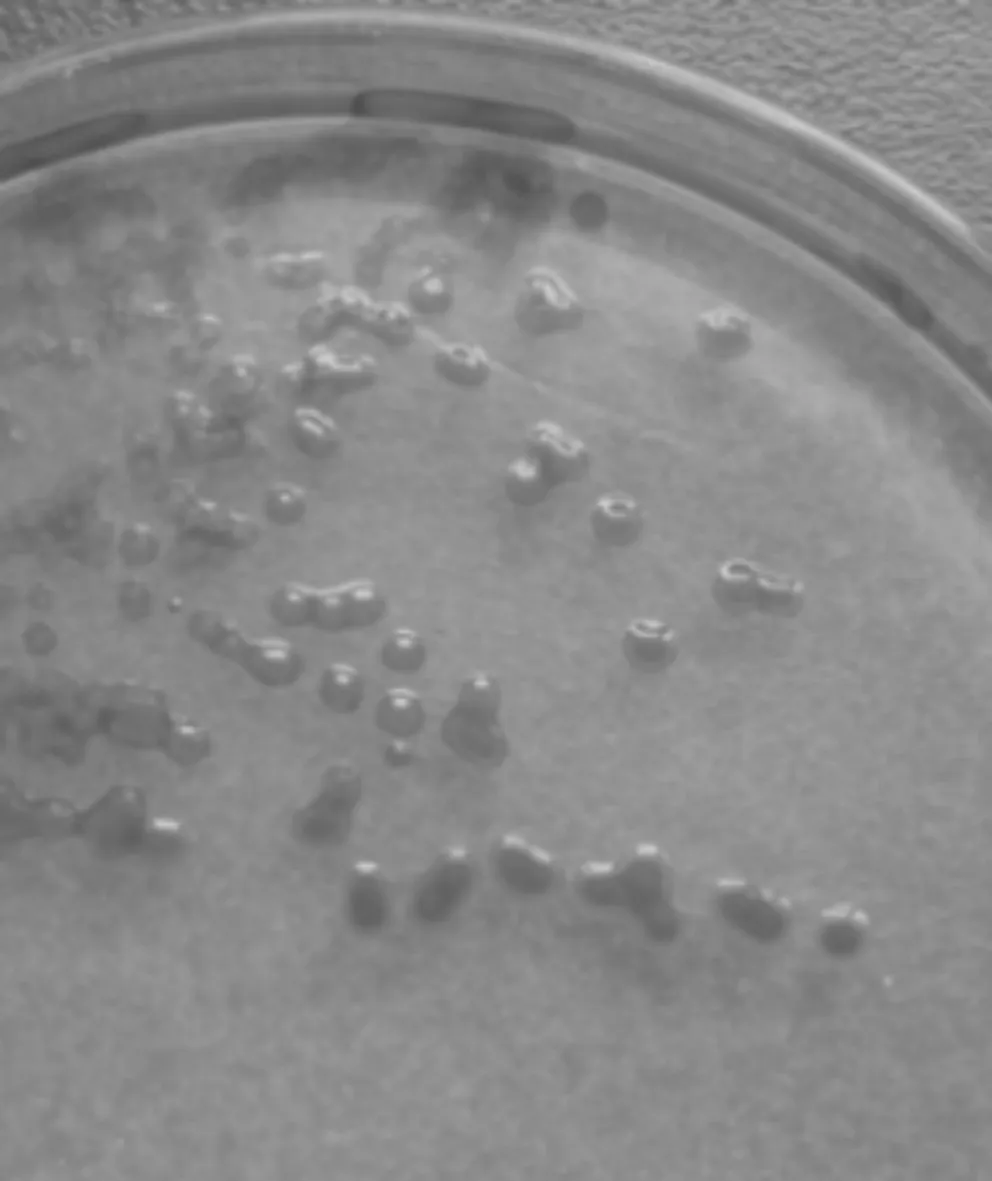
图1 菌株DSFD111菌落形态Fig. 1 The colony morphology of strain DSFD111
1.4.3 系统发育树的构建及分析:PCR产物送上海桑尼生物公司进行序列测定.测序结果提交到GenBank得到基因序列登录号,并通过EzTaxon server 2.1寻找相似性较接近的几个模式菌株的16S rRNA序列,利用MEGA5.0 软件构建系统发育进化树.
1.5 生理生化鉴定
依据文献[11,14]中相关内容进行.
2 结果与分析
2.1 形态学鉴定
将菌株DSFD111于RM平板培养基上划线培养1周后观察,菌落呈红色、圆形、直径约3 mm、表面光滑、边缘整齐、中间有突起(如图1).在光学显微镜下观察菌体形态,呈球状,直径约1.5 μm.革兰氏染色阴性,不产芽胞,细胞内含类脂粒.
2.2 系统发育分析
扩增菌株DSFD111的16S rRNA基因序列,将PCR产物进行序列测定,测序结果提交至 GenBank得到基因序列登录号为JQ068948.查找与该菌株亲缘关系较近的已发表的10个模式菌株的16S rRNA序列,采用MEGA 5.0软件包中的Kimura 2-Parameter model及Neighbor-Joining法进行聚类分析和构建系统发育进化树,结果如图2所示.
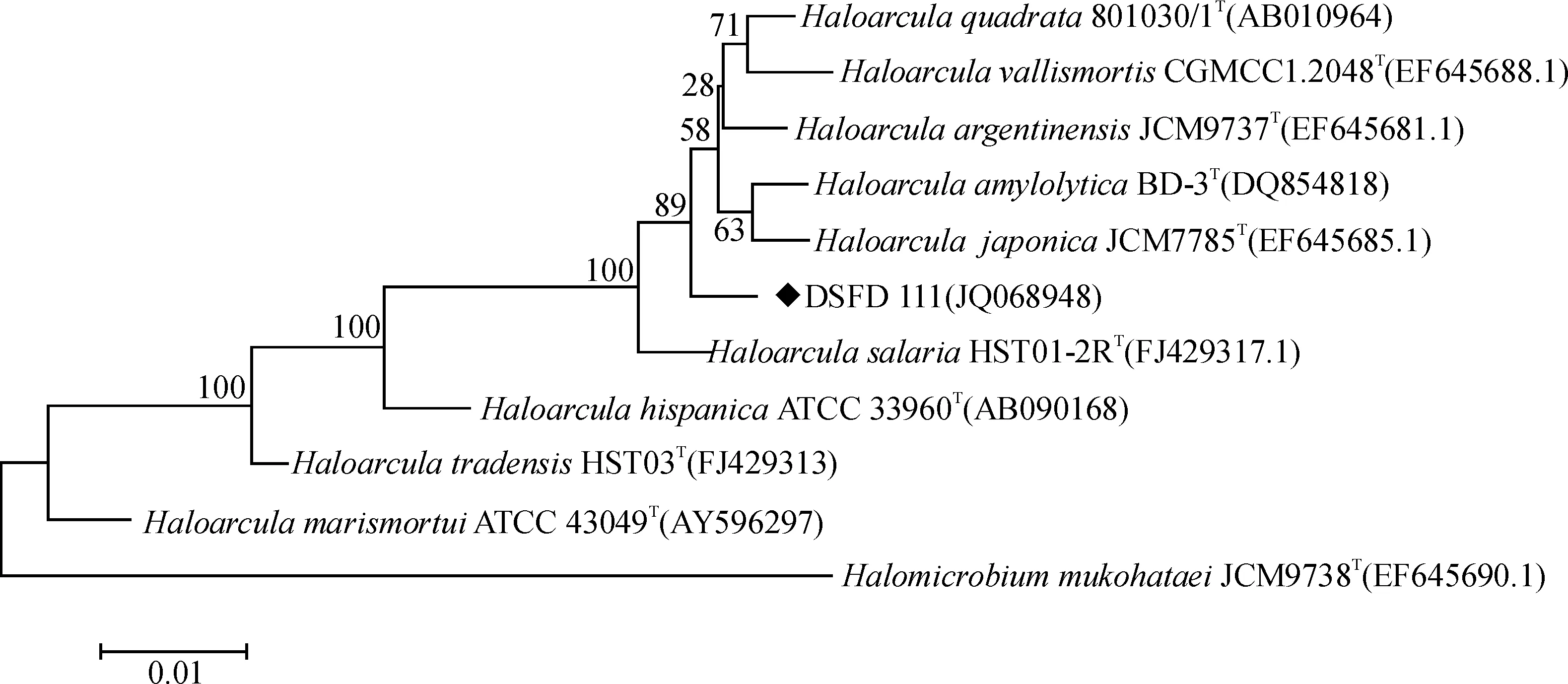
图2 菌株DSFD111 及其近缘种构建的系统发育树Fig. 2 Phylogenetic tree showing the relationship between DSFD111 and the type species in Haloarcula
结果表明,菌株DSFD111与Haloarcula属中的Haloarculaquadrata801030/1T,HaloarculavallismortisCGMCC1.2048T,HaloarculaargentinensisJCM9737T,HaloarculaamylolyticaBD-3T以及HaloarculajaponicaJCM7785T聚为一支,说明与它们都有较近的亲缘关系,其中与最近缘的HaloarculaargentinensisJCM9737T的相似度为99%.因此,分子鉴定结果DSFD111归于Haloarcula属.
2.3 生理生化特征
根据分子鉴定结果,选择模式菌株HaloarculaargentinensisJCM9737[15]为参照菌株,进行了部分的生理生化试验,结果见表1.
表1菌株DSFD111的生理生化特性
Tab.1PhysiologicalandbiochemicalcharacteristicsofstrainDSFD111
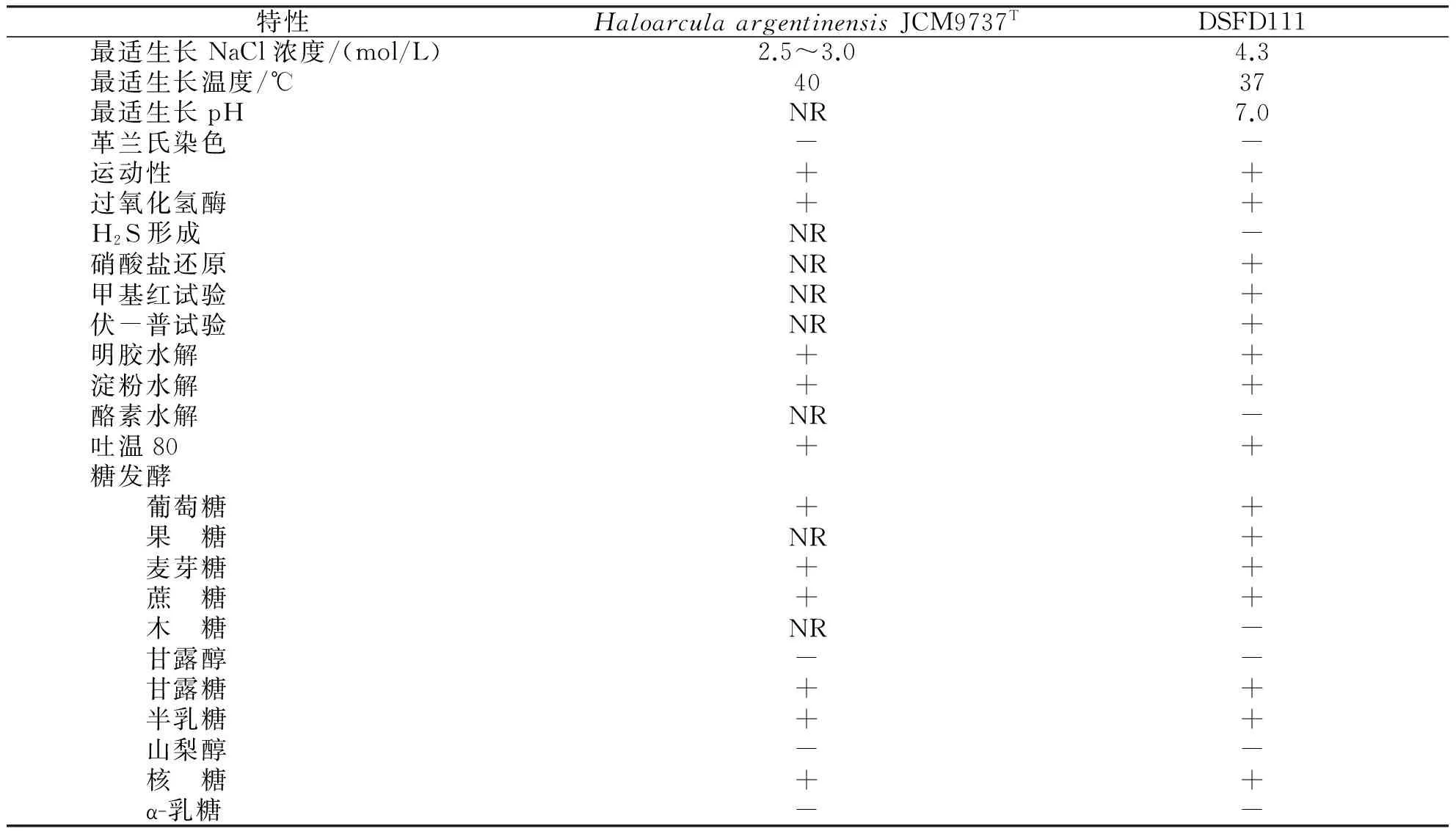
特性HaloarculaargentinensisJCM9737TDSFD111最适生长NaCl浓度/(mol/L)2.5~3.04.3最适生长温度/℃4037最适生长pHNR7.0革兰氏染色--运动性++过氧化氢酶++H2S形成NR-硝酸盐还原NR+甲基红试验NR+伏-普试验NR+明胶水解++淀粉水解++酪素水解NR-吐温80++糖发酵 葡萄糖++ 果 糖NR+ 麦芽糖++ 蔗 糖++ 木 糖NR- 甘露醇-- 甘露糖++ 半乳糖++ 山梨醇-- 核 糖++ α-乳糖--
+:阳性;-:阴性; NR:未见报道.
以上结果表明,DSFD111菌株的主要生理生化特征与Haloarculaargentinensis标准菌株基本吻合.综合上述形态、生理生化特性以及16S rRNA基因测序结果,初步鉴定DSFD111菌株为Haloarculaargentinensis.
3 讨 论
嗜盐古菌一般栖居在海水、日光晒盐池、天然盐湖或人工腌制食品的表面,在接近饱和的高盐环境中,属优势种群.由于早期的分类鉴定多侧重于形态和生理生化特性,嗜盐古生菌的分类地位一直倍受争议.直到20世纪70年代末三域学说(Three domains)[16]的提出,把微生物划分为细菌域(Bacteria)、古菌域(Archaea)、真核生物域(Eukarya)3个分类单元,并从细胞结构、遗传机制等方面与细菌域和真核生物域相比较,发现古生菌确实与真细菌和真核生物存在很大不同[17],同时在方法上建立了以16S rRNA基因为指征的序列分析技术,大大推进了古生菌菌种鉴定的相关研究.
嗜盐古菌的分类地位归属于古菌域(Archaea)、广古菌门(Euryarchaeota)、盐杆菌纲(Halobacteria)、盐杆菌目(Halobacteriales)、盐杆菌科(Halobacteriaceae),目前仅此一科.1957年,Elazari-Volcani提出了盐杆菌科的第一个属,即盐杆菌属(Halobacterium)[18].随后,Gibbons于1974年确立了盐球菌属(Halococcus),是盐杆菌科的第二个属[19].2001年,伯杰氏系统细菌学手册依据16S rRNA基因序列分析结果、极性脂的组分、细胞形态及一些其它的表型特征,将盐杆菌科划分为15个属[20].到2009年,该科已包含27个属,96个正式发表的种[21].
本研究对嗜盐古生菌DSFD111进行了16S rRNA序列同源性分析.扩增结果与GeneBank上的标准序列进行比对,结果显示,DSFD111菌株与Haloarculaargentinensis标准序列比较同源性在99%以上.进一步的生理生化鉴定结果表明,其主要特征与Haloarculaargentinensis标准菌株基本吻合.因此,初步鉴定DSFD111菌株为Haloarculaargentinensis,是一株极端嗜盐古生菌.
[1] 刘志恒.现代微生物学[M].北京:科学出版社,2008:196-201.
[2] Chen Lei, Wang Guangyu, Bu Tong,etal. Phylogenetic analysis and screening of antimicrobial and cytotoxic activities of moderately halophilic bacteria isolated from the Weihai Solar Saltern (China)[J]. World J Microb Biot, 2010, 26(5):879-888.
[3] 迪丽拜尔·托乎提,旭格拉,穆尔特扎,等.新疆罗布泊周边地区极端环境嗜盐菌的研究[J].生物技术,2009,19(5):16-19.
[4] 柴丽红,王涛,崔晓龙,等.青海柯柯盐湖16株细菌的ARDRA筛选及系统发育初步分析[J].云南大学学报:自然科学版,2003,25(6):541-544.
[5] 孟凡旭,吴敏,张会斌,等.阿牙克库木湖嗜盐菌的分离及功能酶的筛选[J].浙江大学学报:理学版,2006,33(6):671-675.
[6] 潘海莲,周成,王红蕾,等.内蒙古锡林浩特地区嗜盐古菌多样性的研究[J].微生物学报,2006,46(1):1-6.
[7] 田新玉,刘洪灿,郭金昌.青岛东风盐场中极端嗜盐古细菌的特性[J].应用与环境生物学报,1998,4(2):175-178.
[8] 周旭华,王勇,吴敏.舟山地区嗜盐菌的分离和产胞外多糖菌株的筛选[J].浙江大学学报:理学版,2007,34(3):335-339.
[9] 何敏艳,邹正中,蔡林,等.连云港台北和盐城三圩盐田土壤嗜盐菌多样性研究[J].微生物学通报,2008,35(5):737-742.
[10] Dussault H P. An improved technique for staining red halophilic bacteria[J]. J Bacteriol, 1955, 70(4):484-485.
[11] 东秀珠,蔡妙英.常见细菌系统鉴定手册[M].北京:科学出版社,2001:349-370.
[12] 陈敏,赵立平.焦化废水处理系统中不同培养基分离的细菌种群多样性[J].微生物学报,2003,43(3):366-371.
[13] 张萌,张晓梅,窦文芳,等.嗜盐脂肪酶产生菌的筛选及其粗酶性质[J].微生物学通报,2009,36(1):14-19.
[14] Holt J G, Krieg N R, Sneath P H A,etal. Bergey’s manual of determinative bacteriology[M]. 9th ed. Baltimore: Williams and Wilkins,1994:175-187.
[15] Ihara K, Watanabe S, Tamura T.Haloarculaargentinensissp. nov. andHaloarculamukohataeisp. nov., two new extremely halophilic archaea collected in Argentina[J]. Int J Syst Evol Microbiol, 1997, 47(1):73-77.
[16] Woese C R, Kandler O, Wheelis M L. Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya[J].Proc Natl Acad Sci, 1990, 87(12):4576-4579.
[17] Preseott L M, Harley J P, Klein D A. Microbiology[M]. Boston: McGraw-Hill,2002:442.
[18] Genus XII.Halobacterium[M]//Elazari-Volcani B. Bergey’s manual of determinative bacteriology. 7th ed. Baltimore: Willians and Wilkins,1957:207-212.
[19] Gibbons N E. Family V. Halobacteriaceae fam. nov[M]//Buchanan R E, Gibbons N E. Bergey’s manual of determinative bacteriology. 8th ed. Baltimore: Willians and Wilkins,1974:269-273.
[20] Boone D R, Castenholz R W, Garrity G. Bergey’s manual of systematic bacteriology [M]. New York: Springer-Verlag,2001:1-251.
[21] Oren A, David R A, Ventosa A. Emended descriptions of genera of the familyHalobacteriaceae[J]. Int J Syst Evol Microbiol,2009,59(3):637-642.
IdentificationofaHalophilicArchaeaDSFD111
CHENG Lu-lu, FU Jin-chen, YANG Dan-dan, ZHANG Han-wei, YU Shan-shan, CHEN Min
(College of Life and Environmental Sciences, Hangzhou Normal University, Hangzhou 310036, China)
A halophilic archaea DSFD111 was identified based on morphology and phylogenetic analysis. The strain is Gram-negative bacteria, no spores and contains lipid particles. The best growth environment of the strain is 37 ℃, pH 7 and 4.3 mol/L NaCl, which indicates that the strain is extreme halophiles. The phylogenetic analysis based on 16S rRNA gene homology shows that the strain belongs toHaloarcula, and shares 99% similarity withHaloarculaargentinensis.
halophilic archaea; strain identification; 16S rRNA
2012-07-03
杭州市科技发展计划项目(20101032B26).
陈 敏(1963—),女,教授,主要从事微生物研究.E-mail:mchen63@163.com
10.3969/j.issn.1674-232X.2012.06.004
Q93
A
1674-232X(2012)06-0495-05

