化脓性链球菌高毒力株特异基因文库的构建和分析*
田福亮,吴剑锋,夏永祥△
(1.南京师范大学生命科学学院 210046;2.南京医科大学附属南京第一医院 210006)
化脓性链球菌高毒力株特异基因文库的构建和分析*
田福亮1,吴剑锋2,夏永祥2△
(1.南京师范大学生命科学学院 210046;2.南京医科大学附属南京第一医院 210006)
目的构建化脓性链球菌高毒力株特异基因文库,克隆和筛选化脓性链球菌高毒力株特异表达基因。方法采用抑制消减杂交技术(SSH),以从患者体内所分离链球菌为毒力菌株,分离特异表达基因,将其与T载体进行T/A连接构建文库,将连接产物转化感受态大肠杆菌TOP10进行文库扩增后,转化菌液涂布于LB固体平板,构建化脓性链球菌毒力株特异基因消减文库,用斑点杂交初步筛选消减文库后,将获得的阳性克隆进行测序和同源性分析。结果酶切产物为100~2 000bp,连接效率大于50%,消减组与非消减组差异明显,成功构建了化脓性链球菌毒力株特异基因消减文库,所得阳性克隆经斑点杂交筛选后测序,与Genebank数据库进行同源性比对,5个未知序列可能为新基因,7个与已知基因有高度的同源性。结论用SSH及T/A克隆技术成功构建了人源化脓性链球菌高毒力株基因文库,5个未知的新序列有待于进一步的研究。
链球菌,化脓;抑制消减杂交;毒力基因;基因消减文库
A群链球菌又称化脓性链球菌[1],主要引起化脓性炎症,以上呼吸道感染为主。引起的主要疾病有扁桃体炎、咽炎、蜂窝组织炎、亚急性细菌性心内膜炎及产褥感染等。因链球菌的M蛋白与心肌、肾小球基底膜有共同的抗原,可引起风湿热和急性肾小球肾炎等超敏反应性疾病;同时,可引起猩红热、毒性休克综合征等中毒性疾病,严重威胁人类的健康[2]。Zhang等[3]研究认为一些菌株的毒性强于其他菌株可能与菌株的毒力变异有关。比较毒力株和标准菌株的基因组DNA,鉴别差异基因,有助于对感染性疾病分子发病机制进一步深入了解,并为临床诊断和治疗提供理论依据。
1 材料与方法
1.1 材料 化脓性链球菌取自南京市第一医院化脓性链球菌感染患者(征得患者同意);标准菌株采购于中国工业微生物菌种保藏管理中心(CICC),菌种编号为10373;抑制消减杂交试剂盒购自Cloth公司;细菌基因组提取试剂盒购自海门市碧云天公司;异丙基-β-D-硫代半乳糖苷(IPTG)、5-溴-4-氯-3-吲哚-β-D-半乳糖(X-GAL)购于Sigma公司;氨苄青霉素(AMP)购自Promega公司;感受态大肠杆菌TOP10购自Takara公司;PMD18T载体购自Takara公司;PCR产物纯化试剂盒购自O-mega公司。
1.2 方法
1.2.1 细菌培养 在血琼脂固体培养基上接种化脓性链球菌,放置于温度培养箱里37℃、5%CO2培养过夜。
1.2.2 细菌基因组DNA的提取 根据细菌基因组提取试剂盒操作手册,分别对毒力菌株和标准菌株提取基因组DNA,并用紫外分光光度计对所提取的基因组DNA进行浓度、纯度测定。
1.2.3 抑制消减杂交技术(SSH) 应用PCR-Select基因组消减试剂盒,并按其说明书进行。对毒力菌株和标准菌株进行基因组消减杂交,包括两个菌株的基因组DNA酶切,接头连接及其效率分析,两轮消减杂交,两轮选择性PCR扩增,并且对比消减组和非消减组,以对消减效率进行检测。
1.2.4 PCR产物的T-A克隆 应用 Advantage PCR cloning试剂盒,并按其说明书进行。取2μL第2轮PCR产物与2μL线性化的PMD18-T载体混合,在1μL T4DNA连接酶的作用下,14℃连接16h,-20℃保存。取10μL连接反应产物与100μL感受态细菌TOP10混匀于离心管中,用热激法将连接产物导入感受态大肠杆菌TOP10,向离心管中加入890 μL LB培养基,混匀后置于摇床,37℃,150r/min培养45 min,涂布200μL菌液在含有100μg/mL氨苄青霉素 X-gal/IPTG琼脂平板上,37℃培养18h。随机挑选直径大于1mm的白色克隆,点种于含100μg/mL氨苄青霉素的LB培养基的96孔板中,37℃培养过夜,加15%甘油,-70℃保存。
1.2.5 菌液斑点杂交筛选 取-70℃保存菌株,点种于含100μg/mL氨苄青霉素LB培养基的96孔板中,37℃培养过夜。取1μL菌液接种于尼龙膜 (Roche),在含100μg/mL氨苄青霉素琼脂平板上37℃培养18h,尼龙膜上的菌斑经变性、中和、80℃固定2h后备用。取纯化的正向和反向消减杂交第2轮PCR产物各80ng(3μL),用5μL[α-32p]d-ATP随机引物法标记纯化后备用。42℃预杂交1h、杂交18h后,洗膜(58℃,2×柠檬酸钠缓冲液(SSC),0.5% 十二烷基磺酸钠(SDS)、,20min 2次,58 ℃,0.2×SSC,0.5%SDS,20min 2次),-70℃放射自显影12h。
1.2.6 阳性克隆的测序与同源性分析 取斑点杂交初步筛选的阳性克隆,用nested primer1和nested primer 2R为引物,进行PCR扩增,2%琼脂糖凝胶电泳分析确认有插入片段后,以M13为正向引物,PE ABI PRISM 3700测序仪测序 (上海英骏生物技术有限公司代理)。测序结果用Primer5.0分析后,在 Genebank(http://www.ncbi.nlm.gov/BLAST)中进行序列同源性检索分析。
2 结 果
2.1 细菌基因组DNA的提取 经过紫外分光光度计测定,浓度分别为120、160ng/μL,光密度值(OD)260/280分别为1.73,1.81,取5μL基因组DNA 2%琼脂糖凝胶电泳。

图1 细菌基因组DNA的提取
2.2 消减效率的分析 将消减和非消减的18、23、28、33个循环产物2%琼脂糖凝胶电泳分析,结果如图2,未消减过的产物在18cycles时就已出现清晰条带,消减过的在28个循环时才出现条带,表明消减效果理想。
2.3 消减杂交文库 200μL转化菌液涂布的平板上约有300多个白色菌落生长,菌落清晰饱满,白色菌落占总菌落的60%以上,说明消减文库构建成功,随机挑取100个白色菌落建成化脓性链球菌毒力株基因消减文库。
2.4 消减杂交文库的构建筛选结果 100μg/mL氨苄青霉素/X-gal/IPTG琼脂平板上,37℃培养18h后,平板可见254个白色及蓝色菌落生长,以白色菌落为主(约占80%),菌落饱满清晰,直径1mm左右。随机挑选175个白色菌落,保存。斑点杂交信号对比,获得 A1、A2、A6、B5、C5、C7、D3、E4、E8、F5、G5、H2共12个差异基因片段,且这12个基因片段均表现为毒力株DNA片段标记的探针信号增强,结果如图3。
2.5 DNA测序与同源性分析结果 对12个含有差异基因片段的阳性克隆进行M13双向引物测序,与Genebank数据库初步比对,结果显示12个差异片段中有5个未检索到同源序列,其余7个序列均与已知基因有高度的同源性,结果如表1。
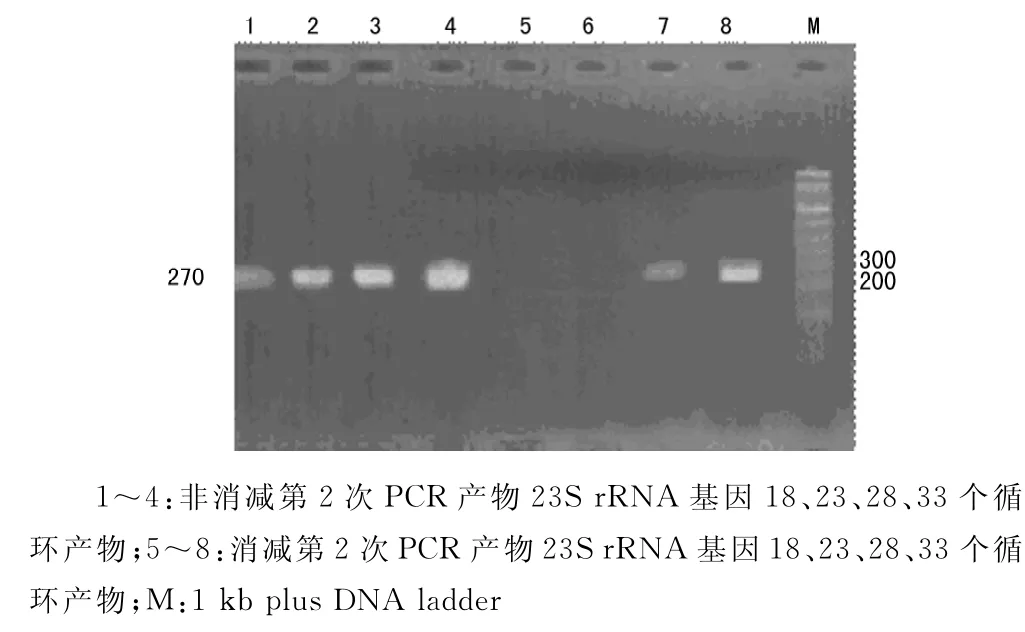
图2 消减效率分析

图3 斑点杂交筛选结果
3 讨 论
研究微生物致病机制的首要前提是识别和分离其毒力基因[4]。Diatehenko等[5]于1996年提出SSH,该技术最初用于肿瘤细胞与正常细胞基因组或cDNA差异的研究,后来被推广到微生物领域。SSH已经应用于许多致病菌毒力基因的发现,SSH被用于嗜水气单胞菌[3]、尿毒性大肠杆菌菌株536[6]、猪链球菌2型强毒株[7]、副猪嗜血杆菌[8]、海德尔堡沙门菌[9]、肺炎链球菌[10]、鸡瘟沙门菌[11]毒力基因的发现,都得到了理想的效果。SSH 目前还被应用于鉴定细菌[12]、鉴定毒力岛[13]、发现插入序列及构建检测探针[14]等多方面,表明该技术是一个简单且有效的鉴定细菌间遗传差异的方法。
本研究首次将SSH技术应用于化脓性链球菌毒力株特异基因的研究,以标准菌株作为对比材料,严格检测基因组DNA提取质量,基因组DNA酶切,接头连接,两轮消减杂交及两次抑制PCR等反应步骤,使Tester与Driver中共有的基因片段得到消减,存在于Tester中的特异基因得到特异性扩增。通过T/A克隆技术,将两轮扩增PCR产物纯化后直接与T载体连接,成功构建化脓性链球菌毒力株特异基因文库,并且通过M13双向引物测序,与Genebank数据库BLAST比对得到了12个差异基因片段,7个序列均与已知基因有高度的同源性,包括了链球菌激酶、链球菌补体抑制剂、C5a肽酶、致热外毒素C、DNA促螺旋酶等,有5个未检索到同源序列,这些未知的序列有待于进一步的研究,期望对治疗化脓性链球菌感染有所帮助。
[1]Lancefield RC.A serological differentiation of human and other groups of hemolytic streptococci[J].J Exp Med,1933,57(4):571-595.
[2]Holm SE.Invasive group A streptococcal infections[J].N Engl J Med,1996,335(8):590-591.
[3]Zhang YL,Ong CT,Leung KY.Molecular analysis of genetie differences between virulent and avirulent strains of Aeromonas hydroPhila isolated from diseased fish[J].Microbiology,2000,146(Pt 4):999-1009.
[4]Strauss EJ,Falkow S.Microbial pathogenesis:genomic and beyond[J].Science,1997,276(5313):707-712.
[5]Diatchenko L,Lau YF,Campbell AP,et al.Suppression subtractive hybridization:a method for generating differentially regulated or tissue-specific cDNA probes and libraries[J].Proc Natl Acad Sci USA,1996,93(12):6025-6030.
[6]Janke B,Dobrindt U,Hacker J,et al.A suppressive subtractive hybridlzation analysis of genomic differences between the uropathogenic E.coli strain 536and the E.coil k-12strain MG1655[J].FEMS Microbial Letters,2001,199(1):61-66
[7]田云,Frank M Aarestrup,陆承平.猪链球菌2型的可能毒力基因的发现[J].微生物学报,2004,44(5):613-616.
[8]Zhou H,Yang B,Xu F,et al.Identification of putative virulence-associated genes of Haemophilus parasuis through suppression subtractive hybridization[J].Vet Microbiol,2010,144(3/4):377-383.
[9]Bronowski C,Winstanley C.Identification and distribution of accessory genome DNA sequences from an invasive African isolate of Salmonella Heidelberg[J].FEMS Microbiol Lett,2009,298(1):29-36.
[10]Suzuki N,Seki M,Nakano Y,et al.Discrimination of Streptococcus pneumoniae from viridians group Streptococci by genomic subtractive hybridization[J].J Clin Microbiol,2005,43(9):4528-4234.
[11]Qiuchun L,Xu Y,Jiao X.Identification of Salmonella pullorum genomic sequences using suppression subtractive hybridization[J].J Microbiol Biotechnol,2009,19(9):898-903.
[12]Radnedge L,Gamez-Chin S,McCready PM,et al.Identification of nucleotide sequences for zhe specific and rapid detection of Yersinia pestis[J].Appl Environ Microbiol,2001,67(8):3759-3762.
[13]Walker JC,Verma NK.Identification of a putatlve pathogenicity island in shigella flexneri using subtractive hybridization of the S.flexneri and Escherichia coli genomes[J].FEMS Microbiol Lett,2002,213(2):257-264.
[14]Parsons YN,Panagea S,Smart CH,et al.Use of subtractive hybridization to identify a diagnostic probe for a cystic fibrosis epidemic strain of Pseudomonas aeruginosa[J].J Clin Microbiol,2002,40(12):4607-4611.
Construction and analysis of virulence genes subtracted library of streptococcus pyogenes by suppression subtractive hybridization*
TianFuliang1,WuJianfeng2,XiangYongxiang2△
(1.DepartmentofLifeSciences,NanjingNormalUniversity,Nanjing,Jiangsu210046,China;2.AffiliatedNanjingFristHospital,NanjingMedicalUniversity,Nanjing,Jiangsu210006,China)
ObjectiveTo construct the virulence genes subtracted library of streptococcus pyogenes and to lay the foundations for screening the virulent genes.MethodsSuppression subtractive hybridization was adopted to isolate the fragments of virulence genes in streptococcus pyogenes.Then these fragments were directly inserted into T/A cloning vector to set up subtractive library and amplication of the library was carried out with transformation of E.coil TOP10.Dot blot was used to screen the subtracted library,the differentially expressed cDNA fragments were sequenced and analyzed in Genebank with Blast search.ResultsA smear ranged from 100~2 000bp was observed.The ligation efficiency of tester DNA with adaptor was at least higher than 50percent.The difference between subtractive group and unsbtractive group was apparently significant.Partial positive clones in the library were randomly selected and successfully sequenced.5/12sequence showed no homology and presumably represent unknown genes,7/12had a high similarity to the known genes.ConclusionThe virulence genes subtracted library of streptococcus pyogenes is constructed successfully with SSH and T/A cloning techniques.The library is efficient and lays solid foundation for screening and cloning new and specific virulence genes of streptococcus pyogenes.
streptococcus pyogenes;suppression subtractive hybridization;virulence genes;subtracted library
10.3969/j.issn.1671-8348.2012.06.004
A
1671-8348(2012)06-0529-03
南京市卫生局医学科技发展基金资助项目 (YKK07047)。△ 通讯作者,E-mail:18951670130@189.cn。
2011-06-11
2011-11-07)

