肺炎克雷伯菌的多药外排泵KetM的解析
高桂凤,李英民,孙佳岩,张晓燕
(1.吉林农业科技学院生物工程学院,吉林吉林 132101;2.吉林省通榆县扶贫开发办公室,吉林通榆 137200;3.吉林省通榆县职教集团,吉林通榆 137200)
肺炎克雷伯菌属于肠道细菌科的革兰氏阴性杆菌,人一旦被该菌感染易患大叶性肺炎,肺脓肿等病症,且容易重度化[1]。
近年来,由于临床上抗生素的大量使用,尤其是不合理使用,使肺炎克雷伯菌对抗菌药获得了耐药性。该菌主要耐药机理报道比较多的是含有超广谱 β 内酰胺酶(ESBL)的 β - 内酰胺酶[2-3]。但近年来,不仅分离出对一种药物产生耐药性的菌株,还分离出对喹喏啉药、氯霉素、四环素、替加环素等结构和作用机理不同的药物产生耐药性的菌株[4-5]。其中,对人类威胁最大的是对多种药物产生耐药性的菌株,由这种细菌引起的耐药性被认为与抗菌药向细胞外能动地排出机制密切相关。据报道对多种药物产生耐药性起关键作用的是多药外排泵[6]。
一般地,多药外排泵使细菌细胞对各种各样的抗菌物质、胆汁酸、重金属离子产生耐药性[7-8]。根据其结构和能量作用的不同,多药外排泵被分为MFS(主要易化超)家族、RND(耐药结节分化)家族、SMR(ATP结合盒)家族、ABC(细小多药耐药)家族、MATE(多药和有毒化合物的排出)家族五个家族。目前,在肺炎克雷伯菌中已报道的多药外排泵仅有属于RND家族 的AcrAB[9]、属于SMR家族的 QacE[10]、属于 MFS 家族的 KmrA[11]。
肺炎克雷伯菌的临床分离株MGH78578是对多种抗菌药产生耐药性的菌株[12]。为了阐明其耐药基因的遗传学和生物化学特性,首先从该菌的染色体组上,克隆了对抗菌药产生耐药性的基因,并制成多个重组质粒[12]。然后从中选择一个重组质粒,并对其上载有的耐药性基因进行解析。
1 材料与方法
1.1 菌株与菌株制备 实验菌株肺炎克雷伯菌MGH78578株和缺失大肠杆菌KAM32、载体pBluescript II来自日本冈山大学药学部分子微生物教研室。首先,从肺炎克雷伯菌MGH78578菌株提取染色体DNA,用限制性内切酶Sau3AI酶切,分离含有4~10 kb的DNA酶切片段;同时,载体pBluescript II用限制性内切酶Bam H I酶切,并且使酶切面脱磷酸化,然后在连接酶的作用下将DNA片段插入到载体上得到重组质粒pDSH8,这个质粒含有一个插入基因命名为ketM。以缺失大肠杆菌KAM32(抗菌药高度敏感性菌株;ΔacrB、ΔydhE、hsd)作为宿主,通过转化方式导入宿主中得到 E.coli KAM32/pDSH8。
1.2 菌株和菌株培养条件 菌株E.coli KAM32/pDSH8用LB液体培养基(1%蛋白胨、0.5%酵母粉、0.5%NaCl,pH 7.0)在温度37 ℃好气条件下振动培养。另外,还使用在液体培养基里加2%琼脂的固体培养基进行培养,同时,在液体培养基和固体培养基里添加终浓度为100μg/mL氨苄青霉素。
1.3 MIC(最小抑菌浓度)的测定 微型培养皿使用96孔U字型微型测试皿。首先,菌种在水解酪蛋白培养基里培养到OD650值为0.7~0.8左右,细菌浓度为4.8 ×109~1.03×1010左右,因为菌种处于对数期时,活力最强。然后,用无菌生理盐水(NaCl 0.85%)进行1000倍稀释作为接种用的菌液。再用这种稀释菌夜5μL接种到100μL含各种浓度抗菌药的MH培养基,在37℃静止培养20 h后,用肉眼确认菌不能生长发育的最低浓度。
1.4 DAPI(4',6 - 二脒基 -2 - 苯基吲哚)排出活性的测定 DAPI一但插入到DNA上,荧光强度就增大,利用这个原理测定细胞内DAPI蓄积量。在含有终浓度为100μL/mL氨苄青霉素的LB培养基使菌液培养到对数期的中期(OD650=0.7),收集 E.coli KAM32/pDSH8 菌体(8000 r/min,5 min,4℃),用tanaka最小盐培养基(34 mmol/L KH2PO4、64 mmol/L K2HPO4、20 mol/L(NH4)2SO4、0.3 mmol/L MgSO4、1 mmol/L FeSO4、1 mmol/L ZnCl2、10 mmol/L CaCl2(pH 7.0))[13]洗 净。用 含 5 μmol/L DAPI 和5 mmol/L DNP的相同培养基悬浮到 OD650值约0.1,并在 37 ℃ 振动10 h,然后集菌(8000 r/min,15 min,4 ℃),用含有0.3 mmol/LMgSO4的吗啉代丙烷磺酸(MOPS)洗净,再用MOPS悬浮,使OD650达到0.4左右。用荧光分光光度计在激发波长332 nm,发射波长463 nm测定荧光强度。
1.5 RT-PCR 从培养、收集 E.coli KAM32/pDSH8的细胞里提取RNA,RNA用QIAGENRNeasyMini kit(QIAGEN Inc)精制。经过DNase处理的RNA最后收集到离心管1 ng,用 QIAGEN One-Step RT-PCR Kit(QIAGEN Inc)进行 RT-PCR。按添加的程序准备原料,并设计了Ket、uncB特异性的引物。引物的碱基顺序:Ket的上游引物是5-CTGGCTATACCCGTCATCCTTG-3,下游引物是5-ATGTATCCGGCGTTCCACA-3;uncB的上游引物是5-ATTGCTCTGCTGCGCC-3,下游引物是5-GTCCTCAGGTCAGTGGCATTA-3。反应结束后,进行3%琼脂糖凝胶电泳,然后切胶回收目的DNA片段。
1.6 同源性(进化过程中源于同一祖先的分支之间的关系)检索 为了调查KetM和已知蛋白质序列的类似性,选用了以下几种已知蛋白质,如大肠杆菌的YdhE、副溶血性弧菌的NorM、霍乱弧菌的VcmA、流感嗜血杆菌的 HmrM、鲍曼不动杆菌的AbeM、霍乱弧菌的 VcmD、VcmB、VcmM,用 BLAST进行同源性检索。
1.7 MATE型多药外排泵的系统树的制作 为了进一步调查KetM与其它已知蛋白质的亲缘关系,选用了以下几种蛋白,有大肠杆菌的YdhE、副溶血性弧菌的NorM、霍乱弧菌的VcmA、流感嗜血杆菌的HmrM、鲍曼不动杆菌的 AbeM、霍乱弧菌的VcmD、VcmB、VcrM、肺炎链球菌的 DinF等通过用Stanford university的计算机系统的ClustalW,Decypher制作了KetM的系统树。
2 结果
2.1 同源性检索结果 肺炎克雷伯菌的KetM与MATE型多药外排泵大肠杆菌的YdhE和副溶血性弧菌的NorM有很高的蛋白质序列的相似性,KetM与MATE型多药外排泵大肠杆菌的YdhE表示有56%的蛋白质分子结构的同一性和96%的蛋白质序列的相似性(表1);与副溶血性弧菌的NorM表示有56%的同一性和96%的相似性。与霍乱弧菌的VcmA表示有54%的同一性和86%的相似性。从这些结果可以得知,肺炎克雷伯菌KetM属于MATE型多种药物外排泵。
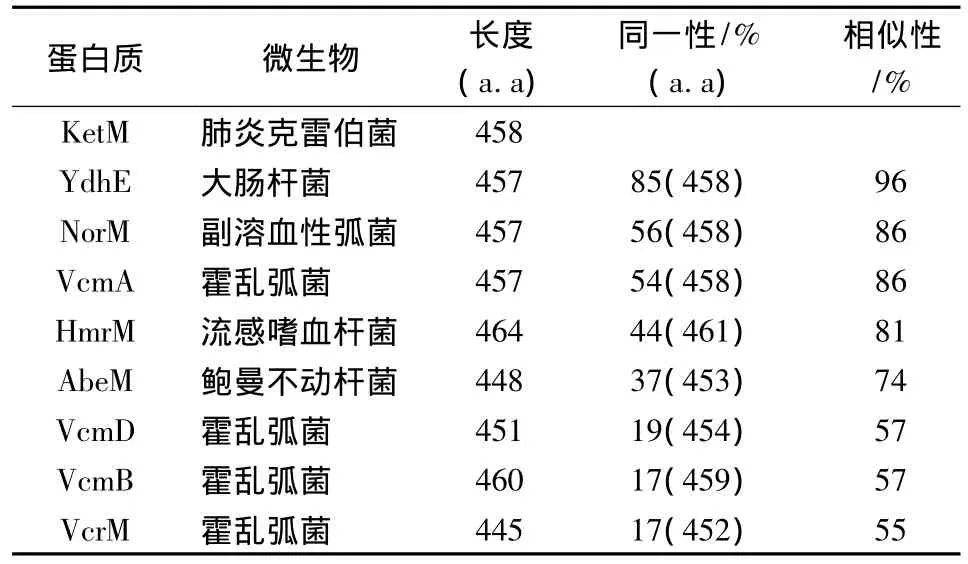
表1 与KetM有相似性的多种药物外排泵
2.2 MATE型多种药物外排泵的系统树的解析据报道MATE家族成员分为3个集团[13]。根据系统树认为KetM与副溶血性弧菌的NorM和大肠杆菌的YdhE属同一集团(图1)

图1 MATE型多种药物外排泵的系统树
2.3 大肠杆菌KAM32/pDSH8对抗菌药敏感性的变化 通过测定MIC调查KetM对耐菌性的影响,结果见表2。大肠杆菌KAM32/pDSH8与对照组大肠杆菌KAM32/pBluescriptII比较,诺氟沙星的MIC升高8倍,吖啶黄、赫希斯特33342、头孢噻肟的MIC上升了2倍,DAPI的MIC上升了64倍。由此可见,含有基因ketM的大肠杆菌 KAM32/pDSH8对多数抗菌药产生耐药性。
2.4 DAPI排出活性的测定 KetM被认为属于MATE家族的外排泵。据报道这个家族的外排泵是利用位于细胞膜的 Na+作为运输基质动力[14-17],如副溶血性弧菌的 NorM[17]和 VmrA[15],大肠杆菌的 YdhE[18]以及霍乱弧菌非醇的 VcmA[14]和VcrM[16]等。另一方面,还有试验数据表明鲍曼不动杆菌的AbeM是利用H+作为运输基质动力。在这里,为了调查KetM是否利用Na+作为排出抗菌药的动力,测定了DAPI排出活性(图2)。结果显示,与对照组比较含有 ketM的大肠杆菌KAM32/pDSH8菌株利用阳离子排出DAPI的能力明显增强。添加能量源乳酸-TMAH(呼吸链的基质)DAPI向细胞外排出,而添加 KCl、NaCl、LiCl的过程,更加促进了DAPI的排出活性。特别是通过添加KCl显著地引起了DAPI排出(图3)。添加KCl、NaCl、LiCl,但未添加乳酸 - TMAH,即使只添加NaCl、LiCl也可增强DAPI的排出活性(图4)。但是只添加KCl时,未见DAPI的排出。因此,认为在乳酸-TMAH和KCl都存在时,KetM拥有最大的排出活性。从以上结果显示,KetM拥有依靠能量排出DAPI的活性。实际上,从以上试验确认了KetM是外排泵。
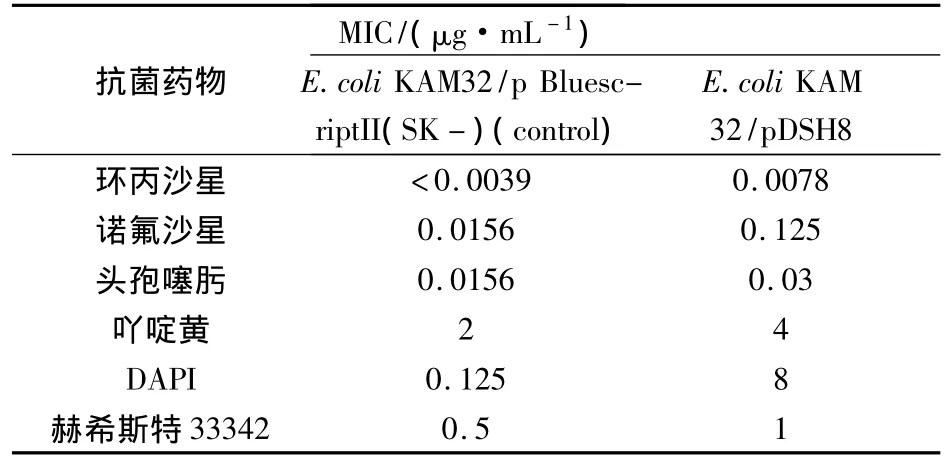
表2 大肠杆菌KAM32/pDSH8的各种抗菌药的最小抑菌浓度(MIC)
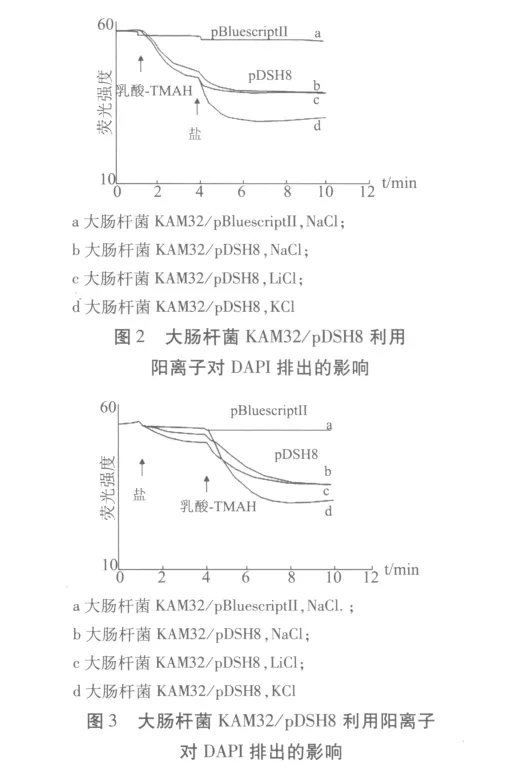
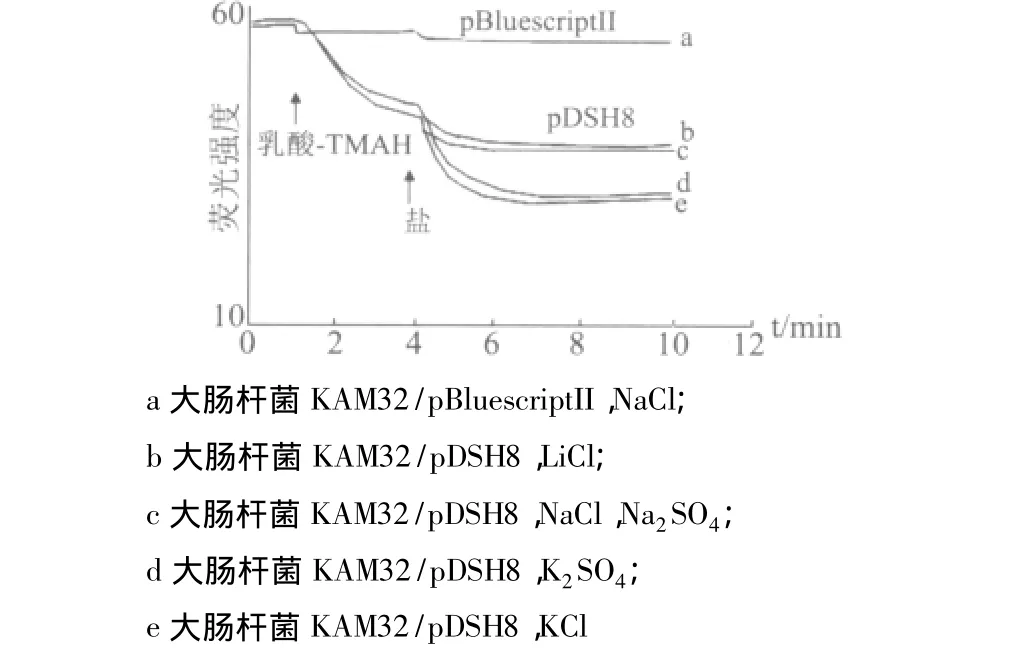
图4 其他盐离子对DAPI排出的影响
2.5 RT-PCR 从上述的调查了解到KetM是属于MATE家族的多种药物外排泵,并且耐药性也特别强,因此,它的基因表达水平也应该很好。为了调查ketM在肺炎克雷伯菌MGH78578内表达状况,用uncB作为内参基因,用 RT-PCR调查了ketM的mRNA的表达水平(图5)。结果ketM与内参基因uncB一样表达良好。

图5 ketM在肺炎克雷伯菌的表达
3 讨论
本实验从遗传学及生物化学上对多种药物外排基因ketM进行了研究。通过同源性检索和系统树分析发现KetM是属于MATE型的泵。通过测定MIC和抗菌药排出活性发现KetM是多药外排泵。用RT-PCR法证明了基因ketM不但耐药性强而且基因的表达水平也很强。
从以上结论得知,大肠杆菌KAM32/pDSH8与对照组大肠杆菌KAM32/pBluescriptII相比耐药性更强。因此,这两种菌株在生物制药研究上可作为供试菌株。MIC测试结果则提供了很好的抑菌参数,所以,本研究为生物制药提供了前期的准备工作,具有重要参考价值。
[1] 日本臨床社.日本临床(上)[M].大阪:日本臨床社出版,2002:802.
[2] Bush K,Jacoby G A,Medeiros A A.A functional classification scheme for beta-lactamases and its correlation with molecular structure[J].Antimicrob Agents Chemother,1995,39:1211 -1233.
[3] Kang C I,Kim SH,Park W B,etal.Bloodstream infections due to extended-spectrumβ-lactamase-producing Escherichia coli and Klebsiella pneumoniae:Risk factors for mortality and treatment outcome,with special emphasis on antimicrobial therapy antimicrob[J].Agents Chemother,2004,48:4574 - 4581.
[4] Hasdemir U O,Chevalier J,Nordmann P,et al.Detection and prevalence of active drug effluxmechanism in variousmultidrug–resistant Klebsiella pneumoniae strains from Turkey[J].JClin Microbiol,2004,42:2701 -2706.
[5] Alexey R,Melissa A,Visalli,David K,et al.Influence of transcriptional activator ramA on expression ofmultidrug efflux pump AcrAB and tigecycline susceptibility in Klebsiella pneumoniae[J].Antimicrob Agents Chemother,2005,49:1017 -1022.
[6] Li X Z,Nikaido H.Efflux-mediated drug resistance in bacteria Drugs[J].JClin Microbiol,2004,64:159 -204.
[7] Corinne R L,Steven A D,Michael K,et al.The NorM efflux of Neisseria gonorrhoeae and Neisseria meningitides recgnizes antimicrobial cationic compounds[J].Journal of Bacteriology,2003,185:1101-1106.
[8] Rogers E E,Guerinot M L.FRD3,a member of the multidrug and toxin efflux family,controls iron deficiency responses in arabidopsis[J].Plant Cell,2002,14:1787 -1799.
[9] Mazzariol A,Zuliani J,Cornaglia G,etal.AcrAB efflux system:expression and contribution to fluoroquinolone resistance in Klebsiella spp[J].Antimicrob Agents Chemother,2002,46:3984 -3986.
[10] Paulsen IT,Littlejohn TG,Rdstrm P,et al.The 3’conserved segment of integrons contains a gene associated withmultidrug resistance to antiseptics and disinfectants[J].Antimicrob Agents Chemother,1993,37:761 -768.
[11] Ogawa W,Koterasawa M,Kuroda T,et al.KmrA multidrug efflux pump from Klebsiella pneumoniae[J].Biol Pharm Bull,2006,29:550-553.
[12] Ogawa W,Li D W,Begum A,et al.Multidrug resistance in Klebsiella pneumoniae MGH78578 and cloning of genes responsible for the resistance[J].Biol Pharm Bull,2005,28:1505 -1508.
[13] Brown M H,Paulsen I T,Skurray R A.The multidrug efflux protein NorM is a prototype of a new family of transporters[J].Mol.Microbiol,1999,31:394 - 395.
[14]土屋有房.シンプル微生物学改定第3版[M].日本:南江堂出版社158-159.
[15] ChenJ,MoritaY,NazmulHudaM,etal.VmrA,amemberofa novelclassofNa+-coupledmultidrugeffluxpumpsfromVibrio parahaemolyticus[J].JBacteriol,2002,184:572 -576.
[16] NazmulHudaM,ChenJ,MoritaY,etal.Genecloningand characterizationofVcrM,aNa+-coupledmultidrugefflux pump,fromVibriocholeraenon - O1[J].MicrobiolImmunol,2003,47:419 -427.
[17] MoritaY,KataokaA,ShiotaS,etal.NorMofVibrioparahaemolyticusisanNa+- drivenmultidrugeffluxpump[J]JBacteriol,2000,182:6694 -6697.
[18] MoritaY,KodamaK,ShiotaS,etal.NorM,aputativemultidrug effluxproteinofVibrioparahaemolyticusanditshomologinEscherichiacoli[J].AntimicrobAgentsChemother,1998,42:1.

