基因靶向筛选药物寡聚酰胺的研究进展
柯 锋
(浙江工业大学药学院,浙江 杭州 310014)
随着人类基因组计划的完成,相关基因与具体疾病之间的相互关系成为后基因组时代的研究热点,如遗传病基因治疗、癌症基因治疗及以艾滋病为代表的重要感染性疾病的治疗。因此,以DNA为药物作用靶标的研发也越来越受到世界各大制药厂商和医药专家的极大关注。20世纪80年代末,美国Dervan研究组受到偏端霉素A结构特点的启发,将研究重点转向以寡聚酰胺(Polyamide)为模板的DNA识别分子调控基因表达的研究。日本京都大学的Sugiyama研究组同Fukuda研究组合作,深入开展了对这类物质的合成和生物活性测试方面的研究。
寡聚酰胺是继核酸反义技术和RNA干扰技术之后,小沟结合物(Minor groove binder,MGB)研究的又一热点,也是迄今为止最具潜力开发成以基因为靶标的治疗药物的合成小分子化合物之一,因此其相关研究具有重要的现实意义。作者在此综述了该类化合物的化学与生物学研究进展。
1 由偏端霉素A到寡聚酰胺对DNA分子的识别作用
自然界里抗生素偏端霉素A是一类天然的DNA识别小分子物质,具有抗病毒和抗肿瘤活性。偏端霉素A的分子结构特点是由芳香环通过酰胺键连接而成的平面共轭体系,其核心结构是寡聚含吡咯的酰胺,这种结构对DNA的 AT富集区有高度选择性识别作用,尤其与5′-AAATT-3′序列的结合性非常好。偏端霉素A能够对称地结合于DNA小沟的中间,分子中的每一个酰胺键均与相邻碱基腺嘌呤的N(3)和另一条链上胞嘧啶的O(2)形成桥型的氢键,并通过范德华力和静电相互作用与DNA结合,形成三明治状结构[1]。 受此启发,美国Dervan研究组设计合成的吡咯-咪唑寡聚酰胺分子能在DNA双螺旋小沟处按1∶1比例识别特定碱基(图1)[2~5];这类化合物一般由2~3种不同的芳香杂环通过酰胺键连接而成的平面共轭体系组成, 普遍用到的是N-甲基吡咯(Py) 和N-甲基咪唑(Im),分别类似于天然抗生素 Netropsin 和Distamycin。为了让寡聚酰胺分子能与G·C碱基对有效结合而引入了咪唑基团,合成的ImPyPyDp分子(Dp=N,N-二甲基丙胺)以2∶l的比例反向平行与DNA小沟处的5′-TGTCA-3′序列结合[6]。接着Dervan等[7]将γ-氨基丁酸(γ)引入寡聚酰胺分子中,设计并合成了一种新型的头尾相连的发夹状(Hairpin)寡聚酰胺,即通过γ-氨基丁酸将两条反向平行寡聚酰胺链的C端与N端连结起来,在分子内引导形成转折。人工合成的寡聚酰胺分子中通常还含有β-氨基丙酸(β)和N,N-二甲基丙胺(Dp)。进一步研究发现,γ-氨基丁酸(γ)和β-氨基丙酸(β)不仅充当连接的角色,也是具有一定序列选择性的重要构件基团;Dp则能使寡聚酰胺分子在人体pH值条件下以阳离子的形式存在,增大分子的水溶性,同时对寡聚酰胺分子识别DNA时的分子取向以及对细胞膜的穿透也有重要意义[8~10]。
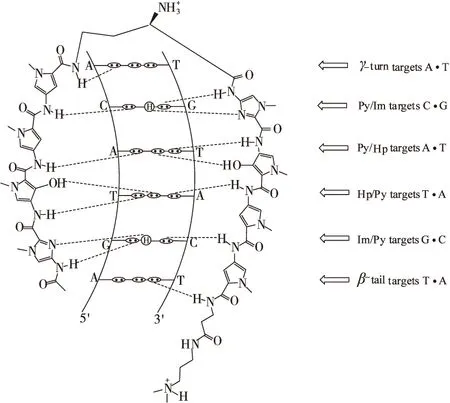
Py.N-methylpyrrole Im.N-methylimidazole γ-turn.γ-aminobutyric acid Hp.3-hydroxy-1-methylpyrrole β-tail.β-alanine dimethylamino propylamide
2 寡聚酰胺与DNA碱基的识别配对规则及结合模式
含吡咯-咪唑寡聚酰胺以其配对的芳香族氨基酸与相应DNA的碱基通过最大可能地形成氢键识别DNA序列。大量实验结果表明:寡聚酰胺分子主要通过反向配对的Py和Im实现对DNA特定序列的识别。据此,Dervan提出了寡聚酰胺识别DNA的配对规则:反向平行成对的Py/Im特异识别C·G碱基对,Im/Py特异识别G·C碱基对;反向平行成对的Hp/Py特异识别T·A碱基对,Py/Hp特异识别A·T碱基对,Py/Py特异识别T·A或A·T碱基对[11]。研究还发现,Im/β和β/Im能够从A·T和T·A中识别G·C和C·G碱基对,而Py/β和β/Py能够从G·C和C·G中识别A·T和T·A碱基对[12]。这类分子可在DNA的小沟处连续识别4~5个d(AoT)碱基对,由此1分子的寡聚酰胺与1条双链DNA通过结构匹配和氢键作用形成特异性识别作用(1∶1模型,图2a)[4]。多维核磁数据也证明在一定浓度下寡聚酰胺以反向平行肩并肩的结合方式(即2分子结合到DNA双螺旋小沟处且分别与2条链上DNA通过结构匹配和氢键形成)识别DNA的碱基序列(2∶1模型,图2b),通过γ-氨基丁酸(γ) 连接2分子形成发夹状寡聚酰胺(1∶1模型,图2c)[2,13,14]。利用这个识别规律,科学家们可以针对某一特定序列设计出相应的寡聚酰胺识别分子对其进行特异性识别,进而调控相关基因而达到治疗相关疾病的目的。
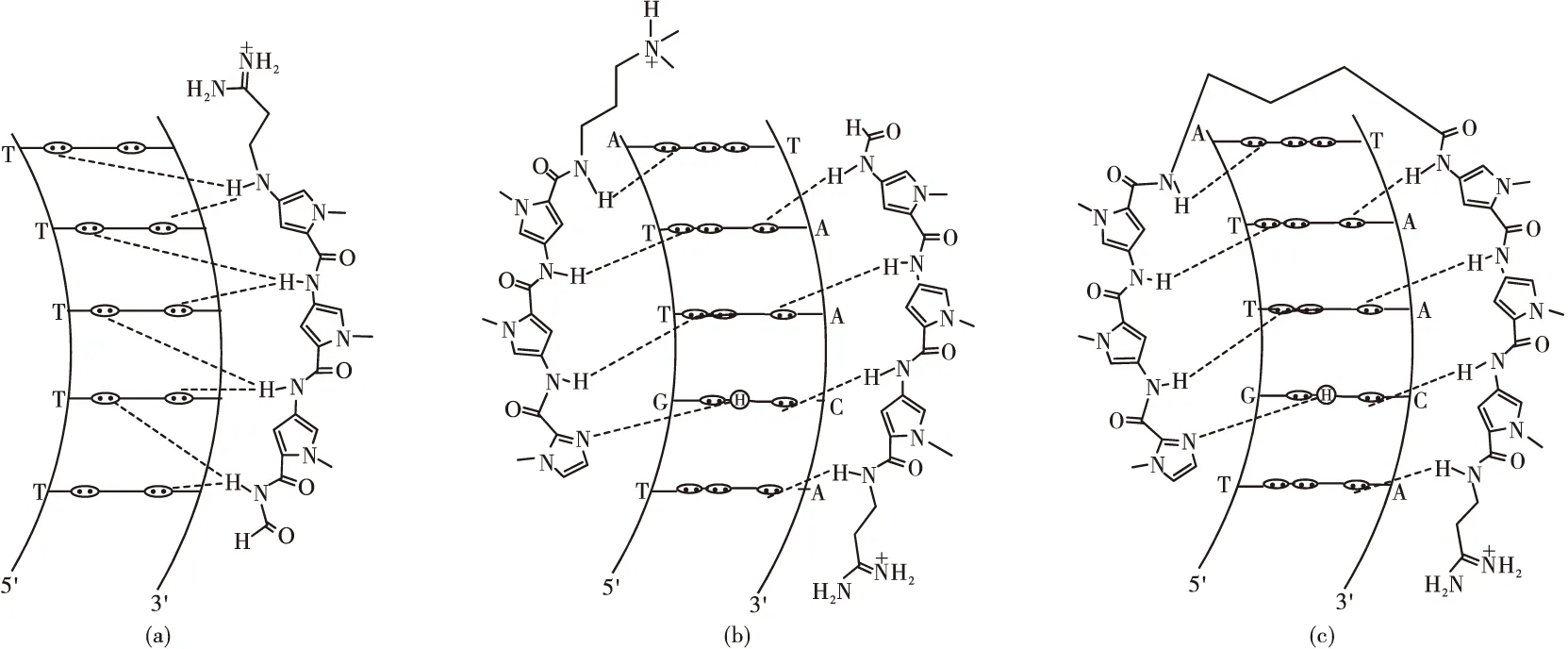
图2 吡咯-咪唑寡聚酰胺与DNA双螺旋作用的1∶1模型(a)、 吡咯-咪唑寡聚酰胺与DNA双螺旋作用的2∶1模型(b)、发夹状寡聚酰胺与DNA双螺旋作用的1∶1模型(c)
3 寡聚酰胺的透膜能力
根据特定DNA序列设计的寡聚酰胺要发挥对特定基因的调控作用,能否穿过细胞膜进入细胞核是关键的一步。Janssen等[15]和Sun等[16]最先开展了这方面的研究,他们分别先用聚甲醛固定果蝇Kc细胞和人类结肠癌细胞系DLD-1,再加入用荧光素标记的寡聚酰胺进行共培养,荧光素标记定位显示寡聚酰胺都能顺利进入这两种细胞的细胞核。Belitsky等[17]、Best等[18]和Dudouet等[19]利用激光共聚焦显微镜观察了多种活细胞中寡聚酰胺的透膜能力。结果表明寡聚酰胺能否进入细胞核主要取决于细胞的类型,例如在SKBR-3(人类乳腺癌细胞)、NB4(人类白血病细胞)、293(成纤维细胞)、Sf9(昆虫细胞)和Kc(果蝇细胞)中,寡聚酰胺都能进入胞质,但未能透过核膜进入细胞核;而在T细胞系CEM、MT-2、PM1及髓系细胞KYO1中则可以顺利进入细胞核。此外,寡聚酰胺的结构(如分子中是否含有β-氨基丙酸和咪唑基团及其位置和数量)明显影响到寡聚酰胺进入细胞核的能力[20]。
4 体外细胞水平寡聚酰胺调控基因表达
为考察寡聚酰胺类识别分子顺利进入细胞核后,是否与特定DNA序列特异性识别并调控其转录,研究者对体外细胞培养体系进行了研究。
Dickinson等[21]根据HIV-1病毒核酸序列增强子/启动子中多种转录因子如TBP、ETs-1和LEF-1等的识别序列,合成了分别与HIV-1基因TATA盒、LEF-1元件和ETs-1元件侧翼序列相匹配和错配的寡聚酰胺分子,结果发现匹配的寡聚酰胺能够抑制转录因子与靶序列结合从而抑制RNA聚合酶Ⅱ的转录。而错配的寡聚酰胺没有这样的效果。Lai等[22]合成了与转化生长因子-β1(TGF-β1)启动子FSE2元件侧翼碱基序列(-545~-539 bp)匹配及错配的寡聚酰胺,将匹配的寡聚酰胺与人平滑肌细胞培养体系共同孵育48 h,TGF-β1启动子的活性显著降低,TGF-β1的mRNA和蛋白转录表达受到明显抑制。研究表明,联合应用多个匹配的寡聚酰胺以阻断病毒的转录和复制是治疗疑难疾病的新策略。
5 体内动物模型中寡聚酰胺调控基因表达
寡聚酰胺分子在动物体内调控基因表达的机制,是通过特异性和某段基因序列相结合,从而对与该段基因序列结合的某些关键性的酶产生竞争性抑制作用来实现的(图3)[13]。相关研究的最初实验对象是以果蝇为主,其后研究寡聚酰胺对胚胎发育的影响、在小鼠模型上针对特定基因的转录抑制实验都取得了不错的结果。

图3 含吡咯-咪唑寡聚酰胺分子抑制基因表达作用模式
Dickinson等[23]设计的一种寡聚酰胺-瘤可宁结合物可作用于组蛋白H4C基因编码区,抑制多种肿瘤细胞系的增殖,且该结合物在荷瘤小鼠体内同样具有生物活性。Matsuda等[24]以Dahl-S小鼠为模型,设计了一种针对TGF-β1基因启动子AP-1元件的寡聚酰胺,能明显抑制小鼠肾小球系膜细胞中该基因启动子的活性。荧光素标记的寡聚酰胺静脉注射小鼠后可分布到肾脏,显著抑制肾脏皮质中TGF-β1的mRNA和蛋白的表达,并且不依赖血压变化就能减少尿蛋白。这种寡聚酰胺分子有可能治疗TGF-β1高表达相关的疾病。最近,Washio等[25]、Matsuda等[26]建立了小鼠增生性疤痕的动物模型,设计了针对其TGF-β1启动子区的配对的寡聚酰胺分子作为筛选药物并以错配的寡聚酰胺分子作为参照,分别从外观和微观切片的角度进行观察并在mRNA和蛋白质水平进行检测,结果显示配对的寡聚酰胺分子较好地抑制了TGF-β1的高表达,而错配的寡聚酰胺的抑制效果不明显,这些成果将这类化合物走向临床靶向治疗推进了一大步。
6 展望
DNA是遗传信息的载体,也是药物作用的重要靶点。随着人们对遗传信息越来越深入的认识,利用已知的遗传信息开发出治疗相关重大疾病的药物是科学家一直努力的方向。寡聚酰胺是一种新型的调控基因表达的DNA识别分子,与反义寡聚核苷酸和肽核酸相比更稳定、细胞通透性能更好,而且具有高度的序列选择性与结合力,能有效地抑制或激活相关基因的转录和表达。此外,寡聚酰胺识别分子不仅可以应用于抗肿瘤和抗病毒研究,还在医学诊断、生物标记、抗细菌、抗真菌等方面有着广泛的应用潜能,是一种很有前途的基因靶向筛选药物。
[1] Pindur U,Jansen M,Lemster T.Advances in DNA-ligands with groove binding,intercalating and/or alkylating activity:Chemistry,DNA-binding and biology[J].Curr Med Chem,2005,12(24):2805-2847.
[2] 江世坤.手性修饰的寡聚酰胺的构建及其对B-DNA碱基T的特异性识别[D].杭州:浙江工业大学,2011.
[3] Pelton J G,Wemmer D E. Structural characterization of a 2∶1 distamycin A.d (CGCAAATTTGGC) complex by two-dimensional NMR[J].Proc Natl Acad Sci USA,1989,86(15):5723-5727.
[4] Wade W S,Mrksich M,Dervan P B. Design of peptides that bind in the minor groove of DNA at 5′-(A,T)G(A,T)C(A,T)-3′ sequences by a dimeric side-by-side motif[J].J Am Chem Soc,1992,114(23):8783-8794.
[5] White S,Szewczyk J W,Turner J M,et al. Recognition of the four Watson-Crick base pairs in the DNA minor groove by synthetic ligands[J].Nature,1998,391(6666):468-471.
[6] Kelly J J,Baird E E,Dervan P B.Binding site limit of the 2∶1 pyrrole-imidazole polyamide-DNA motif[J].Proc Natl Acad Sci USA,1996,93(14):6981-6985.
[7] Dervan P B,Doss R M,Marques M A.Programmable DNA binding oligomers for control of transcription[J].Curt Med Chem Anticancer Agents,2005,5(4):373-387.
[8] Cho J Y,Parks M E,Dervan P B. Cyclic polyamides for recognition in the minor-groove of DNA[J].Proc Natl Acad Sci USA,1995,92(22):10389-10392.
[9] Herman D M,Turner J M,Baird E E,et al. Cycle polyamide motif for recognition of the minor groove of DNA[J].J Am Chem Soc,1999,121(6):1121-1129.
[10] Turner J M,Swalley S E,Baird E E,et al. Aliphatic/aromatic amino acid pairings for polyamide recognition in the minor groove of DNA[J].J Am Chem Soc,1998,120(25):6219-6226.
[11] Wemmer D E,Dervan P B.Targeting the minor groove of DNA[J].Curr Opin Struct Biol,1997,7(3):355-361.
[12] Surveyor G A,Wilson A K,Brigstock D R. Localization of connective tissue growth factor during the period of embryo implantation in the mouse[J].Biol Reprod,1998,59(5):1207-1213.
[13] Dervan P B. Molecular recognition of DNA by small molecules[J].Bioorg Med Chem,2001,9(9):2215-2235.
[14] Dervan P B,Edelson B S. Recognition of the DNA minor groove by pyrrole-imidazole polyamides[J].Curr Opin Struct Biol,2003,13(3):284-299.
[15] Janssen S,Durussel T,Laemmli U K.Chromatin opening of DNA satellites by targeted sequence-specific drugs[J].Mol Cell,2000,6(5):999-1011.
[16] Sun X H,Baltimore D.An inhibitory domain of E12 transcription factor prevents DNA binding in E12 homodimers but not in E12 heterodimers[J].Cell,1991,64(2):459-470.
[17] Belitsky J M,Leslie S J,Arora P S,et al. Cellular uptake ofN-methyl pyrrole/N-methyl imidazole polyamide-dye conjugates[J].Bioorg Med Chem,2002,10(10):3313-3318.
[18] Best T P,Edelson B S,Nickols N G,et al.Nuclear localization of pyrrole-imidazole polyamide-fluorescein conjugates in cell culture[J].Proc Natl Acad Sci USA,2003,100(21):12063-12068.
[19] Dudouet B,Burnett R,Dickinson L A,et al. Accessibility of nuclear chromatin by DNA binding polyamides[J].Chem Biol,2003,10(9):859-867.
[20] Edelson B S,Best T P,Olenyuk B,et al. Influence of structural variation on nuclear localization of DNA-binding polyamide-fluorophore conjugates[J].Nucleic Acids Res,2004,32(9):2802-2818.
[21] Dickinson L A,Gulizia R J,Trauger J W,et al. Inhibition of RNA polymerase Ⅱ transcription in human cells by synthetic DNA-binding ligands[J].Proc Natl Acad Sci USA,1998,95(22):12890-12895.
[22] Lai Y M,Fukuda N,Ueno T,et al. Synthetic pyrrole-imidazole polyamide inhibits expression of the human transforming growth factor-beta1 gene[J].J Pharmacol Exp Ther,2005,315(2):571-575.
[23] Dickinson L A,Burnett R,Melander C,et al. Arresting cancer proliferation by small-molecule gene regulation[J].Chem Biol,2004,11(11):1583-1594.
[24] Matsuda H,Fukuda N,Ueno T,et al. Development of gene silencing pyrrole-imidazole polyamide targeting the TGF-beta1 promoter for treatment of progressive renal diseases[J].J Am Soc Nephrol,2006,17(2):422-432.
[25] Washio H,Fukuda N,Matsuda H,et al. Transcriptional inhibition of hypertrophic scars by a gene silencer,pyrrole-imidazole polyamide,targeting the TGF-β1 promoter[J].J Invest Dermatol,2011,131(10):1987-1995.
[26] Matsuda H,Fukuda N,Ueno T,et al. Transcriptional inhibition of progressive renal disease by gene silencing pyrrole-imidazole polyamide targeting of the transforming growth factor-β1 promoter[J].Kidney International,2011,79(1):46-56.

