江蓠属和龙须菜属5种海藻ITS序列分子系统学分析*
李婷婷,陈 斌,陈省平,刘 翠,姚 雪,陈伟洲,赖学文,刘 涛
(1.中国海洋大学海洋生命学院,山东 青岛 266003;2.中山大学中山医学院,广东 广州510080;3.汕头大学,广东 汕头 515063;4.汕头市水产研究所,广东 汕头 515000)
江蓠科Gracilariaceae在自然分类系统上属于红藻门Rhodophyta江蓠目Gracilariales,包括江蓠属Gracilaria、龙须菜属Gracilariopsis、蓠生藻属Gracilariophila、Hydropuntia、Curdiea、Melanthalia和Congracilaria7个属。在红藻门中,江蓠科无论是解剖学层面[1-3]还是分子学层面[4-6],都是很明确的一个科,但是因缺乏足够多的形态学和生殖结构特征,其属间种间存在着复杂的分类关系[7-10]。在过去的20年里,分子生物学技术不断的应用到江蓠科分类研究中,揭示江蓠科种水平上的系统发生关系。目前江蓠科分子系统学研究多采用18S rDNA[10-11]、Rubisco spacer[11-13]、ITS(internal transcribed spacer)[10,12,14]、rbcL[15-18]、cox2-3[13,19]和psbA[16]等序列。
ITS序列包括ITS1和ITS2,是分别介于18S与 5.8S、5.8S 与28S rDNA之间的非编码序列,呈ITS1-5.8S-ITS2结构(本文将ITS1-5.8S -ITS2简称为ITS)。此结构中5.8S 是高度保守的基因序列,而ITS1和ITS2是进化速度较快的序列,利用ITS序列可以从不同分类水平上探讨系统进化关系。本文通过对来自江蓠属和龙须菜属总5个物种23个种群的ITS序列进行扩增,并与GenBank中江蓠科海藻ITS序列进行比对分析,从分子水平上阐明江蓠科海藻系统发育和种群水平的遗传变异,为江蓠科海藻的分类和鉴定提供更多数据。
1 材料与方法
1.1 实验材料
本实验所用江蓠科海藻5个物种23个群体由中国海洋大学大型海藻种质库提供(见表1)。每个群体选取2~3株个体进行实验,总66株实验材料。

表1 实验材料信息1)
1.2 实验方法
1.2.1 总DNA提取 总DNA提取采用改进的CTAB法[20]。所提取DNA用w=1%琼脂糖凝胶进行电泳检测,使用NanoDrop ND-1000仪器,测定A260/A280、A260/A230比值,对DNA浓度和纯度进行质量评价,并将DNA模板浓度统一调整到50 ng/μL。
1.2.2 PCR扩增 用于ITS序列扩增的引物为TW81:5’-GGGATCCGTTTCCGTAGGTGAACCTGC-3’;AB28:5’-GGGATCCATATGCTTAAGTTCAGCGGGT-3’[21],引物由上海博尚生物技术有限公司合成。PCR反应体系为25 μL,反应体系:ddH2O 16.8 μL,10×Buffer 2.5 μL, MgCl2(25 mmol/L) 2.3 μL,dNTP 2.0 μL,正反引物(10 μmol/L)各0.1 μL,DNA模板 (50 ng/μL) 1.0 μL,Taq DNA 聚合酶 (5 U/μL) 0.2 μL。PCR扩增程序:94 ℃预处理6 min后, 94 ℃变性1 min,51 ℃复性1 min,72 ℃延伸1 min,总35个循环,最后在72 ℃延伸10 min,4 ℃保存。
1.2.3 PCR产物纯化、克隆及测序 PCR产物用TIANgel Midi Purification Kit 试剂盒(北京天根生化科技有限公司)胶回收纯化后与pMD19-T载体(TaKaRa 宝生物工程有限公司)连接后转化至感受态E.coliTOP 10(北京天根生化科技有限公司)中,经氨苄青霉素抗性筛选,每份PCR产物挑取3个阳性克隆进行扩大培养,菌液PCR扩增并利用w=1%琼脂糖凝胶电泳检测插入片段大小,委托上海英骏生物技术有限公司公司进行双向测序。
1.2.4 数据分析 利用SeqMan 7.1.0软件对测序数据进行拼接和质量矫正,并与GenBank中下载到的江蓠科16个物种的19条ITS全长序列进行对比分析,选择长心卡帕藻Kappaphycusalvarezii(GQ869849)作为外群(GenBank获取号以及详细信息见表1和表2)。用MEGA 4.0计算其碱基含量和变异位点,并选择Kimura 2-parameter和Complete Deletion计算遗传距离。选择不同物种和不同地理种群的序列用ClustalX 1.83软件进行序列比对,用PAUP 4.0 软件在Modeltest 3.7中计算不同核酸替代模型的似然值并筛选出最适核酸替换模型,最终按Akatke Information Criterion(AIC)结果得到的最适核酸替换模型为GTR+I+G(参数:Base frequencies:A=0.268 6,C=0.170 8,G=0.237 2,T=0.323 4;substitution rate matrix: A-C=1.19 8,A-G=2.385 8,A-T=1.334 1,C-G=1.143 2,C-T=2.868 0,G-T=1.000 0;proportion of invariable sites=0.093 0,gamma parameter=2.510 4),利用以上数据在PAUP4.0中依据邻接法(Neighbor joining,NJ)和最大似然法(Maximum likelihood,ML)构建系统树,自举值Bootstrap为1 000次重复。依据最大简约法(Maximum parsimony,MP)构建系统树时在PAUP 4.0中采用启发式搜索(Heuristic search)的逐步加入式算法(Stepwise addition)分支交换法(Branch swapping algorithm)设定为树二等分再连接算法(Tree bisection reconnection,TBR),每轮搜索最大尝试次数为1 000次, 每步保留20个树,自举值Bootstrap为1 000次重复。
2 结果与分析
2.1 ITS序列长度和GC含量分析
ITS序列长度和GC含量分析结果见表2。江蓠科海藻ITS存在较为丰富的长度变异,长度在893~1 508 bp之间,其中龙须菜属龙须菜Gp.lemaneiformis和Gp.tenuifrons的ITS序列长度较其他物种短,分别为1 065(1 066) bp和893 bp;江蓠科海藻ITS1序列长度在121~568 bp之间,ITS2序列长度在585~984 bp之间,同一物种的ITS2序列明显长于ITS1序列,且其GC含量明显高于ITS1序列。
相比ITS1和ITS2序列,江蓠科海藻的5.8S序列长度较为稳定,除无管篱生藻Gp.oryzoides、细基江蓠G.tenuistipitata和智利江蓠G.chliensis3个物种外,其他物种5.8S序列长度均为159 bp;5.8S序列GC含量也明显高于ITS1和ITS2序列的GC含量。细基江蓠G.tenuistipitata5.8S序列为139 bp,在60 bp位点后缺失19个碱基,在114 bp处缺失一个碱基;智利江蓠G.chliensis5.8S序列为140 bp,在60 bp位点后缺失19个碱基;蓠生藻属无管篱生藻Gp.oryzoides的5.8S序列长162 bp,在102位点之后增加了3个T碱基(见图1)。

表2 江蓠科物种ITS序列长度及碱基组成1)

图1 江蓠科海藻5.8S核苷酸序列比对
2.2 ITS序列比对分析
对所有江蓠科的ITS 序列用MEGA 4.0进行比对,比对后序列长度为1 781 bp(including gaps),计算遗传距离结果显示,江蓠科各属内的种间遗传距离明显大于种内遗传距离(见表3)。江蓠属各物种的种内遗传距离在0.000~0.012之间,存在0~36个变异位点;细基江蓠繁枝变种和细基江蓠G.tenuistipitata种间遗传距离为0.004,存在25个变异位点,包括了14个种间区分位点;江蓠属其他物种的种间遗传距离在0.041~0.411之间,存在111~611个变异位点。龙须菜属的种内遗传距离为0.000,龙须菜和Gp.tenuifrons种间遗传距离为0.382。Hydropuntia中H.caudata种内遗传距离为0.009,存在14个变异位点;H.caudata和H.crassissima种间遗传距离为0.128,存在252个变异位点。江蓠属与龙须菜属物种间的遗传距离在0.401~0.554之间。江蓠属与Hydropuntia物种间的遗传距离在0.193~0.409之间。江蓠属与篱生藻属物种间的遗传距离在0.527~0.600之间。龙须菜属与Hydropuntia物种间的遗传距离在0.382~0.535之间。龙须菜属与篱生藻属物种间的遗传距离在0.267~0.406之间。Hydropuntia与篱生藻属物种间的遗传距离在0.533~0.578之间。
在本文研究的江蓠属和龙须菜属5个物种的23个群体中,龙须菜8个群体24株个体的平均遗传距离为0.000,存在2个变异位点;芋根江蓠2个群体6株个体的平均遗传距离为0.003,存在12个变异位点;脆江蓠2个群体6株个体的平均遗传距离为0.000,存在1个变异位点;细基江蓠繁枝变种6个群体17株个体的平均遗传距离为0.004,存在16个变异位点;上述物种不存在群体区分位点。真江蓠5个群体13株个体的平均遗传距离为0.002,13个变异位点,且中国、俄罗斯和美国三地的种群间存在9个稳定有效的信息位点,可区分三地的群体(见图2),但在中国两野生真江蓠群体间存在6个变异位点,均为个体的随机变异,并无稳定有效的信息位点。
单独对江蓠科海藻的5.8S序列进行分析,比对后序列长度为162 bp(including gaps),存在36个变异位点,其中包括了14个信息位点,序列比对结果见图1。江蓠属与龙须菜属物种间的遗传距离在0.046~0.084之间,存在7~11个变异位点,其中4个位点可进行属的鉴别。江蓠属与Hydropuntia物种间的遗传距离在0.006~0.026之间,存在1~4个变异位点,其中有1个位点可进行属的鉴别。江蓠属与蓠生藻属物种间的遗传距离在0.099~0.116之间,存在13~17个变异位点,其中12个位点可进行属的鉴别。龙须菜属与Hydropuntia物种间的遗传距离在0.052~0.080之间,存在8~12个变异位点,其中5个位点可进行属的鉴别。龙须菜属与蓠生藻属物种间的遗传距离为0.073~0.101,存在11~15变异位点,其中7个位点可进行属的鉴别。Hydropuntia与蓠生藻属物种间的遗传距离为0.123,存在18个变异位点,均可进行属的鉴别。在江蓠属中,除细基江蓠G.tenuistipitata、智利江蓠G.chilensis和G.pacifica外,其他物种的5.8S序列完全一致;细基江蓠G.tenuistipitata和智利江蓠G.chilensis除碱基缺失外,两者与其他江蓠属物种都存在一个位点的变异;G.pacifica与细基江蓠G.tenuistipitata、智利江蓠G.chilensis的遗传距离均为0.029,与其他江蓠属物种间的遗传距离均为0.019,存在3个变异位点。龙须菜属的龙须菜种内遗传距离为0.000,与Gp.tenuifrons物种间的遗传距离为0.060,存在10个变异位点。Hydropuntia中H.caudata和H.crassissima两物种5.8S序列完全一致。所有江蓠科物种的5.8S序列种内遗传距离都为0.000。

图2 真江蓠不同地理群体间 ITS序列稳定有效信息位点

表 3 江蓠科海藻的种间遗传距离1)
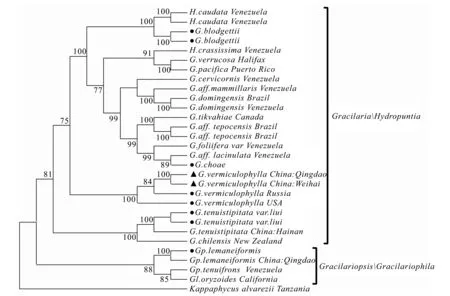
图3 江蓠科海藻ITS 序列系统进化树(MP)
2.3 ITS系统树构建
根据江蓠科物种ITS序列用NJ、MP和ML法构建系统树,3种方法聚类结果一致,仅列出MP法构建的系统树(见图3)。比对后总1772位点矩阵,302个稳定位点,91个变异位点但无信息的位点,1 379个信息位点,启发式搜寻后最终获得1个最优简约树,步长为5 435,一致性指数(CI)和严格性指数(RI)分别是0.586和0.765。从系统树结构中可以看出,20种江蓠科海藻可基本分为2大分支,包括龙须菜属/蓠生藻属分支,江蓠属/Hydropuntia分支。其中龙须菜属和蓠生藻属处在系统树的最基部,说明其分化时间可能早于江蓠属/Hydropuntia。江蓠属/Hydropuntia形成了3个小的分支,分别是细基江蓠繁枝变种G.tenuistipitatavar.liui分支、真江蓠G.vermiculophylla分支、脆江蓠G.chouae与芋根江蓠G.blodgettii分支;青岛与威海的野生真江蓠关系最近,其次是俄罗斯和美国的养殖真江蓠,支持率分别为100、84和100。
3 讨 论
江蓠科4个属20个物种ITS长度差异较大,遗传距离和系统聚类分析结果显示,不同种、属之间存在较为广泛的变异。Yamamoto等[24]根据精子囊窠的发生位置将江蓠属分为江蓠属、Hydropuntia和龙须菜属。但在本研究中用ITS序列构建系统树,结果显示Hydropuntia物种聚类到江蓠属内,并未形成单独的分支,不存在明显的属间遗传分化,但与龙须菜属/蓠生藻属分别聚在两大分支上,存在明显的属间遗传分化。这与之前报道的SSU rDNA、Rubisco spacer和rbcL序列系统聚类的结果是一致的[10,12,15]。在江蓠属系统分类中,江蓠属和Hydropuntia海藻精子囊窠包括了“V”型、“T”型和“P型”3种类型,而龙须菜属精子囊窠则属于“C型”[25],在系统树中可以将其分为两组;但江蓠属和Hydropuntia海藻并未按其精子囊窠排列方式的类型而形成单独的分支,也没有形成与地域分布相关的分支,这可能与这两个属存在着并系起源关系有关。篱生藻属寄生于江蓠属、Hydropuntia和龙须菜属海藻,其藻体内部构造和生殖结构同于江蓠属、Hydropuntia和龙须菜属[26];蓠生藻属海藻是从最初的宿主藻体进化而来[27],在寄生过程中可从宿主获取叶绿体,而保留自身的核基因组和线粒体基因组[28],Bellorin等[10]认为篱生藻属与龙须菜属海藻属于并系起源,至少有一个篱生藻属物种是由龙须菜属演化而来;本研究也显示了同样的结果。但是蓠生藻属海藻寄生过程中是否会与宿主发生遗传物质的水平转移,以及在不同寄主中是否多次发生遗传物质的水平转移还有待证实。
ITS序列无论是在序列长度还是变异程度上都能很好地区分和鉴定江蓠科物种。细基江蓠和细基江蓠繁枝变种的形态学主要差异是前者分枝1~2次,后者具有更多纤细的分枝;尽管细基江蓠和细基江蓠繁枝变种的ITS序列遗传距离低于江蓠科全部海藻的种间差异,属于种内差异水平,但其存在着25个变异位点,包括了14个种间区分位点,同时细基江蓠5.8S序列较江蓠属海藻缺失20个碱基,显示出独特的序列结构,这可能意味着细基江蓠与细基江蓠繁枝变种之间的分类地位还需要进一步的研究确认。
江蓠科海藻5.8S序列长度相对ITS1和ITS2保守,且种内和种间变异很小,存在着特定的稳定属间区分位点,且对于具有并系关系的属(江蓠属/Hydropuntia和龙须菜属/蓠生藻属)也具有稳定的区分位点,因此利用这些位点可以进行江蓠科属水平上的分类鉴定。同样已报道的江蓠科18S rDNA序列,也存在这特定的属间区分位点,能够很好地区分属[12]。
本研究显示,龙须菜、脆江蓠和真江蓠(美国和俄罗斯)的养殖群体的群体间和群体内变异相比野生群体遗传变异较小,江蓠科海藻养殖主要通过无性繁殖进行,不发生有性生殖过程的杂交以及无性繁殖的遗传重组,这种养殖群体变异水平降低的情况可能是由于养殖筛选的影响;但在细基江蓠繁殖变种的养殖群体存在相对较高的遗传变异,这可能是由于其自然分布和养殖主要集中在亚热带和热带海域,生长速率和养殖继代频率高,导致其群体ITS序列变异幅度高于其他江蓠种群。在来自中国、俄罗斯和美国三地的真江蓠群体中,存在着9个与地理分布相关的信息位点,可以进行群体的有效区分。这与在真江蓠、提克江蓠G.tikvahiae、绳江蓠Gp.chorda、张氏江蓠G.changii的cox1和rbcL基因序列存在的与地理位置相关的单倍型是一致的[29-32]。但在青岛和威海的真江蓠野生群体间并不存在种群间的稳定区分位点,因此,信息位点在地理群体识别的方面应用需要考虑到群体数量以及地理距离的因素。
4 前景展望
随着分子生物学研究的不断深入发展,核酸数据的不断积累,利用单个或多个DNA序列并结合物种形态学研究物种分类与系统进化,逐渐成为现代生物学研究的有力工具。目前,DNA序列分析在藻类分类鉴定中得到了广泛的应用,对阐明系统进化关系提供了重要的证据。但从分子系统学研究手段本身而言,也存在很多需要进一步研究和探讨的问题。不同类群同一DNA序列的进化速率有所差异,同一类群中不同DNA序列的进化速率也不相同,另外由于藻类早期演化中存在着线粒体和叶绿体内共生现象,其在平行演化中又存在着并系起源和直系起源现象,这为研究藻类系统发育与分化增添了较大的难度。从多个层次以及多个基因位点上系统地研究藻类形态结构进化、生态适应以及基因进化的内在关系则显得更为重要和迫切。
参考文献:
[1]FREDERICQ S,HOMMERSAND M H.Comparative morphology and taxonomic status ofGracilariopsis(Gracilariales,Rhodophyta) [J].J Phycol,1989,25: 228-241.
[2]FREDERICQ S,HOMMERSAND M H.Proposal of the Gracilariales ord.nov.(Rhodophyta) based on an analysis of the reproductive development ofGracilariaverrucosa[J].J Phycol,1989,25: 213-227.
[3]FREDERICQ S,HOMMERSAND M H.Diagnoses and key to the genera of the Gracilariaceae (Gracilariales,Rhodophyta) [J].Hydrobiologia,1990,204/205: 173-178.
[4]FRESHWATER D W,RUENESS J.Phylogenetic relationships of some EuropeanGelidium(Gelidiales,Rhodophyta) species based onrbcL nucleotide sequences analysis[J].Phycologia,1994,33: 187-194.
[5]SAUNDERS G W,KRAFT G T.A molecular perspective on red algal evolution: focus on the Florideophycidae[J].Plant Systematics and Evolution,1997,11: 115-138.
[6]HARPER J T,SAUNDERS G W.Molecular systematics of the Florideophyceae (Rhodophyta) using nuclear large and small subunit rDNA sequence data[J].J Phycol,2001,37: 1073-1082.
[7]BIRD C J,RICE E L,MURPHY C A,et al.Phylogenetic relationships in the Gracilariales (Rhodophyta) as determined by 18S rDNA sequences[J].Phycologia,1992,31: 510-522.
[8]BIRD C J.A review of recent taxonomic concepts and developments in the Gracilariaceae (Rhodophyta) [J].J Appl Phycol,1995,7: 255-267.
[9]BIRD C J,RAGAN M A,CRITCHLEY A T,et al.Molecular relationships among the Gracilariaceae (Rhodophyta): further observations on some undetermined species[J].Europ J Phycol,1994,29: 195-202.
[10]BELLORIN A M,OLIVEIRA M C,OLIVEIRA E C.Phylogeny and systematics of the marine algal family Gracilariaceae (Gracilariales,Rhodophyta) based on small subunit rDNA and ITS sequences of Atlantic and Pacic species[J].J Phycol,2002,38: 551-563.
[11]IYER R,TRONCHIN E M,BOLTON J J,et al.Molecular sysytematics of the Gracilariaceae (Gracilariales) with emphasis on southern Africa[J].J Phycol,2005,41: 672-684.
[12]GUILLEMIN M L,AKKI S A,GIVERNAUD T,et al.Molecular characterisation and development of rapid molecular methods to identify species of Gracilariaceae from the Atlantic coast of Morocco[J].Aquatic Botany,2008,89: 324-330.
[13]BYRNE K,ZUCCARELLO G C,WEST J,et al.Gracilariaspecies (Gracilariaceae,Rhodophyta) from southeastern Australia,including a new species,Gracilariaperplexasp.nov.: Morphology,molecular relationships and agar content[J].Phycological Research,2002,50: 295-311.
[14]WEINBERGER F,GUILLEMIN M L,DESTOMBE C,et al.Defense evolution in the Gracilariaceae (Rhodophyta):substrate-regulated oxidation of agar oligosaccharides ia more ancient than the oligoagar-activated oxidative burst [J].J Phycol,2010,46: 958-968.
[15]GARGIULO G M,MORABITO M,GENOVESE G,et al.Molecular systematics and phylogenetics of Gracilariaceaen species from the Mediterranean Sea[J].J Phycol,2006,18: 497-504.
[16]KIM M S,YANG E C,BOO S M.Taxonomy and phylogeny ofattened species ofGracilaria(Gracilariceae,Rhodophyta) from Korea based on morphology and protein-coding plastidrbcL andpsbA sequences[J].Phycologia,2006,45 (5): 520-528.
[17]GURGEL C F D,FREDERICQ S.Systematics of the Gracilariaceae(Gracilariales,Rhodophyta): A critical assessment based onrbcL sequence analyses[J].J Phycol,2004,40: 138-159.
[18]GURGEL C F D,LIAO L M,FREDERICQ S,et al.Systematics ofGracilariopsis(Gracilariales,Rhodophyta) based onrbcL sequence analyses and morphological evidence[J].J Phycol,2003,39: 154-171.
[19]ZUCCARELLO G C,URGER G B,WEST J A,et al.A mitochondrial marker for red algal intraspecic relationships[J].Molecular Ecology,1999,8: 1443-1447.
[20]孙晓宇,罗丹,赵翠,等.不同保存条件下五种大型海藻的DNA提取和PCR分析[J].分子植物育种(online),2011,9(95): 1680-1691.
[21]COLEMAN A W,VACQUIER V D.Exploring the phylogenetic utility of ITS sequences for animals: A test case for abalone (Haliotis)[J].Journal of Molecular Evolution,2002,54: 246-257.
[22]GOFF L J,MOON D A,COLEMAN A W.Molecular delineation of species and species relationships in the red algal agarophytesGracilariopsisandGracilaria(Gracilariales)[J].J Phycol,1994,30(3): 521-537.
[23]李敏,隋正红,易恒,等.龙须菜5.8S rRNA和ITS区的克隆与系统学分析[J].中国海洋大学学报: 自然科学版,39(1): 77-83.
[24]YAMAMOTO H.The relationship betweenGraciariopsisandGraciariafrom Japan[J].Bull Fac Fish.Hokkaido Univ,1975,26: 217-222.
[25]ISABELLA A A,ZHANG Junfu,XIA Bangmei.Graciariamixta,sp.nov.and other western Pacific species of the genus (Rhodophyta: Gracilariaceae) [J].Pacific Science,1991,45(1): 12-27.
[26]夏邦美,张峻甫.中国海藻志[M].北京: 科学出版社,1999:17.
[27]GOFF L J,ZUCCARELLO G.The evolution of parasitism in red algae: Cellular interactions of adelphoparasites and their hosts[J].J Phycol,1994,30:695-720.
[28]GOFF L J,COLEMAN A W.Fate of parasite and host organelle DNA during cellular transformation of red algae by their parasites[J].The Plant Cell Online,1995,7(11): 1899-1911.
[29]YANG E C,KIM M S,GERALDINO P J L,et al.Mitochondrialcox1 and plastidrbcL genes ofGracilariavermiculophylla(Gracilariaceae,Rhodophyta)[J].J Appl Phycol,2008,20: 161-168.
[30]GURGEL C F D,FREDERICQ S.Phylogeography ofGracilariatikvahiae(Gracilariaceae,Rhodophyta):A study of genetic discontinuity a continuously distributed species based on molecular evidence[J].J Phycol,2004,40: 138-159.
[31]KIM M S,YANG E C,KIM S Y,et al.Reinstatement ofGracilariopsischorda(Gracilariaceae,Rhodophyta) based on plastidrbcL and mitochondrialcox1 sequences[J].Algae,2008,23(3): 209-217.
[32]YOW Y Y,LIM P E,PHANG S M.Genetic diversity ofGracilariachangii(Gracilariaceae,Rhodophyta) from west coast,Peninsular Malaysia based on mitochondrialcox1 gene analysis[J].J Appl Phycol,2011,23: 219-226.

