应用微卫星标记研究Dunkin Hartley豚鼠封闭群的遗传背景
朱 亮,蔡月琴,屠 珏,应华忠,徐剑钦,陈民利
(浙江中医药大学动物实验研究中心,杭州 310053)
应用微卫星标记研究Dunkin Hartley豚鼠封闭群的遗传背景
朱 亮,蔡月琴,屠 珏,应华忠,徐剑钦,陈民利
(浙江中医药大学动物实验研究中心,杭州 310053)
目的 检测我国现有Dunkin Hartley豚鼠封闭的遗传背景,分析评估其遗传多样性水平和遗传分离情况,为建立标准化的豚鼠封闭群监测方法提供基础资料。方法 应用筛选获得的8个微卫星位点,从一个数量为1000的豚鼠封闭群中随机选择72个个体,通过PCR扩增和聚丙烯酰胺凝胶电泳的方法,进行等位基因检测。并根据检测结果分析评估了该豚鼠封闭群的遗传现状。结果 共检测到28个等位基因,每个座位的等位基因数为2~5个,有效等位基因数为1.5191~3.4422,平均2.3093。平均期望杂合度为0.5294。各位点多态信息含量在0.3154~0.6545之间,平均值为0.4687。有5个位点显著偏离Hardy-Weinberg平衡。结论 豚鼠封闭群的遗传多态性处于中等水平,遗传平衡检测结果提示种群的繁殖过程未能实现完全随机交配,近交现象一定程度上存在。本研究的结果将为豚鼠封闭群遗传监测方法和标准的建立提供基础。
豚鼠;封闭群;微卫星标记;遗传多样性
浙江中医药大学动物实验研究中心豚鼠繁殖中心于1998年引进Dunkin Hartley豚鼠封闭群,饲养保种至今,已形成1000个个体的种群。中心研究人员通过磁珠富集法筛选获得了17个豚鼠微卫星位点[1]。本研究将以这些位点为工具,对Dunkin Hartley豚鼠封闭群进行微卫星位点检测,以期获得现有实验豚鼠种群的遗传概貌信息,并从分子遗传角度对现有繁育方式做出评价。
1 材料与方法
1.1 实验动物及主要试剂和仪器
Dunkin Hartley豚鼠72只,从浙江中医药大学动物实验研究中心豚鼠繁殖中心饲养的封闭群中随机抽取[SCXK(浙)2008-0037]。PCR相关试剂购自宝生物工程(大连)有限公司。聚丙烯酰胺凝胶电泳相关试剂购自上海生工生物工程有限公司。PTC-200 PCR扩增仪(Bio-Rad);水平电泳系统和垂直电泳系统(GE);凝胶成像系统(UVP)。
1.2 基因组的提取
取豚鼠新鲜肌肉组织(0.3~0.5)cm3,在 1.5 mL离心管中用消毒后的剪刀剪成小碎块。然后参照《分子克隆实验指南》方法提取基因组DNA[2]。
1.3 引物的选择及筛选
通过预实验,从已有的17个豚鼠微卫星位点中筛选出8个多态性好的位点(表1)进行本实验。所有引物由上海生工生物技术有限公司合成。
1.4 PCR扩增
10 μL 反应体系:1 μL 模板 DNA(10 ~30 ng),0.5 U Taq DNA 聚合酶,1 μL 10×PCR 缓冲液,1 μL MgCl2(25 mmol/L) ,0.75 μL dNTPs(20 mmol/L),上下游引物(10 μmol/L)各 0.1 μL。反应条件如下:94℃预变性3 min;94℃变性30 s,每对引物的最佳退火温度(表 1)退火30 s,72℃延伸30 s,35个循环;然后72℃延伸10 min。
1.5 等位基因的分离
从扩增产物中取2 μL在1%琼脂糖凝胶上进行电泳检测,并根据检测结果调整产物浓度。利用6%变性聚丙烯酰胺凝胶电泳对PCR扩增产物进行等位基因分离。电泳条件:恒压250 V,0.5×TBE缓冲液中电泳3~4 h。经 Gelred染色,使用凝胶成像系统拍照记录。然后对等位基因电泳图谱进行分析。
1.6 数据统计分析
在 Cervus 3.0遗传分析软件上[3],根据 Bostein等的公式[4],计算微卫星位点的多态信息含量(polymorphic information content,PIC); 根 据Chakraborty的公式,分别计算单位点非父排除概率(exclusion probability,PE)和多位点非父累积排除概率 (cumulative exclusion probability,CPE)。根 据Fisher的公式[5]:DPi=1 -,计算单位点个体识别率(discrimination power,DP),n表示某位点的基因型数,Pi表示该位点第i个基因型的频率;根据公式:CDP=1-(1-DPi),计算多位点累积个体识别率(cumulative DP),其中,m表示微卫星位点数,DPi表示第 i个位点的个体识别率。评估本研究使用的微卫星位点的质量及其反映群体遗传水平的有效性。
使用 POPGENE version 1.32 软件[6]统计计算各个微卫星位点在豚鼠群体中的观察等位基因数(number of alleles per locus,Na)、有效等位基因数(number of effective alleles,Ne)、观察杂合度 (observed heterozygosity,Ho)、期望杂合度(expected heterozygosity,He)等指标。从而评估各微卫星位点及豚鼠群体的遗传多样性。
使用GENEPOP软件对各位点进行连锁分析,根据位点等位基因的数目多少,应用完全计数或马尔科夫链法检验群体的Hardy-Weinberg遗传平衡状态,同时计算Fis值用于评估群体的近交程度。
2 结果
2.1 微卫星扩增结果及多态性
8个微卫星位点均呈现出多态性,共检测到28个等位基因,各等位基因片段大小在149~349 bp之间。每个座位的等位基因数在2~5个之间,平均等位基因数3.5个。各基因位点的有效等位基因数为1.5191~3.4422不等,平均为2.3093个。等位基因最多的座位是 Cpo8,有5个等位基因;等位基因最少的座位是Cpo6,有2个等位基因。基因型种类最多的位点是 Cpo8,检测到9种基因型;位点Cpo6显示的基因型只有2种,是所有位点中最少的。各个基因位点的等位基因数、等位基因频率和有效等位基因数的统计结果见表2。
2.2 群体遗传多样性分析
利用POPGENE version 1.32计算出各座位的观察杂合度(Ho)、期望杂合度(He)、多态信息含量等指标,评估位点及群体的遗传多样性,结果见表3。各个基因位点的观察杂合度在0.2222~0.5833之间,群体平均值为0.4531;期望杂合度在0.3441~0.7159之间,群体平均值为0.5294;8个位点的多态信息含量数值在0.3154~0.6545之间波动,群体平均值为0.4687。期望杂合度和多态信息含量最低的座位是Cpo3,分别为0.3441和0.3154;最高的座位是 Cpo11,分别为0.7159和0.6545。这8个位点的个体识别率从0.5403至0.8606不等,累积个体识别率达到了1.0000;各位点非父排除概率1在0.0584~0.2833之间,八位点累积值为0.7570;各位点非父排除概率2在0.1728~0.4519之间,八位点累积值为0.9415。
2.3 Hardy-Weinberg遗传平衡检验
采用 Fisher's 精确检验(Fisher's exact test)或马可夫链模型计算豚鼠群体每个微卫星位点Hardy-Weinberg遗传平衡检验的精确 P值,并计算相应的Fis值[Fis(W&C)]。结果显示(表 3),在本研究检测的一共 8个位点中,有 5个位点(Cpo2、Cpo4、Cpo6、Cpo8、Cpo11)显著偏离了 Hardy-Weinberg 平衡。在5个偏离 Hardy-Weinberg平衡的位点中,有4 个位点(Cpo2、Cpo4、Cpo8、Cpo11)的 Fis值为正值。以上结果提示本研究检验的豚鼠群体存在杂合子缺失的现象。
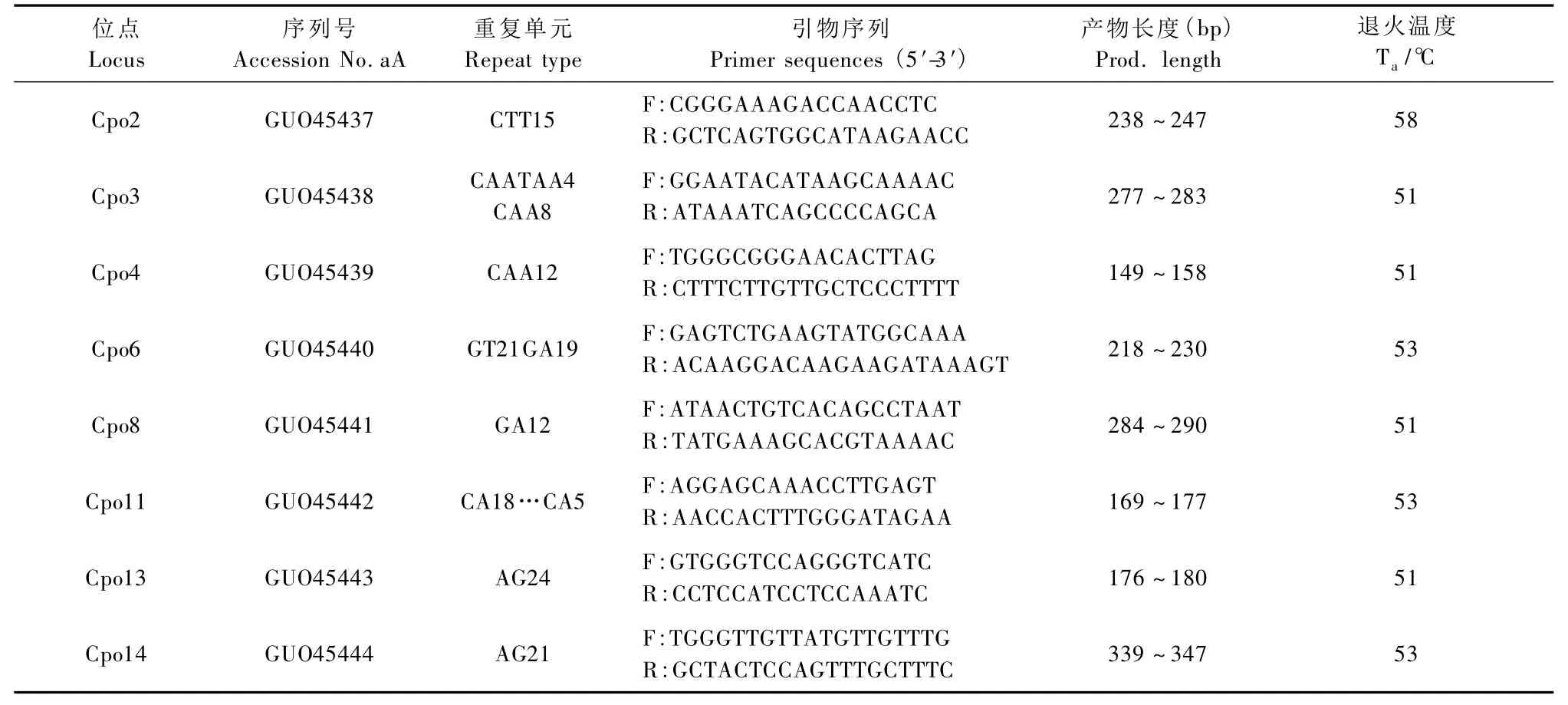
表1 8个豚鼠微卫星位点和相关信息Tab.1 Charecteristics of eight microsatellite loci from guinea pig(Cavia porcellus)

表2 豚鼠封闭群在8个微卫星位点上的等位基因信息Tab.2 Allele information at 8 microsatellite loci of guinea pig outbred colony

表3 豚鼠封闭群在8个微卫星位点上的遗传多态性Tab.3 The genetic diversity measured at 8 microsatellite loci of guinea pig outbred colony
3 讨论
3.1 微卫星标记在实验动物封闭群遗传检测中的应用价值
作为一大类常用的实验动物,封闭群动物在遗传学上有显著的特点。封闭群个体的遗传结构呈杂合性,在同一基因位点上包含更多的等位基因,即具有更高的基因多态性。封闭群动物的遗传背景一般通过等位基因个数、等位基因频率和基因型频率来体现。要获得以上参数的准确数值,需要一个合适的遗传检测工具。该遗传检测工具要能够检测多位点并体现各位点的高多态性。而目前常用的实验动物遗传检测方法是以生化标记检测为主的表型检测方法,主要针对近交系动物。其检测位点较少体现多态性差,不适合于遗传多态性高的封闭群动物,无法反映封闭群动物种群真实的遗传概貌。而微卫星标记具有以下特点:①分布广,覆盖整个基因组;②数量多,揭示的多态性高;③呈现多等位基因的特性,提供的信息量大;④引物通用、容易扩增。以上特点使微卫星标记很适合用于封闭群动物的遗传检测。
本研究利用8个微卫星标记,采用PCR-聚丙烯酰胺凝胶电泳分型技术对一个Dunkin Hartley豚鼠封闭群的遗传背景进行了检测。涉及的8个微卫星位点的多态信息含量(PIC)从0.3154至0.6545不等。多态信息含量(PIC)是根据 Botstein et al(1980)提出的公式计算出来的,它是衡量微卫星DNA座位变异程度高低的指标。当微卫星座位的PIC>0.5时,为高度多态座位;当0.5>PIC>0.25时,为中度多态座位;当PIC<0.25时,为低度多态座位。本研究涉及的8个豚鼠微卫星位点中,3个为高度多态位点,5个为中度多态位点,平均多态信息含量为0.467。同时,这8个位点的个体识别率从0.5403至0.8606不等,累积个体识别率达到了1.0000;其累积非父排除概率1和累积非父排除概率2分别达到了0.7570和0.9415。以上结果表明本研究使用的8个微卫星位点能够提供充足的遗传多样性信息,可以满足封闭群动物遗传背景检测的要求,同时还可以作为动物个体间亲缘关系检测及新品系培育等工作的有力辅助工具。
3.2 豚鼠封闭群的遗传多样性
每个位点等位基因的数目及全部位点等位基因的平均数是表征遗传多态性的重要参数。有效等位基因数是基因纯合度的倒数,反映了等位基因的相互影响,也可作为群体遗传变异的一个指标[7]。本研究各座位等位基因数在2~5个之间,平均3.5个,各位点有效等位基因平均2.3093。
观察杂合度和期望杂合度都是群体遗传多样性的反映。前者反映的是群体中实际的杂合比例,容易受样本大小等因素的影响。而期望杂合度是假定各基因座位符合Hardy-Weinberg平衡而得到的杂合度,它受样本取样的影响较小,常被用来度量群体的遗传多样性,其高低可反映群体的遗传一致性程度[7],期望杂合度的数值越高,所在群体的遗传多样性就越丰富。本研究中各个微卫星位点期望杂合度在0.3441~0.7159之间,群体平均值为0.5294。
由于豚鼠种群的遗传多样性水平目前还没有报道,我们将本研究获得的豚鼠种群遗传多样性指标与分类地位相近的实验动物做了比较。昆明小鼠封闭群的平均等位基因数为6.1333,平均有效等位基因数为 3.1029,平均期望杂合度为 0.5721[8];Wistar大鼠和SD大鼠的平均等位基因数为4.1667和4.3333,平均有效等位基因数为2.6717和2.5629,平均期望杂合度为 0.5982和 0.5859[9];长爪沙鼠的平均等位基因数为2.6,平均有效等位基因数为2.1756[10]。日本大耳白兔、新西兰兔和 WHBE兔的平均有效等位基因数分别为3.6077、2.6537和2.0402[11]。通过比较可见,本研究中 的 Dunkin Hartley豚鼠种群遗传多样性处于中等水平。
Fis是种群内个体之间的近交系数,它的取值范围为-1至1。当Fis值为极显著的正值时,表示群体内近交程度较严重;当Fis值为极显著的负值时,群体内观测杂合度大于期望杂合度,则表示群体内存在远交[12]。本研究得到的 Fis值在 4个位点(Cpo2、Cpo4、Cpo8、Cpo11)为正值,说明该豚鼠种群内杂合体较少,提示群体内存在近交现象。
根据Hardy-Weinberg遗传平衡检验结果,豚鼠种群在 5 个位点(Cpo2、Cpo4、Cpo6、Cpo8、Cpo11)偏离Hardy-Weinberg平衡,在3个位点处于遗传平衡状态(Cpo3、Cpo13、Cpo14),大部分偏离遗传平衡的位点都表现出杂合子缺失的现象。导致这一现象的原因除了无效等位基因之外,大部分的偏离可能是封闭群内存在近交导致的。同时,偏离遗传平衡的位点相应的Fis值绝大多数为正值,也揭示了种群内存在一定程度近交的可能性。实验动物封闭群Hardy-Weinberg平衡偏离现象在实验动物繁育研究中常见报道的一个问题[13,14]。
封闭群动物个体的遗传结构呈杂合性,而整个动物群体内全部杂合型基因的分布频率,即遗传组成在每一代保持稳定性。杂合性有利于群体携带更多的基因,稳定性则保证群体对实验反应呈现最大的重复性[15]。如果随机交配因素在种群内持续存在的话,会导致等位基因的丢失和种群基因频率的改变,无法保持封闭群的遗传特性,从而影响到试验结果的可比性。以目前封闭群实验动物的繁育过程中普遍缺乏相应的遗传监测的现状来看,除了要遵守严格的繁育程序操作以避免近交现象,还有必要对封闭群进行定期的遗传检测,并且根据检测结果评估和调整繁育方式。
本研究利用8个微卫星标记,初步确定了豚鼠群体的遗传概貌,揭示了豚鼠封闭种群的遗传多样性处于中等水平。同时,Hardy-Weinberg遗传平衡检验结果提示种群的繁殖过程未能实现完全随机交配,近交现象一定程度上存在。本研究的结果将为豚鼠封闭群遗传监测方法和标准的建立提供基础。
[1] 朱亮,蔡月琴,屠珏,等.磁珠富集法筛选实验豚鼠微卫星分子标记[J]. 中国比较医学杂志,2006,20(6):29-34.
[2] J.萨姆布鲁克,D.W.拉塞尔.分子克隆实验指南[M].第三版,北京:科学出版社,2002,463-483.
[3 ] Marshall TC,Slate J,Kruuk LE,et al.Statistical confidence for likelihood-based paternity inference in natural populations[J].Mol Ecol,1998,7:639-655.
[4] Botstein D,White RL,Skolnick M,et al.Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J].Am J Human Genet,1980,32(3):314-331.
[5] Fisher RA.Standard calculations for evaluating a blood group system[J].Heredity,1951,5:95-102.
[6 ] Yeh FC,Yang RC,Boyle-Timothy B,et al.POPGENE,the userfriendly shareware for population genetic analysis[M].Molecular Biology and Biotech nology Centre,University of Alberta,Canada,1997.
[7] Frankham R,Ballou JD,Briscoe DA.Introduction to Conservation Genetics[M].Cambridge University Press,2002,29-62.
[8] 商海涛,魏泓,岳秉飞,等.应用微卫星标记对三个昆明小鼠封闭群的遗传学研究[J].实验动物科学,2009,26(2)1-6.
[9] 商海涛,魏泓,岳秉飞,等.应用微卫星DNA标记对Wistar和SD大鼠封闭群的遗传学研究[J].分子细胞生物学报,2008,41(1):28-34.
[10] 刘月环,吴旧生,萨晓婴,等.Z:ZCLA长爪沙鼠微卫星标记的遗传多态性研究[J]. 兽类学报,2005,25(2):168-174.
[11] 陈民利,蔡月琴,陶涛,等.微卫星标记对 WHBE兔封闭群、日本大耳白兔和新西兰兔的遗传分析[J].中国比较医学杂志,2008,18(9):21-27.
[12] Weir BS,Cockerham CC.Estimation F-statistics for the analysis of population structure[J].Evolution,1984,38:1358-1370.
[13] Degorge J,Genetic evaluation of outhred rats[C].In:Microbial Status and Genetic Evaluation of Mice and Rats.Proceedings of the 1999 US/Japan Conference,2000,47-50.
[14] KlotingI,Nitschke C,Van den Brandt J.Impact of genetic profiles on experimental studies:outbred versus wild rats[J].Toxicol Appl Pharmacol,2003,189(1):68-71.
[15] 蒋建敏,陈民利.实用医学实验动物学[M].浙江:浙江人民出版社,2009,23.
Assessment of the genetic background in Dunkin Hartley guinea pig outbred stock using microsatellite DNA markers
ZHU Liang,CAI Yue-qin,TU Jue,YING Hua-zhong,XU Jian-qin,CHEN Min-li
(Laboratory Animal Research Center,Zhejiang Chinese Medical University,Hangzhou 310053,China)
Objective To estimate the genetic diversity and genetic differentiation of a Dunkin Hartley guinea pig outbred stock and providing valuable information and references for standardization of the population.Methods A total of 72 samples from a Dunkin Hartley guinea outbred pig stock were genotyped using 8 microsatellite DNA markers by PCR-denaturing polyacrylamide gel electrophoresis.Genetic variation of the population was evaluated and Hardy-Weinberg equilibrium was tested for each locus.Results 28 alleles were found in the outbred stock,2-5 alleles each locus,and the effective number of alleles each locus ranged from 1.5191 to 3.4422,with an average of 2.3093.The average of unbiased expected heterozygosity was 0.5294.The polymorphic information content(PIC)ranged from 0.3154 to 0.6545,with an average of 0.4687.Five of 8 loci showed significant deviations from Hardy-Weinberg equilibrium(HWE).Conclusions The results indicate that the genetic diversity of the Dunkin Hartley guinea pig outbred stock is moderate.Inbreeding has some shortcomings in comparison with outbred stock.Genetic information provided by this reserch will be useful in developing genetic monitoring methods and standardization of the outbred guinea pigs.
Guinea pig;Outbred colony;Microsatellite mark;Genetic diversity
Q95-33,R394
A
1005-4847(2011)01-0051-05
10.3969/j.issn.1005-4847.2011.01.012
豚鼠作为一种实验动物被用于生物医学研究已经有很长的历史。目前,在速发型过敏性呼吸道疾病研究、各种抗结核病药物的筛选、实验性坏血病研究、听觉和内耳疾病研究等方面,豚鼠都是最佳选择。
Dunkin Hartley豚鼠是英国种豚鼠的白化封闭群,因为其生长快、抗病力强,繁殖性能好,是我国使用最广泛的封闭群豚鼠。
浙江省科技厅实验动物公共服务平台项目(2008F80024)。
朱亮(1978-),女,助理研究员,博士,研究方向:动物分子遗传。E-mail:tozhuliang@126.com
2010-09-30

