生活垃圾堆肥过程中细菌群落演替规律
党秋玲,刘 驰,席北斗,魏自民,李鸣晓,杨天学,李 晔
1.东北农业大学生命科学院,黑龙江 哈尔滨 150030
2.中国环境科学研究院水环境系统工程研究室,北京 100012
生活垃圾堆肥过程中细菌群落演替规律
党秋玲1,2,刘 驰2,席北斗2,魏自民1*,李鸣晓1,杨天学2,李 晔1
1.东北农业大学生命科学院,黑龙江 哈尔滨 150030
2.中国环境科学研究院水环境系统工程研究室,北京 100012
应用PCR-DGGE技术研究生活垃圾堆肥过程中的细菌群落演替规律,对堆肥不同时期的宏基因组DNA进行提取,扩增16S rDNA的V3区,分析生活垃圾堆肥过程中细菌群落的变化.DGGE图谱表明,随着堆体温度的升高,DNA条带表现出了明显的动态变化,降温期出现了新的优势条带并趋于稳定,说明堆肥不同时期的细菌群落发生了更替.对条带分布进行聚类分析,结果表明:以55℃为界,将14个堆肥样品划分为2个族,族间的相似性仅为13%,说明堆肥过程中常温期(<55℃)和高温期(>55℃)微生物群落结构差别较大.对优势条带回收测序的结果表明:在升温期,堆肥堆体中检测到H.obtusa和人类排泄物中的细菌;但随着温度的升高,具有纤维素降解功能的嗜热微生物Clostridium thermocellum成为堆肥高温期的优势细菌;当堆体温度小于55℃时出现了大量的未培养微生物.
生活垃圾;堆肥;PCR;DGGE;细菌群落演替
Abstract:The PCR-DGGE technique was used to study the dynamic succession law of bacterial communities during composting of Municipal Solid Waste(MSW).The microbialmetagenom ic DNA was extracted from samples in different periods of composting,and the V3 region of 16S rDNA was amplified for analyzing the change of the bacterial community during the composting process. DGGE prints showed that the bacterial community changed dramatically with the rise of temperature.New predominant bacteria appeared at the end of the composting process.This indicated that the bacterial community changed in different composting periods.Clustering analysis results showed that 14 samples were divided into two families at 55℃.The similarity of the two families was only 13%,indicating that the bacterial community structure was different in the normal temperature process(<55℃)and high temperature process(>55℃),The results of DNA sequencing showed that:caterpillar pathogenH.obtusaand human waste samp le gene were detected in the rising temperature period;thermophilic microbeClostridium thermocellu,which can decompose cellulose,was the dominant group in the high-temperature composting;and,lots of uncultured bacterial appeared when the temperature was lower than 55℃.
Keywords:domestic waste;compost;PCR;DGGE;bacteria community succession
生活垃圾好氧堆肥是实现其资源化、无害化利用的一种重要方式,其本质是由群落结构演替非常迅速的微生物群体协同作用于堆肥基质的生理生化过程[1-3].对堆肥过程中微生物群落结构变化规律的研究是了解堆肥过程中微生物作用机理和为生物强化堆肥提供技术支撑的必要前提,然而传统的微生物分离培养方法不能很好地反映堆肥过程中的微生物多样性变化的本质[4].基于16S rDNA的聚合酶链式反应 -变性梯度凝胶电泳(PCR-DGGE)技术可通过图谱特征分析系统中微生物的群落变化规律,目前该技术已被广泛应用于固体废物、水体和土壤等研究中[5-8],但用于研究堆肥过程中微生物动态变化规律的较少.
笔者应用PCR-DGGE技术研究生活垃圾好氧堆肥过程中细菌群落演替规律,以期为堆肥过程微生物的作用机理及堆肥工艺参数的优化提供理论依据.
1 材料和方法
1.1 堆肥原料
将从北京市某生活垃圾综合处理厂取回的生活垃圾进行分拣,剔除难降解物质,如塑料(所占比例6.5%)、玻璃和砖石(所占比例4.2%)等,将可降解有机生活垃圾粉碎至粒径≤5 cm.将中国环境科学研究院收割的杂草晾干,剁碎至2~3 cm,用其调节堆肥碳氮比.有机成分的理化性质见表1.
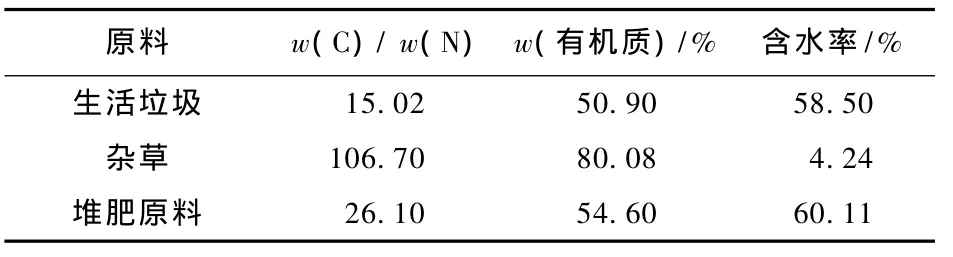
表1 堆肥原料基本理化参数Table 1 Some basic characteristics of composting materials
1.2 堆肥试验及样品采集
调节初始含水率为60.11%,碳氮比为26.1,pH为7.13,通风量为0.5 L/(min·kg).从堆肥开始每隔24 h分别在距堆心15,20和25 cm深度处取等量样品200 g混匀,平均为2份.其中一份样品用于测定实时pH和含水率等理化参数,另一份保存于-20℃冰箱用于后续分析.
1.3 PCR-DGGE分析
1.3.1 堆肥样品基因组DNA的提取和PCR扩增
堆肥样品经脱腐缓冲溶液洗涤去除腐殖质,用Omega土壤 DNA提取试剂盒进行总基因组DNA的提取,然后对16S rDNA的V3区进行PCR扩增. 扩增引物采用细菌通用引物 534r (ATTACCGCGGCTGCTGG)和 341 f(CCTACGG GAGGCAGCAG)[9],由上海生工合成,其中正向引物5'端连接有GC夹板(CGCCCGGGGCGCGCC CCGGGCGGGGCGGGGGCACGGGGGG),以提高在后期DGGE电泳时的解链范围[10].PCR扩增体系为:10μmol/L正反向引物各1μL,10×Taq Buffer 5μL,25 mmol/L的MgCl24μL,10 mmol/L的dNTP溶液1μL,2.5 U/μL的Taq DNA聚合酶0.5μL,模板DNA 2μL,无菌去离子水补足至50μL.扩增条件:采用降落式(touchdown)PCR,94℃预变性5 m in,94℃变性1 min,68℃退火30 s,每个循环降1℃,经过10个循环后,退火温度降至58℃,72℃延伸1 m in,94℃变性1 m in,58℃退火30 s,72℃延伸1 m in,25个循环,72℃终延伸10 min,最后4℃保温.PCR扩增产物采用1.5%琼脂糖凝胶电泳检测.
1.3.2 DGGE凝胶电泳及图谱分析
DGGE试验条件参考 YU等[11-12]的试验并进行了改进.采用Bio-Rad公司 DcodeTM的基因突变检测系统对PCR产物进行电泳分离.DGGE聚丙烯酰胺凝胶浓度为 8%,变性梯度为 35% ~60%,电泳缓冲液为1×TAE,电压80 V,60℃条件下电泳16 h.电泳结束后用SYBR GreenⅠ(1∶10 000)染色30 min,用UVP凝胶成像系统观察结果并拍照.
利用BIO-RAD QUANTITY ONE 4.6.2软件对所得 图谱进行处 理,用戴斯系数 (Dice Coefficient,CS)计算微生物群落的相似性:
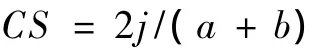
式中,j为样品A和B共有的条带;a和b分别为样品A和B中各自的条带数.戴斯系数为0(没有相同条带)~1(所有的条带相同).依据Complete Linkage算法对DGGE条带进行聚类分析.
1.3.3 优势条带的切胶回收和测序分析
用无菌的手术刀在345 nm波长下将优势条带切下,放在2 mL无菌离心管中,并用 tip头捣碎,加入60μL TE缓冲溶液,4℃放置24 h,取10 ng DNA进行第2轮PCR扩增,引物和扩增条件见1.3.1节,扩增产物交由北京三博远志公司测序.将测序结果提交到 Genebank中利用BLAST软件与Genebank数据库中的序列进行比对(http:// www.ncbi.nlm.nih.gov),获得同源性最大的菌属16S rDNA序列[13].
2 结果与分析
2.1 堆肥过程理化参数变化
图1为堆肥过程中的主要理化参数的动态变化.由图1可知,堆体温度在第2天就上升到54℃,并在50℃以上维持了10 d,最高温度达74℃,能有效杀灭堆肥中的致病菌;堆肥体系的pH始终呈弱碱性,最大值出现在高温期后期,堆肥结束时在7.5以下;堆肥的含水率始终维持在60%左右,可能是由于整个堆肥过程在3月进行,室温较低,反应器内外温差较大所致;w(有机质)由初始的 58.81% 降 到 32.31%;堆 肥 过 程 中w(水溶性有机碳)/w(水溶性有机氮)呈下降趋势,由初始的 34.7降到 5.4,李国学等[14]研究认为, 堆 肥 腐 熟 时w(水溶性有机碳)/w(水溶性有机氮)在4~6之间,该研究在第16天即降到了6以下,可以认为堆肥已经腐熟.
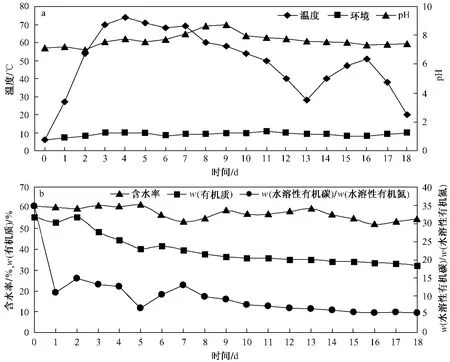
图1 堆肥理化参数变化Fig.1 The change of physical and chem ical parameters
2.2 堆肥样品基因组DNA的提取和PCR扩增结果
由于堆肥过程中会产生大量的腐殖质类物质,且腐殖质会严重影响DNA的提取和PCR的扩增效果[15],因此在用土壤DNA试剂盒提取前对样品进行了预处理,即分别用 PBS缓冲溶液和添加了PVP的脱腐缓冲溶液进行充分洗涤,直至样品溶液呈较清的颜色.如图2所示,经试剂盒提取后的基因组DNA片段大小在23 kb左右,无需纯化可直接进行PCR扩增.扩增产物片段在230 bp左右,经PCR产物纯化试剂盒纯化回收后的各样品均适宜进行后续DGGE分析.
2.3 DGGE指纹图谱分析
PCR-DGGE图谱中处于不同位置的DNA条带及其亮度的强弱代表细菌群落中某一特定微生物及其在群落中的相对丰度[16],条带数目越多表示细菌种类越丰富.如图3所示,随着堆肥的进行,堆肥样品中的细菌呈现出不同的条带分布.第1天的堆肥样品条带呈 smart状,表明堆肥初期细菌数量虽多但优势种群不明显,可能是由于堆肥初期的微生物多样性很高,导致PCR产物太少而不能形成明显的条带[17].随着堆肥温度的升高,出现了优势条带,并伴有优势条带的更迭现象.这说明在堆肥过程中微生物表现出了明显的动态变化.条带a,b,c和d只出现在图谱的前2天,且条带较亮,为升温期的优势种属,后期由于不适应堆肥的高温环境而逐渐在DGGE图谱上消失,条带e,f,g,h,i,j和 k一直出现在堆肥高温期(>55℃)的DGGE图谱上,较为清晰,说明该类微生物在此阶段数量较多,为高温期的优势微生物.降温期和二次发酵期又出现了新的细菌种群(如条带l,m,n和o),一直持续到堆肥结束.这些微生物的出现和持续存在,为 ISHII等[18]提出的以出现特征微生物作为堆肥腐熟标志的设想提供了试验依据.
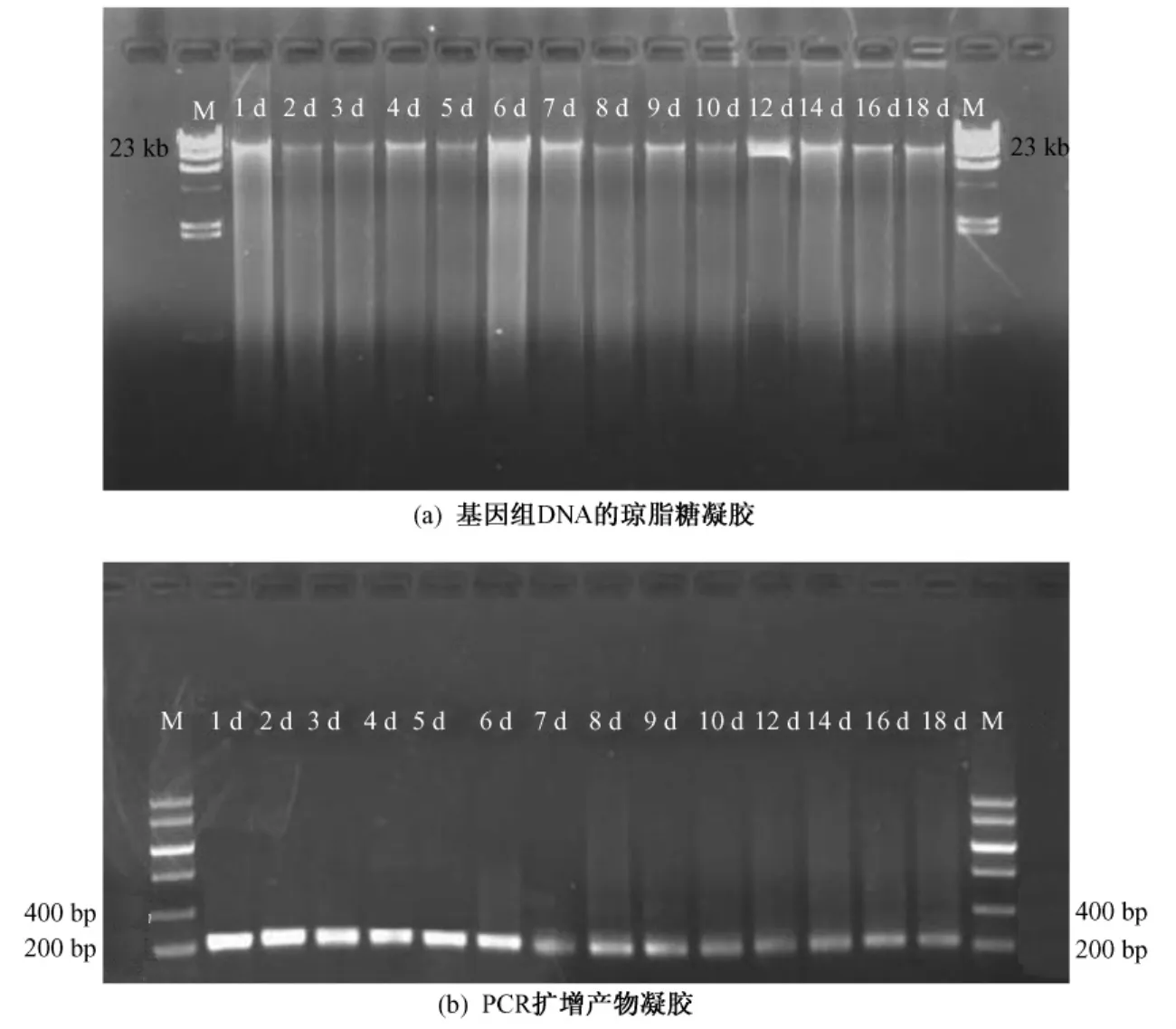
图2 基因组DNA的琼脂糖凝胶和PCR扩增产物凝胶电泳图像Fig.2 Agarose gel electrophoresis of genomic DNA of different samples and Agarose gel electrophoresis of PCR amplified 16SrDNA

图3 DGGE凝胶电泳图像Fig.3 DGGE patterns produced from different sample
2.4 DGGE图谱相似性分析
用Quantity one 4.6.2软件将DGGE图谱数字化后,按照 Complete Linkage算法进行聚类分析.由图4可知,依据堆体温度(55℃),将14个堆肥样品划分为2个族,族间的相似性仅为13%.表明温度变化对堆肥中微生物种群结构影响较大,可以将55℃作为堆肥微生物群落结构跃迁的一个标志性温度.
各时期堆肥样品中微生物 DNA序列的相似性见表2.表2中的数字表示各样品间的相似程度,数字越大表明其微生物种类和数量越接近,数字越小,说明其差别越大[19].从表2可知,不同时期的微生物种群相似性较低,升温期和高温期的相似性仅为18%(第1天和第10天),高温期和降温期的相似性仅为22.4%(第8天和第12天),高温期和二次发酵期的相似性仅为12.6%(第8天和第18天).说明好氧堆肥过程中,堆体内微生物的群落演替激烈,这可能和堆肥过程中堆体的温度区间跨度较大(6~74℃)有关,而每种微生物都有其生长的最适温度范围,超出这个范围其生长繁殖都将受到抑制,这也说明了通过高温好氧堆肥可以杀灭常温状态下存在于堆体内的病原微生物,实现生活垃圾无害化处理的目的.同一堆肥阶段的微生物种群表现出相对较好的相似性,其中堆肥第4天和第5天的相似性达到72%,说明当堆体温度变化不大时,对微生物的群落结构影响也较小.因此,温度变化对微生物的群落演替起到了重要的决定性作用.

图4 DGGE图谱的聚类分析Fig.4 The clustering analysis for DGGE patterns

表2 基于戴斯系数(CS)的相似性矩阵Table 2 Similarity matrix based on Dice cofficient for DGGE patterns
2.5 堆肥过程中优势细菌的测序分析
将图3中标记的DGGE条带回收测序后得到8个较好的结果,将该测序结果提交到 Genebank中进行比对.由表3可知,其中大部分为未培养细菌的16S rDNA V3区片段,其同源性均大于92%.与堆肥升温期(<55℃)条带b和d所代表的微生物种属同源性最大的细菌为H.obtusa和人类排泄物中细菌,最大相似性分别为95%和94%.H. obtusa是一种能够感染纤毛虫并在其体内寄生的G+病原菌[20],条带b和d所代表的微生物对复杂变化的堆肥条件适应能力较差,当温度高于55℃时就在DGGE图谱上消失了,说明堆肥高温期能有效地杀灭致病菌.条带 e所代表的微生物菌属一直是堆肥高温期(第3~12天)的优势细菌,与其同源性最大的细菌为Clostridium thermocellum,最大相似性为98%.Clostridium thermocellum是具有纤维素降解功能的嗜热细菌,说明该菌在堆肥高温期对生活垃圾中纤维素的降解起到了重要作用.解开治等[21]在猪粪堆肥过程中也检测到了该种细菌;在堆肥高温期的前期(第3~7天)出现了大量未培养的Bacillussp.和堆肥细菌,并成为该时期的优势菌群.Bacillussp.有木质素降解能力[22],表明在进入高温期已经有大量降解木质素的微生物存在.NIISAWA等[23]在研究海洋动物资源堆肥产品时也检测到了未培养堆肥细菌.腐熟期在堆体中出现了大量未培养的Clostridiumsp.和未培养的Firmicutesbacterium.
由于用于DGGE研究的细菌16S rDNA序列只有193 bp,在种属鉴定中只能鉴定到属,无法进行种的鉴定,因此,在今后的研究中还需要结合其他分子生物学方法对堆肥过程中微生物进行更全面的分析.

表3 条带序列的比对结果Table 3 Blast results of the band sequences
3 结论
a.经 PVP缓冲溶液洗涤过的堆肥样品可直接用土壤基因组DNA提取试剂盒进行有效提取,提取后的DNA可直接用于后续分析,且能得到较好效果.
b.生活垃圾好氧堆肥过程中细菌多样性较为丰富,堆肥不同阶段细菌群落演替较快,温度对微生物的群落演替起到了重要的限制作用,微生物和温度间的相互制约关系推进了堆肥的进程.
c.测序结果表明,在堆肥的升温期检测到了H.obtusa和人类排泄物中的细菌;在高温期检测到了纤维素降解菌Clostridium thermocellum,作为该时期的优势微生物,其对堆肥过程中纤维素的降解发挥了重要作用.在整个堆肥过程中检测到了大量的不可培养微生物,并作为堆肥腐熟期的优势菌群.
[1]牛俊玲,高军侠,李彦明,等.堆肥过程中的微生物研究进展[J].中国生态农业学报,2007,15(6):185-189.
[2]席北斗,刘鸿亮,孟伟,等.垃圾堆肥高效复合微生物菌剂的制备[J].环境科学研究,2003,16(2):58-64.
[3]胡春明,姚波,席北斗,等.堆肥复合功能菌剂的优化组合研究[J].环境科学研究,2010,23(8):1039-1043.
[4]杨朝辉,曾光明,蒋晓云,等.城市垃圾堆肥过程中的生物学问题研究[J].微生物学杂志,2005,25(3):57-61.
[5]CALLIA B,MERTOGLUA B,ROESTB K,et al.Comparison of long-term performances and final microbial compositions of anaerobic reactors treating land fill leachate[J].Bioresour Technol,2006,97(4):641-647.
[6]ZWART G,HUISMANS R,VAN AGTERVELD M P,et al. Divergentmembers of the bacterial division Verrucomicrobiales in a temperate freshwater lake[J].FEMS Microbiology Ecology,1998,25:159-169.
[7]PHILIPS S,VERSTRAETEW.Effect of repeated addition of nitrite to semi-continuous activated sludge reactors[J]. Bioresour Technol,2001,80(1):73-82.
[8]WANG G H,JIN J.Bacterial community structure in a mollisol under long-term natural restoration,cropping,and bare fallow history estimated by PCR-DGGE[J].Soil Science Society of China,2009,19(2):156-165.
[9]MUYZER G,WAAL E C,UITTERLINDEN A G.Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplfied genes coding for 16S rRNA[J].Appl Environ Microbiol,1993,59(3):695-700.
[10]NǔBEL U,ENGELEN B,FELSKE A,et al.Sequence heterogeneities of genes encoding 16S rRNAs in Paenibacillus polymyxa detected by temperature gradient gel electrophoresis[J].Bacteriol,1996,178(19):5636-5643.
[11]YU Z T,MARK M.Comparisons of different hypervariable regions of rrs genes for use in fingerprinting of microbial communities by PCR-denaturing gradient gel electrophoresis[J].Appl Environ M icrobiol,70(8):4800-4806.
[12]BONITO G, ISIKHUEMHEN O S, VILGALYS R. Identification of fungi associated with municipal compost using DNA-based techniques[J].Bioresource Technology,2010,101(3):1021-1027.
[13]刘振静,李璐滨,庄彩云,等.青藏铁路沿线土壤可培养微生物种群多样性分析[J].环境科学研究,2008,21(6): 176-181.
[14]李国学,黄懿梅,姜华.不同堆肥材料及引入外源微生物对高温堆肥腐熟度影响的研究[J].应用与环境生物学报,1999,5(S1):139-142.
[15]杨朝辉,肖勇,曾光明,等.用于分子生态学研究的堆肥DNA提取方法[J].环境科学,2006,27(8):1613-1617.
[16]MUYZER G,SMALLA K.App lication of Denaturing Gradient Gel Electrophoresis(DGGE)and Temperature Gradient Gel Electrophoresis(TGGE)in microbial ecology[J].Antonie van Leeuwen2hoek,1998,73(1):127-141.
[17]刘有胜,杨朝晖,曾光明,等.PCR-DGGE技术对城市餐厨垃圾堆肥中细菌种群结构分析[J].环境科学学报,2007,27(7):1151-1156.
[18]ISHII K,FUKUI M,TAKII S.Microbial succession during a composting process as evaluated by denaturing gradient gel electrophoresis analysis[J].JAppli Microbiol,2000,89(5): 768-777.
[19]谢冰,徐亚同.锌离子对活性污泥微生物DNA序列多样性的影响[J].环境科学研究,2003,16(4):18-31.
[20]HANSD G,NORBERT A,HORST R.Ultrastructure of the infectious and reproductive forms of holospora obtusa,a bacterium infecting the macronucleus of paramecium caudatum[J].Journal of General Microbiology,1989,135:3079-3085.
[21]解开治,徐培智,张发宝,等.接种微生物菌剂对猪粪堆肥过程中细菌群落多样性的影响[J].应用生态学报,2009,20(8):2012-2018.
[22]李凤,曾光明,范长征,等.农业有机废物与城市生活垃圾堆肥高温期微生物种群结构比较[J].微生物学通报,2009,36(11):1657-1663.
[23]NIISAWA C,OKA S,KODAMA H,et al.Microbial analysis of a composted product ofmarine animal resources and isolation of bacteria antagonistic to a plant pathogen from the compost[J].JGen App l Microbiol,2008,54(3):149-158.
Dynam ic Succession Law o f Bacterial Comm unities during Dom estic Waste Com posting
DANG Qiu-ling1,2,LIU Chi2,XIBei-dou2,WEI Zi-m in1,LIMing-xiao1,YANG Tian-xue2,LIYe1
1.College of Life,Northeast Agricultural University,Harbin 150030,China
2.Water Environment System Project Laboratory,Chinese Research Academy of Environmental Sciences,Beijing 100012,China
X705
A
1001-6929(2011)02-0236-07
2010-08-16
2010-10-22
国家自然科学基金项目(50878201);国家“十一五”科技支撑计划项目(2009BADC2B04,2006BAC06B04);农业科技成果转化资金项目(2008GB24420470)
党秋玲 (1982 -), 女, 黑 龙 江 克 山 人,dangling819@126.com.
*责任作者,魏自民(1970-),男,黑龙江依安人,教授,博士,博导,主要从事固体废弃物处理处置研究,weizm691120@163.com

