广西地区副猪嗜血杆菌ERIC-PCR指纹图谱分型研究*
彭 昊,李 军,谢宇舟,马春霞,禤雄标,胡 帅,杨 威,许力干,谢永平,陈泽祥,潘 艳
(广西壮族自治区兽医研究所,广西南宁 530001)
副猪嗜血杆菌病又名猪革拉泽氏病,是由副猪嗜血杆菌(Haemophilus parasuis,Hps)引起的一种以纤维素性多发性浆膜炎、关节炎和脑膜炎为典型特征的传染病[1]。本病呈世界性分布,在养猪发达国家都有此病的流行和发生,我国不少省市亦有该病报道[2-4],常与猪繁殖与呼吸综合征病毒、多杀性巴氏杆菌、支气管败血波氏杆菌等猪的呼吸道病原微生物混合或继发感染,使猪的呼吸道传染病更为复杂和严重,已成为重要的细菌性传染病,对养猪业的危害日益严重。由于副猪嗜血杆菌传统的检测方法是病原分离和生化鉴定,按照KRG血清分型方法,目前副猪嗜血杆菌可分为15个血清型,但大约15%~41%的菌株无法进行血清分型[5],且血清分型尚有不同血清型常存在严重的交叉反应、耗时、操作繁琐等问题,不利于流行病学规律的掌握,从而也阻碍了对该病的预防与治疗。应用分子生物学方法对副猪嗜血杆菌分型有助于克服传统分类方法的局限性。目前国内外学者[6-13]利用 ERIC-PCR 技术获取特异性的细菌DNA指纹图谱来进行基因分型,逐渐成为分型研究的热点。本研究通过对广西地区不同猪场分离的22株副猪嗜血杆菌进行ERICPCR指纹图谱分析,旨在为副猪嗜血杆菌分子流行病学研究提供一种实用性强、费用低的方法。
1 材料与方法
1.1 材料
1.1.1 阳性血清 副猪嗜血杆菌1~15型标准阳性血清为武汉科前动物生物制品有限责任公司产品。
1.1.2 培养基及试剂 胰蛋白胨大豆琼脂培养基(TSA)为中国检验检疫科学研究院产品;烟酰胺腺嘌呤二核苷酸(NAD)、新鲜特级马血清为北京索莱宝科技有限公司产品;肠道菌科细菌微量生化鉴定管为杭州天和生化仪器公司产品;PCR Taq mix、DNA Marker 3为广州东盛生物科技有限公司产品;细菌基因组DNA提取试剂盒为北京天根生物公司产品。
1.1.3 供试菌株 供试菌株分离自广西壮族自治区不同规模化猪场临诊发生以纤维素性多发性浆膜炎、关节炎为主要特征病变猪,具有“卫星生长现象”且不溶血、各种生化鉴定结果均符合副猪嗜血杆菌标准菌株特征。
1.2 方法
1.2.1 供试菌株血清型鉴定 以副猪嗜血杆菌1~15型标准阳性血清对这22株细菌进行血清型鉴定。
1.2.2 细菌DNA的提取 参照细菌基因组DNA提取试剂盒说明书进行。
1.2.3 引物设计与合成 参照文献[14]的方法,设计副猪嗜血杆菌ERIC-PCR引物。上游引物为:5'-ATGTAAGCTCCTGGGGATTCAC-3',下 游 引 物为:5'-AAGTAAGTGACTGGGGTGAGCG-3',由广州invitrogen生物工程有限公司合成。
1.2.4 22株副猪嗜血杆菌的ERIC-PCR鉴定PCR反应条件为95℃2min;94℃30s,50℃1 min,72℃2.5min,共进行35个循环;72℃10min。反应结束后取20μL产物进行20g/L琼脂糖凝胶电泳分析。
1.2.5 ERIC-PCR指纹图谱的聚类分析 对副猪嗜血杆菌的ERIC-PCR多态性电泳图谱进行扩增条带的数量和大小进行统计,有条带记为“1”,无条带记为“0”,在SPSS软件中,采用组间均联法进行聚类分析,绘出聚类树状图,以直观了解各菌株的亲缘关系。
2 结果
2.1 供试菌株血清型鉴定结果
对双株供试菌用血清学方法进行血清型鉴定,其中13株的血清型被确定,分别为血清2、4、5、9、10、11、13、14和15型,剩余9株供试菌株未被鉴定出血清型,结果见表1。
2.2 副猪嗜血杆菌ERIC-PCR扩增结果
22株副猪嗜血杆菌通过ERIC-PCR扩增后,产物的琼脂糖凝胶电泳图谱呈现出清晰可见的多态性条带,大小范围在200bp~2 500bp之间。重复性试验证实该方法具有良好的重复性。之前无法通过血清分型的菌株也得到充分分型 (图1)。
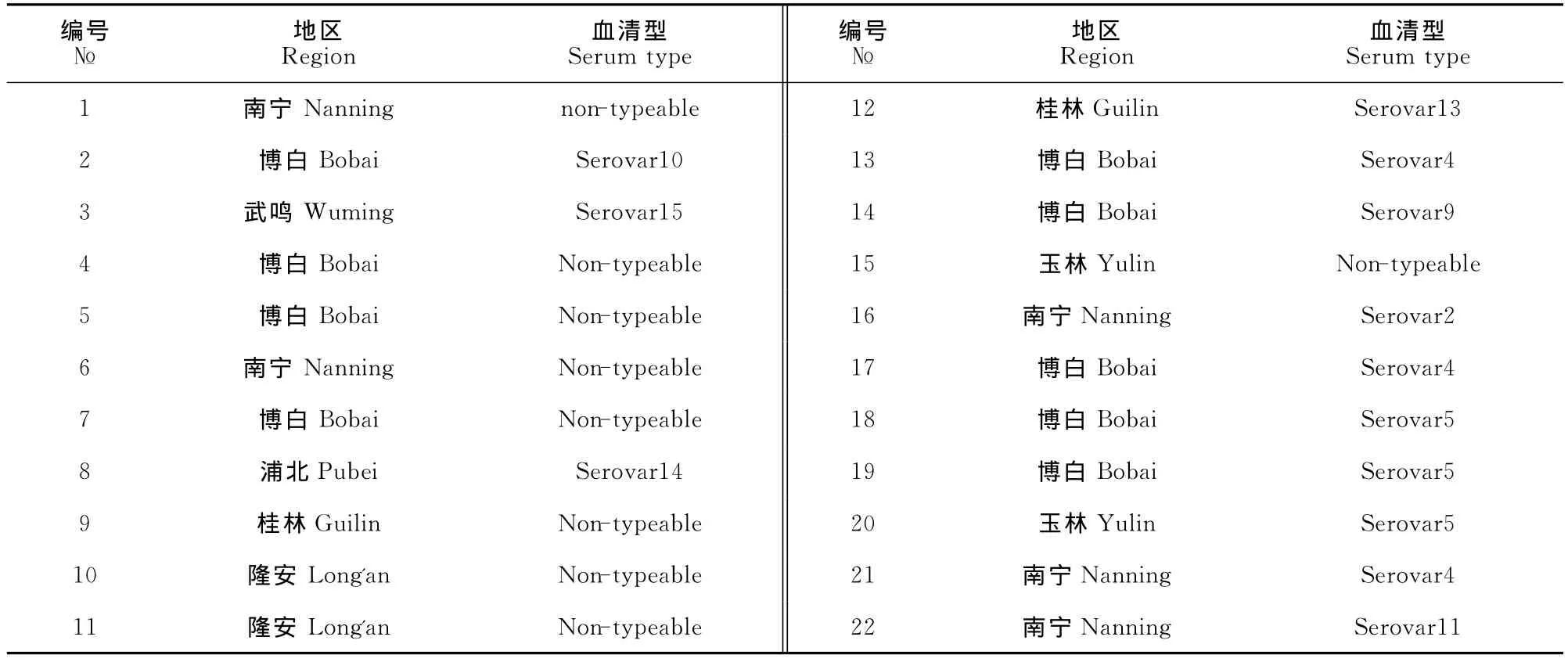
表1 副猪嗜血杆菌的血清型及来源Table 1 Serum type of Haemophilus parasuis and distribution in different regions
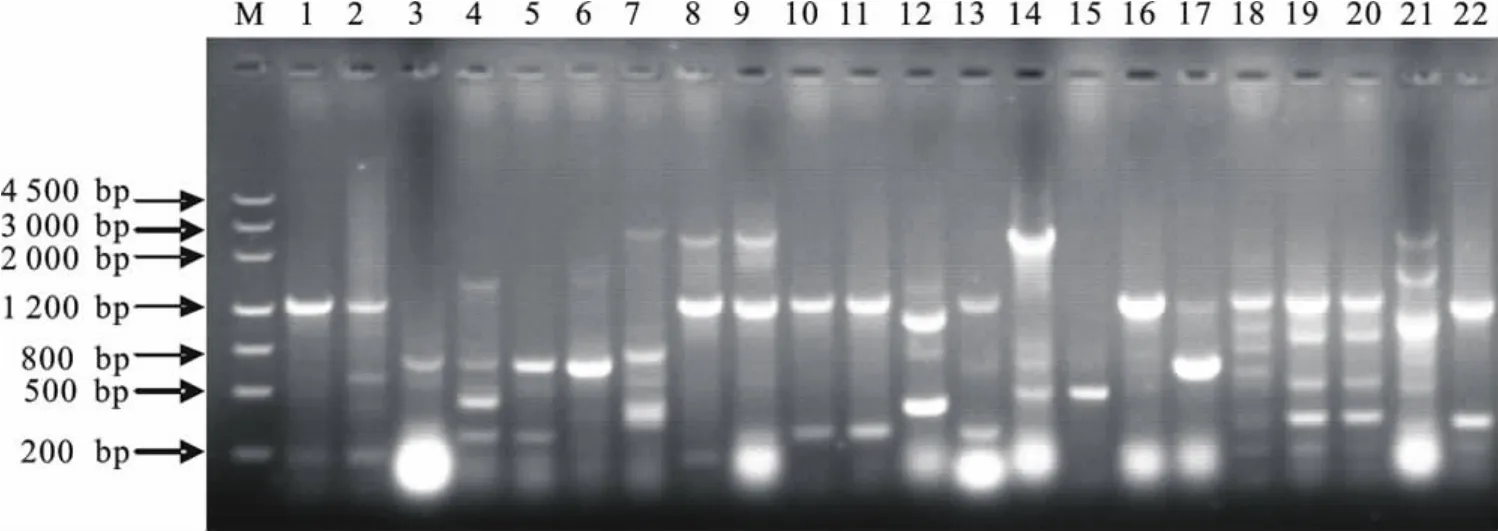
图1 22株副猪嗜血杆菌ERIC-PCR指纹图谱Fig.1 ERIC-PCR fingerprints of 22 Haemophilus parasuis isolates
2.3 电泳图谱的比较及数据分析
对每株副猪嗜血杆菌的ERIC-PCR多态性电泳图谱进行扩增条带的数量和大小统计后,有条带记为“1”,无条带记为“0”,在SPSS软件中,采用组间均联法进行聚类分析,绘出聚类分析树状图(图2)。由图可看出,22株供试菌株可分为12种谱型,谱型相似的菌株各自聚为一类,如其中第1号、第16号菌株ERIC-PCR图谱带型完全一致,聚为一类。而第18号菌株与其余各株差异较大,在较远的分支上,提示该菌株与其他菌株相比遗传背景差异较大,该病原与引起其他猪场发病的病原亲缘关系较远。
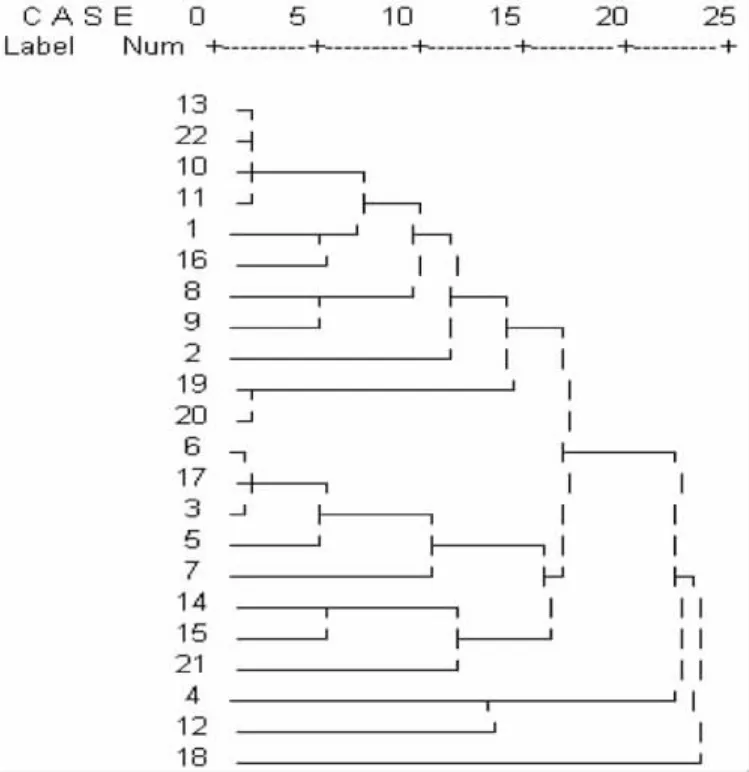
图2 22株副猪嗜血杆菌的ERIC-PCR聚类分析图Fig.2 Cluster analysis by ERIC-PCR fingerprinting of 22 Haemophilus parasuis isolates
3 讨论
Olvera A等[15]报道至少有15%~41% 的副猪嗜血杆菌不能用传统的血清分型法进行分型。本试验临床分离的22株副猪嗜血杆菌中仅有13个血清型被检出,未能分型菌株占总菌株数的40.91%。另外使用血清型分型方法需要具备15种标准血清,国内的实验室具有此条件的并不多。因此,用血清分型法对副猪嗜血杆菌进行分型存在很大的局限性。而ERIC-PCR技术一般分子生物学实验室均有条件进行,操作步骤较简便,分型速度快。
副猪嗜血杆菌间存在很高的遗传多样性,不仅存在于同一个血清型的不同菌株之间,而且存在于大量的不可分型菌株之间。相对于血清学分型,ERIC-PCR技术对菌株的描述和比较的区分能力更高[16]。本研究未发现血清分型与基因分型之间有明显的相关性,究其原因可能是因为血清分型的交叉反应未能消除,结果判定主要靠经验,主观性较大,因此试验数据的比对造成一定的影响。
ERIC-PCR为副猪嗜血杆菌的流行病学调查提供了一个快捷的工具,有利于快速判断出地区流行菌株及其传播情况,从而为防控疫病传播提供重要参考。从ERIC-PCR聚类分析图可见第10、11、13、22号菌株虽来自不同猪场,但带型完全一样,聚为一类,提示流行于这几个猪场的菌株亲缘关系非常相近,很有可能是同一种菌株,类似的还有第3、6、17号菌株等,疑似同种菌株。这为弄清副猪嗜血杆菌在地区间的传播流行情况提供了直观的信息,有助于了解猪场感染菌株的背景及传染源的推测和追溯,说明ERIC-PCR技术可以为副猪嗜血杆菌病的防控工作提供重要的辅助作用。
[1]赵德明,张中秋,沈建忠,译.猪病学[M].北京:中国农业大学出版社,2008:407-416.
[2]赵 萍,贺 英,储岳峰,等.江西省部分猪场副猪嗜血杆菌病血清学调查[J].动物医学进展,2009,30(12):107-109.
[3]仇微红,张盼锋,郭世宁.副猪嗜血杆菌的分离鉴定及中药药敏试验[J].动物医学进展,2009,30(2):53-55.
[4]陈金山,李任峰,马金友,等.副猪嗜血杆菌病的诊断和治疗[J].动物医学进展,2011,32(2):117-119.
[5]Kielstein P,Rapp-Gabrielson V J.Designation of 15serovars ofHaemophilusparasuison the basis of immunodiffusion using heatstable antigen extracts[J].J Clin Microbiol,1992,30:862-865.
[6]金莉莉,王秋雨,王 芳,等.食源性单核增生李斯特氏菌重复序列-PCR分型与鉴定研究[J].中国人兽共患病学报,2008,24(8):712-717.
[7]倪学勤,郑晓丽,曾 东,等.采用AFLP和ERIC-PCR技术研究规模化鸡场健康鸡群中产气荚膜梭菌的遗传多样性[J].畜牧兽医学报,2009,40(5):717-724.
[8]易明梅,朱建国,华修国,等.奶牛乳房炎样品金黄色葡萄球菌分离株的基因分型[J].畜牧与兽医,2010,42(2):62-65.
[9]何海建,陆国林,熊永忠.浙江地区副猪嗜血杆菌分离株的生物学特性[J].中国预防兽医学报,2010,32(12):941-944.
[10]Rafiee M,Bara M,Stephens C P,et al.Application of ERICPCR for the comparison of isolates ofHaemophilusparasuis[J].Australian Vet J,2000,78:846-849.
[11]Cao S Y,Wang M S,Cheng A C,et al.Comparative analysis of intestinal microbial community diversity between healthy and orally infected ducklings withSalmonellaenteritidisby ERIC-PCR[J].World J Gastroenterol,2008,14(7):1120-1125.
[12]Mondal K K ,Mani C.ERIC-PCR-generated genomic fingerprints and their relationship with pathogenic variability ofXanthomonascampestrispv.punicae,the incitant of bacterial blight of pomegranate[J].Current microbiol,2009,59(6):616-620.
[13]Nath G,Maurya P,Gulati A,et al.A new approach to variable metric algorithms ERIC PCR and RAPD based fingerprinting ofSalmonellatyphistrains isolated over a period of two decades[J].Infection Genetics Evolution,2010,10(4):530-536.
[14]Oliveira S,Blackall P J,Pijoan C.Characterization of the diversity ofHaemophilusparasuisfield isolates by use of serotyping and genotyping[J].Am J Vet Res,2003,64:435-442.
[15]Olvera A,Cerda-Cuellar M,Nofrarias M,et al.Dynamics ofHaemophilusparasuisgenotypes in a farm recorvered from an outbreak of Glasser's disease[J].Vet Microbiol,2007,123:230-237.
[16]Macedo N R,Oliveira S R,Lage A P,et al.ERIC-PCR genotyping ofHaemophilusparasuisisolates from Brazilian pigs[J].Vet J,2011,188(3):362-364.

