山核桃脂肪代谢相关cDNA文库的构建
黄银芝,周 秦,黄有军,曾燕如
(浙江农林大学 亚热带森林培育国家重点实验室培育基地,浙江 临安 311300)
植物油脂在植物生长、发育和繁衍过程中扮演着重要的角色。合成后的植物油脂主要有2个去向:一是用来构成生物膜的甘油脂和磷脂,另一个是储藏在种子中,即储藏态脂,常以甘油三酯(TAG)的形式存在。储藏态脂是一种封闭的相对惰性的光合产物,其主要成分的改变不影响植物总的分解功能,因此,植物油脂的生物合成途径特别适合遗传操作[1]。山核桃Carya cathayensis为浙江省重要的经济树种,其种仁的含油率平均达到70.0%,远远超过油菜Brassica campestris新品系 “超油2号”(52.8%)和 “世界油王”油棕Elaeis guineensis(55.0%)[2],为高含油量的树种,且其油质多为不饱和脂肪酸。因此,推测山核桃果实发育的过程中可能存在其特有的脂肪代谢机制。互补脱氧核糖核酸(cDNA)文库的构建基于基因表达过程产生的信使核糖核酸(mRNA)。通过构建cDNA文库能直接分离到生命过程中的一些调控基因,了解这些基因编码蛋白质的相互作用关系,估计大多数基因的表达水平,并发现一些组织特异的基因。本研究以糖类转变成油脂并积累时期的山核桃果实为材料构建cDNA文库,以期深入研究脂肪代谢的分子调控机制,挖掘和鉴定更多的脂肪代谢及调控的关键基因。
1 材料与方法
1.1 材料
cDNA文库构建用的山核桃果实采自山核桃产区浙江省临安市板桥乡罗塘村(30°N,119°E)1株约40年生正值盛果期的山核桃树,现场采集的果实置于液氮速冻后带回实验室-70℃保存备用。
1.2 方法
1.2.1 文库构建用果实采集时间的确定 生物体每时每刻都有大量不同的基因因生命活动的需要在不同的时间启动表达。要构建山核桃糖类转变成油脂相关的cDNA文库,首先要确定采样时间,以便在后续的研究中获得感兴趣的基因片段。周秦等[3]根据山核桃种仁中粗脂肪含量的变化规律,利用差异显示反转录-聚合酶链式反应(DDRT-PCR)方法对果实成熟期(8月1日至9月4日)基因的表达进行了分析,获得了30条与脂肪酸代谢相关的基因(包括油体钙蛋白、脂肪酶、脂肪酸去饱和酶、脂肪酰辅酶A结合蛋白和苹果酸合成酶)高度同源的cDNA片段,并发现这些片段多出现在8月11日至8月23日采集的样品中,推断这个时期是油脂形成、转化、积累的关键时期[4]。在此基础上,本研究以同年8月11日至8月23日共6次采集的果实样品为材料,构建cDNA文库。
1.2.2 mRNA的提取 周秦等[5]参考Laura等[6]提取总RNA的方法,在酸酚 ∶氯仿 ∶异戊醇(25∶24∶1)抽提、沉淀核酸的温度和时间、溶解初次沉淀物的方式等方面进行了改进。本研究采用周秦等改进的方法提取总RNA,提取的总RNA用 DNase I(RNase Free;TaKaRa)消化 DNA,并进行电泳检测。确认总RNA未降解后,将6次样品提取的总RNA等量混合。用mRNA Isolation Systems Kit(Promega)从混合的总RNA中分离mRNA,并进行电泳检测,用紫外分光光度计(ND-1000,NanoDrop)确定浓度。
1.2.3 文库的构建 以提取的mRNA为材料,用SMARTTMcDNA Library Construction Kit(Clontech)构建cDNA文库,但实际操作时有所不同。在构建文库过程中,分级后的双链cDNA先与λTriplEx2 Vector连接,然后参照周祥明[7]的方法,用聚合酶链式反应(PCR)法检测各连接混合物的连接效率,取200 bp处特异扩增条带信号最弱的连接产物用于噬菌体包装。文库的宿主菌为Escherichia coli‘HST08’。按照试剂盒说明书计算未扩增文库的滴度,并用试剂盒提供的测序引物 (5’sequencing primer TCCGAGATCTG GACGAGC/3’sequencing primer TAATACGACTCACTATAGGG)检测插入片段的大小。初级文库经扩增后的文库即为λ噬菌体文库。参照文库构建试剂盒的说明,将λTriplEx 2克隆转变成pTriplEx 2,以pTriplEx 2测序引物序列(5’sequencing primer TCCGAGATCTGGACGAGC/3’sequencing primer TAATACGACTCACTATAGGG)直接对含有pTriplEx2的宿主菌(大肠肝菌Escherichia coli)液进行PCR,检测文库插入片段的大小。
2 结果与分析
2.1 总RNA及mRNA的提取
提取的总RNA经电泳检测,28 s rRNA条带与18 s rRNA条带都很清楚,且亮度比例约为2∶1,而5 s rRNA条带亮度较暗,表明提取的总RNA完整性较好,不存在降解现象。经过基因组DNA消化后,可完全除去总RNA中的DNA污染,且总RNA也基本未发生降解(图1)。
高质量的mRNA是构建cDNA文库的基础。在不同的物种、不同组织mRNA弥散范围的大小有所不同[8]。本研究中对获得的mRNA电泳检测,发现其在0.5~2.0 kb呈均匀弥散状分布(图2),分光光度计检测其D(λ)260/D(λ)280=1.86,说明纯度较高,也即mRNA质量很好,可以用于后续cDNA文库的构建。
2.2 cDNA的合成与分级分离检测
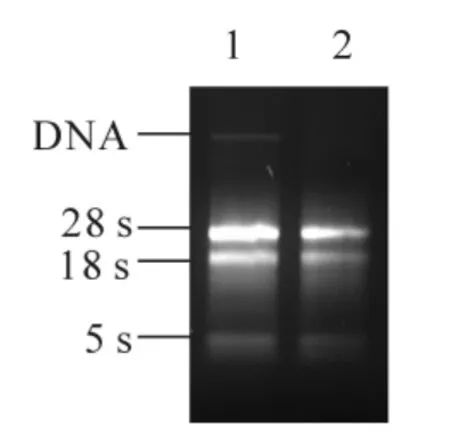
图1 总RNA电泳图泳道1:总RNA;泳道2:消化基因组DNA后的总RNAFigure1 Electrophoresis of the total RNA extractedlane 1:total RNA extracted;and lane 2:total RNA after DNA is digested
mRNA反转录后合成双链cDNA。双链cDNA在0.1~3.0 kb呈弥散状,集中分布在0.5~2.0 kb,与试剂盒所带的对照cDNA大小相近(图3)。cDNA集中弥散的范围与分离的mRNA相似,说明反转录较为成功,双链cDNA的质量能够满足构建cDNA文库的要求。
由于cDNA与载体连接的过程中往往小片段优先连接。为了得到高质量的cDNA文库,必须去除双链cDNA中的小片段。本研究中使用CHROMA SPIN-400对cDNA片段进行分级分离,舍弃400 bp以下的小片段。分级分离的结果电泳如图4所示,将图4中与泳道6,7,8相对应的分离产物合并后进行后续的操作。

图2 mRNA电泳图泳道1:mRNA;泳道2:λ-hindⅢ消化DNA标记Figure2 Electrophoresis of isolated mRNAlane 1:mRNA;and lane 2:λ-hindⅢ digest DNA marker

图3 双链cDNA电泳图泳道1:双链cDNA;泳道2:对照cDNA;泳道3:λ-hindⅢ DNA消化标记 (从下到上标记的大小依次为:125 bp,564 bp,2027 bp,2322 bp,4361 bp,6557 bp,9416 bp,23130 bp);泳道 4:100 bp 以上DNA梯度标记Figure3 Electrophoresis of ds cDNAslane 1:ds cDNA;lane 2:control cDNA;lane 3:λ-Hind Ⅲ digest DNA marker;and lane 4:100 bp plus DNA ladder marker
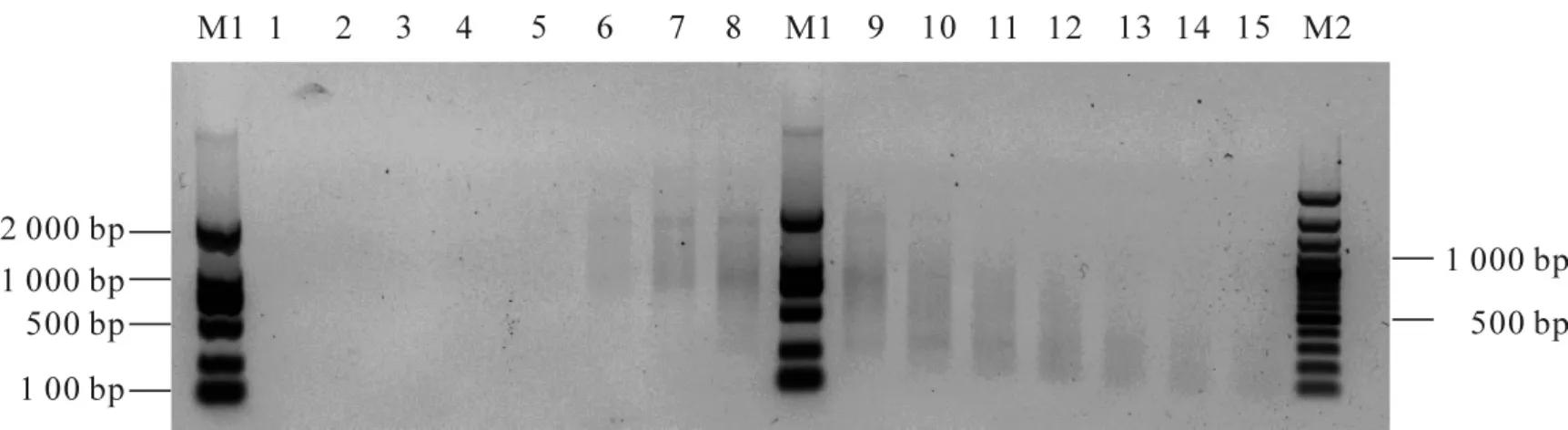
图4 双链cDNA大小片段的分级分离泳道M1:DL2000标记;泳道M2:100 bp以上DNA梯度标记;泳道1~15:双链cDNAFigure4 The ds cDNA size fractionslane M1:DL2000 marker;lane M2:100 bp plus DNA ladder marker;and lanes 1-15:cDNA
2.3 连接效率的检测
cDNA与载体λTripIEx 2连接时采用了3种cDNA:λTripIEx 2连接比例 (0.5∶1.0,1.0∶1.0,1.5∶1.0)。参照周祥明[7]的方法,用PCR法检测各连接混合物的重组率(图5)。取200 bp处特异扩增条带信号最弱的连接产物 (图5中的泳道3)用于噬菌体包装,因为200 bp处信号越弱,说明与载体连接的小片段越少,也即连接效率最好。因此,在本文库构建中采用1.5∶1.0的cDNA∶λTripIEx 2的连接比例。
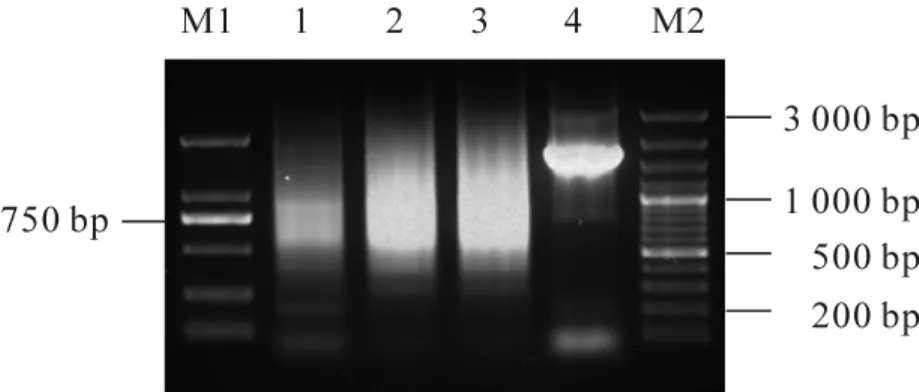
图5 ds cDNA与载体连接产物的电泳图泳道M1:DL2000标记;泳道M2:100 bp以上DNA梯度标记;1~3:连接混合物;4.对照Figure5 Electrophoresis of ds cDNA ligated with vectorslane M1:DL2000 marker;lane M2:100 bp plus DNA ladder marker;and lanes 1 - 3:mixed,ligated products;4.control
2.4 文库的质量检测
经检测,未扩增文库(或初级文库)的滴度为5.0×105个噬菌斑形成单位(pfu)·mL-1。随机挑取16个噬菌斑PCR检测插入片段的大小,结果显示cDNA插入片段大小都在500 bp以上,且70.0%左右的片段在1000 bp以上(图6)。
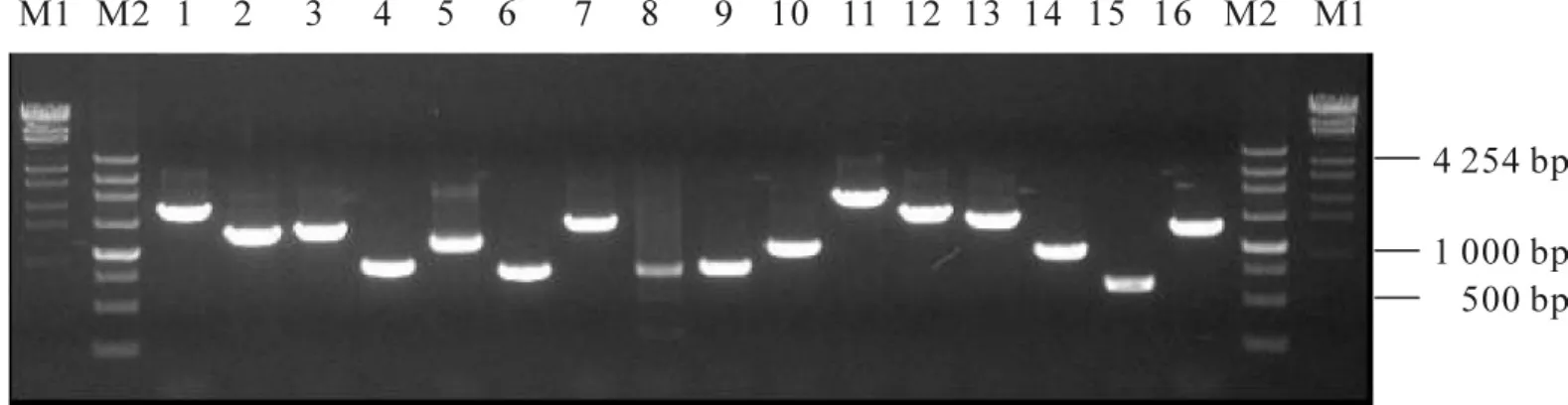
图6 未扩增文库中cDNA插入片段大小的电泳结果 (部分)泳道M1:λ-EcoT14Ⅰ标记 (从下到上标记的大小依次为74 bp,421 bp,925 bp,1489 bp,1882 bp,2690 bp,3472 bp,4254 bp,6223 bp,7743 bp,19329 bp);泳道M2:250 bp DNA梯度标记 (从下到上标记的大小依次为250 bp,500 bp,750 bp,1000 bp,1500 bp,2250 bp,3000 bp,4500 bp);泳道 1~16:噬菌斑扩增结果Figure6 Electrophoresis of cDNA inserts(partial)lane M1:λ-EcoT14Ⅰmarker;lane M2:250 bp DNA ladder marker;and lanes 1-16:amplified cDNA fragments
经检测,扩增文库的滴度为5.0×109噬菌斑形成单位(pfu)·mL-1。本文库在将λTripIEx 2转换为pTrip-IEx 2时采用的是大肠杆菌Escherichia coli BM25.8菌株,该菌株不能用蓝白斑筛选的方法判断重组子,只能用PCR的方法来操作[9]。随机挑取200个克隆进行PCR插入片段大小的检测,发现片段大小都在500 bp以上,最大的有2500 bp,平均大小在1000 bp左右,200个克隆中有11个空载,重组率达94.5%(图7),大于1000 bp的片段占66.0%左右,质量较好。
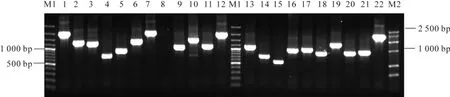
图7 扩增文库中cDNA插入片段大小的电泳检测(部分)泳道M1:100 bp以上DNA梯度标记;泳道M2:DNA标记G;泳道1~22:扩增文库中插入片段大小的随机检测Figure7 Electrophoresis of cDNA inserts (partial)lane M1:100 bp plus DNA ladder marker;lane M2:DNA marker G;and lanes 1-22:cDNA inserts
3 讨论
传统的cDNA文库构建方法用oligo(dT)或随机引物作逆转录引物合成cDNA,通过酶切削平末端,并连接到适当的载体[10-11]。SMART(switching mechanism at 5’ end of RNA transcript)技术是近年发展起来的一项利用较少mRNA甚至总RNA建立cDNA文库的方法[12-14],其主要原理是应用5’SMART引物得到全长cDNA,并利用长距离聚合酶链式反应(LD-PCR)特异扩增全长cDNA,排除了不完整的cDNA片断,并通过基因组内极其稀有的酶切位点SfiⅠ切割cDNA,得到接头并定向连接到载体中,大大提高了连接效率。同时,运用LD-PCR对cDNA进行合成,使得低丰度的基因表达序列在文库中得到了富集,便于稀有mRNA扩增和克隆,从而使库容量得到了保证。
构建高质量的cDNA文库,前提是提取并分离出高质量的mRNA,所获得mRNA越完整,种类越多,构建的cDNA文库就越完整,质量就越高。因此,提取纯度高、完整性好的mRNA就成为cDNA文库构建的核心步骤,直接关系到文库质量[15]。植物组织中RNA含量较低。因此,采集山核桃果实后用液氮速冻,并存放于-70℃,以保证RNA不降解;另外,在提取过程中严格防止RNA酶的污染,这是实验成败的关键,尤其是几个mRNA样品混合时。常用的mRNA的纯化方法有oligo(dT)-纤维素柱层析法、 oligo(dT)-纤维素液相离心法、 oligo(dT)-磁珠法[16]。本研究采用 oligo(dT)-磁珠法,分离得到的mRNA质量较好,是构建高质量的cDNA文库的一个重要因素。
此试剂盒利用长距离聚合酶链式反应(LD-PCR)特异扩增全长cDNA,所以控制LD-PCR的循环数对于cDNA的合成大小以及基因在文库中拷贝数分布很重要[17],适当的循环次数可以防止低拷贝数的基因丢失;同时也可以防止高拷贝数的基因过分放大,试验中应严格按照mRNA起始量,合理安排PCR循环数[18]。本实验中根据获得的mRNA的量(0.055 μg),选用了26个循环数,效果较好。
cDNA片段与载体连接之前除去过短的cDNA片段也非常重要,若不能有效地去除短cDNA片段,将会降低文库平均插入片段长度。本研究采用CHROMA SPIN-400将小于400 bp的片段去除,保证了文库中具有较大分子的cDNA,减少短片段优先连接的机率,提高长片段的连接效率。此外,我们将分级分离后的cDNA片段与载体λTripIEx 2按不同的比例分别进行连接,且取连接效率较好的进行文库包装。有研究表明,将cDNA片段分级分离,去除小片段后再与载体连接可以提高大片段克隆的比例[19]。因此,本研究建立的cDNA文库插入片段都大于500 bp,平均长度较长,大于1000 bp的片段占66.0%左右,质量较好。在文库插入片段大小检测方面,周秦等[4]曾就质粒双酶切、质粒PCR和含有质粒菌液的PCR等3种方法进行了比较,3种方法的结果一致。因此,在研究中可以直接用菌液PCR的方法来检测插入片段的大小,不用提取质粒,方便快捷。
山核桃脂肪代谢相关cDNA文库的成功构建,为后续基于cDNA文库的表达序列标签(EST)测序分析及单核苷酸多态性(SNP)与简单序列重复(SSR)标记开发奠定了良好的基础。
[1]邬贤梦,官春云,李栒,等.油菜脂肪酸品质改良的研究进展[J].作物研究,2003,17(3):152-158.WU Xianmeng,GUAN Chunyun,LI Xun,et al.Advances in breeding for improved fatty acids in rapeseed [J].Crop Res,2003,17 (3):152 - 158.
[2]黄坚钦,郑炳松,黄有军,等.木本油料植物山核桃新品种的选育[J].生物质化学工程,2006,40(12):178-181.HUANG Jianqin,ZHENG Bingsong,HUANG Youjun,et al.Selection and breeding of new varieties of the oil-bearing tree hickory (Carya cathayensis Sarg.)[J].Biom Chem Eng,2006,40 (12):178 - 181.
[3]解红恩,黄有军,薛霞铭,等.山核桃果实生长发育规律[J].浙江林学院学报,2008,25(4):527-531.XIE Hong’en,HUANG Youjun,XUE Xiaming,et al.Growth and development of the Carya cathayensis nut [J].J Zhejiang For Coll,2008,25 (4):527 - 531.
[4]周秦.山核桃果实成熟期间基因表达的初步分析[D].临安:浙江农林大学,2010.ZHOU Qin.Fruit Ripening-associated Gene Expression in Carya cathayensis [D].Lin’an:Zhejiang A&F University,2010.
[5]周秦,黄有军,曾燕如,等.山核桃胚和胚乳总RNA的提取与cDNA的合成[J].浙江林业科技,2009,29(1):36-39.ZHOU Qin,HUANG Youjun,ZENG Yanru,et al.Isolation of total RNA from embryo and endosperm of hickory nut and synthesis of cDNA [J].J Zhejiang For Sci Technol,2009,29 (1):36 - 39.
[6]LAURA J,ANNA M P,MINNA H.Isolation of high quality RNA from bilberry (Vaccinium myrtillus L.) fruit[J].Mol Biotechnol,2001,19:201-203.
[7]周祥明.美洲黑杨雄性花芽cDNA文库构建及花发育相关基因的克隆与鉴定[D].北京:北京林业大学,2007.ZHOU Xiangming.Construction of cDNA Library and Identification of Genes Involved in Floral Development in Male Floral Buds of Populus deltoids [D].Beijing:Beijing Forestry University,2007.
[8]PROMEGA.Technical Manual:PolyATtract® mRNA Isolation Systems [R].Madison:Promega,2009.
[9]CLONTECH.SMARTTM cDNA Library Construction Kit User Manual[R].San Francisco:Clontech,2009.
[10]SUZUKI Y,YOSHITOMO-NAKAGAWA K,MARUYAMA K,et al.Construction and characterization of a fulllength-enriched and a 5’-end-enriched cDNA library [J].Gene,1997,200 (1):149 - 156.
[11]HEALY V.RT-PCR in cDNA library construction [J].Methods Mol Biol,2002,193 (3):349 - 361.
[12]WELLENREUTHER R,SCHUPP I,POUSTKA A,et al.The German cDNA consortium SMART amplification combined with cDNA size fractionation in order to obtain large full-length clones [J].BMC Genomics,2004,36 (5):1-8.
[13]CHENCHIK A,MOQADAM F,SIEBERT P.A new method for full-length cDNA cloning by PCR [M]//KRIEG P A.A Laboratory Guide to RNA Isolation,Analysis and Synthesis.New York:Wiley-Liss,1996:273 - 32l.
[14]SEHULER G D.Pieces of the puzzle:expressed sequence tags and the catalog of human genes [J].J Mol Med,1997,75:694- 698.
[15]方花,魏小勇.铁皮石斛cDNA文库的构建及分析[J].生命科学研究,2005,9(3):263-266.FANG Hua,WEI Xiaoyong.Construction and analysis of a cDNA expression library of Dendrobium candidum [J].Life Sci Res,2005,9 (3):263 - 266.
[16]姚知行.分子生物学技术讲座(三)真核细胞mRNA的提取,纯化,分析及cDNA文库的构建[J].生物学通报,1997,32 (8):23-24.YAO Xingzhi.Lecture on molecular biotechnology(three):extraction,purification,analysis of eukaryotic mRNA and cDNA library construction [J].Bull Biol,1997,32 (8):23 - 24.
[17]朱金鑫,李小方.酵母双杂交技术及其在植物研究中的应用[J].植物生理学通讯,2004,40(2):235-240.ZHU Jinxin,LI Xiaofang.Yeast two-hybrid technology and its application in the research of plants [J].Plant Physiol Commun,2004,40 (2):235-240.
[18]方玉楷,许丽艳,麦瑞琴.酵母双杂交技术的影响因素及其实验策略[J].中国实验诊断学,2005,9(1):70-74.FANG Yukai,XU Liyan,MAI Ruiqin.Factors effecting on yeast two hybrid experiments and their responding strategies [J].Chin J Lab Diagn,2005,9 (1):70 - 74.
[19]WELLENREUTHER R,SCHUPP I,POUSTKA A,et al.The German cDNA consortium SMART amplification combined with cDNA size fractionation in order to obtain large full-length clones [J].BMC Genomics,2004,36 (5):1-8.

