盐胁迫下疏叶骆驼刺根基因表达差异及功能注释
赖黎丽,赵 洁,张 博,王 涛
(1.中国农业大学 农业生物技术国家重点实验室,北京 100193;2.新疆农业大学 草地资源与生态重点实验室,新疆 乌鲁木齐 830052)
盐分是影响植物生长和产量的一个重要环境因子,根据国内外研究资料显示,盐胁迫对植物的生长、水分关系、叶片解剖学、光合色素及蛋白、脂类、离子水平、抗氧化酶及抗氧化剂、氮素代谢、苹果酸盐代谢、叶绿体超微结构以及光合作用的机制等方面都有重要的影响[1]。植物的耐盐机制是一个非常复杂的问题,涉及到生理、生化和分子生物学等方面,各种途径可能是单独作用,也可能是协同作用[2]。随着功能基因组学的发展,植物的耐盐性研究已深入到耐盐相关基因的克隆、基因的结构分析以及基因表达特性等领域[3]。对于这些基因的表达方式及其在耐盐反应中的作用已逐步被了解,植物耐盐性是由多基因控制的数量性状。当植物受到盐胁迫后,一部分基因的表达量增加,协同完成胁迫的应答,这类基因称作盐诱导基因[4]。目前,大 量的盐诱导基因已经在拟南芥[5]、截型苜蓿[6,7]、烟草[8]、小麦[9]、水稻[10]中分离克隆,并用于遗传转化。
基因芯片技术既可以高敏度的定量、定性检测基因表达水平的差异,也能够提供某一基因或基因群的表达模式与其他基因表达模式之间关联的信息[11]。在小麦[12]和拟南芥[13]中,已经利用cDNA 微阵列大规模地分析盐胁迫相关基因表达谱。疏叶骆驼刺(Alhagi sparsifolia)是多年生豆科植物,具有很强的抗旱、抗霜冻、耐盐碱能力[13-15],与基因组序列测定的豆科模式植物截形苜蓿(Medicago truncatula,2n=16)染色体组相同[16],是理想的研究植物耐盐机制的材料。目前,利用微列阵研究疏叶骆驼刺耐盐相关基因表达谱的研究尚未见报道。试验利用Affymetrix公司的MedicagoGenome Array,检测疏叶骆驼刺在盐处理0h和24h根部的基因表达差异,寻找耐盐相关基因,为进一步克隆耐盐基因奠定基础。
1 材料和方法
1.1 实验材料和培养条件
实验以疏叶骆驼刺为材料,材料由新疆农业大学牧草育种与生产实验室提供。
将种子在4℃冰箱中放置2d进行低温春化,用98%的浓硫酸处理30min,用移液器将浓硫酸吸干,放在自来水下冲洗30min。在超净工作台上,用70%乙醇浸泡1min,然后用0.1%升汞溶液灭菌10min,无菌水清洗5次,放在铺上2层滤纸培养皿上,黑暗条件下,进行种子萌发。将萌发2d后长势一致的幼苗,用220mmol/L的NaCl溶液进行盐处理,做3个生物学重复。分别取胁迫时间为0h和24h的根为试验材料,液氮速冻后,保存于-80℃备用。
1.2 基因芯片分析
利用Affymetrix公司的MedicagoGenome Array,检测疏叶骆驼刺幼根在盐处理0h和24h的基因表达谱。截型苜蓿基因组芯片包括61 278个探针。基因芯片是由博奥生物有限公司生物检测实验室操作完成。
1.2.1 RNA提取和探针合成 植物总RNA用天根生化科技有限公司的植物总RNA提取试剂盒方法提取,反转录用Eukaryotic Poly-A RNA Control Kit试剂盒(Affymetrix)进行反转录,体外转录用 Message-AmpTMⅡ(Ambio)进行转录,同时合成生物素标记的cRNA。生物素标记的cRNA于94℃保温35min进行片段化。取2μL片段化产物用甲醛变性电泳检测片段化cRNA大小分布范围,经片段化的cRNA即为杂交用的探针。
1.2.2 芯片预杂及杂交 每张芯片注入250μL预杂交液,置于杂交炉中,45℃,60r/min旋转预杂交10 min,吸出预杂交液,注入相应体积澄清杂交液,将芯片平衡置于杂交炉中,45℃,60r/min旋转杂交16h。
1.2.3 芯片清洗染色及扫描 杂交结束后,吸净杂交液,注入与杂交液等体积的Wash Buffer A。利用Affymetrix公司的Fluidics Station 450进行芯片清洗染色,90min,将芯片放入扫描仪中进行扫描。
1.3 基因芯片检测数据的处理
利用Microarray sulteversion 5.0等软件对芯片扫描所得数据进行计算和处理。
2 结果与分析
2.1 基因芯片检测的质量分析
以盐处理0h和24h疏叶骆驼刺幼根为试验材料,与截型苜蓿基因芯片进行杂交,芯片中间的“+”字、周围的边框以及GenechipMedicago等阳性对照信号正常;看家基因信号值及3′/5′比值正常;平均背景值与噪音值较低(表1);0h和24h的Present Percent分别为8.4%和8.8%,数值正常。样品各项检测参数达到质控要求,达到实验目的。
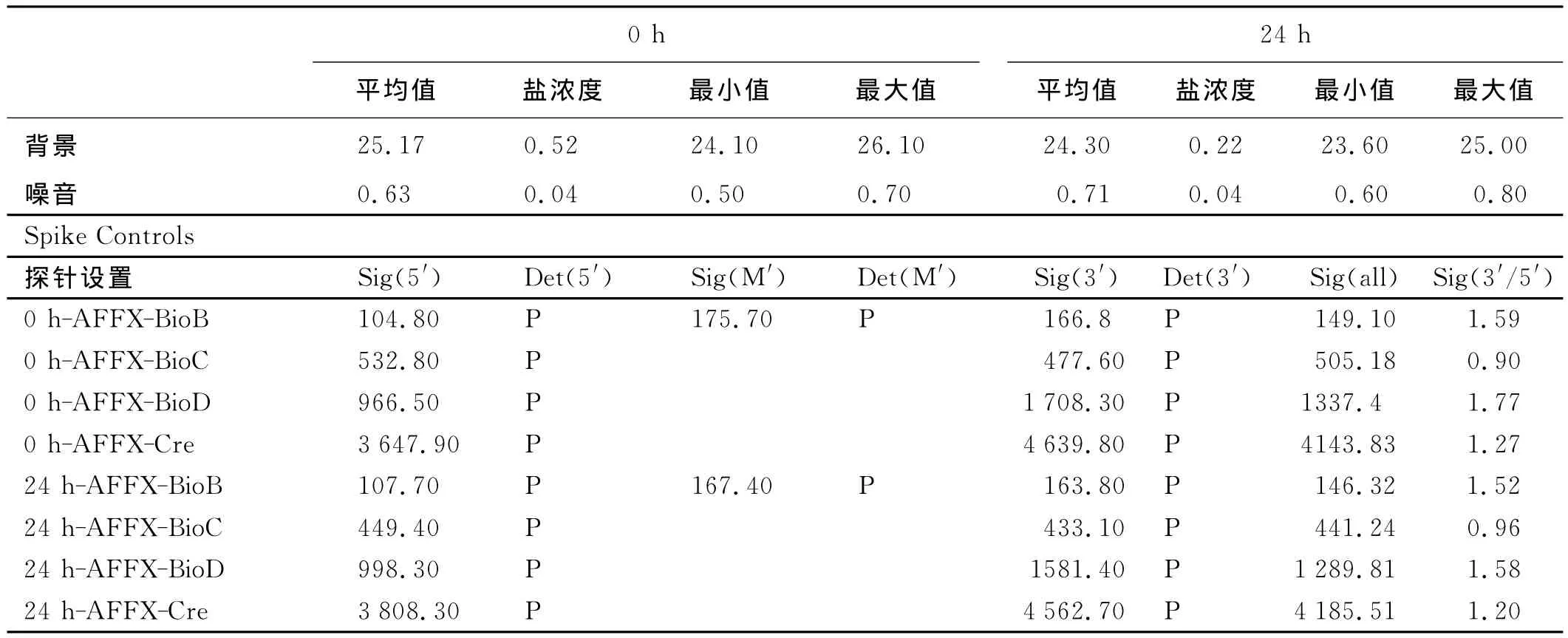
表1 质量检测分析报告Table 1 QC Report
2.2 基因芯片检测结果
利用Affymetrix公司提供的GCOS软件对0h和24h芯片的杂交检测结果进行了扫描并分析。结果显示,在61 278个探针中,0h检测到的有5 133个,占总探针数的8.4%,24h检测到的有5 378个,占总探针数的8.8%(表2)。
以0h样品为CK,对拟盐处理24h样品的差异基因进行检测,分别得到与之比较上调ratio值≥1差异基因127个,下调ratio值≤-1的差异基因119个,上调ratio值≥2倍的差异基因有3个。以截型苜蓿在180mmol/L NaCl溶液拟盐处理24h样品为CK,对疏叶骆驼刺拟盐处理24h样品的差异基因进行比较,共有相同的ratio值≥1和≤-1的差异表达基因50个。其中,在疏叶骆驼刺中表达上调的基因为33个,下调的基因为17个。

表2 疏叶骆驼刺0h和24hcRNA与截型苜蓿芯片杂交结果Table 2 Blotting results of Medicago Genome Array and cRNA of Alhagi sparsifolia under 0hand 24h
2.3 差异表达基因功能初步分析
按照Gene Ontology(GO)分类法则对ratio值≥1和≤-1的差异表达基因以及与截型苜蓿24h的交集差异表达基因在生化过程水平、分子功能水平两方面进行功能分析。(图1)
2.3.1 差异表达(ratio值≥1和≤-1)基因的功能分析 由图1可以看出,经盐胁迫后,在生化过程水平上,主要表达的差异基因参与新陈代谢和细胞壁形成。参与新陈代谢的上调基因43个,下调基因33个。参与细胞壁形成的上调基因44个,下调基因37个。在分子功能水平上,主要表达的差异基因具有催化活性和绑定活性。具有催化活性的上调基因45个,下调基因37个。具有绑定活性的上调基因34个,下调基因37个。参与细胞壁形成的上调基因50个,下调基因43个。参与细胞器形成是上调基因39个,下调基因31个。上下调一倍以上的基因还有63个未知功能的基因,有待进一步的研究。
2.3.2 上调一倍以上与截型苜蓿24h的交集差异表达基因功能分析 由表3中的推测基因经GO分析可知,经盐胁迫后,与信号转导相关的基因有4个,与物质能量运输相关的基因有9个,与新陈代谢相关的基因有5个,与逆境相关的基因有6个。


图1 差异基因GO分析Fig.1 Differently expressed genes based on GO analysis

表3 上调交集差异表达基因功能分析Table 3 Function annotation of intersect up-regulated expressed genes
上调最高的基因是亚铁血红素基因(Mhb1,Msa.878.1.S1_at),参与物质运输,编码非共生的血色素蛋白,在组织缺氧的情况下诱导表达,与CO信号传导路径相关[17]。富含脯氨酸蛋白基因(PRP2,Mtr.40069.1.S1_x_at)是PRPs家族成员,被假设为植物生长调控、导管分化、抗逆的分子构架[18]。甲酸脱氢酶基因(FDH,Mtr.37779.1.S1_at)属于 D-2-羟基酸脱氢酶类,可以将甲酸氧化为CO2,同时将NAD+还原为NADH,维持新陈代谢稳定进行[19,20]。
分子在质膜和液泡膜中的调节运输是植物响应非生物胁迫的主要特征[21]。由表4可知,具有运输功能的基因有7个,占下调基因的63.64%,主要是转运蛋白和质膜蛋白。转运蛋白在质膜和液泡膜上都存在,一般能够转运离子、有机大分子和小分子,保证植物体内的离子平衡,渗透平衡,物质交换与转运。运输功能的基因大量下调,说明疏叶骆驼刺幼根响应盐胁迫后,积累了大量渗透物质和能量。
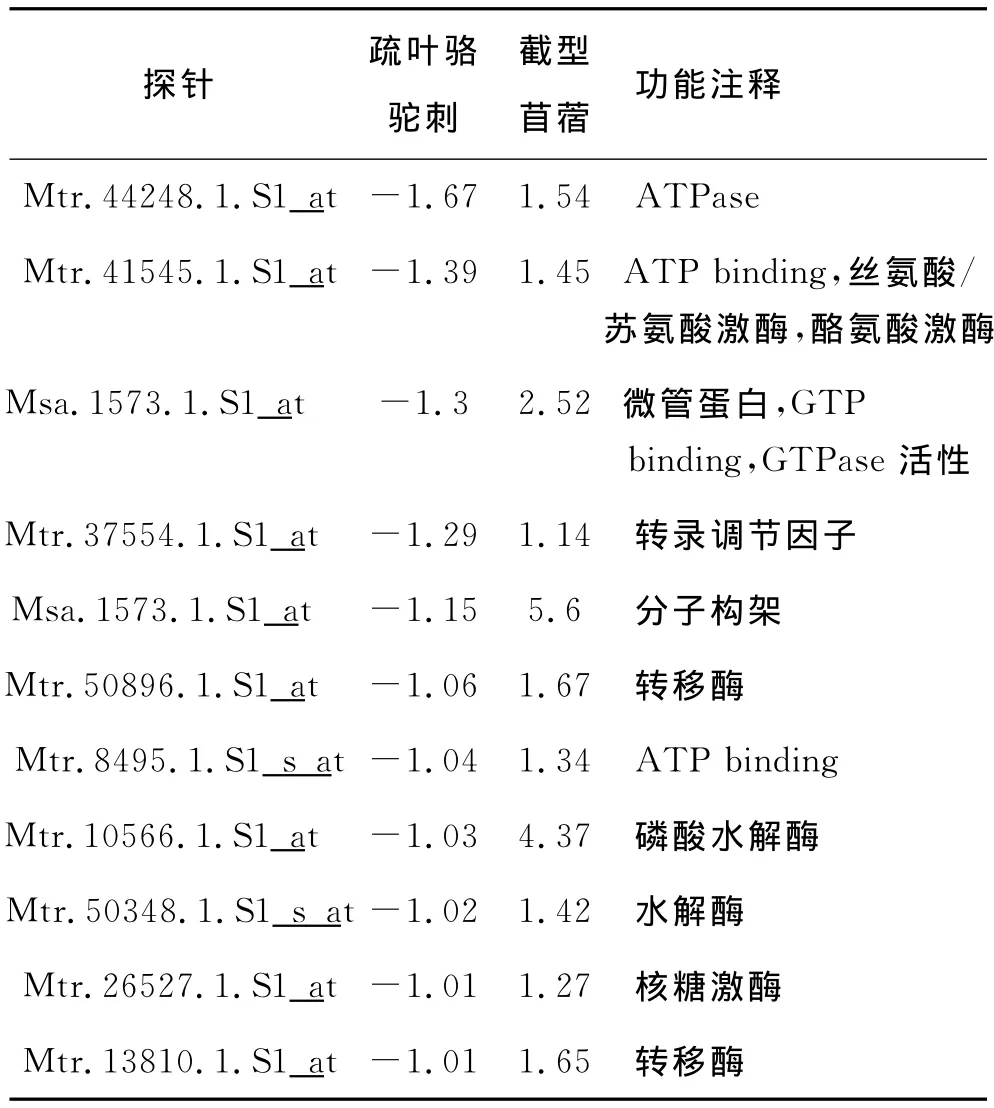
表4 下调交集差异表达基因功能分析Table 4 Function annotation of intersect down-regulated expressed genes
3 讨论
基因表达芯片的检测能力很大程度上取决于芯片上的探针数量、代表性和特异性[22],本研究采用Affymetrix公司的苜蓿基因组芯片,包括48 116个截型苜蓿、1 850个紫花苜蓿和8 226个苜蓿根瘤菌已知表达序列。Affymetrix公司芯片独特的PM-MM探针对复杂样品背景的检测具有高灵敏度和特异性,所获得的数据翔实可靠[23]。同时,在芯片杂交过程中质控严格,数据将更加可靠。盐胁迫后,检测到基因5 378个,占总探针数的8.8%,这可能因为探针序列与目标序列同源性低有关。现有研究认为,通过调整探针信号阈值范围可以解决这一问题[23],但疏叶骆驼刺与截型苜蓿亲缘关系较远,疏叶骆驼刺中或许存在特有基因,苜蓿基因芯片将无法检测到这些基因,建立疏叶骆驼刺基因组芯片将会解决这一问题。
通过GO分析了上下调差异表达基因,对其功能进行了分类,发现与信号转导相关的基因有4个,与物质能量运输相关的基因有16个,与新陈代谢相关的基因有5个,与逆境相关的基因有6个。其中非共生的血色素蛋白,在组织缺氧的情况下诱导表达,与CO信号传导路径相关;脯氨酸蛋白基因被假设为植物生长调控、导管分化、抗逆的分子构架;甲酸脱氢酶基因可以将甲酸氧化为CO2,同时将NAD+还原为NADH,维持新陈代谢稳定。此外,我们可以将这些基因在盐胁迫应答和耐盐反应中的作用利用分子生物学手段使其过量表达或降低表达加以验证,从中筛选出具有实际意义上的耐盐基因,为新型的耐盐转基因植物的研究奠定基础。
[1]杨少辉,季静.盐胁迫对植物影响的研究进展[J].分子植物育种,2006(3):139-142.
[2]Asish K P,Anath B D.Salt tolerance and salinity effects on plants a review[J].Ecotoxicology and Environment safety,2004,60:324-349.
[3]马雅琴,翁跃进,赵勇,等.植物耐盐相关基因克隆的研究进展[J].植物遗传资源学报,2004,5(1):81-86.
[4]赵宝存,赵芊,葛荣朝,等.利用基因芯片研究小麦耐盐突变体盐胁迫条件下基因的表达图谱[J].中国农业科学,2007,40(10):2355-2360.
[5]Hong S W,Jon J H,Kwak J M,et al.Identification of a receptor-like protein kinase gene rapidly induced by abscisic acid,dehydration,high salt,and cold treatments in Arabidopsis thaliana[J].Plant Physiol,1997,113:1203-1212.
[6]Analysis of regulatory pathways involved in the reacquisition of root growth after salt stress in Medicago truncatula[J].Plant J,2007,51:1-17.
[7]Gruber V.Identification of transcription factors involved in root apex responses to salt stress inMedicago truncatula[J].Mol Genet Genomics,2009,281:55-66.
[8]He P M,Zhang D B,Liang W Q,et al.Expression of choline oxidase gene(codA)enhances salt tolerance of tobacco[J].Acta Biochim Biophy sin,2001,33(5):519-524.
[9]Brini F.Cloning and characterization of a wheat vacuolar cation/proton antiporter and pyrophosphatase proton pump[J].Plant physiology and biotechnology,2005,43:347-354.
[10]Kawasaki S.Gene expression profiles during the initial phase of salt stress in rice[J].The Plant Cell,2001,13:889-905.
[11]李同祥.基因芯片与植物基因差异表达分析[J].植物研究,2002,3:310-313.
[12]Seki M,Narusaka:Monitoring the expression profiles of 7000Arabidopsis genes under drought,cold and high-salinity stresses using a full-length cDNA microarray[J].Plant Journal,2002,31(3):279-292.
[13]时永杰.疏叶骆驼刺[J].中兽医医药杂志,2003(增刊):144-145.
[14]曾凡江,张希明,李小明.骆驼刺植被及其资源保护与开发的意义[J].干旱区地理,2002,25(3):286-288.
[15]董志国.干旱荒漠区骆驼刺的经济价值及其利用[J].塔里木农垦大学学报,2000,12(3):58-60.
[16]司马义·巴拉提.两种国产骆驼刺的染色体核型分析[J].中国草地学报,1992,6:56-59,55.
[17]Seregēlyes C,Mrstárdy L,Ayaydin F.Nuclear localization of ahypoxia-inducible novel non-symbiotic hemoglobin in cultured alfalfa cells[M].FEBS Letters,2000:125-130.
[18]Robert C,Wilson.Characterization of PRPl and PRP2 fromMedicago truncatula[J].Plant Physiol,1994:105:445-446.
[19]黄志华.甲酸脱氢酶用于辅酶NADH再生的研究进展[J].过程工程学报,2006,6(6):1011-1016.
[20]Zhang Y P.Introduction of an NADH regeneration system intoKlebsiella oxytocaleads to an enhanced oxidative and reductive metabolism of glycerol[J].Metabolic Engineering,2009,11:101-106.
[21]Sahi C,Singh A,Blumwald E,et al.Beyond osmolytes and transporters:novel plant salt-stress tolerance-related genes from transcriptional profiling data[J].Physiologia Plantarum,2006,127(1):1-9.
[22]Wang J.Clustering of the SOM easily reveals distinct gene expression palIeres:Results ofa reanalysis of lymphoma study[J].BMC Bioinformatics,2002,3:36-44.
[23]Cheng L.Model-based analysis of oligonucleotide arrays:Expression index computation and outlier detection[J].PNAS,2001,98(1):31-36.

