野牛草种质基于SRAP标记的遗传多样性研究
周莹洁,王显国,张新全
(1.四川农业大学草业科学系,四川 雅安 625014;2.中国农业大学草地研究所,北京 100193)
野牛草(Buchloedactyloides)别名水牛草、牛毛草,禾本科画眉草亚科野牛草属多年生暖季型C4草本植物,原产于北美中部半干旱温带和亚热带地区,是北美干草原的特有草种[1]。由于它具有抗逆性强,易养护管理等优良的坪用特性,因此从最初主要被用作牧草,逐渐被用作园林绿化植物。20世纪40年代作为水土保持植物引种到我国,已在西北、华北及东北地区广泛种植。近年来,随着冷季型草坪草耗水量大、养护强度高等问题的突显,野牛草成为北方和缺水地区绿化及水土保持的首选草种。
至今,关于野牛草遗传多样性的研究多见于国外的报道。Huff等[2]用RAPD标记发现野牛草自然异性杂交群体内多样性大于群体间多样性。Zhang等[3]对野牛草遗传多样性RAPD分析得出10份野牛草种质相似系数分布范围为0.30~0.89。Budak等[4-5]利用SRAP标记分析53份野牛草基因型之间的遗传多样性及表型关系,以及不同倍性水平野牛草的遗传多样性,结果表明控制性状的基因型极易区分草坪草的品质和颜色,各倍性基因型之间的遗传距离为0.33~0.99,说明 SRAP标记是遗传多样性评价、鉴别特异基因型及从分子水平上分析野牛草栽培种的进化与培育的有效工具;同时还利用ISSR、SSR、RAPD和SRAP四种分子标记分析了种子繁殖类型和营养体繁殖类型野牛草之间的遗传关系,这些标记都可区分研究材料,其中SRAP标记多态性丰富,适合应用于野牛草近缘栽培种遗传多样性研究。刘莉等[6]利用ISSR分子标记和表型性状研究野牛草实生苗群体内的多样性,研究发现野牛草群体内的遗传多样性较高,相似系数为0.58~0.82,ISSR标记聚类结果与形态特征没有明确的对应关系。
相关序列扩增多态性 (sequence-related amplified polymorphism,SRAP)标记自从2001年开发以来,已被广泛应用于多种植物的图谱构建、基因定位、比较基因组学和遗传多样性分析等研究领域[7]。在草业科学领域中,已有关于鸭茅(Dactylisglomerata)[8]、狗牙根(Cynodondactylon)[9]和结缕草属[10]等植物SRAP标记遗传多样性的报道。
目前国内还未见有关野牛草SRAP遗传多样性研究的报道,本研究采用SRAP分子标记技术对来自31份国外野牛草种质资源进行分析,为野牛草新品种的培育提供理论依据。
1 材料与方法
1.1供试材料 供试材料为中国农业大学草业科学系收集的31份材料,包括28份野生材料和3个栽培品种。其中29份来自美国,1份来自加拿大,另1份来自日本(表1)。
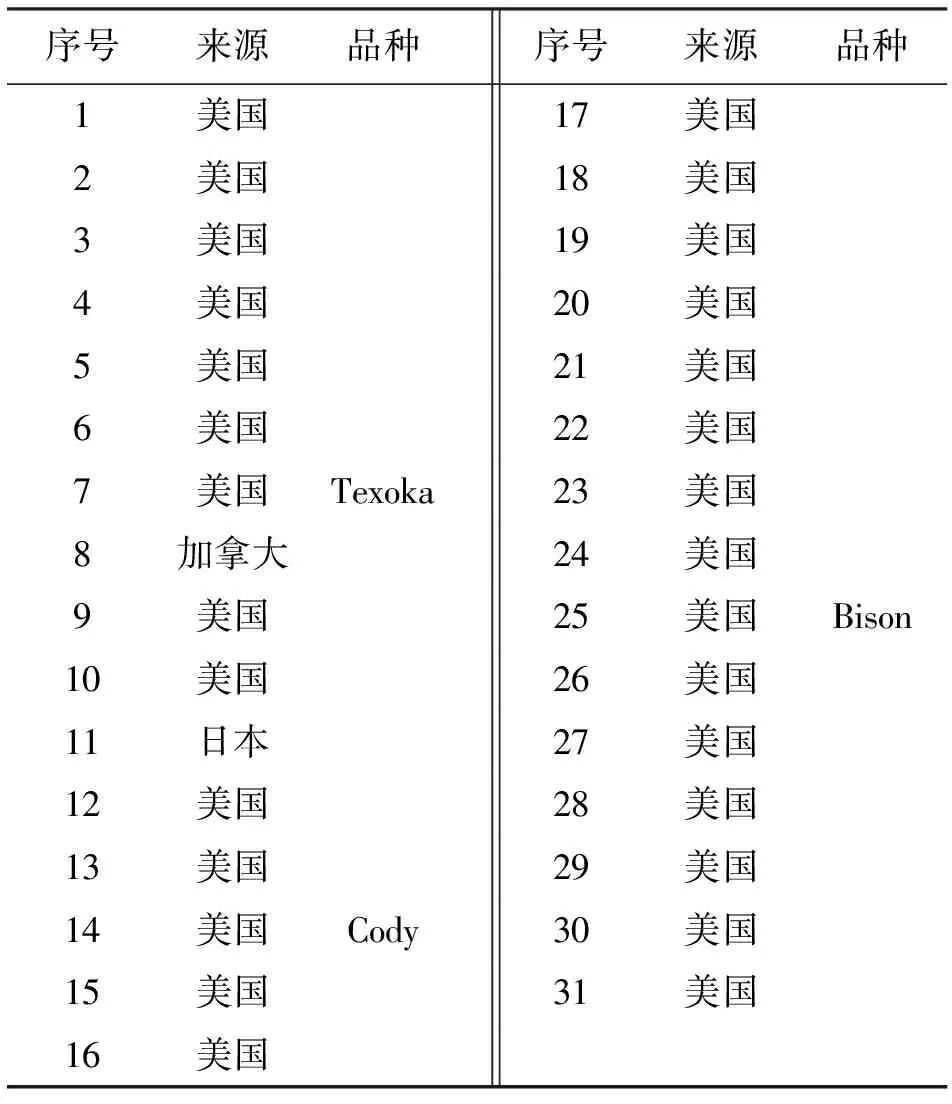
表1 供试材料
1.2基因组DNA提取与检测 采用CTAB方法提取DNA[11],提取的DNA储存于4 ℃冰箱中备用。在0.8%的琼脂糖凝胶上,1×TBE缓冲液条件下电泳,以已知含量的λDNA为对照,检测DNA的浓度[12-13]。
1.3PCR扩增体系 SRAP标记引物序列来自Li和Qurios[14]及Guo和Luo[15]的报道,12个上游引物和19个下游引物组合成228对引物序列,由上海生工生物公司合成。选取4个DNA质量较高且田间试验性状表现差异较大的材料对引物进行筛选,选出19对条带清晰,多态性好的引物组合用于野牛草SRAP遗传多样性研究,引物序列见表2。
反应总体积为15 μL,反应最终浓度:30 ng DNA,0.4 μmol/L引物,0.75 U Taq DNA 聚合酶,250 μmol/L dNTPs,1.5 mmol/L MgCl2。PCR扩增反应在Biometra PCR仪上进行,扩增程序为:94 ℃预变性4 min;94 ℃变性1 min,35 ℃退火1 min,72 ℃延伸1 min,5个循环;94 ℃变性1 min,55 ℃退火1 min,72 ℃延伸1 min,35个循环;72 ℃延伸10 min;4 ℃保存。
扩增产物用非变性聚丙烯酰胺凝胶(丙烯酰胺∶甲叉=39∶1,1×TBE)电泳分离。在200 V电压下电泳120~150 min,电泳结束后快速银染检测,凝胶用数码相机拍照并保存[16]。
1.4数据统计及分析 对清晰可重复的DNA条带进行统计,在相同迁移位置,有带记为1,无带记为0,不同引物扩增的结果构成原始的“0,1”二元数据矩阵。按Nei-Li[17]方法计算供试材料间遗传相似系数(GS,即Dice系数),GS=2Nij/(Ni+Nj),式中,Ni为材料i中出现的谱带数,Nj为材料j中出现的谱带数,Nij为材料i和材料j共有的谱带数。利用NTSYS-pc软件按Nei-Li遗传相似系数的非加权成对算术平均法(UPGMA)进行各种质材料间的聚类分析[18]。进行基于遗传相似系数的主成分分析(principal components analysis,PCA),依据引起变异的最大第1主向量、第2主向量作出各份种质材料的2-D散点分布图。

表2 用于野牛草SRAP分析的引物序列
2 结果与分析
2.1供试材料SRAP扩增产物的多态性 利用19对引物组合对31份野牛草基因组DNA进行SRAP扩增,共扩增出312条清晰的条带。其中多态性条带271条,多态性条带比率(percentage of polymorphic band,PPB)为85.79%,扩增DNA片段大小集中在50~1 500 bp。引物组合Me6-Em16扩增出最多的26条带,Me1-Em16扩增的条带最少,仅有7条。平均每对引物扩增出16.42条带,其中14.26条具有多态性,每对引物扩增的多态性位点百分率为69.23%~100%(表3)。
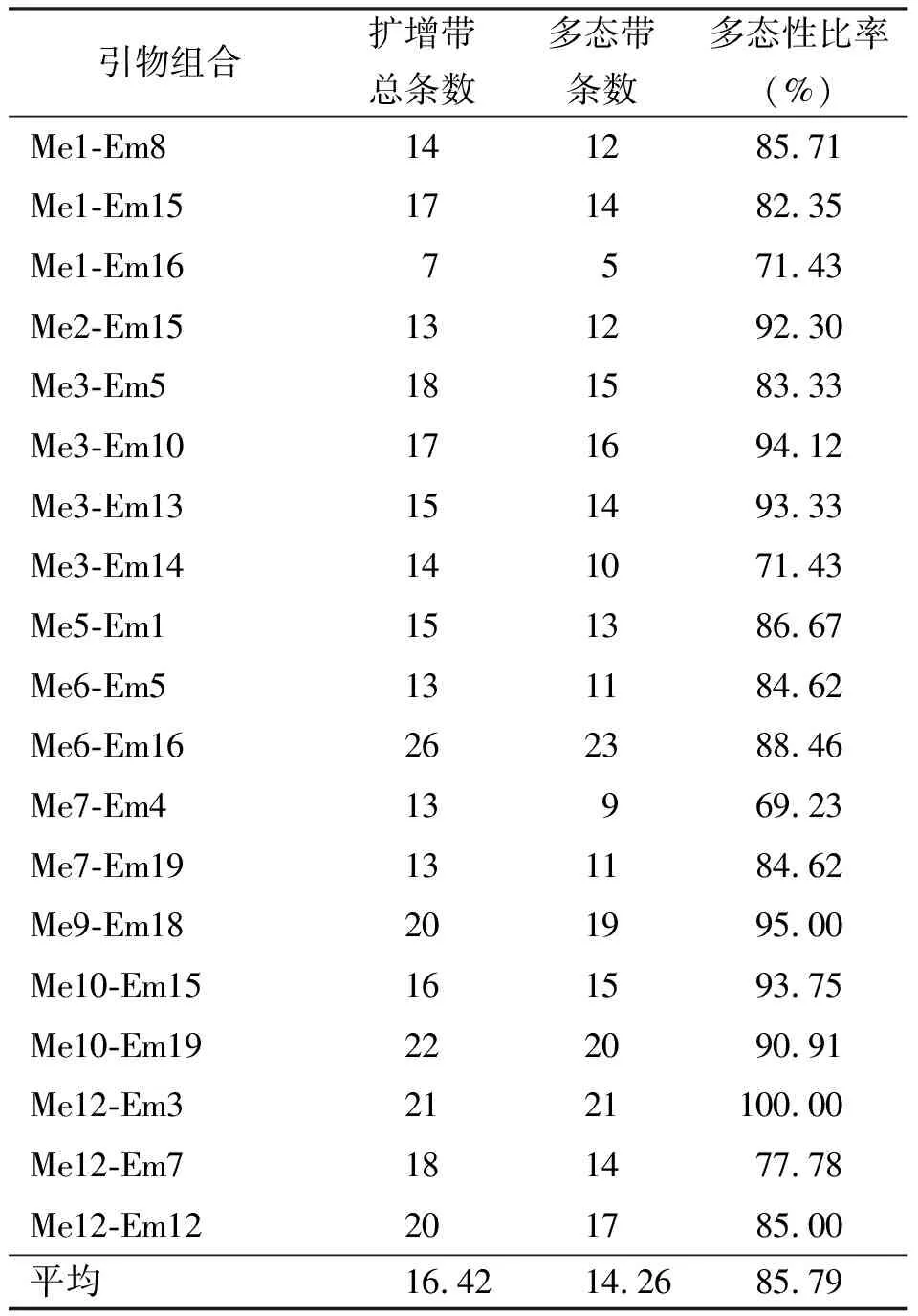
表3 SRAP标记引物扩增结果
2.2野牛草SRAP标记特异性缺失标记及特异性标记 在31份野牛草基因型中,15号野生材料在Me3-Em13引物组合扩增时缺失一条50 bp左右扩增带,可以作为15号材料种质鉴定的特异标记。在Me10-Em15扩增产物中,品种Texoka在50 bp附近出现特异性条带,其他材料都没有此条带,同时Me3-Em5对15号材料能扩增出400 bp处的特异带。这些特异性缺失标记和特异性标记都可以作为野牛草品种鉴定的重要依据。
2.3供试材料的遗传相似性 对扩增结果采用Nei-Li相似系数的计算方法,得到相似性矩阵。31份野牛草供试材料的GS变异范围为0.586 5~0.916 7,平均GS值为0.669 1。从遗传相似矩阵可以看出,27号和31号材料的遗传相似性最高,亲缘关系最近,18号和29号材料之间的遗传相似性最小,遗传距离最大表明亲缘关系最远。这些结果说明供试野牛草之间差异明显,具有较为丰富的遗传多样性。
2.4供试材料的聚类分析和主向量分析 基于Nei-Li相似系数,采用NTSYS-pc绘制出UPGMA聚类图(图1)。从聚类图可以看出,把31份材料聚为两大类,第一类共包括了25份材料,其中包括20份美国野生材料,3份栽培品种和1份加拿大和1份日本的野生材料,第二类包括了6份美国野生材料。同时,基于Nei-Li相似系数,利用NTSYS-pc对这些材料进行主向量分析,绘制出前两个主向量2-D散点分布图(图2)。从图2 可知,第一主向量和第二主向量分别能够接受总的遗传变异的21.98%和1.06%。主向量分析的结果和聚类分析结果基本一致,两类材料可明显的区分开来。
3 讨论
通过SRAP标记对31份野牛草材料进行PCR扩增,分析其遗传状况。本研究得到的多态性比率为85.79%。虽然低于此前Budak等[4-5]利用SRAP技术对野牛草遗传多样性研究的结果,但是高于ISSR和RAPD两个标记的结果。本研究利用16个引物组合共扩增出271条多态性带,平均每个引物组合扩增多态性带14.26条,说明SRAP对野牛草的扩增效率较高,可用于标记评价野牛草基因型的遗传分析,也适用于对大量种质的遗传变异检测。
由于SRAP标记的引物特点,此标记在重要基因性状标记的辅助选择上起到了重大的作用。Li等[19]在大白菜(Brassicacampestris)中发现与雄性不育基因有关的SRAP标记,并且筛选到与芹菜(Apiumgraveolens)病毒抗性基因紧密连锁的SRAP标记。本研究在对31份野牛草材料进行SRAP标记分析时发现,某些材料用特定的引物组合扩增以后,会出现一些特异性缺失的条带及特异性的条带,这些特异性缺失条带和特异性的条带可为野牛草的品种鉴定及亲本选择提供必要的理论依据。此外,野牛草是比较少见的雌雄异株禾本科草坪草,如能利用SRAP标记筛选到与其性别相关的基因,可为野牛草性别早期鉴定和揭示性别分化程序表达的机理提供理论基础和技术支持。
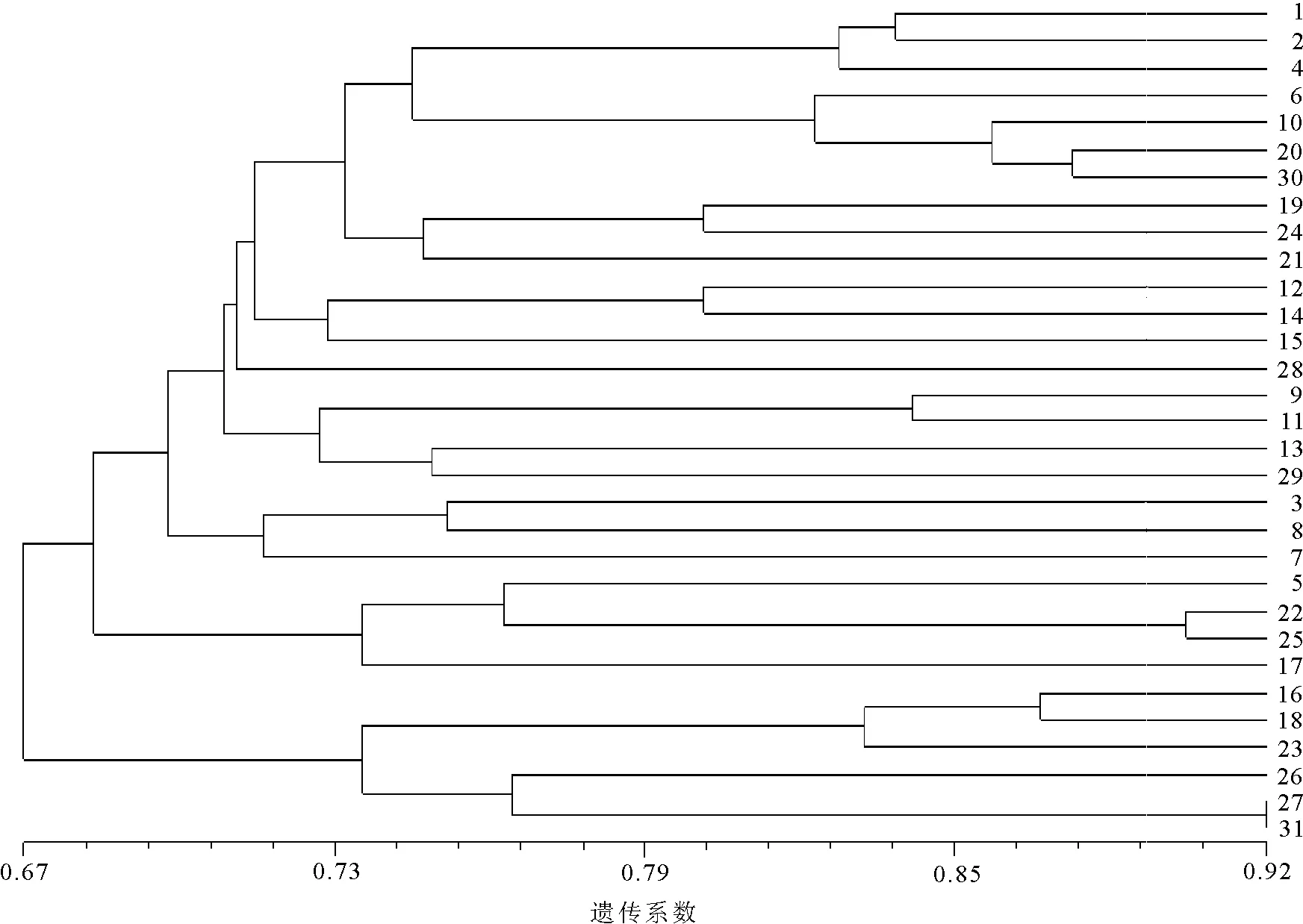
图1 31份野牛草材料基于Nei-Li相似系数的UPGMA聚类
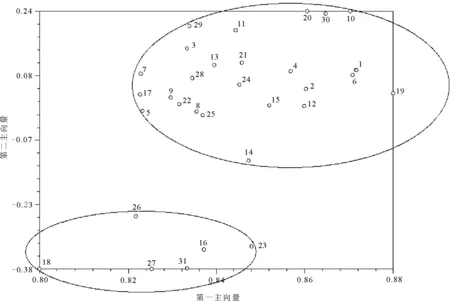
图2 基于SRAP 数据的野牛草材料主向量分析
31份材料中,27号和31号材料的遗传关系最近,18号和29号材料之间的遗传相似性最小,亲缘关系最远。从供试材料的聚类图和主向量分析可看出,地理来源相同的野牛草材料没有聚为一类,与预期相比,来自美国的29份材料并没有单独的聚在一类,不符合地理生态环境的分布。有较多关于牧草或草坪草遗传多样性的文献都报道相似生境或地理来源的类群会优先聚为一类[8,20-21]。本研究这种聚类不符合地域性分布规律的原因可能有以下几种:一是样本的数量有限,来自日本和加拿大的种质数量太少,各自只有一份,导致其特异带在总条带中的比例很小,可能对各群体遗传多样性指数的比较产生了影响,有研究[22]指出,在尽量多的起源地取代表性的种源进行研究才能得到更加准确的结果;二是与野牛草混合生殖方式和基因突变有关;三是这些材料都是在20世纪80和90年代引入中国,相同的生境条件产生了具有相同的选择压力,形成了相似的生态类型。
[1]Quinn J A,Engel J L.Life-history strategies and sex ratios for a cultivar and wild population ofBuchloedactyloides(Nutt.)Engelm [J].American Journal of Botany,1986,73(6):874-881.
[2]Huff D R,Peakall R,Smouse P E.RAPD variation within and among natural populations of outcrossing buffalograss (Buchloedactyloides(Nutt.) Engelm.)[J].Theoretical and Applied Genetics,1993,86(8):927-934.
[3]Zhang X B,Su D R,Ma L Y,etal.Analysis of genetic diversity in buffalograss determined by random amplified polymorphic DNA markers [J].HortScience,2007,42(3):474-477.
[4]Budak H,Shearman R C,Parmaksiz I,etal.Molecular characterization of buffalograss germplasm using sequence-related amplified polymorphism markers [J].Theoretical and Applied Genetics,2004,108(2):328-334.
[5]Budak H,Shearman R C,Parmaksiz I,etal.Comparative analysis of seeded and vegetative biotype buffalograsses based on phylogenetic relationship using ISSRs,SSRs,RAPDs,and SRAPs[J].Theoretical and Applied Genetics,2004,109:280-288.
[6]刘莉,邓春婷,包满珠.野牛草实生群体多样性的表型及ISSR分析[J].草业科学,2008,25(1):100-105.
[7]海燕,何宁,康明辉,等.新型分子标记SRAP及其应用[J].河南农业科学,2006(9):9-12.
[8]Zeng B,Zhang X Q,Lan Y,etal.Evaluation of genetic diversity and relationships in orchardgrass (DactylisglomerataL.) germplasm based on SRAP markers[J].Canadian Journal of Plant Science,2008,88(1):53-60.
[9]易杨杰,张新全,黄琳凯,等.野生狗牙根种质遗传多样性的SRAP研究[J].遗传,2008,30(1):94-100.
[10]郭海林,郑轶琦,陈宣,等.结缕草属植物种间关系和遗传多样性的SRAP标记分析[J].草业学报,2009,18(5):201-210.
[11]Saghai-Maroof M A,Soliman K M,Jorgensen R A,etal.Ribosomal DNA spacer-length polymorphisms in barely: ribosomal inheritance, chromosomal location, and population dynamics[J].Proceedings of the National Academy of Sciences of the United States of America,1984,81(24):8014-8018.
[12]黄智伟,黄琛.DNA检测技术研究现状[J].传感器世界,2001(1):10-14.
[13]解新明,云锦凤.植物遗传多样性及其检测方法[J].中国草地,2000(6):51-59.
[14]Li G,Qurios C F.Sequence-related amplified polymmphism (SRAP),a new marker system based on a simple PCR reaction:Its application to mapping and gene tagging in Brassica[J].Theoretical and Applied Genetics,2001,103:455-461.
[15]Guo D L,Luo Z R.Genetic relationships of some PCNA persimmons (DioispyroskakiThunb.) from China and Japanre-vealed by SRAP analysis [J].Genetic Resources and Crop Evolution,2005(1):1-7.
[16]赵培,王振英,彭永康,等.琼脂糖和聚丙烯酰胺凝胶电泳技术检测小麦基因组DNA RAPD扩增产物的方法学比较[J].中国生物工程杂志,2003,23(8):99-104.
[17]Nei M,Li W H.Mathematical model for studying genetic variation in terms of restriction endonucleases [J].Proceeding of the National Academy Sciences,1979,76:5269-5273.
[18]Rohlf F J.NTSYS-pc,Numerical Taxonomy and Multivariate Analysis System (Version 2.1)[M].New York:Exeter Software,2001:16-29.
[19]Li G,Gao M,Yang B,etal.Gene for gene alignment between theBrassicaandArabidopsisgenomes by direct transcriptome mapping[J].Theoretical and Applied Genetics,2003,107(1):168-180.
[20]凌瑶,张新全,齐晓芳,等.西南五省区及非洲野生狗牙根种质基于SRAP标记遗传多样性分析[J].草业学报,2010,19(2):196-203.
[21]鄢家俊,白史且,张新全,等.青藏高原老芒麦种质基于SRAP标记的遗传多样性研究[J].草业学报,2010,19(1):173-183.
[22]Wu Y Q,Taliaferro C M,Bai G H,etal.AFLP analysis ofCynodondactylon(L.) Pers.var.dactylongenetic variation[J].Genome,2004,47:689-696.

