木材形成的分子生物学研究1)——“多组学”在应力木系统中的应用
李坚 石江涛
(生物质材料科学与技术教育部重点实验室(东北林业大学),哈尔滨,150040)
木材具有复杂的生物学特征,并最终展现出优劣不易的表型与品质。经典的木材科学研究,主要集中在立木采伐后的加工利用方面,尽可能发挥木材的优良特性并抑制其缺陷;但是,整个过程都可以看作是一种后天的补救措施,不论是物理还是化学的处理方法都将给能源和环境带来不小的压力。木材行业自身,应该在低碳经济及人类可持续发展的生产实际中发挥积极作用。选择生物技术方法,可以从源头上改良木材品质,提高其在低碳经济中的效率。对于木材品质性状的生物技术改良,目前尚处于原始材料积累阶段[1]。要真正实现对木材品质进行遗传调控,必须将传统的木材科学研究方法与现代分子生物学方法相结合,把研究对象从传统的“死”的木材转变为有生命的“活”立木上来;重在考察林木群体性状的遗传变异,挖掘木质部发育中的关键基因,探索木材形成的分子网络调节结构,掌握木材形成的分子调控基础,指导木材品质的定向遗传改良。
1 木材形成的生物学过程
木材形成过程,可以看作是发育生物学上的木质部发生过程[2]。该过程是一个有机的、复杂的生物学过程,是树木将空气中的CO2和水在光能及叶绿素条件下转化成碳水化合物,并通过各种代谢途径,将每种成分组装在一起而形成具有特殊结构和功能的天然高分子材料。木材形成,起源于维管形成层,依次经过细胞分裂、细胞增大和伸长、次生壁形成、木质化、程序性死亡和心材形成生命活动循环而生[3]。木材形成,是一系列复杂的遗传和生理因素的交互作用的结果。木质部发生过程中,各个不同阶段在分子水平上,都是在特定的时空条件下将细胞内的基因组群有序表达。所有的生命过程,都是由相应的一群基因编码的程序所控制。同一植物体,每一个细胞,都拥有相同的遗传基因。它们之所以能发育成结构和功能各异的组织或器官,是由于某些基因得以表达,另一些基因受到抑制而没有表达。而树木生长产生不同结构性质的木材组织,最终表现出多变的木材特征性质。这种可塑性,有可能是由木材形成中特异基因或蛋白质的差异表达引起的[4]。
探讨处于不同生理、病理条件下的有机体之间的未知基因的差异表达,是研究生命本质的有效途径。应力木,是树木中的非正常组织,表现在树木横切面上特殊偏心的偏宽部分。通常是指,外部环境或条件的改变诱导形成的弯曲或倾斜的树干,试图恢复原来垂直高生长或维持树枝的自然角度方向,从而形成的在解剖(图1)、化学组成及物理力学性质明显不同的木材[4-7]。阔叶树材偏心横切面的宽边,位于倾斜树干的上侧或受拉一侧,称为应拉木;针叶树材偏心横切面的宽边,位于倾斜树干的下侧或受压一侧,称为应压木[8]。针叶材和阔叶材在次生木质部结构和性质存在很多差异,所以要对两类木材形成途径进行同步研究。杨属和桉属作为阔叶树基因改良研究的模式树种[9-10],而松属为针叶树的模式树种[11]。相对于阔叶树,针叶树有较大的基因组碱基数(10~40 Gb)和较长的生长周期,这些因素限制了其分子遗传分析和育种发展[12]。又由于裸子植物和被子植物进化距离较远,阻碍了两种基因资源的比对和共享。应力木系统,不仅是研究木材构造与性质关系、细胞壁化学组成和构建、木材性质与培育及加工利用关系的最佳实验材料,同时也是研究木质部发育形成的最佳实验系统。
2 分子生物学基础知识
细胞是构成所有生命体的基本单元。植物的生活细胞有细胞壁、细胞质和细胞核组成。细胞核包含了主要的染色体,携带着植物发育和生长所必需的遗传信息。染色体是成对存在的,称为同源染色体对。同一种物种正常个体中正常同类型细胞都具有相同数目的染色体数。如:花旗松有13对,26条;杉木有11对,22条[13]。染色体上的遗传物质是DNA,它是由碱基,脱氧核糖和磷酸组成。带有遗传信息的DNA片段称为基因,是遗传的基本功能单位。遗传信息包含在DNA序列绘制的“天书”里,它又是如何被生命体读懂,并和外界环境相协调的呢?基因将遗传信息从DNA传递给RNA,再从RNA传递给蛋白质,即完成遗传信息的转录和翻译过程。遗传物质在DNA和RNA之间可以可逆的传递,但是只能从RNA单向传递给蛋白质。这就是所有具有细胞结构的生物所遵循的中心法则。它是现代生物学中最重要的基本规律之一,在探索生命现象的本质及普遍规律方面起着巨大的作用[14]。蛋白质是生命的物质基础,没有蛋白质就没有生命。它是与生命及各种形式的生命活动紧密联系在一起的物质,它几乎参与了所有的生命活动和反应。蛋白质是一种复杂的有机化合物,其组成的基本单元是氨基酸。氨基酸序列完全是由细胞核内对应基因编码的。双链DNA分子将存贮的遗传信息转录成信使核糖核酸(mRNA),后者移动并透过核膜,到达细胞质中的核糖体,在转运核糖核酸(tRNA)调控下翻译转录的遗传信息合成蛋白质。蛋白质链的生成经过起始、延伸和终止,在每个阶段都有特定的三联密码子。比如:AUG是起始密码子,UAG、UAA、UGA是终止密码子。总之,细胞核内的DNA序列是遗传信息的贮存载体,其严格遵循半保留复制法则和中心法则,将遗传信息转录翻译,最终产生形态迥异的万千物种。
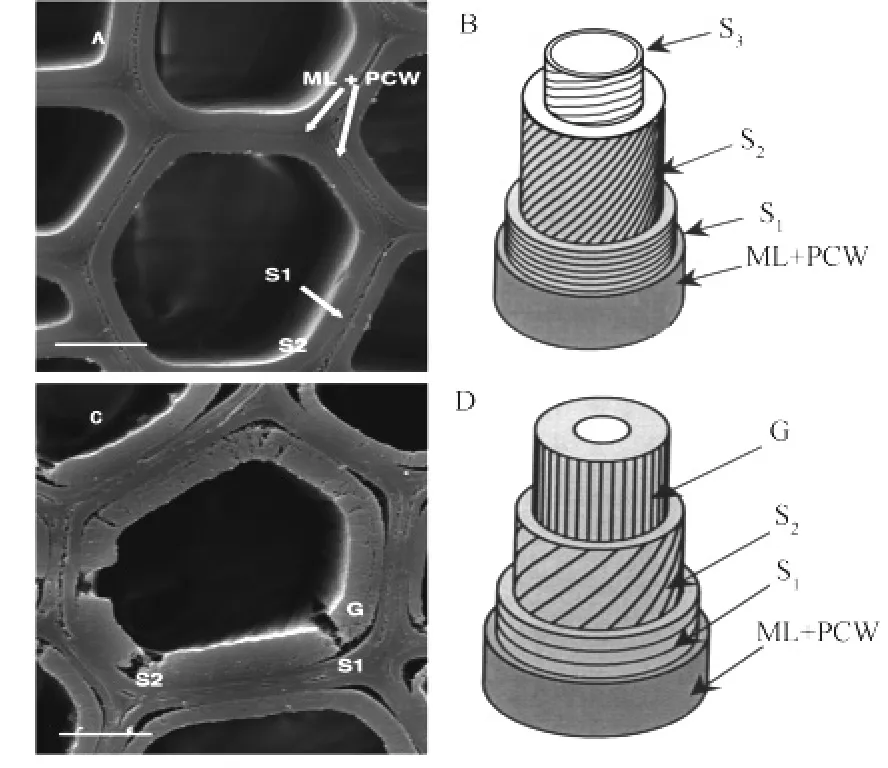
图1 木材次生壁的多层结构(杨木正常木(A、B)与应拉木(C、D)纤维不同壁层扫描电镜图和示意图)
3 木材形成的“多组学”技术
分子生物学的总体目标是确定基因及其产物的功能,并将它们与代谢途径和调控网络联系起来,深入认识生物系统的运行机制。随着高通量、大规模的信息技术发展,在整体水平上研究生物系统成为可能。首先是对重要生物的全基因组测序、基因图谱绘制;接下来就要确定这些基因在生物系统中的具体功能及相互作用。这就要通过转录组、蛋白质组以及代谢组水平研究来实现。基因组是一个静态的信息源,一般情况不因细胞类型和环境条件的变化而发生波动;而转录组、蛋白质组以及代谢组水平物质是动态的,相对丰度随着生境条件和自身生理条件的变化而波动。将不同组学研究联系起来即成为“多组学”技术。通过该技术阐述每个基因的功能,以及基因彼此之间的协调作用网络,才不至于将辛苦得来的基因组数据当做“天书”。就像木材发育一样,我们不仅要了解每个组分的组成和作用,还要深入掌握组分之间相互协调的网络作用,只有这样我们才能真正意义上认知木材形成的生命过程。
3.1 基因组学研究
基因组学,从生物体所有遗传信息着手,从整体水平上测定全部核酸、蛋白质、糖类等生物分子复合体的一级结构,计算或模拟其空间结构,并在细胞中定位,以便确定在各种生物代谢途径、生理途径、信号传导中系统水平上的分子结构图,从而在整体水平上理解生命的本质。树木基因组学研究最初是采用表达序列签(EST)测序技术,探索松树和杨树木材形成过程中基因表达情况[11,15];从此以后,开展了大量不同组织和树种的EST研究。从标准化的桉树全长次生木质部cDNA文库和幼龄材与成熟材的差减文库克隆得到10 000个高质量的ESTs[16]。通过从杨树木质部组织的cDNA文库中大量获取序列表达签,对其进行基因作图[17],并可应用于重要材性性状定位分析和图谱克隆[18]。随后,模式树种杨树的全基因组也测序成功[19]。应力木基因组研究已经在火炬松和杨树中展开,并获得了大量表达序列签。接下来要对基因在染色体上、细胞中定位、确定功能,还需要进行大量的功能验证。
3.2 转录组学研究
转录组,是指在特定时空条件下,生物体将其所有编码序列DNA分子的遗传信息表达,产生的所有RNA分子。转录水平研究是基因功能验证的重要手段之一,主要有以下几种分析方法:基于微阵列技术(cDNA或oligonucleotides)、基于测序技术的基因表达系列分析(SAGE)、大规模平行信号测序(MPSS)、基于差异表达技术(DD-PCR)。
在林木中应用较多的是微阵列分析。应力木组织与正常木在基因表达水平上呈现差异。桉树树枝应拉木中阿拉伯半乳糖束状蛋白编码基因EgrFLA2表达差异最大,弯曲部位上部表达相对丰度是下部的27倍[20]。Paus等[10]通过玫瑰桉弯曲试验,鉴定出一个纤维素合酶基因,并发现其在应拉木中表达丰度较高。许多参与木质素生物合成的基因,在弯曲应拉木中的瞬时表达量增加,但在弯曲回复后又趋于正常。欧洲山杨×欧洲颤杨应拉木转录物分析发现,蔗糖合酶活性增加,因此促使更多的碳元素参与纤维素生物合成。但是,不同次生壁特异纤维素合酶基因,在表达水平上存在很大差异[21]。免疫化学实验,也验证美国枫香树和朴树应拉木中阿拉伯半乳糖束状蛋白编码基因表达丰度升高[22]。最近研究发现在胶质壁层形成中有木葡聚糖内源转糖基酶(XET)表达,推测其可能参与木葡聚糖交联结构的修复[23]。
针叶树种在转录水平上,针对不同表型木材形成的分子基础做了大量研究[24-27]。Steven G Palph等[28]获得阿拉斯加云杉200 000个ESTs和6464个高质量的全长cDNA,为其建立了新的基因资源。裸子植物中参与细胞壁合成的基因,像3种MYB基因[29]、阿拉伯半乳糖蛋白编码基因、α-和β-微管蛋白、木质素合成相关基因等[24,30-31],在应压木中有相对较高的表达量。Yoshida M[32]采用荧光差异显示技术,检测日本扁柏应压木形成中cDNA片段表达,在转录水平上的变化与木材材性变异的相关性。解剖结构观察表明,应压木在树木茎干基部比顶端区域形成更严重,显示出应压木形成与基因表达有相关性。差异表达基因,主要参与细胞分裂、细胞扩张和细胞壁加厚等生物学过程。Saori Yamashita等[33]研究应压木形成程度与相关转录物的关系。随着弯曲角度增加,木质素含量和一个与漆酶高度相似的转录物相对丰度增加;管胞长度随弯曲角度增大而减小,同时一个与抗坏血酸盐氧化酶高度相似的转录物相对丰度降低。木质部发生是包含不同发育阶段,不同细胞类型以及千变万化基因表达的复杂生物学过程[34]。迄今为止,很难将不同发育时期,分解的极其准确并进行基因表达分析。也许,随着单细胞技术的发展,将有可能对树木形成层细胞分化的基因表达,进行更准确的阐述。
3.3 蛋白质组学研究
蛋白质组,是在一定条件下,由特定细胞或生物体产生的所有蛋白质[35]。细胞通过调控其蛋白质的表达水平和活性来适应内外环境的改变,所以蛋白质组无论是在质或量上的改变,都反映了处于生活中的细胞的状况。蛋白质组,是一个复杂而动态的统一体,它是依据其组分的序列、结构、丰度、定位、修饰、相互作用和生化功能来确定的。蛋白质组学研究,也是首先从基本序列的认识开始,鉴定其组成和结构;依次是分析蛋白质在不同生理状态下的表达丰度,蛋白质之间遗传和物理的相互作用及其核酸或其它小分子间的相互作用;最后通过直接功能或蛋白质芯片结合生物信息学技术,验证确定蛋白质功能。目前,常规蛋白质组学的实验流程和主要研究方法如图2所示。
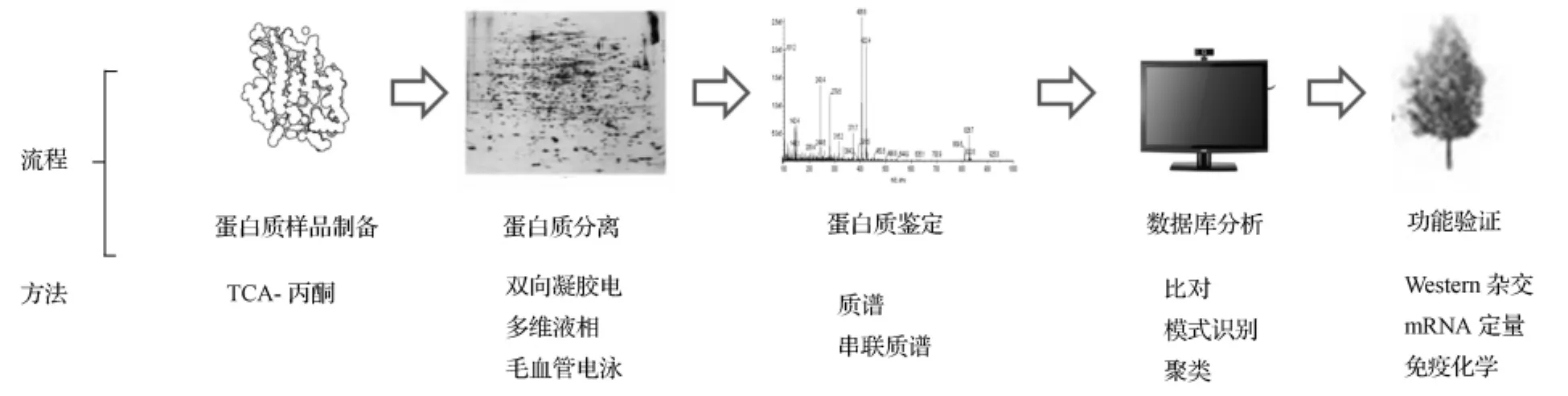
图2 蛋白质组学实验流程及主要方法
应压木与正常木木材形成过程中,蛋白质表达丰度存在差异。蛋白质表达水平,也影响木材的宏观性质。Plomion C.[30]利用双向聚丙烯酰胺凝胶电泳首次分析应压木应答蛋白,32个蛋白质表达点与应力有关。1-环丙胺-1-羧酸氧化酶、咖啡-O-甲基转移酶和咖啡醇甲基转移酶辅酶A表达上调。而后2种是木质素合成途径中的重要中间产物,这与应压木中木质素含量增加相吻合。海岸松木材形成组织蛋白质组学研究[36]发现上千个蛋白质点,分别经过串联质谱(MS/MS)和电离质谱(MALDI-MS)分析部分蛋白质,发现其主要参与防御、碳水化合物和氨基酸代谢、基因和蛋白质表达、细胞骨架、细胞壁生物合成以及新陈代谢。
木材形成组织非胞质蛋白质组分析,也可成为探索木材形成分子机制的强力工具。木质部质膜蛋白质中,纤维素合酶和蔗糖合酶表达丰度较高,特有许多参与木质素合成的酶类,可以推测,这些蛋白质交联成复合体并和质膜有关联[37]。Mast S.[38]等利用高效液相色谱-串联质谱,分析辐射松应压木形成组织细胞膜蛋白,鉴定175个蛋白点,大多数为细胞膜整合蛋白或发源于细胞成分(如细胞核、原质体、内质网、质膜和高尔基体)。经生物信息学分析,预测大多数蛋白参与木材形成调控或细胞壁生物合成。
3.4 代谢组学研究
代谢水平分析,将成为功能基因组学和系统生物学的有力工具和重要组成部分。代谢组学,是对某一生物或细胞在特定生理时期内所有低相对分子质量量代谢产物同时进行定性和定量分析的一门新学科[39]。代谢物质是细胞调控过程的终产物,它们的种类和数量变化被视为生物系统对基因或环境变化的最终响应[40]。代谢物质成分复杂,物理化学性质存在很大差异,需要通过多种分析手段才能获得较完整的信息。目前应用的主要有:气相色谱质谱联用(GC-MS)、液相色谱质谱联用(LC-MS)、核磁共振质谱联用(NMR-MS)。气质联用可以同时检测出数百种化合物,包括糖类、有机酸、氨基酸、脂肪酸和大量不同的次生代谢物[41-42]。木材形成组织中,代谢物质的变化是树木对生长环境的应答。通过鉴定代谢物质种类,描述代谢途径,可以发现关键调控因子,解释生物应答的分子机制。采用GC-MS技术分析火炬松幼龄材和应压木木材形成组织中极性代谢物质,发现应压木中果糖和葡萄糖相对表达丰度明显低于正常木,而参与木质素生物合成的重要中间产物:莽草酸、松柏苷含量增加[43]。这就可以在代谢水平上解释应压木中高木质素、低纤维素含量的化学组成。红松应压木木材形成组织极性代谢产物中,果糖和葡萄糖含量也明显降低。总体看,针对木材形成组织的代谢物质分析研究相对较少。木材形成组织代谢水平研究还要对非极性部分分析,同时要对同一样品进行多种方法分析,像结合气质联用、液质联用和核磁质谱联用3种或更多种平台,这样才能得到相对较完整、充分的认识。
3.5 生物信息学数据处理
生物信息学,是将生物遗传物质的化学信息转变成计算机存储的数字信息,为系列性、大规模研究与分析生命活动奠定基础。基因组、转录组、蛋白质组、代谢组的生物信息,主要以数据库的形式储存。主要数据库有:通用蛋白质资源UniProt、美国的GenBank、欧洲的EBI和日本的DDBJ。还有一些实验室,根据自己的实验结果,建立的小型、单一物种的数据库。木材形成组织相关的数据库,主要集中在这种类型。像桉树ESTs数据库[16]、杨树转录因子数据库[44]、杨树综合功能基因组数据库、云杉的ESTs和全长cDNA数据库[28]等。随着生物信息骤增,必须加强数据库的管理和维护,提炼分支、清理冗余信息、增加资源共享度。生物信息,只有在生物体生命活动中才能真正体现它的功能,它又与生物体结构相互依托。简单将其信息化、孤立出来,其处理结果必然有局限性。期望从生命进化起源的视角,结合理性思维,来看待实验结果。
4 结语与展望
研究现状表明,通过生物技术,鉴定潜在参与木材形成和调控其化学组成与物理性质的关键因子和候选基因,是科学的、有效的策略。应力木系统在该研究中具有不可替代的优势。大量理论证明,大多数植物的生物化学过程、细胞发育、新陈代谢及发生过程,都是相似的。因此,关于木质部发生过程,也已在基因工程模式植物拟南芥(Arabidopsis)和百草目(Zinnia elegans)中展开研究[45-46]。采用“多组学”技术,在整体水平上研究木材形成的生物学过程,是必要的,也是必然的,将会更加全面和精确地阐述木材形成的生物学过程。从植物样品的采集、实验技术的运用、数据的分析、深层次生命信息的发掘,都是互相联系、相辅相成的。木材形成中多组学实验的开展,应该平行进行、横向拓展,将所得信息联系起来,不能使之孤立。
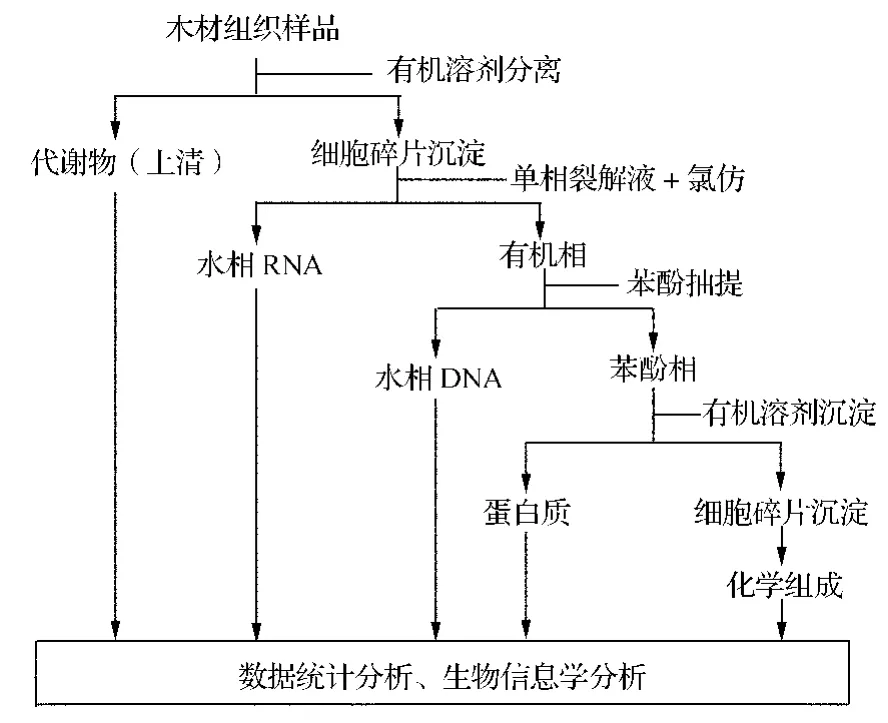
图3 木材形成组织多组学分析流程
以后研究中应该注意的问题:①实验材料多样化,使得实验方法凌乱,难以标准化。②取样标准难以统一;如何科学缜密地获取,需要专业的技术和丰富的经验。③实验周期短、样本少,难以提供完整、充分的验证数据;树木生长生命周期较长,某一性状的形成需要长时间的遗传积累。④实验室之间数据难以共享。对一种或多种材料的研究,是很多学者关注的,平行进行的;但由于材料取样、技术方法、实验设备以及实验人员操作等条件的限制,致使所得结果之间无法相互交流。一旦出现断层,那么这些结果将变得毫无意义。⑤多组学实验数据之间的联系、标准化。⑥分子水平与更高层次认识水平的结合。木材具有如此的细胞结构体系,不仅仅是生物大分子在分子水平上活动的结果。只在分子水平上认识木材细胞壁各组分的发育形成,是远远不够的;应该从发育进化的角度,去探索揭示木材形成的生命本质和奥妙。
[1]王明庥.林木遗传育种学[M].北京:中国林业出版社,2001.
[2]崔克明.植物发育生物学[M].北京:北京大学出版社,2007.
[3]Plomion C,Leprovost G,Stokes A.Wood formation in trees[J].Plant Physiology,2001,127(4):1513-1523.
[4]Déjardin A,Laurans F,Arnaud D.Wood formation in angiosperms[J].Comptes Rendus Biologies,2010,333(4):325-334.
[5]Timell T E.Studies on opposite wood in conifers part(Ⅰ):chemical composition[J].Wood Science and Technology,1973,7(1):1-5.
[6]Timell T E.Studies on opposite wood in conifers part(Ⅱ):histology and ultrastructure[J].Wood Science and Technology,1973,7(2):79-91.
[7]Timell T E.Studies on opposite wood in conifers part(Ⅲ):distribution of lignin[J].Wood Science and Technology,1973,7(3):163-172.
[8]Du S,Yamamoto F.An overview of the biology of reaction wood formation[J].Journal of Integrative Plant Biology,2007,49(2):131-143.
[9]Sterky F,Bhalerao R R,Unneberg P,et al.A populus EST resource for plant functional genomics[J].Proc Natl Acad Sci USA,2004,101(38):13951-13956.
[10]Paux E,Carocha V,Marques C,et al.Transcript profiling of eucalyptus xylem genes during tension wood formation[J].New Phytol,2005,167(1):89-100.
[11]Lev-Yadun S,Sederoff R.Pines as model gymnosperms to study evolution wood formation and perennial growth[J].Journal of Plant Growth Regulation,2000,19(3):290-305.
[12]Allona I,Quinn M,Shoop E,et al.Analysis of xylem formation in pine by cDNA sequencing[J].Proc Natl Acad Sci USA,1998,95:9693-9698.
[13]李坚,栾树杰.生物木材学[M].哈尔滨:东北林业大学出版社,1993.
[14]朱玉贤,李毅,郑晓峰.现代分子生物学[M].3版.北京:高等教育出版社,2007.
[15]Sterky F,Regan S,Karlsson J,et al.Gene discovery in the woodforming tissues of poplar:analysis of 5692 expressed sequence tags[J].Proc Natl Acad Sci USA,1998,95:13330-13335.
[16]David Rengel,Hélène San Clemente,Florence Servant,et al.A new genomic resource dedicated to wood formation in eucalyptus[J].BMC Pant Biology,2009,9(1):1-14.
[17]Cervera M T,Storme V,Ivens B.Dense genetic linkage maps of three populus species(P.deltoids,P.nigra and P.trichocarpa)based on AFLP and microsatellite markers[J].Genetics,2001,158(2):787-809.
[18]Frewen B E,Chen T H,Howe G T,et al.Quantitative trait loci and candidate gene mapping of bud set and bud flush in populus[J].Genetics,2000,154(2):837-845.
[19]Tuskan G A,DiFazio S,Jansson S,et al.The genome of black cottonwood populus trichocarpa(Torr.&Gray)[J].Science,2006,313:1596-1604.
[20]Lafarguette F,Leplé J C,Déjardin A,et al.Poplar genes encoding faciclin-like arabinogalactan proteins are highly expressed in tension wood[J].New Phytologist,2004,164(1):107-121.
[21]Andersson-Gunner。as S,Mellerowicz E J,Love J,et al.Biosynthesis of cellulose-enriched tension wood in Populus:global analysis of transcripts and metabolites identifies biochemical and developmental regulators in secondary wall biosynthesis[J].Plant J,45:144-165.
[22]Andrew J Bowling,Kevin C Vaughn.Immunocytochemical characterization of tension wood:gelatinous fibers contain more than just cellulose[J].American Journal of Botany,2008,95(6):655-663.
[23]Nobuyuki Nishikubo,Tatsuya Awano,Alicja Banasiak,et al.Xyloglucan endo-transglycosylase(XET)functions in gelatinous layers of tension wood fibers in poplar:a glimpse into the mechanism of the balancing act of trees[J].Plant Cell Physiology,2007,48(6):843-855.
[24]Whetten R,Sun Y H,Zhang Y,et al.Functional genomics and cell wall biosynthesis in loblolly pine[J].Plant Molecular Biology,2001,47(1/2):275-291.
[25]Gregoire Le Provost,Raúl Herrera,Jorge Ap Paiva,et al.A micromethod for high throughput RNA extraction of forest trees[J].Biological Research,2007,40(3):291-297.
[26]Egertsdotter U,van ZyI L M,MacKay J,et al.Gene expression during formation of earlywood and latewood in loblolly pine:wxpression profiles of 350 genes[J].Plant Biology,2004,6(6):654-663.
[27]Cato S,McMillan L,Donaldson L,et al.Wood formation from the base to the crown in Pinus radiate:gradients of tracheid wall thickness,wood density,radial growth rate and gene expression[J].Plant Molecular Biology,2006,60(4):565-581.
[28]Steven G Ralph,Hye Jung E Chun,Natalia Kolosova,et al.A conifer genomics resource of 200000 spruce(Picea spp.)ESTs and 6464 high-quality,sequence-finished full-length cDNAs for Sitka spruce(Picea sitchensis)[J].BMC Genomics,2008,9:484-501.
[29]Frank Bedon,Jacqueline Grima-Pettenati,John Mackay.Conifer R2R3-MYB transcription factors:sequence analyses and gene expression in wood-forming tissue of white spruce(Picea glauca)[J].BMC Plant Biology,2007,7:17-34.
[30]Plomion C,Pionneau C,Brach J,et al.Compression wood-responsive proteins in developing xylem of maritime pine(Pinus pinaster Ait.)[J].Plant Physiol,2000,123:959-969.
[31]Koutaniemi S,Warinowski T,Karkonen A,et al.Expression profiling of the lignin biosynthetic pathway in Norway spruce using EST sequencing and real-time RT-PCR[J].Plant Molecular Biology,2007,65(3):311-328.
[32]Saori Yamashita,Masato Yoshida,Hiroyuki Yamamoto,et al.Screening genes that change expression during compression wood formation in Chamaecyparis obtuse[J].Tree Physiology,2008,28(9):1331-1340.
[33]Yamashita S,Yoshida M,Yamamoto H.Relationship between development of compression wood and gene expression[J].Plant Science,2009,176:729-735.
[34]Du J,Groover A.Transcriptional regulation of secondary growth and wood formation[J].J Inter Plant Biol,2010,52(1):17-27.
[35](英)Twyman R M.蛋白质组学原理[M].王恒樑,袁静,刘先凯,等译.北京:化学工业出版社,2007.
[36]Jean-Marc Gion,Céline Lalanne,Grégoire Le Provost,et al.The proteome of maritime pine wood formation tissue[J].Proteomics,2005,5:3731-3751.
[37]Robert Nilsson,Katja Bernfur,Niklas Gustavsson,et al.Proteomics of plasma membranes from poplar trees reveals tissue distribution of transporters,receptors,and proteins in cell wall formation[J].Molecular&Cellular Proteomics,2010,9:368-387.
[38]Mast S,Peng L,Jordan T W,et al.Proteomic analysis of membrane preparations from developing Pinus radiate compression wood[J].Tree Physiology,2010,30(11):1456-1468.
[39]Goodacre Royston.Metabolic profiling:pathways in discovery[J].Drug Discovery Today,2004,9(6):260-261.
[40]Fiehn O.Metabolomics:the link between genotypes and phenotypes[J].Plant Molecular Biology,2002,48(1/2):155-171.
[41]Taylor J,King R D,Altmann T,et al.Application of metabolomics to plant genotype discrimination using statistics and machine learning[J].Bioinformatics,2002,18(2):241-248.
[42]Tolstikov V V,Fiehn O.Analysis of highly polar compounds of plant origin:Combination of hydrophilic interaction chromatography and electrospray ion trap mass spectrometry[J].Analytical Biochemistry,2002,301(2):298-307.
[43]Yeh T,Morris C,Goldfarb B,et al.Utilization of polar metabolite profiling in the comparison of juvenile wood and compression wood in loblolly pine(Pinus taeda)[J].Tree Physiology,269(11):1497-1503.
[44]Zhu Q H,Guo A Y,Gao G,et al.DPTF:a database of poplar transcription factors[J].Bioinformatics,2007,23(10):1307-1308.
[45]Chaffey N,Cholewa E,Regan S,et al.Secondary xylem development in Arabidopsis:a model for wood formation[J].Physiologia Plantarum,2002,114(4):594-600.
[46]Reiter W D.Arabidopsis thaliana as a model system to study synthesis,structure,and function of the plant cell wall[J].Plant Physiology and Biochemistry,1998,36(1/2):167-176.

