潮霉素A的生物合成研究进展
李军,朱清华,马俊英,王博,刘静,张云,鞠建华*
(1.中国科学院海洋生物资源可持续利用重点实验室广东省海洋药物重点实验室中国科学院海洋微生物中心中国科学院南海海洋研究所,广东广州510301;2.中国科学院研究生院,北京100049;3.德州学院生物系,山东德州253023)
潮霉素A是一种天然的抗生素,1953年第一次从吸水链霉菌NRRL 2388的发酵产物中被分离得到[1],由5-脱氢-α-L-岩藻糖、(E-3-(3,4-二羟苯基)-2-甲基丙烯酸和2L-2-氨基-2-脱氧-4,5-O-亚甲基肌醇3个结构单元组成。早期的研究显示,潮霉素A具有广泛的生物学活性,如抗菌活性[2](G+、G-)、抗红细胞凝聚活性和抗密螺旋体活性[3]及免疫抑制活性[4]等。进一步的研究表明,潮霉素A抗菌作用机理为其通过与核糖体50S亚基结合,抑制核糖体肽基转移酶,从而抑制蛋白质的合成[5];潮霉素A具有抗红细胞凝聚活性和抗密螺旋体活性,暗示其在猪密螺旋体引起的痢疾治疗方面的潜在应用价值[3]。最新的研究结果表明其结构类似物(甲氧潮霉素A)具有去除杂草的生物活性[6]。正因为潮霉素A具有新颖的结构特点,良好的生物学活性,独特的作用机制及潜在应用价值,引起了国内外众多学者对其研究开发的兴趣。本文就潮霉素A的结构特点、生物合成途径以及生物合成基因的工程改造等方面进行综述。
1 潮霉素A的化学结构及其类似物
在吸水链霉菌NRRL 2388(Streptomyces hygroscopicus NRRL 2388)的发酵产物中发现了潮霉素A(Ⅰ)。从发酵产物中将其分离,并进行化学手段分析,得出它的化学结构是由3个结构单元构成:5-脱氢-α-L-岩藻糖(结构单元A)[5-dehydro-α-L-fucofuranose]、(E)-3-(3,4-二羟苯基)-2-甲基丙烯酸(结构单元B)[(E)-3-(3,4-dihydroxyphenyl)-2-methylacrylic acid]和2L-2-氨基-2-脱氧-4,5-O-亚甲基肌醇(结构单元C)[2L-2-amino-2-deoxy-4,5-O-methylene-neo-inositol][7-8]。在同一发酵产物中也分离得到了甲氧潮霉素A(methoxyhygromycin A,Ⅱ),其化学结构见图1。
利用以潮霉素A为基础所进行的化学半合成[9],得到了大量的潮霉素A的结构类似物,比如:5''-二氢甲氧基-潮霉素A[5''-dihydromethoxyhygromycin A,Ⅲ]、5''-二氢潮霉素A(DHHA)[5''dihydrohygromycin A,Ⅳ]和潮霉素A苷元(hygromycin A aglycon,Ⅴ)等。通过化学结构与生物功能之间的关系研究表明氨基环醇结构模块是其显示抗菌活性的药效基团,而5-脱氢-α-L-岩藻糖模块则不是其活性的必需基团,它可以被疏水性的烯丙基团所取代。如果(E)-3-(3,4-二羟苯基)-2-甲基丙烯酸模块中的甲基被丙基或烯丙基等基团所取代的话,整个化合物的抗菌活性会降低。核苷类抗生素A201A(Ⅵ)有着与潮霉素A相类似的结构单元,它也具有广谱的抗菌活性[10]。

图1 潮霉素A及其类似物的化学结构Fig.1 Chemical structures of hygromycin A and its structurally related analogues
2 潮霉素A的生物合成基因簇及其生物合成过程
潮霉素A的相关生物合成基因簇已被成功克隆。潮霉素A的生物合成基因簇约长31.5 kb,有29个开放阅读框(图2),分别命名为hyg1~29,其中包括调控基因、抗性基因和负责3个结构单元合成的结构基因[11]。通过生物信息学分析,各基因的功能如表1所示。其中,基因hyg1和hyg3是调控基因;另外几个基因如hyg6或hyg29、hyg19、hyg21和hyg28,它们分别编码甲基转移酶、跨膜蛋白、磷酸转移酶、ABC运输蛋白,这些蛋白质都与菌株本身抵抗潮霉素A的作用有关[12]。
对hyg基因簇进行同源比对及功能分析(表1)后,推测有几个基因可能参与从NDP-甘露糖到5-脱氢-α-L-岩藻糖的生物合成[13]。hyg5被认为编码GDP-D-甘露糖-4,6-脱水酶(MDH),负责催化活化的NDP-甘露糖转化为NDP-4-甲基-6-脱氧甘露糖;潮霉素A合成的重要步骤是NDP-L-岩藻糖的生成,此过程可能由基因hyg23编码产物所催化,因为hyg基因的推测产物与L-岩藻糖合成酶有超过60%的同源性;接下来是由吡喃六环糖转变为呋喃五环糖,这步反应可能由与葡萄糖转移酶同源性很高的基因hyg20编码的蛋白催化完成[14];5-脱氢岩藻糖合成的最后一步可能是基因hyg26编码的脱氢酶来完成。此外,基因hyg16编码葡萄糖基转移酶,负责5-脱氢-α-L-岩藻糖结构单元和(E)-3-(3,4-二羟苯基)-2-甲基丙烯酸结构单元之间的糖苷键的形成。巧合的是,在抗生素A201A的生物合成中也涉及到葡萄糖苷键的形成,该反应可能由生物合成基因簇中的结构基因ata5编码的蛋白催化执行的,Ata5蛋白与Hyg16蛋白有63%的相似度[10]。

图2 潮霉素A的生物合成基因簇的组织结构Fig.2 Organization of hygromycin A biosynthetic gene cluster in Streptomyces hygroscopicus NRRL 2388

表1 潮霉素A生物合成基因簇中各基因的功能[11]Table 1 Deduced functions of open reading frames in the hygromycin A biosynthetic gene cluster[11]

续表
在结构单元B合成途径中,涉及到基因hyg9~15、hyg27、hyg4、hyg22。该模块的合成前体是4-羟基苯甲酸,它由基因hyg4编码的分支酸裂合酶催化分支酸而得到。分支酸产生于莽草酸途径,这条途径的关键步骤是由DAHP(3-deoxy-D-arabino-heptulosonate-7-phosphate,3-脱氧-D-阿拉伯酸-庚酮糖-7-磷酸)合成酶催化反应[15]的,芳香族酸通过变构机制对DAHP(的活性或含量)进行反馈抑制调节[16]。实验表明,在潮霉素A生物合成中,基因hyg27编码一种与DAHP合成酶同源的功能蛋白;基因hyg2编码的蛋白质与4-羟基苯甲酸羟化酶相似,可能催化了3,4-二羟基苯甲酸的合成,目前不能确定羟化反应的底物是4-羟基苯甲酸还是之后的反应中间体;然后,3,4-二羟基苯甲酸(或4-羟基苯甲酸)可能在基因hyg12的蛋白产物的催化下形成一个硫酯类化合物,基因hyg12的蛋白质产物与乙酰辅酶A连接酶家族相类似;之后,通过基因hyg9、hyg13(编码的蛋白产物与酰基载体蛋白ACP类似),基因hyg22(蛋白产物是酰基转移酶AT[17-19]),基因hyg10(编码β-酮脂酰合酶KS[20])以及基因hyg11的协同作用,得到化合物3-(3,4-二氢苯基)-3-羟基-2-甲基-丙酰ACP。最后,通过基因hyg14和hyg15编码的功能蛋白3-羟酰基ACP脱水酶(3-hydroxylacyl ACP dehydratase)和3-酮脂酰ACP还原酶(3-ketoacyl ACP reductase)的催化作用,转变为(E)-3-(3,4-二羟苯基)-2-甲基丙烯酸ACP。
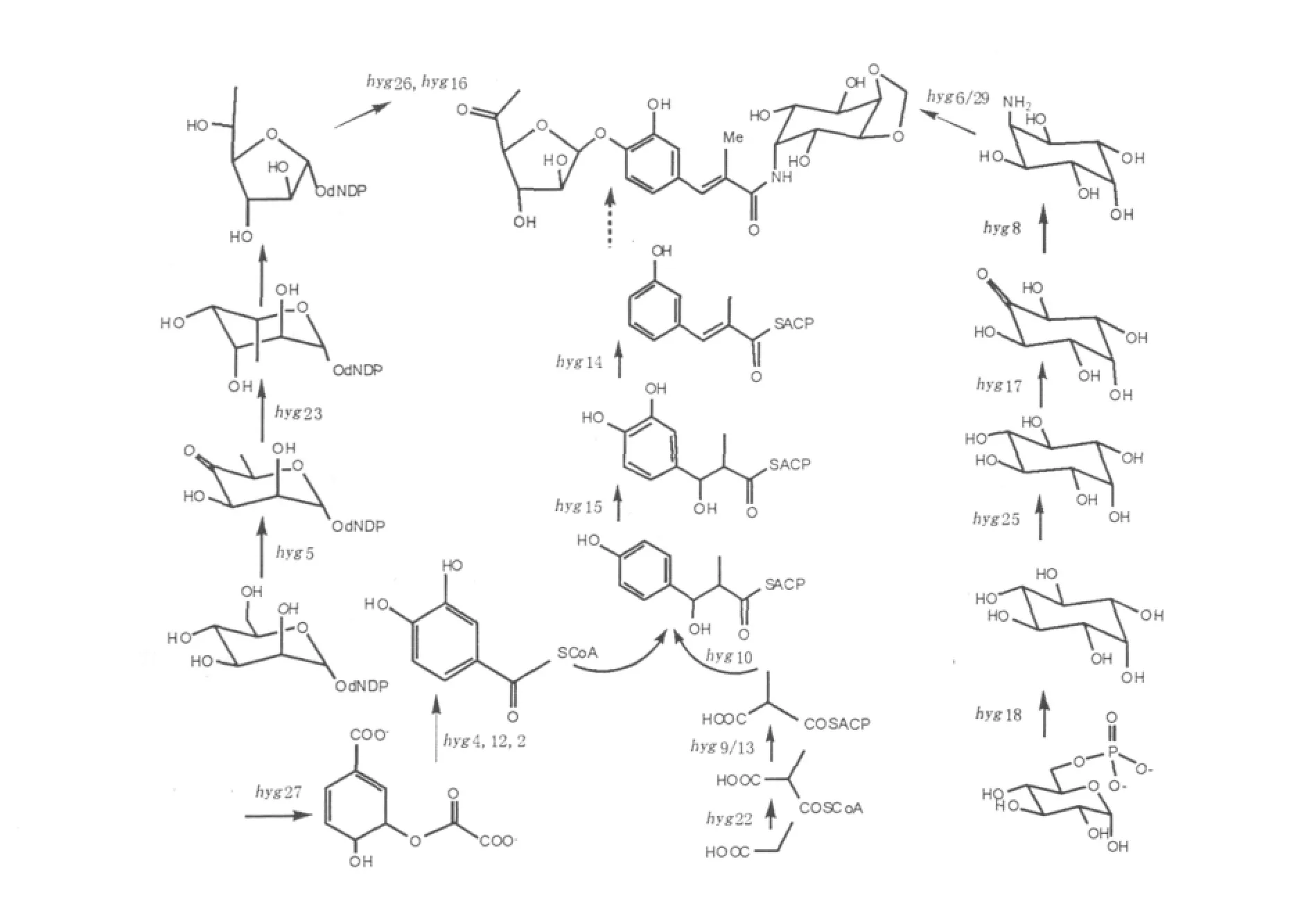
图3 潮霉素A在NRRL 2388中可能的生物合成过程Fig.3 Proposed biosynthetic pathway of hygromycin A in Streptomyces hygroscopicus NRRL 2388
潮霉素A结构单元C的合成起始于6-磷酸-葡萄糖。该底物依次经过基因hyg18编码的1-磷酸肌醇合酶和基因hyg25编码的肌醇单磷酸酶的催化形成肌醇。然后,在基因hyg17编码产生的肌醇脱氢酶的催化下,肌醇C5位置上发生氧化反应,再通过hyg8基因编码的氨基转移酶的作用,形成2L-2-氨基-2-脱氧新肌醇[21]。另外,在潮霉素A的化学结构中有1个在C4和C5之间的亚甲基桥,现在还不确定它形成于哪个阶段的,推测有1个甲基在这条合成途径的倒数第2个步骤上被转移到了C5上,形成了2L-2-氨基-2-脱氧-5-O-甲基新肌醇,而2L-2-氨基-2-脱氧-4,5-O-亚甲基肌醇的最后形成还需要发生一个独特的氧化反应。而这类氧化反应在糖类反应中也是少见的。
3 潮霉素A生物合成基因簇的基因功能验证及其自抗性机制
目前,一些分子生物学实验证明了hyg生物合成基因簇的功能,为进一步弄清潮霉素A的生物合成过程提供了很多信息[11,22]。同源重组就是一个研究基因功能的良好方法。
利用同源重组的方法,将两端与hyg26基因序列同源而中间具有阿普拉霉素抗性标记的DNA片段导入野生型吸水链霉菌NRRL 2388中,用以替换hyg26基因,得到SCH30突变株[11]。将野生型和突变型菌株发酵处理,通过HPLC检测,可以看到,在野生型中既有潮霉素A又有甲氧潮霉素A;而突变株SCH30中这2种化合物都不存在,但出现了另外3个较高的峰段。经分离纯化后,3种化合物被证明是潮霉素A类似物:5''-二氢甲氧基-潮霉素A[5''-dihydromethoxy-hygromycin A]、5''-二氢潮霉素A(DHHA)[5''-dihydrohygromycin A]、(E)-3-(3-羟基-4-O-α-岩藻呋喃苯基)-2-甲基丙烯酸[(E)-3-(3-hydroxy-4-O-α-fucofuranosylphenyl-2-methylacrylicacid]。根据对SCH30发酵产物的研究,推测:形成5-脱氢-α-L-岩藻呋喃糖模块的最后一步是由Hyg26催化的α-L-岩藻呋喃糖的氧化过程,但无法确定这步氧化过程发生于2模块之间糖苷键的形成顺序。
任何一种产生抗生素的微生物都会具备一种或几种自身保护机制,而编码具有自我保护功能的蛋白质的基因一般就存在于抗生素生物合成基因簇内部,并作为其中的一部分,与其他基因起协调作用[23]。在潮霉素A的生物合成基因簇中,hyg6或hyg29编码甲基转移酶可能使潮霉素A失活;hyg19和hyg28编码跨膜运输蛋白,可能将潮霉素A转运到膜外而进行保护自身;hyg21编码磷酸转移酶,使潮霉素A发生磷酸化而使其失活。体外试验表明磷酸化的潮霉素A(HA-P)失去了抑菌活性,另外,对hyg21基因双交突变株进行HPLC分析,结果表明突变株的发酵产物中存在潮霉素A,但含量较野生型下降了90%以上,这说明Hyg21对潮霉素A的合成产量有重要影响。但突变株与野生型菌株对潮霉素A有同等的抗性,这说明在吸水链霉菌NRRL 2388中存在多种自我保护机制,将潮霉素A磷酸化使其失活只是其中之一。
基因hyg19编码质子梯度依赖型转运蛋白,它能将潮霉素A转运到细胞外,保护菌株不被伤害。基因hyg19的存在对于潮霉素A的合成是必要的。有研究表明敲除基因hyg19会有两种结果,其一是突变株中的潮霉素A的产量会减少,并且有DHHA中间体产生。DHHA对大肠埃希菌有微弱的抗菌活性和微弱的体外蛋白合成抑制剂活性。其二是突变株没有增加对潮霉素A的敏感性,而基因hyg19和hyg21均被敲除的突变株对潮霉素A的敏感性则大大增强。这些资料表明,在合成和转运潮霉素A方面,Hyg21和Hyg19是起协同作用,同时Hyg19蛋白兼有参与合成潮霉素A和转运潮霉素A的双重功能[24]。
4 结语与展望
潮霉素A表现出的广泛的生物学活性,新颖的结构特征,独特的作用机制及其潜在的应用价值,显示出对其进行生物合成过程和合成机制研究的重要意义。组合生物合成是上世纪90年代中后期发展起来的新技术,随着后来基因测序技术、基因重组新技术(如PCR-targeting)和异源基因表达技术的发展,组合生物合成已在生物学领域带来了革命性的变化,也给化学学科创造新的化学结构和构建化合物库带来了新的方法[25-27]。利用生物信息学技术,潮霉素A生物合成相关的基因簇已被克隆,并根据潮霉素A的结构,推测了该抗生素的生物合成途径。另外,利用同源重组等手段,已得到功能基因的缺失多个突变菌株,并结合HPLC、LC-MS等技术,已经初步确定了部分基因的功能。而随着对其生物合成各基因功能研究的深入,各基因在其生物合成过程中的作用将会得到进一步的阐明,功能新颖、作用机制独特的酶将会被发现并加以利用。
[1] Pittenger R C,Wolfe R N,Hoehn P N,et al.Hygromycin.I.Preliminary studies in the production and biologic activity on a new antibiotic[J].AntibiotChemother,1953,3:1268-1278.
[2] Mann R L,Bromer W W.The isolation of a second antibiotic from Streptomyces hygroscopicus[J].J Am Chem Soc,1958,80:2714-2716.
[3] Omura S,Nakagawa A,Fujimoto T,et al.Hygromycin A,an antitreponemal substance.I.Screening method and therapeutic effect for Treponema hyodysenteriae-caused infection in CF-1mice[J].J Antibiot,1987,40:1619-1626.
[4] Uyeda M,Mizukami M,Yokomizo K,et al.Pentalenolactone I and hygromycin A,immuosuppressants produced by Streptomyces filipensis and Streptomyces hygroscopicus[J].Biosci Biotechnol Biochem,2001,65:1252-1254.
[5] Guerrero M D,Modolell J.Hygromycin A,a novel inhibitor of ribosomal peptidyltransferase[J].Eur J Biochem,1980,107:409-414.
[6] Lee H B,Kim C-J,Kim J-S,et al.A bleaching herbicidal activity of methoxyhygromycin(MHM)produced by an actinomycete strain Streptomyces sp.8E-12[J].Lett Appl Microbiol,2003,36:387-391.
[7] Katsumi K,Yoshio S.Nuclear magnetic resonance and structure of hygromycin(homomycin)[J].Agric Biol Chem,1978,42:279-286.
[8] Katsumi K,Setsuko K,Kimihiro W.On the structure of hygromycin,the location of a methylene substituent and the anomeric configuration of the arabino-hexoside moiety[J].J Antibiot,1976,50:771-773.
[9] Hayashi S F,Norcia L J,Seibel S B,et al.Structure-activity relationships of hygromycin A and its analogs:protein synthesis inhibition activity in a cell free system[J].J Antibiot,1997,50:514-521.
[10] Irene S,Eloisa S,Antonio J.Identification of a set of genes involved in the biosynthesis of the aminonucleoside moiety of antibiotic A201A from Streptomyces capreolus[J].Eur J Biochem,2002,269:5527-5535.
[11] Palaniappan N,Ayers S,Reynolds K A.Production of hygromycin A analogs in Streptomyces hygroscopicus NRRL 2388 through identification and manipulation of the biosynthetic gene cluster[J].Chem biol,2006,13:753-764.
[12] Cundliffe E.Resistance to macrolides and lincosamides in Streptomyces lividans and to aminoglycosides in Micromonospora purpurea[J].Gene,1992,115:75-84.
[13] Habib el S E,Scarsdale J N,Reynolds K A.Biosynthetic origin of hygromycin A[J].Antimicrob Agents Chemother,2003,47:2065-2071.
[14] Sanders D A,Staines A G,McMahon S A,et al.UDP-galactopyranose mutase has a novel structure and mechanism[J].Nat Struct Biol,2001,8:858-863.
[15] Bentley R.The shikimic pathway—a metabolic tree with many branches[J].Crit Rev Biochem Mol Biol,1990,25:307-383.
[16] Shumilin I A,Zhao C,Bauerle R,et al.Allosteric inhibition of 3-deoxy-D-arabino-heptulosonate-7-phosphatesynthasealters the coordination of both substrates[J].J Mol Biol,2002,320:1147-1156.
[17] Haydock S,Aparico J F,Molnar I,et al.Divergent structural motifs correlated with the substrate specificity of(methyl)malonyl-CoA:acyl carrier protein transacylase domains in modular polyketide synthases[J].FEBS Lett,1995,374:246-248.
[18] Lau J,Hong F,Cane D E,et al.Dissecting the role of acyltransferase domains of modular polyketide synthases in the choice and stereochemical fate of extender units[J].Biochemistry,1999,38:1643-1651.
[19] Reeves C D,Murli S,Ashley G W,et al.Alteration of the substrate specificity of a modular polyketide synthase acyltransferase domain through site-specific mutations[J].Biochemistry,2001,40:15464-15470.
[20] Huang W,Jia J,Edwards P,et al.Crystal structure of beta-ketoacyl-acyl carrier protein synthase II from E.coli reveals the molecular architecture of condensing enzymes[J].EMBO J,1998,17:1183-1191.
[21] Ahlert J,Distler J,Mansouri K,et al.Identification of stsC,the gene encoding the L-glutamine:scyllo-inosose aminotransferase from streptomycin-producing Streptomycetes[J].Arch Microbiol,1997,168:102-113.
[22] Palaniappan N,Dhote V,Ayers S,et al.Biosynthesis of the aminocyclitol subunit of hygromycin A in Streptomyces hygroscopicus NRRL 2388[J].Chem Biol,2009,16:1180-1189.
[23] Vidya D,Shuchi G,Reynolds K A.An O-phosphotransferase catalyzes phosphorylation of hygromycin A in the antibiotic-producing organism Streptomyces hygroscopicus[J].Antimicrob A-gents Chemother,2008,3580-3588.
[24] Vidya D,Agata L S,Reynolds K A.The final step of hygromycin A biosynthesis,oxidation of C-5''dihydrohygromycin A,is linked to a putative proton gradient-dependent efflux[J].Antimicrob Agents Chemother,2009,53(12):5163-5172.
[25] Baltz,R.H.Molecular engineering approaches to peptide,polyketide and other antibiotics[J].Nat Biotechnol,2006,24:1533-1540.
[26] 刘文,唐功利.以生物合成为基础的代谢工程和组合生物合成[J].中国生物工程杂志,2005,25(1):1-5.
[27] McDaniel,R.;Welch,M.;Hutchinson,C.R.Genetic approaches to polyketide antibiotics[J].Chem Rev,2005,105:543-558.

