3种水产病原菌简型基因芯片检测技术的建立*
李 晨,黄 倢,谢国驷,赵 培,王秀华
(1.农业部海洋渔业资源可持续利用重点开放实验室中国水产科学研究院黄海水产研究所,山东青岛266071;2.上海海洋大学水产与生命学院,上海200090)
3种水产病原菌简型基因芯片检测技术的建立*
李 晨1,2,黄 倢1**,谢国驷1,2,赵 培1,2,王秀华1
(1.农业部海洋渔业资源可持续利用重点开放实验室中国水产科学研究院黄海水产研究所,山东青岛266071;2.上海海洋大学水产与生命学院,上海200090)
根据水产养殖中常见的3种病原菌鳗弧菌(Vibrio anguillarum)、嗜水气单胞菌(Aeromonas hydrophila)及迟缓爱德华氏菌(Edwardsiella tarda)的研究资料,筛选3个毒力相关基因toxR、aerA、evpA设计引物和探针,构建简型基因芯片,并使用对虾白斑病综合征病毒(WSSV)variable region的PCR荧光标记产物作为表面化学质控。通过已构建和优化的多重PCR反应条件,获得了3个目的基因的PCR产物;经过芯片制作过程的优化,将探针以终浓度20μmol/L溶于50%DMSO,在室温、相对湿度45%的条件下点印于醛基基片表面。扩增的PCR产物与杂交液混合后,在42℃杂交2 h就可检测到理想的杂交信号。芯片的灵敏度试验结果表明,可以检测到的3种水产病原菌的最低模板DNA为:鳗弧菌3×102拷贝、嗜水气单胞菌5×103拷贝、迟缓爱德华氏菌6×101拷贝。该芯片可成功应用于患病大菱鲆内脏的细菌分离物的鉴定。
基因芯片;水产病原菌;检测
基因芯片(Gene chip)又称DNA芯片(DNA chip)、微阵列(Microarray),是1980年代产生,1990年代迅速发展起来的1种高科技手段,它具有高度并行性、高通量、微型化、自动化以及较高的灵敏度和直观性等优点[1],如今它已经应用于多种领域[2]。
随着水产养殖业规模的不断扩大和集约化程度的不断提高,各种病害也接踵而至,给海水养殖生产造成了重大损失,将基因芯片技术应用于水产病原微生物的鉴定、毒性分析等研究具有重要意义。美国学者Warsen等根据细菌16S rDNA设计寡核苷酸探针构建基因芯片,对18种细菌进行检测,其中包括嗜水气单胞菌、爱德华氏菌、葡萄球菌、链球菌等15种鱼类常见病原菌,结果能100%特异性地检测出这些病原菌[3];Georg Mitterer等根据23S rDNA设计特异引物和探针,建立固相PCR-芯片检测技术,可以检测出多种葡萄球菌、大肠杆菌、假单胞菌等[4];日本水产综合研究中心2005年成功开发出同时检测38种鱼类细菌的新技术,其中包括20种淡水鱼病原菌和18种海水鱼病原菌[5];国内学者李国军等根据大肠埃希氏菌、伤寒沙门氏菌、铜绿假单胞菌、李斯特氏菌等10种细菌的23S rDNA的高度保守和中间变异的特性,设计引物和探针,制备出可快速检测临床常见致病菌的寡核苷酸芯片[6]。另外基因芯片技术在水产食品中常见微生物的特异性检测中也得到了应用[7]。
由于基因芯片技术荧光标记引物和氨基化探针合成、PCR扩增、芯片点样等方面成本显著,很多情况下限制了基因芯片的应用,基因芯片技术的精简化在水产养殖领域十分必要。鳗弧菌和迟缓爱德华氏菌是感染海水养殖鱼类最常见的病原菌,嗜水气单胞菌和迟缓爱德华氏菌是淡水养殖中最常见的病原菌,而鳗弧菌、嗜水气单胞菌又是中国明对虾及斑节对虾主要的致病菌[8-9],建立可同时检测这3种病原菌的基因芯片,可以以较低的成本对淡海水及半咸水的广泛养殖区域的3种细菌性疾病进行有效诊断。在此基础上,进一步选择3种病原菌稳定而特异的毒力相关基因,不仅可以实现对3种病原菌的快速检测,还可提供病原菌的毒力信息。基于此思想,本文针对上述3种病原菌,构建基因芯片检测技术,尝试开展简化、低成本的基因芯片检测技术的研究。
1 材料与方法
1.1 菌种来源
鳗弧菌菌株ATCC43306购自美国标准菌种收藏所(American Type Culture Collection,ATCC);嗜水气单胞菌、迟缓爱德华氏菌,均为本实验室保种。
1.2 引物探针的设计与合成
根据GenBank数据库中的鳗弧菌调控毒力蛋白表达的toxR基因、嗜水气单胞菌编码气溶素的aerA基因、迟缓爱德华氏菌中编码分泌系统装置蛋白的evpA基因对应的序列设计引物,筛选合适引物,再在同一条件下设计对应的探针,注意排除各引物及探针间存在的发卡结构、二聚体、交叉配对和错配等情况。所设计的引物、探针及阳性质控variable region的引物一起由Takara(宝生物有限公司)合成,并且在正向引物的5′端标记HEX,探针3′端作氨基修饰。
1.3 细菌基因组DNA的提取
细菌基因组DNA提取采用细菌基因组DNA提取试剂盒(TIANamp bacteria DNA kit,北京天根生化科技有限公司)提取,并用基因分析仪(NanoDrop ND-2000c,美国NanoDrop)测定DNA浓度,-20℃保存备用。
1.4 多重PCR扩增
利用已构建并优化的三重PCR体系[10],25μL PCR体系中含模板1.0μL,1.5 U ExTaq(5U/μL,Takara),1×ExTaqPCR buffer,2 mmol/L Mg2+,0.2 mmol/L dNTP。扩增程序94℃4 min;94℃30 s,58℃30 s,72℃40 s,30个循环;72℃10 min。分别以单一的鳗弧菌、嗜水气单胞菌、迟缓爱德华氏菌的基因组DNA及3种细菌基因组DNA的混合物为模板进行toxR、aerA、evpA基因片段的扩增,产物用于芯片杂交试验以测试探针的特异性。将3种细菌基因组DNA的混合物进行101~1010倍梯度稀释,并以此为模板进行三重PCR扩增,扩增产物用于测试芯片检测的灵敏度。
1.5 芯片的制备
利用1.4中PCR体系及扩增程序,不加Mg2+,并调整退火温度到55.5℃,以WSSV的核酸为模板扩增variable region产物。用二甲基亚砜(DMSO,Amresco)等比例稀释探针toxR、aerA、evpA至终浓度20 μmol/L。利用Personal Arrayer 16点样仪(Capital-Bio)将探针及variable regionPCR产物点印于醛基基片(CapitalBio)表面,点的直径为0.15 mm,形成5×5方阵。每个子阵的布局为:1,QC1(variable region)×5;2,tox R×5;3,aerA×5;4,evpA×5;5,QC2(variable region)×5。按照醛基基片说明书进行芯片的固定,37℃湿盒内水合12 h以上,0.2%十二烷基硫酸钠(SDS)洗涤,NaBH4溶液封闭5 min,清洗后离心甩干。
1.6 芯片的杂交及检测
在杂交盒内放入200μL的灭菌水,以保证杂交环境的湿度。将制备好的芯片贴上围栏,放入杂交盒,盖上盖片,使整个小子阵都包含在1个杂交空间里。将多重PCR的产物与杂交液混合均匀后热变性5 min,冰骤1 min,加入杂交空间里,装好杂交盒,42℃杂交2 h。杂交体系为15μL,除PCR(混合)产物7μL外,还有20×saline sodium citrate(SSC)2.25μL、10%SDS 0.3μL、去离子甲酰胺3.75μL、50×Denhardt’s 1.5 μL。洗涤参照醛基基片说明书操作。离心甩干后,采用Luxscan 10K扫描仪(CapitalBio)检测杂交结果,分辨率为10μm,该扫描仪的光电倍增管(PMT)是16位的,形成TIFF图片的灰阶是216,即最高信号值就是216-1=65535。各个点在532 nm处的信号值与周围背景值之比是信噪比(Signal to Noise Ratio,SNR),SNR越大表示阳性信号越强。
1.7 芯片特异性和灵敏性的试验
将1.4中得到的单一细菌的基因组DNA及3种细菌混合基因组DNA为模板的多重PCR产物,分别与杂交液混匀,加入子阵,用于检验探针的特异性;将1.4中各浓度梯度混合基因组DNA的多重PCR产物,分别与杂交液混匀,加入子阵,用于检测芯片的灵敏性。芯片杂交及检测过程同1.6。
1.8 患病大菱鲆内脏组织中分离菌的鉴定
从某养殖场患病大菱鲆的内脏组织中分离出的2株细菌,分别命名09071501、09071502,提取基因组DNA为模板,进行多重PCR,体系及扩增程序同1.4。扩增产物一部分按照1.6中程序进行芯片杂交试验,另一部分送去Takara测序。
2 结果与分析
2.1 细菌基因组DNA的提取及3种基因目的产物的扩增
成功提取了鳗弧菌、嗜水气单胞菌、迟缓爱德华氏菌的基因组DNA,基因分析仪测定三者的核酸浓度分别为125.4、256和25.8 ng/μL。利用条件优化后的多重PCR体系,成功扩增出了3种特异性基因的目的片段及混合PCR产物,如图1所示,3种目的产物分别为鳗弧菌的toxR368 bp、嗜水气单胞菌aerA545 bp、迟缓爱德华氏菌evpA250 bp。
2.2 芯片上的固定及特异性检验
芯片固定后,扫描信号展示如图2,亮点处为各子阵中表面化学质控variable region的信号,由于2种产物的模板浓度不同而形成的信号强度不同,但数据表明质控信号很好,说明芯片固定成功。

图2 芯片固定后的信号展示及3种病原菌探针方阵的示意图Fig.2 Displaying the signal after the chip fixed and diagram of probe square of three pathogens
探针特异性的检验结果如图3所示,与单一细菌基因组DNA为模板的多重PCR产物杂交,结果分别在相应地鳗弧菌探针tox R、嗜水气单胞菌aerA、迟缓爱德华氏菌evpA位点处出现杂交阳性信号,而且很强。与混合基因组DNA为模板的多重PCR产物杂交后,相应的3种探针位点处都为阳性信号,说明这些探针特异性良好,可以用于多重PCR产物的基因芯片检测。
2.3 基因芯片检测灵敏性的检验
利用图像扫描得到的各个位点的信号值、背景值计算得到该点的SNR值,将所得数据形成折线图,可以直观地判断出由多重PCR方法处理的样品,用简型基因芯片技术可以检测到的最低限度是多少。

图3 芯片上3种病原菌的探针特异性的检验结果Fig.3 Test results of probe specificity of three pathogens on chip

图4 模板浓度未被稀释的多重PCR产物与芯片杂交后的信号展示及信噪比折线图Fig.4 Display signals and line chart of SNR after hybridized within products of m-PCR and chip while not diluted
如图4所示为模板在未稀释时的多重PCR产物与芯片杂交后的信号展示图,其信噪比值很高,数据折线图也说明3种信号都为阳性,而且evpA的信号最强。当稀释倍数增至104时,aerA基因5个信号值的信噪比中出现0点,因此将25.6 pg视为嗜水气单胞菌的检测阈值,根据摩尔常数公式计算出拷贝数为5×103,如图5所示;同样的方式判读出对鳗弧菌toxR的检测阈值是1.254 pg,即3×102拷贝;但很奇怪的是直到稀释倍数增至106时,evpA基因的5个信号值的信噪比中才有0点出现,怀疑有引物二聚体的影响。通过煮沸引物的方法抑制引物二聚体的形成,以消除其在芯片杂交中的影响,重复试验后判定,迟缓爱德华氏菌最低检测阈值是0.258 pg,即6×101拷贝。利用多重PCR方法处理样品后,由此简型基因芯片进行检测,检测限比用1%琼脂糖凝胶电泳查看PCR产物的方式,提高了至少10倍。
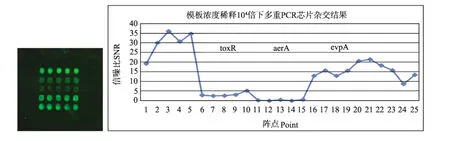
图5 模板被稀释104倍后的多重PCR产物与芯片杂交后的信号展示及信噪比折线图Fig.5 Display signals and line chart of SNR after hybridized within products of m-PCR and chip while diluted 104times
2.4 患病大菱鲆内脏组织中分离菌的鉴定
2种细菌的多重PCR产物与基因芯片杂交后的信号展示如图6-1和图6-2。图6-1中,09071501的PCR产物与芯片上toxR探针杂交后出现阳性信号,而与其他2种探针没有杂交信号;图6-2中,09071502的PCR产物与芯片上3个特征基因的探针都没有出现杂交信号。将两者的测序结果在GenBank中BLAST,与09071501 PCR产物的测序序列同源的有3条序列:AY065624、AY042547及AJ299739,且都为鳗弧菌toxR基因序列,同源率为98%~99%,可以确定扩增出的产物是鳗弧菌的toxR片段;09071502没有测序结果。这2种细菌是实验室谢国驷从某养殖场患病大菱鲆的内脏组织内分离出的,用16S rRNA片段测序、API细菌鉴定试剂条实验的结果显示09071501为鳗弧菌、09071502为交替假单胞菌,这与本研究中用基因芯片鉴定的结果相符,从而可以确定此患病大菱鲆内脏组织中感染有鳗弧菌,用这种基因芯片检测具有一定的特异性。

图6 患病鱼内脏组织中病原菌的芯片检测结果Fig.6 The microarray results of pathogens in visceral tissue of sick fish
3 讨论
一般细菌检测基因芯片的设计多采用细菌学上较保守的基因,比如被称为细菌进化的“活化石”16S rRNA基因及核糖体大亚单位23S rRNA等作为靶分子。但是16S rRNA和23S rRNA序列的保守性又是相对的,因为在此段序列上存在着每种细菌特异性的片段,就降低了rRNA用于鉴定和分类的精确度,所以特异性的靶分子有必要加入基因芯片的布局,用于病原菌的检测或毒性分析。西班牙学者选用7种染色体上的特异性基因及2种质粒上的特异性基因,通过多重PCR和短核苷酸探针构建成DNA芯片,可同时检测创伤弧菌、鳗弧菌、副溶血弧菌、杀鲑气单胞菌等5种海水鱼病原菌,灵敏度是琼脂糖电泳的4倍[11];许拉等利用弧菌的特异性保守基因toxR特异性地区分出水体中副溶血弧菌、创伤弧菌、哈维氏弧菌和溶藻胶弧菌等主要水产病原菌[12];Panicker等用多重PCR和DNA芯片的方法可以灵敏地检测和鉴定出墨西哥湾地区牡蛎的创伤弧菌、霍乱弧菌、副溶血弧菌[13]。
本试验选择鳗弧菌的保守基因toxR不仅可以调节毒力蛋白的表达,而且具有一定的种特异性,重要的是在它在鳗弧菌的强毒株及弱毒株中都可以检出[14];嗜水气单胞菌的aerA作为其主要的毒力基因,已经是该菌的快速诊断、流行病调查及公共卫生检测、检疫等方面的重要标准[15-16];迟缓爱德华氏菌的毒力机制比较复杂,这里选择的其中1个重要的分泌系统毒力蛋白编码基因evpA[17]。另外本研究构建的多重PCR体系不与弧菌属的其他几种弧菌、交替假单胞菌、对虾的基因组DNA等有扩增反应[10],而且构建的多重PCR-基因芯片不仅可用于3种病原菌的检测,将检测限提高至少10倍,还可用于它们的毒性分析,为临床上的3种病原菌的检测提供了一种可行的手段。
基因芯片检测技术一方面可以代替测序对PCR产物进行验证,以排除PCR的假阳性[18];另一方面可以提高检测的灵敏度,但是基因芯片还不能用于绝对定量。同时在构建此简型基因芯片的过程中,有些问题也凸显出来,如引物和探针的设计、PCR扩增、芯片的制备、杂交及杂交后的处理过程的各个环节都会影响基因芯片检测技术的稳定性及灵敏性。目前人们正在进行更多种病原菌的平行检测基因芯片技术的构建,相信随着技术的不断跟进,研究的不断深入,这些方面都将得到优化,并最终形成一种完整的、精确的、稳定的实用技术。
[1] 刘垣,郑文杰,刘伟,等.转基因番茄DNA检测芯片的研究[J].食品研究与开发,2005,26(4):109-112.
[2] 李谨.基因芯片技术的发展与应用[J].中国兽医杂志,2007,8(43):87-89.
[3] Warsen A E,Krug M J,LaFrentz S.et al.Simultaneous discrimination between 15 fish pathogens by using 16S ribosomal DNA PCR and DNA microarrays[J].App1 Environ Microbiology,2004,70(7):4216-4221.
[4] Georg M,Martin H,Ernst L,et al.Microarray-based identification of bacteria in clinical samples by solid-phase PCR amplification of 23S Ribosomal DNA sequences[J].Journal of clinical microbiology,2004,42(3):1048-1057.
[5] 许拉,黄倢,杨冰.病原检测基因芯片应用及在水产病害检测的前景[J].海洋水产研究,2008,29(1):109-114.
[6] 李国军,王卫华,李洪涛,等.细菌核糖体23S亚单位基因芯片在感染性疾病快速诊断中的应用研究[J].中华医院感染学杂志,2005,15(9):969-972.
[7] 高爽,谢明杰,金大智,等.运用基因芯片技术建立检测水产食品中常见病原微生物方法的研究[J].生物技术通讯,2007,18(1):072-076.
[8] 李天道,于佳,俞开康.四种弧菌对中国对虾的致病性研究[J].海洋湖沼通报,1998,1:57-64.
[9] 徐晓津,王军.我国养殖对虾细菌性疾病[J].河北渔业,2001,(3):22-25,41.
[10] 李晨,黄倢.三种主要水产病原菌多重PCR检测方法的建立[J].渔业研究进展,2010,31(3):100-106.
[11] Gonzalez S F,Krug M J,Nielsen M E,et al.Simultaneous detection of marine fish pathogens by using multiplex PCR and a DNA microarray[J].Clinica1 Microbiology,2004,42(4):14l4-1419.
[12] 许拉,黄倢,戈蕾,等.同时检测两种对虾病毒和4种弧菌的同步PCR方法的建立[J].海洋水产研究,2008,29(4):39-45.
[13] Panicker G,Call D R,Krug M J,et al.Detection of pathogenic vibrio spp.in sheltfish by using muhiplex PCR and DNA mieroarrays[J].App1 Environ.Microbio1,2004,70(12):7436-7444.
[14] Okuda J,Nakai T,Chang P S,et al.The toxR gene ofVibrio(Listonella)anguillarumcontrols expression of the major outer membrane proteins but not virulence in a natural host model[J].Infection and Immunity,2001,69(10):6091-6101.
[15] 朱大玲,李爱华,钱冬,等.嗜水气单胞菌毒力基因的研究进展[J].水生生物学报,2004,28(1):80-84.
[16] 李莲瑞,罗红斌,卢强,等.嗜水气单胞菌Aer毒素的研究进展[J].塔里木农垦大学学报,2004,16(3):46-51.
[17] Srinivasa P S,Rao Y Y,Yuen P T,et al.Use of proteomics to identify novel virulence determinants that are required for Edwardsiella tarda pathogenesis[J].MolecularMicrobiology,2004,53(2):573-586.
[18] 赵锦,耿艺介,何建凡,等.弓形虫乙肝病毒多重PCR及芯片杂交技术的研究[J].中国热带医学,2005,5(5):937-939.
Establishment of a Reduced Microarray for Detection of Three Aquatic Bacterial Pathogens
LI Chen1,2,HUANGJie1,XIE Guo-Si1,2,ZHAO Pei1,2,WAN G Xiu-Hua1
(1.Key Laboratory of Sustainable Utilization of Marine Fisheries Resources Certificated by the Ministry of Agriculture,Yellow Sea Fisheries Research Institute,Chinese Academy of Fishery Sciences,Qingdao 266071,China;2.College of Fisheries and Life Science,Shanghai Ocean University,Shanghai 200090,China)
Based on the reported information of three major aquatic pathogensVibrio anguillarum,Aeromonas hy drophila,andEdwardsiella tarda,three primer pairs and oligo probes were designed against the genetoxR,aerAandevpAto construct a simple and convenient multiplex PCR-microarray.The gene variable regionof shrimp white spot syndrome virus(WSSV)was used as the surface chemistry quality control on the microarray.The target products which were amplified by multiplex PCR were hybridized to the microarray.The optimized preparation protocol employed an Aldehyde slide and 20μmol/L aminogroup DNA probes in 50%DMSO to be spotted on the slide at 45%humidity.The prepared microarray slide was hybridized with fluorescent PCR products at 42℃for 2 h.The overall LDLs(lowest detection level)of the microarray were tested with the genomic DNA of three bacterial strains around 3×102copies ofV.anguillarum,5×103copies ofA.hy drophila,and 6×101copies ofE.tarda.The technology was successfully demonstrated in the identification of bacterial isolates from a diseased turbot.
microarray;aquatic bacterial pathogens;establishment
S917
A
1672-5147(2011)03-037-06
农业公益性行业科研专项(200803012);国家现代产业技术体系建设(nycytx-46);国家高技术研究发展计划项目(2006AA100306);基本科研业务费专项资金项目(2007-GY-03)资助
2010-06-03;
2010-07-14
李 晨(1984-),女,硕士生。E-mail:lichen1010@sina.com
**通讯作者:E-mail:huangjie@ysfri.ac.cn
责任编辑 于 卫

