光皮桦SSR分子标记体系的建立
尤卫艳,黄华宏,程龙军,童再康,朱玉球
(浙江林学院 林业与生物技术学院,浙江 临安 311300)
光皮桦Betula luminifera属桦木科Betulaceae桦木属Betula[1],为中国特有优良用材树种,天然分布于秦岭淮河流域以南的河南、四川、贵州、云南、安徽、湖北、湖南、广东、广西、江西、浙江、福建等10多个省(自治区)。其木材呈淡黄色或红褐色,材质细腻坚韧,切面光滑,不挠不裂,干燥性能良好,广泛应用于航空、建筑、家具、造纸等各行业,也可用于提取木醇和芳香油等物质。近年来光皮桦人工造林发展迅速,有关光皮桦的研究也不断增多。在分子方面,谢一青等[2-3]通过对比和优化实验,建立光皮桦DNA提取及其随机扩增多态性DNA(random amplified polymorphic DNA,RAPD)技术分析体系。同时陈伟[4]应用RAPD和简单序列重复区间扩增多态性(inter-simple sequence repeat,ISSR)技术对福建省光皮桦天然群体的的遗传多样性进行分析,发现11个群体明显分为两大类。童再康等[5]建立了光皮桦的扩增片段长度多态性(amplified fragment length polymorphism,AFLP)反应体系。简单重复序列[6](simple sequence repeat,SSR)或者微卫星序列[7](microsatellite,MS),是一类由几个到十几个核苷酸串联重复组成的DNA序列。其长度一般在100 bp以下,广泛分布于生物体基因组的不同位置,由于重复次数不同及重复程度不完全造成的每个位点的多态性。微卫星序列两端多是相对保守的单拷贝序列,因此可以根据两端序列设计特异引物对基因组DNA进行PCR扩增,扩增片段的长度多态性可用作分子标记。SSR标记具有共显性、高度重复性、高度丰富的多态性等优点,成为构建遗传连锁图谱[8],研究群体遗传学[9],进行分子标记辅助育种[10],绘制品种指纹图谱[11],检测品种纯度[12]等的理想工具。SSR标记的众多优点使它成为最理想的分子标记之一。本研究利用近缘种的引物在光皮桦中进行扩增,建立光皮桦的SSR反应体系,为应用SSR分子标记对光皮桦群体进行研究奠定基础。
1 材料与方法
1.1 材料与试剂
光皮桦材料来自浙江林学院智能温室,取正常生长的嫩叶,置-70℃冰柜保存备用。Taq DNA聚合酶(83.35 mkat·L-1),脱氧核糖核苷酸 dNTPs(各 2.5 mol·L-1),氯化镁 MgCl2(25.0 mol·L-1)等试剂均购于由宝生物工程(大连)有限公司,引物由GenScript公司合成。
1.2 方法
1.2.1 光皮桦DNA的提取和检测 本研究采用十六烷基三甲基溴化铵(CTAB)-硅珠吸附法提取光皮桦叶片DNA,用NanoDrop微量分光光度计(ND-1000)测定DNA的纯度和浓度。同时每个样品取5 μL,加1 μL上样缓冲液混合后,于10.00 g·L-1琼脂糖凝胶(含 0.05 g·L-1溴化乙锭)进行电泳检测,在凝胶成像系统(Gel DocTM,Bio-Rad)下观察并拍照。将样品稀释至20.0 mg·L-1,置于-20℃下保存。
1.2.2 PCR扩增及其产物检测 聚合酶链式反应(PCR)扩增在ABI 9700 PCR仪上进行。扩增程序为:94℃,2 min;94℃,30 s;51~60℃(各引物退火温度不同),30 s;72℃,30 s(30个循环);72℃,5 min。反应总体积为20 μL。体系建立过程中,分别对Mg2+浓度、dNTPs浓度、Taq DNA聚合酶浓度、引物浓度和模板DNA用量等设置不同的浓度梯度,以确定最佳反应体系。
PCR产物经15.00 g·L-1琼脂糖凝胶电泳检测,在凝胶成像系统(Gel DocTM,Bio-Rad)上观察、拍照和分析。
1.2.3 PCR反应因素水平的确定为了确定PCR反应中5个因素(模板DNA用量、dNTPs浓度、Mg2+浓度、引物浓度、Taq DNA聚合酶浓度)的水平,选用引物AB084473采用单因素法进行试验,实验重复2次)。PCR反应的因素水平见表1。
1.2.4 引物合成 SSR引物的序列来源于近缘种日本白桦Betula platyphylla var.japonica[13]和欧洲白桦 B.pendula[14]的 36 对 SSR 引物。
1.2.5 电泳检测 PCR扩增产物在80 g·L-1聚丙烯酰胺凝胶上电泳检测,染色方法参照胥猛[15]等的方法,功率恒为75 W,电泳1.0~1.5 h。

表1 聚合酶链式反应的因素水平Table1 Factors and levels of PCR
1.2.6 引物通用性研究 随机选取引物AF310851,AB084479,AB084480(GenBank accession number),PCR扩增产物在80.00 g·L-1聚丙烯酰胺凝胶上电泳,参照李明芳[16]的方法,割取多态性片段,进行扩增。扩增后在琼脂糖上电泳检测,用试剂盒回收扩增产物,4℃连接过夜,转入大肠杆菌Escherichia coli培养12 h以上,如蓝白斑区别不明显,4℃放置1 h,挑单克隆,培养8 h进行菌液PCR检测,测序。
2 结果和分析
2.1 不同DNA用量对SSR反应结果的影响
当其他条件一定 (0.200 mmol·L-1dNTPs,1.50 mmol·L-1Mg2+,2.50 mmol·L-1引物,1 × 16.67 nkat Taq酶),由图1可知,模板DNA用量为20或40 ng时,扩增出的条带较弱;而用量为60或80 ng时,有较好的结果;当用量为100 ng时,扩增条带反而变弱,可能是模板DNA浓度较大,影响反应体系浓度,进而影响PCR反应等因素造成的。为了节约试剂和减少非特异性条带的产生,所以本实验选用的模板DNA用量为60 ng。
2.2 不同dNTPs浓度对结果的影响
当其他条件一定(模板 DNA 用量为 60 ng,1.50 mmol·L-1Mg2+,2.50 mmol·L-1引物,1 × 16.67 nkat Taq酶),6个dNTPs浓度均可扩增出条带。图2显示,当dNTPs浓度为0.125 mmol·L-1时,扩增出的条带较弱;而dNTPs浓度为0.200,0.175,0.150 mmol·L-1时,有较好的结果;当浓度为0.250和0.225 mmol·L-1时,扩增条带反而变弱,可能是扩增的片段较小,当dNTPs浓度增大时扩增产物不随之增加造成的。体系选用0.175 mmol·L-1为dNTPs最终浓度。
2.3 不同Mg2+浓度对结果的影响
当其他条件一定(模板 DNA 用量为 60 ng,0.175 mmol·L-1dNTPs,2.50 mmol·L-1引物,1 × 16.67 nkat Taq酶),Mg2+浓度对PCR反应有很大影响,Mg2+浓度过高容易引起非特异性扩增,导致阳性克隆较多,影响数据分析结果。当Mg2+浓度为1.00和1.25 mmol·L-1时,扩增条带很弱;而Mg2+浓度为1.50 mmol·L-1时,有较好的结果;当浓度为1.75 mmol·L-1时,扩增主条带反而变弱;当Mg2+浓度为2.00和2.25 mmol·L-1时,主条带亮度又增强,可能此时已经在进行非特异片段的扩增(图3)。所以选用 Mg2+最终浓度为 1.50 mmol·L-1。
2.4 不同引物浓度对结果的影响
当其他条件一定(模板 DNA 用量为 60 ng,0.175 mmol·L-1dNTPs,1.50 mmol·L-1Mg2+,1 × 16.67 nkat Taq酶),引物浓度为1.00,1.50,2.00 mmol·L-1时,扩增的条带极其微弱,几乎检测不到;当引物浓度增加到 2.50和 3.00 mmol·L-1时,条带亮度增高(图 4)。所以选用 2.50 mmol·L-1为最终浓度。
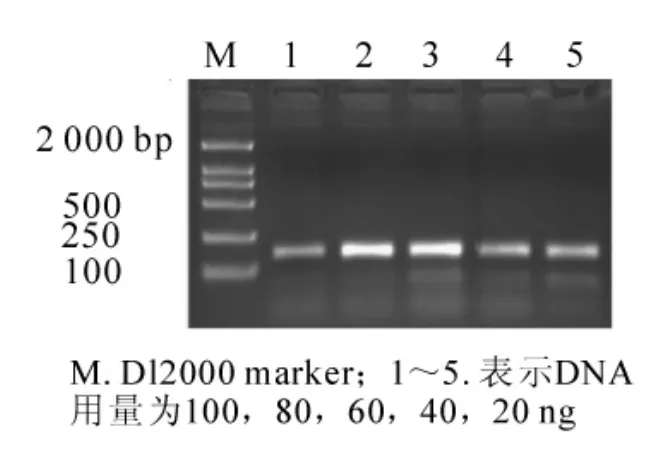
图1 DNA用量对SSR反应结果的影响Figure1 Effects of DNA dosages on result of SSR reaction
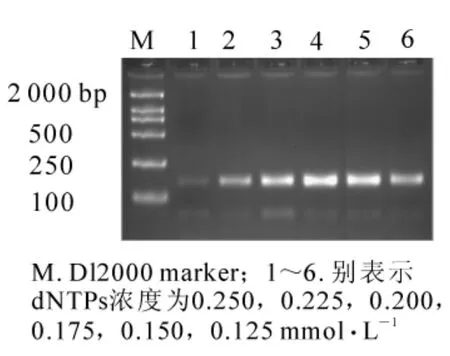
图2 不同dNTPs浓度对SSR反应结果的影响Figure2 Effects of different dNTPs concentrations on result of SSR reaction
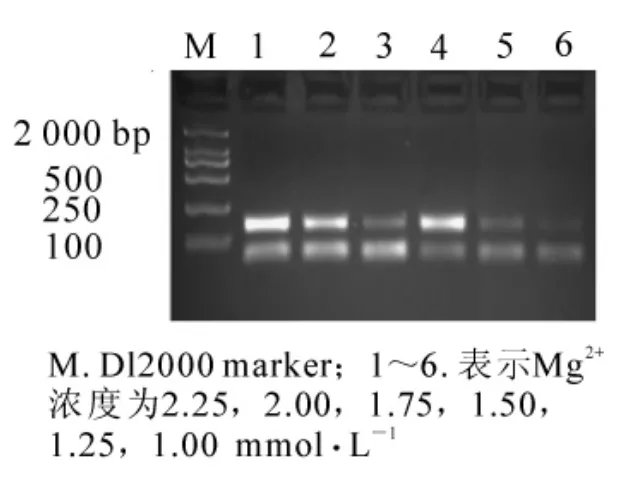
图3 不同Mg2+浓度对SSR反应结果的影响Figure3 Effects of different Mg2+concentrations on result of SSR reaction

图4 不同引物浓度对SSR反应结果的影响Figure4 Effects of different primers concentrations on result of SSR reaction
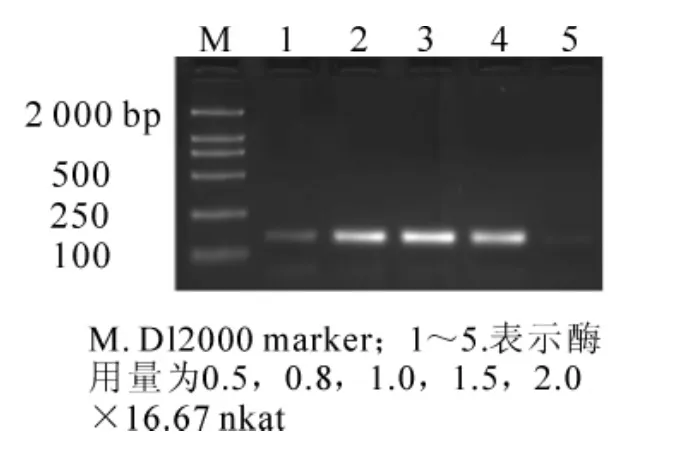
图5 不同酶用量对SSR反应结果的影响Figure5 Effects of different enzyme dosages on result of SSR reaction
2.5 不同酶用量对结果的影响
当其他条件一定(模板 DNA 用量为 60 ng,0.175 mmol·L-1dNTPs,1.50 mmol·L-1Mg2+,2.50 mmol·L-1引物),Taq DNA聚合酶用量是PCR反应中最为重要的影响因子之一。Taq DNA聚合酶浓度高时,极易产生非特异性扩增。如图5所示,5个处理均有扩增产物,但当Taq DNA聚合酶浓度为0.5×16.67 nkat时,扩增条带很弱;浓度0.8×16.67,1.0×16.67,1.5×16.67 nkat时,谱带亮且清晰;2.0×16.67时,条带反而变弱。因此,确定Taq DNA聚合酶适宜用量为16.67 nkat。
2.6 退火温度的影响
退火温度的高低与PCR扩增特异性有显著的相关性,而退火温度主要取决于引物的解链温度(melting temperature,Tm)值、引物与模板的配对程度等因素。当温度较低时,非特异性条带增加,退火温度较高时,条带变少(图6)。所以体系选用的退火温度比在日本白桦(其引物退火温度为56℃)中提高了1℃。
2.7 欧洲白桦和日本白桦引物通用性检验

图6 部分引物的在不同退火温度下扩增产物在80 g·L-1聚丙烯酰胺凝胶上电泳检测图Figure6 Amplified product of some pairs of primer at different annealing temperature detecting in 80 g·L-1PAGE

表2 引物AF310851,AB084479,AB084480序列在GeneBank比对结果Table2 Blast results of AF310851,AB084479,AB084480 in GeneBank
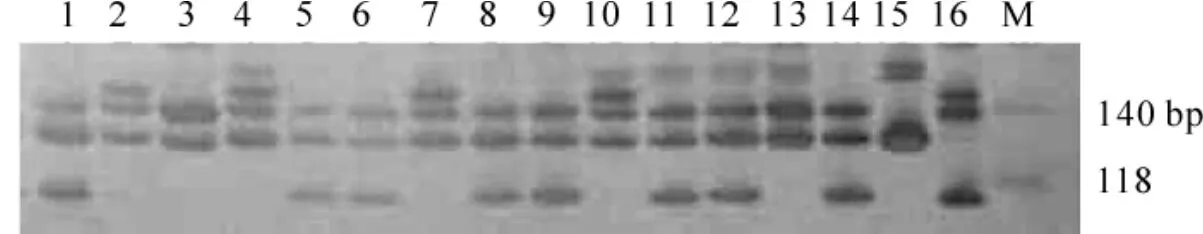
图7 引物AB084473扩增产物在80 g·L-1聚丙烯酰胺凝胶上电泳检测图Figure7 Amplified product of AB084473 detecting in 80 g·L-1PAGE
克隆序列经比对后,与原物种序列相似度(max ident值)都在95%以上(表2),说明欧洲白桦和日本白桦的引物可以应用于光皮桦。体系建立后用引物AB084473对光皮桦16个植株的DNA进行扩增,得到较好的扩增结果(图7)。
3 结论与讨论
通过单因素法确定了光皮桦SSR反应体系中模板DNA用量、Mg2+浓度、dNTPs浓度、引物浓度、Taq DNA聚合酶浓度,建立光皮桦SSR反应体系。在体系建立过程中,当各个因素水平较低或较高时,条带都比较弱,这与柳晓磊等[17]对椰子Cocos nucifera的研究结果一致。其中退火温度是影响PCR反应的重要因素之一,实验结果表明:较小的温度变化对最后的扩增结果影响不明显,但较大的温度跨度,影响扩增条带的多寡及其清晰程度。所选的SSR引物各解链温度值相差很大,最大跨度达10℃,特别需要对每一对引物进行梯度退火实验,以期得到各引物最佳退火温度。
SSR分子标记因具有诸多优点,成为现代分子生物学最理想的标记之一,但其昂贵的引物开发费用,令人望而却步。而微卫星序列的又具有保守性,使其引物在近缘中可以通用。同时在植物分类学上,光皮桦、日本白桦和欧洲白桦同属于桦木科桦木属,亲缘关系较近,微卫星序列也趋于一致,故日本白桦和欧洲白桦的SSR引物在光皮桦中应具有较高通用性。本研究结果也表明:3个随机选取的SSR引物在光皮桦中的扩增序列,与原物种白桦的扩增序列经比对,最大相似度值均达到95.0%以上。
[1]郑万钧.中国树木志:第2卷[M].北京:中国林业出版社,1985:2124-2131.
[2]谢一青,李志真,黄儒珠,等.光皮桦基因组DNA提取方法的比较[J].浙江林学院学报,2006,23(6):664-668.XIE Yiqing,LI Zhizhen,HUANG Ruzhu,et al.Comparison of methods of extracting genomic DNA from Betula luminifera [J].J Zhejiang For Coll,2006,23 (6):664-668.
[3]谢一青,黄儒珠,李志真,等.光皮桦RAPD分析体系优化设计方案比较[J].福建林学院学报,2006,26(1):73-77.XIE Yiqing,HUANG Ruzhu,LI Zhizhen,et al.Comparison between single factor design and uniform design to optimize the reaction system for RAPD of Betula luminifera [J].J Fujian Coll For,2006,26 (1):73 - 77.
[4]陈伟.光皮桦天然群体遗传多样性研究[J].北京林业大学学报,2006,28(6):28-34.CHEN Wei.Genetic diversity of the natural populations of Betula luminifera [J].J Beijing For Univ,2006,28 (6):28-34.
[5]尤卫艳,黄华宏,童再康,等.光皮桦AFLP分子标记体系的建立[J].生物技术,2008,18(6):42-45.YOU Weiyan,HUANG Huahong,TONG Zaikang,et al.Establishment of AFLP molecular labeling technique system for Betula luminifera [J].Biotechnology,2008,18 (6):42 - 45.
[6]FRAGEAU C J,FOURNEY R M.DNA typing with short tandem repeat:A sensitive and accurate approach to human identification[J].Biotchniques,1993,15:100 - 119.
[7]SHARON D,ADATO A,MHAMEED S,et al.DNA fingerprint in plant using simple sequence repeat and minisatellite probes [J].HortScience,1995,30 (1):109 - 112.
[8]宋健,韩明玉,赵彩平,等.桃‘秦光2号’ב曙光’F1代SSR遗传连锁图谱的构建[J].西北植物学报,2008,28(5):895 - 900.SONG Jian,HAN Mingyu,ZHAO Caiping,et al.Construction of a general genetic linkage map for peach using a‘Qinguang 2’ × ‘Shuguang’ F1 progeny by SSR markers [J].Acta Bot Boreal-Occident Sin,2008,28 (5):895 - 900.
[9]徐小林,徐立安,黄敏仁,等.栓皮栎天然群体SSR遗传多样性研究[J].遗传,2004,26(5):683-688.XU Xiaolin,XU Li’an,HUAN Minren,et al.Genetic diversity of microsatellites (SSRs) of natural populations of Quercus variabilis [J].Hereditas,2004,26 (5):683 - 688.
[10]黄烈健,苏晓华,张香华.与杨树木材密度、纤维性状相关的SSR分子标记[J].遗传学报,2004,31(3):299-304.HUANG Liejian,SU Xiaohua,ZHANG Xianghua,et al.SSR molecular markers related to wood density and fiber traits in poplar [J].Acta Gen Sin,2004,31 (3):299 - 304.
[11]艾呈祥,张力思,魏海蓉.甜樱桃品种SSR指纹图谱数据库的建立[J].中国农学通报,2007,23(5):55-58.AI Chengxiang,ZHANG Lisi,WEI Hairong,et al.Construction of molecular fingerprinting database for sweet cherry using SSR markers [J].Chin Agric Sci Bull,2007,23 (5):55 - 58.
[12]李菊芬,许玲,马国斌.应用SSR分子标记鉴定甜瓜杂交种纯度[J].农业生物技术学报,2008,16(3):494-500.LI Jufen,XU Ling,MA Guobin.Identification of melon hybrid purity by SSR markers [J].J Agric Biotechnol,2008,16 (3):494 - 500.
[13]WU B,LIAN C,HOGETSU T.Development of microsatellite markers in white birch(Betula platyphylla var.japonica)[J].Mol Ecol Notes,2002,2:413 - 415.
[14]KULJU K K,PEKKINEN M,VARVIO S.Twenty-three microsatellite primer pairs for Betula pendula (Betulaceae)[J].Mol Ecol Notes,2004,4:471 - 473.
[15]胥猛,李火根.鹅掌楸EST-SSR引物开发及通用性分析[J].分子植物育种,2008,6(3):615-618.XU Meng,LI Huogen.Development and characterization of microsatellite markers from expressed sequence tags for Liriodendron [J].Mol Plant Breed,2008,6 (3):615 - 618.
[16]李明芳.荔枝SSR标记的研究及其对部分荔枝种质的遗传多样性[D].儋州:华南热带农业大学,2003.LI Mingfang.Development of SSR Markers in Litehi chinensis and Application for Analysis of Genetic Diversity within Litchi Varieties [D].Danzhou:South China University of Tropical Agriculture,2003.
[17]柳晓磊,汤华,李东栋,等.椰子SSR反应体系的建立和优化[J].华中农业大学学报,2007,26(5):676-679.LIU Xiaolei,TANG Hua,LI Dongdong,et al.Optimization and establishment of SSR analysis method in coconut [J].J Huazhong Agric Univ,2007,26 (5):676 - 679.

