中国陆生蓝藻新记录属
摘 要:蓝藻具较强的适应能力,不仅能生活在淡水和海洋等各种水体生境中,也能在土壤、沙漠、极地、温泉等极端生境中生存.通过野外样品采集,从中国广西壮族自治区桂林市九马画山生物土壤结皮中成功分离到1株蓝藻藻株JM-1001,采用形态学、生态学和分子生物学相结合的多相分析方法对其进行了系统发育分析和分类特征研究.该藻株为单细胞、丝状体或群体生活,具有明显的生活史,具异形胞和厚壁孢子,与科马雷克藻属Komarekiella形态特征极其相似.基于16S rRNA基因系统发育分析表明,该藻株聚集在科马雷克藻属支系中,并且JM-1001和大西洋科马雷克藻Komarekiella atlantica的序列紧密聚为一个系统进化分支,具有很高的步展值(98/93/1).藻株JM-1001与模式种大西洋科马雷克藻K. atlantica的16S rRNA基因相似性99.9%.同时,藻株JM-1001和大西洋科马雷克藻ITS二级结构中的D1-D1′、Box-B和V3螺旋结构一致.这些结果共同支持了本研究分离的藻株属于模式种大西洋科马雷克藻,该属是中国首次发现的新记录属.
关键词:蓝藻;新记录属;系统发育;分类
中图分类号:X52;Q949.22""""" 文献标志码:A文章编号:1000-2367(2025)02-0073-09
蓝藻是一类古老的光合放氧原核生物,为地球有氧环境做出巨大的贡献,在地球生命进化过程中扮演着重要角色[1].蓝藻是许多生态系统的重要组成部分,其适应性很强,能够在淡水、海洋甚至是极端生境中生存[2].正是蓝藻这种较强的适应能力、巨大的扩散能力和较快的生长速度[3],使其在生物土壤结皮和亚氧湿壁组合中也频繁出现[4].
在过去的20多年里,基于多相分析方法,蓝藻分类学研究发现了比以前更多的属,如果仅根据藻株的形态结构特征评估,那些最新描述的属都将被错误地认定为传统属的成员[5].近年来的研究中,DNA序列及其二级结构在鉴定蓝藻类群和解决其系统发育关系等问题中起到了关键作用.16S rRNA基因序列是区分蓝藻属或种的重要标识,也是重组和修订整个蓝藻分类体系的要素[6].但是,单靠该基因也并不能充分地在物种水平上解决蓝藻系统发育问题[7-9],ENGENE等[8]也发现了16S rRNA基因内的差异性并不能作为蓝藻分类的绝对标准,也不能准确反映蓝藻内的物种形成.因此,16S-23S ITS作为新的基因组位点被应用于系统发育树的构建和ITS区域二级结构的分析中[10].许多研究证实了16S rRNA基因系统发育和16S-23S ITS二级结构相结合的综合性方法对于复杂的蓝藻分类系统来说是非常有用和可靠的[11-12].目前,应用分子系统发育学、细胞形态学和生态学等相结合的多相分析法进行蓝藻新类群的鉴定已成为蓝藻分类学研究的最佳
收稿日期:2023-11-07;修回日期:2023-12-15.
基金项目:国家自然科学基金(32000166).
作者简介:陈嘉欣(1999-),女,河南南阳人,武汉轻工大学硕士研究生,研究方向为蓝藻的分类与系统发育学研究,E-mail:2407525663@qq.com.
通信作者:蔡芳芳(1991-),女,湖北黄冈人,武汉轻工大学讲师,博士,研究方向为蓝藻的系统发育、分子进化与生态学,蓝藻有害产物的研究,E-mail:fangfangcai@whpu.edu.cn.
引用本文:陈嘉欣,席雨,李姝衡,等.中国陆生蓝藻新记录属——科马雷克藻属(Komarekiella)[J].河南师范大学学报(自然科学版),2025,53(2):73-81.(Chen Jiaxin,Xi Yu,Li Shuheng,et al.Komarekiella(Nostocale,Cyanobacteria):a terrestrial novel recorded genus from China[J].Journal of Henan Normal University(Natural Science Edition),2025,53(2):73-81.DOI:10.16366/j.cnki.1000-2367.2023.11.07.0002.)
方法[13-15].
科马雷克藻属(Komarekiella)隶属于念珠藻目(Nostocales),念珠藻科(Nostocaceae)[16].科马雷克藻属在形态特征上与念珠藻属相似,都具有异形胞和厚壁孢子的分化,藻丝聚集形成群体.但是系统发育分析发现科马雷克藻属与念珠藻属在系统进化树上不聚集在同一个分支,亲缘关系相对较远,因此,HENTSCHKE等[17]把科马雷克藻属Komarekiella从念珠藻属中分离出来,形成了一个新的蓝藻属.科马雷克藻属的模式种是大西洋科马雷克藻Komarekiella atlantica,是SCOTTA等[17]在2017年从大西洋热带雨林(巴西沿海森林)的树木上首次分离出来的.此模式种的特征如下:藻体最初呈亮蓝绿色,随着增长逐渐变成较暗的蓝绿色,并由许多不规则藻丝体聚集组成,长度可达1 cm;起初藻丝体排列不可见,后来慢慢明显且分散开来形成多列藻丝体,宽度可达10 μm;成熟的藻丝体再聚集变成球形或压缩成亚球形,在一个以上的平面上进行分裂,平铺开来.目前已报道的科马雷克藻属有5个物种[15],分别是Komarekiella atlantica[17],K. chia[18],K. delphini-convector[19],K. globosa[20],K. gloeocapsoidea[20].Komarekiella atlantica发现于大西洋热带雨林(巴西沿海森林),K. chia发现于希腊俄斯岛洞穴内,K. delphiniconvector发现于美国佛罗里达州杰克逊维尔的圣约翰河,K. globosa发现于西班牙加那利群岛、K. gloeocapsoidea发现于捷克某地区的岩石上,这5个种均发现于国外,而国内目前并无该属的相关报道.
本研究从中国广西壮族自治区桂林市九马画山采集并分离到1株蓝藻藻株,在形态特征上与科马雷克藻属的物种相似.基于16S rRNA基因序列相似性和系统发育分析表明,该藻株与科马雷克藻属的模式种聚为紧密的一个系统进化分支,且基因序列相似性99.9%;进一步通过ITS二级结构对比分析,确定该藻株为科马雷克藻属的模式种:大西洋科马雷克藻Komarekiella atlantica.这也是我国首次对科马雷克藻属的报道.
1 材料与方法
1.1 样品采集与藻株的分离、纯化与培养
1.1.1 藻株样品的采集
2023年5月,在中国广西壮族自治区桂林市九马画山潮湿的山体墙壁上(110.496 821°E,24.915 794°N)用镊子刮取,采集到该藻株的陆生样品,然后将样品放入滤纸袋内保存,运回实验室.将陆生样品放在50 mL容量瓶中复苏.
1.1.2 藻株样品的分离纯化与培养
首先将采集到的藻株样品置于光学显微镜(NEXCOPE,Ningbo,China)下观察,待藻体或者藻丝分散开来后,用经典的毛细管分离法[21]分离纯化单根藻体或藻丝,然后转移至装有无菌BG11液体培养基[22]的24孔培养板中进行培养.待藻丝长成肉眼可见的群体时,置于光学显微镜(NEXCOPE,Ningbo,China)下观察,若为纯藻株,即可将24孔培养板中的样品转移至装有15 mL无菌BG11培养基的螺旋管中培养.24孔培养板和螺旋管的培养条件均为:培养温度25 ℃,光照周期12h∶12h,光照强度2 000 lx.获得的纯藻株编号为JM-1001,保存于武汉轻工大学藻类实验室.
1.2 藻株的形态学观察
待藻株生长至对数生长期,具有一定的生物量时,即可进行形态学观察.藻株于光学显微镜(NEXCOPE,Ningbo,China)下观察,用自带的拍照软件拍摄获得该藻株的显微照片,同时用自带的辅助软件(Imageview)测量并记录细胞的形态特征数据,如厚壁孢子、营养细胞、异形胞的长与宽,进行60~100次测量.
1.3 DNA提取、PCR扩增、克隆及测序
分离得到的藻株采用改良后的十六烷基三甲基溴化铵(CTAB)法[23]进行 DNA的提取.16S rRNA基因和16S-23S ITS片段的扩增引物为 PA(5′-AGAGTT-TGATCCTGGCTCAG-3′)和B23S(5′-CTTCGCCTCTGTGTGCCTAGGT-3′)[24-25].PCR扩增体系为:25 μL Premix Taq,2 μL模板DNA,1 μL上游引物,1 μL下游引物,最后加入ddH2O至50 μL.PCR扩增程序为:在95 ℃预变性3 min;94 ℃变性30 s,58 ℃退火30 s,72 ℃延伸2 min,共34个循环反应.PCR产物通过质量分数1%琼脂糖凝胶电泳分析,目的条带使用胶纯化试剂盒(Omega,USA)进行纯化回收,然后将得到的PCR反应产物用于克隆测序.
1.4 16S rRNA基因序列比对与系统发育分析
获得的序列首先在NCBI网站上进行BLAST对比分析,检测序列是否正确扩增为所需要的目的片段.为进一步确定藻株JM-1001的系统发育位置,将目的序列与NCBI网站上下载的念珠藻科的其他种的相关序列进行比对分析,使用MAFFT v7.312[26]将序列对齐,并使用MEGA v.7.0[27]手工检查并校正,校正后的序列用于系统进化树的构建.
用于系统发育分析的类群包括86条序列,选择Gloebacter vioiaceus PCC8105作为外类群.系统进化树的构建采用邻接法(neighbor joining,NJ),最大似然法(maximum likelihood,ML)和贝叶斯推理法(bayesian inference,BI).其中ML基于GTR+I+G 核酸替代模型,采用PAUP v4.0b10[28]软件构建.BI分析使用软件CIPRES Science Gateway V.3.3[29]构建.最后系统进化树使用软件TreeView 1.6.6进行显示,用Adobe Photoshop CS6 V 13.0进行编辑.16S rRNA的基因序列用MEGA v.7.0[27]软件进行计算,确定16S rRNA数据的序列一致性(100×(1-p)).
藻株JM-1001的16S rRNA和16S-23S ITS基因序列已上传NCBI GenBank数据库,登录号为OR758489,OR758490,OR763295,OR763296.
1.5 16S-23S ITS二级结构分析
选取所研究藻株的16S-23S ITS进行二级结构的分析.采用RNA structure ver.6.4软件[30]测定该藻株的D1-D1′,Box-B和V3螺旋的ITS二级结构.并与该属的2个种类的二级结构进行比较分析.二级结构是在理想条件下创建的,默认温度固定在37 ℃,结构分配为untangle loop fix.然后使用Adobe Acrobat DC重新绘制二级结构.
2 结 果
2.1 形态学观察
科马雷克藻属(念珠藻目,念珠藻科).
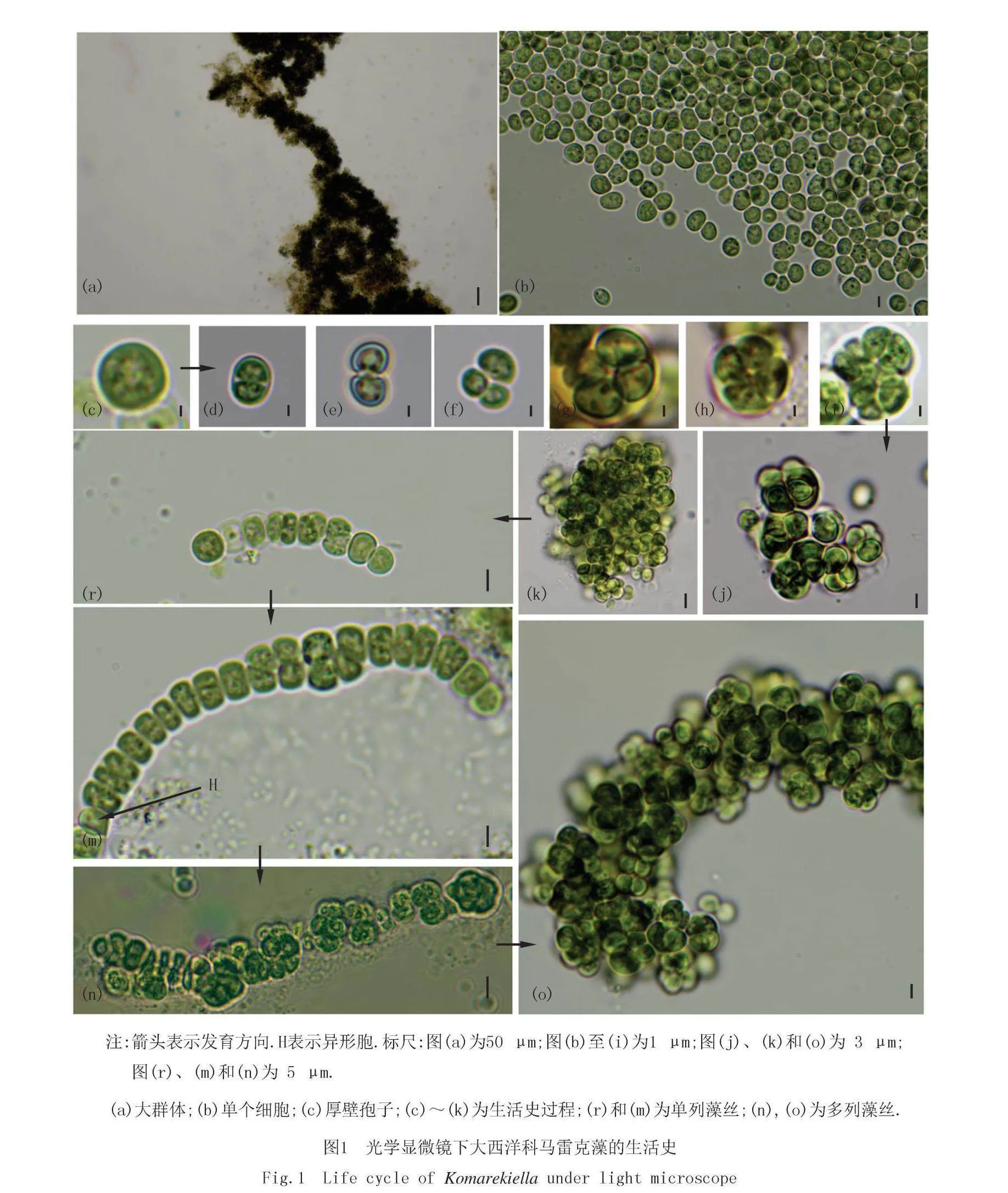
大西洋科马雷克藻Komarekiella atlantica Hentschke,G. S.,Johansen,J. R. et Sant′Anna,C. L. 2017(图1).
形态描述:单细胞、丝状体或群体.群体肉眼可见,小型群体聚集形成大的不规则群体.藻丝颜色最初呈蓝绿色,随着藻丝生长逐渐变成较暗的蓝绿色,生活史前期藻丝细胞单列、双列或多列排列,宽度可达5~10 μm;后期藻丝紧密聚集形成群体.群体起初可看到多列藻丝,后逐渐无法分辨单个细胞和单列藻丝.成熟的藻丝再变成球形或压缩成亚球形细胞,在1个以上的平面上进行分裂,营养细胞球形或半球形,直径为3.4~5.5 μm.异形胞直径为2.0~3.0 μm,厚壁孢子直径为3.5~5.0 μm.
实验藻株:JM-1001.
采样地区:中国广西壮族自治区桂林市九马画山.
生境:在潮湿的山体壁上自由生活.
标本号:WHPU-GXGL202305.
2.2 系统发育分析
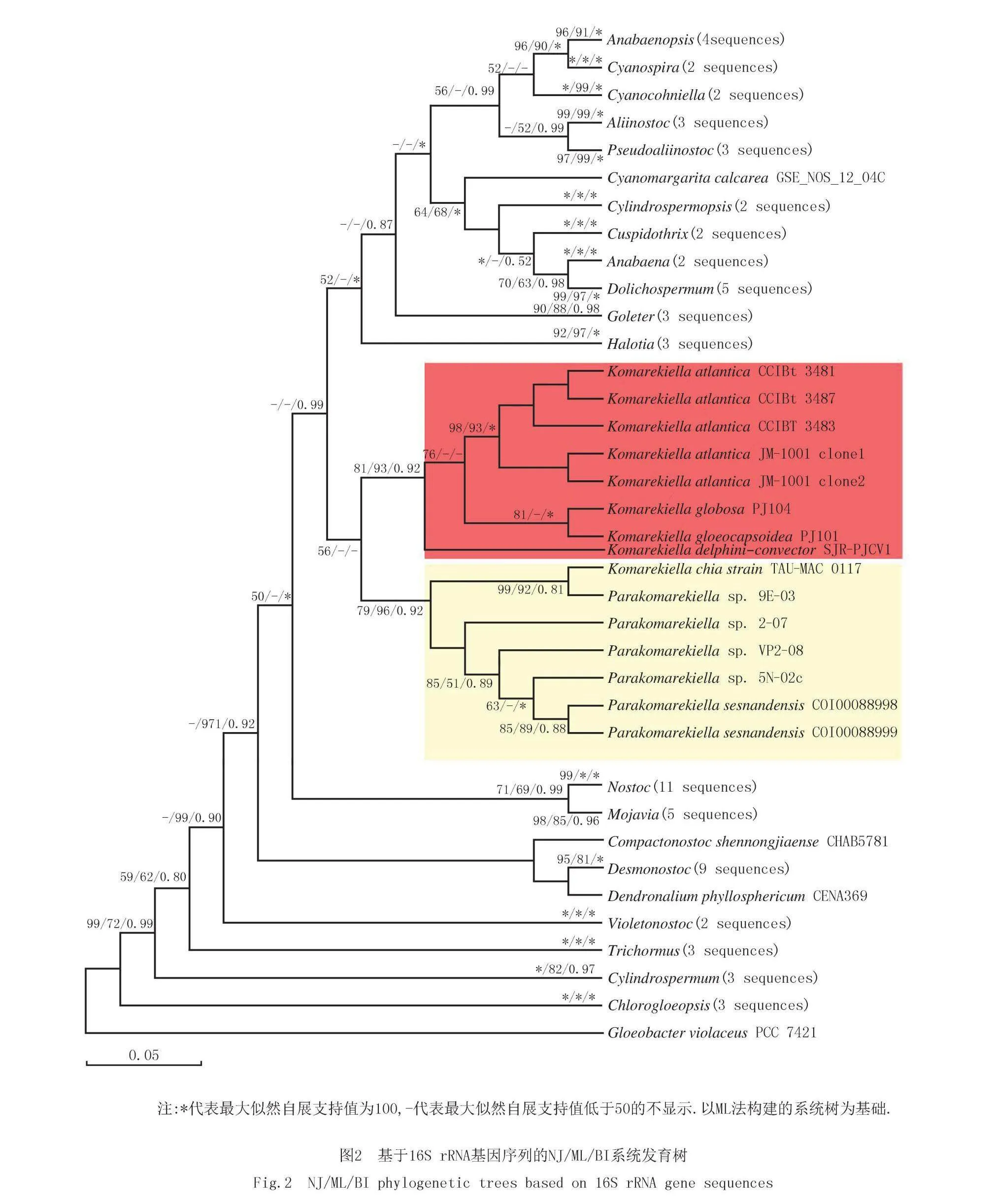
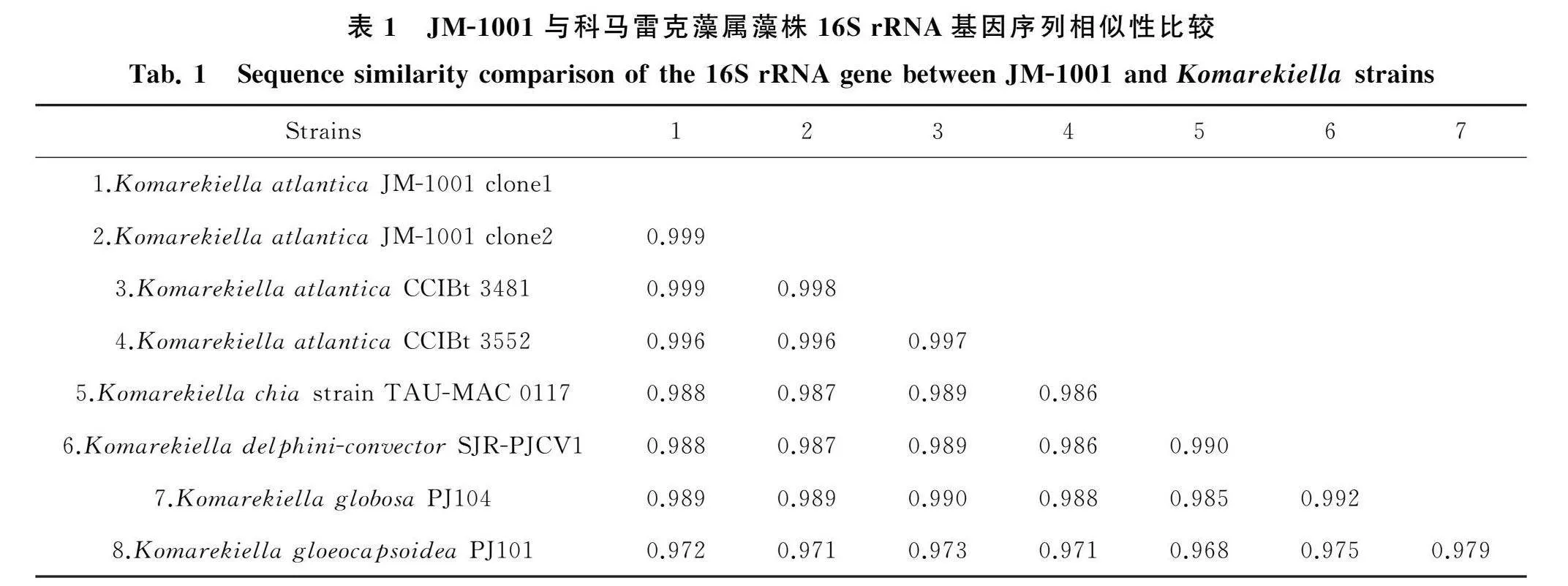
通过测序,获得本研究藻株16S rRNA基因的2个克隆序列,全长分别为1 484 bp和1 483 bp.结果显示,藻株与科马雷克藻属下的5个种基因序列相似性为96.8%~99.9%,与模式种K. atlantica藻株16S rRNA 基因序列相似性高达99.9%(表1).
基于邻接法、最大似然法和贝叶斯法3种方法构建的16S rRNA基因系统发育树(图2)可知,藻株JM-1001与科马雷克藻属下的K. atlantica、K. globosa、K. gloeocapsoidea和K. delphini-convector的序列聚为1个系统进化分支,具有较高的自展支持值(81/93/0.92),说明藻株JM-1001属于科马雷克藻属的类群.藻株JM-1001与模式种K. atlantica在系统进化树中具有更近的亲缘关系,自展支持率更高(98/93/1),说明JM-1001属于模式种K. atlantica.
2.3 16S-23S ITS二级结构分析
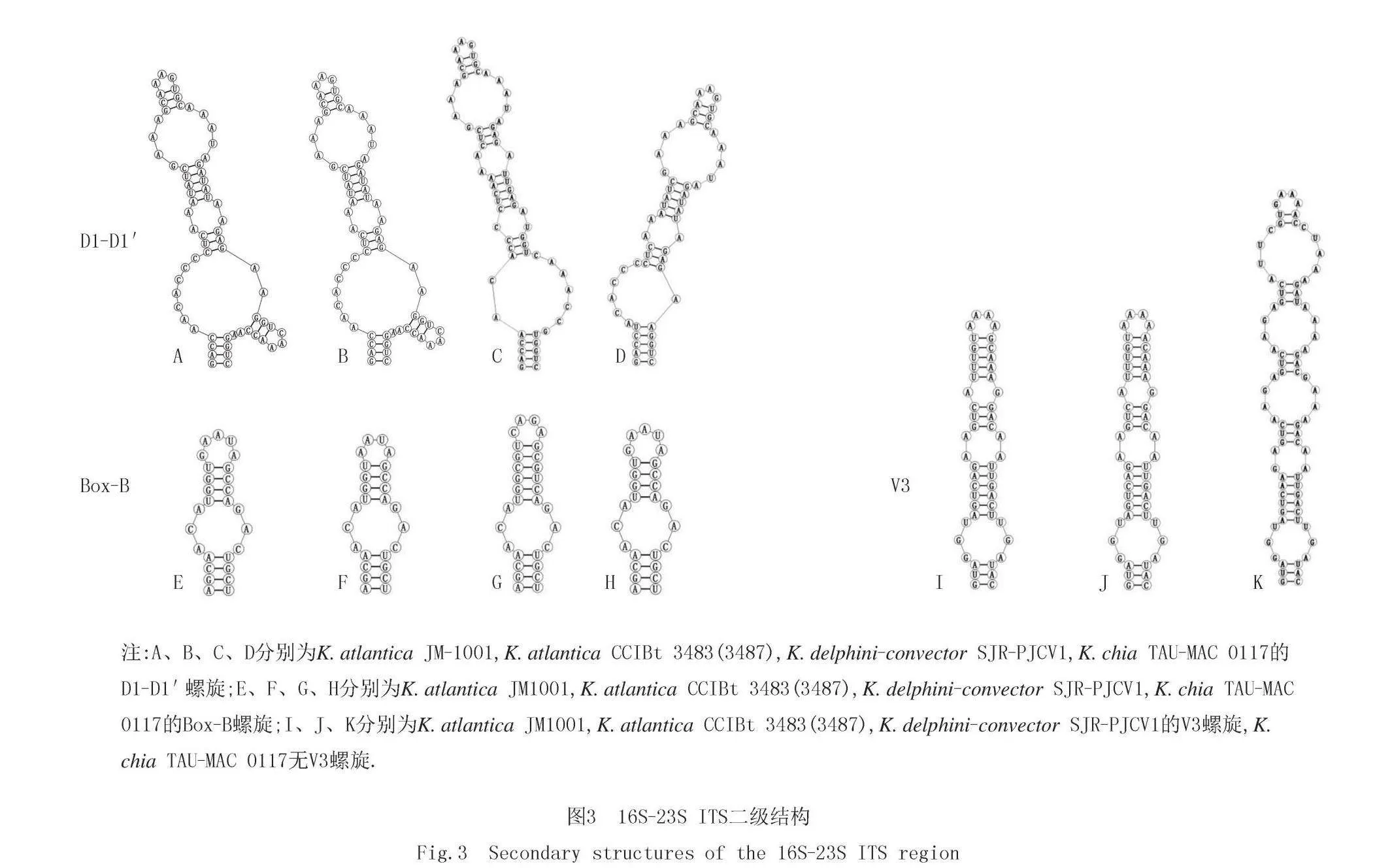
将本研究的藻株JM-1001,与K. atlantica CCIBt 3483、K. delphini-convector SJR-PJCV1和K. chia TAU-MAC 0117(无V3螺旋)的ITS二级结构进行比较分析(图3).因K. globosa和K. gloeocapsoidea没有16S-23S ITS基因序列,相关文献也未记录ITS二级结构,所以没有与这2个种的ITS二级结构进行比较分析.
D1-D1′螺旋分析表明(图3),实验藻株JM-1001的D1-D1′'螺旋有66个核苷酸,基部茎包含1个4 bp的螺旋,紧接着为一个右侧带有9 bp茎环结构的双侧凸起,1个双侧对称凸起和1个4∶5的双侧凸起,末端为3 bp的环状结构.K. atlantica CCIBt3483和K. atlantica CCIBt3487的D1-D1′'螺旋也有66个核苷酸,基部茎包含1个4 bp的螺旋,紧接着为一个右侧带有9 bp茎环结构的双侧凸起,1个双侧对称凸起和1个4∶5的双侧凸起,末端为3 bp的环状结构.与实验藻株JM-1001的D1-D1′螺旋一致.K. delphini-convector SJR-PJCV1的D1-D1′螺旋有65个核苷酸,基部茎包含1个5 bp的螺旋,其上为1个2∶7的双侧凸起,紧接着为1个1∶2的双侧凸起和1个2∶1的双侧凸起,其后是1个4∶5的双侧凸起,末端为3 bp的环状结构.与实验藻株JM-1001的D1-D1′螺旋的基部碱基对有差别.K. chia TAU-MAC 0117的D1-D1′螺旋有54个核苷酸,基部茎包含1个5 bp的螺旋,其上为1个6∶1的双侧凸起,1个2∶1的双侧凸起,紧接着为1个4∶5的双侧凸起,末端为3 bp的环状结构.与实验藻株JM-1001的D1-D1′螺旋的差别较大.
Box-B螺旋分析表明(图3),实验藻株JM-1001的Box-B螺旋有27个核苷酸,基部茎包含1个4 bp的螺旋,其后为1个3 bp双侧对称凸起,紧接着为1个4 bp螺旋,末端含有1个5 bp环状结构(GAAUA).K. atlantica CCIBt 3483和K. atlantica CCIBt 3487的Box-B螺旋有26个核苷酸,基部茎包含1个4 bp的螺旋,紧接着为1个3 bp双侧对称凸起和1个4 bp螺旋,末端为1个4 bp环状结构(AAUA).K. delphini-convector SJR-PJCV1的Box-B螺旋有30个核苷酸,基部茎包含1个4 bp的螺旋,紧接着为1个3 bp双侧对称凸起和1个6 bp螺旋,末端为1个4 bp环状结构(CAGA).K. chia TAU-MAC 0117的Box-B螺旋有27个核苷酸,基部茎包含1个4 bp的螺旋,紧接着为1个3 bp双侧对称凸起和1个4 bp螺旋,末端为1个5 bp环状结构(GAAUA).
V3螺旋分析表明(图3),实验藻株JM-1001的V3螺旋的基部茎包含1个3 bp螺旋,其后紧接着3个双侧对称凸起,末端环状结构含有4 bp.K. atlantica CCIBt 3483和K. atlantica CCIBt 3487的V3螺旋的基部茎也包含1个3 bp的螺旋,紧接着为3个双侧对称凸起,末端为1个4 bp环状结构.K. delphini-convector SJR-PJCV1的V3螺旋的基部茎包含1个3 bp的螺旋,紧接着为4个双侧对称凸起和1个4∶5的双侧凸起,末端含1个4 bp环状结构.K. chia TAU-MAC 0117无V3螺旋.
3 讨 论
蓝藻是生物土壤结皮群落的主要组成部分,它们是土壤表层的第一批栖息者[31],为创造土壤稳定环境并为下一个生物的产生提供条件[32].研究表明,形态上相似的蓝藻并不属于同一个类群,而且其命名方式也不符合“藻类、真菌和植物国际命名规则”[33].近年来,随着分子生物学的不断发展,利用16S rRNA基因描述新的蓝藻分类群变得越来越广泛,出现了许多新属和新种,从目到属和种,揭示了蓝藻的遗传多样性远高于其形态多样性[5].通过蓝藻新类群的不断发现或对一些已知类群的重新解释,蓝藻类群的分类体系不断更新和完善[34].
念珠藻属是一多系、形态学上难以区分的类群,科马雷克藻属是从念珠藻属中分离出来[35-36].科马雷克藻属在形态上与微观的念珠藻属相似,因为它们有重叠的特征,藻丝都具有厚壁孢子和异形胞分化,且藻丝能形成群体.然而,科马雷克藻属与典型的念珠藻属有明显的不同,它不形成念珠藻属中发现的大量藻丝聚集或紧密缠绕形成的由胶被包裹的肉眼可见的圆球形群体.科马雷克藻属的独特之处还在于其特殊的厚壁孢子的萌发方式,在第1次分裂时会产生1个较大的营养细胞和1个较小的异形胞.其模式种Komarekiella atlantica的典型特征是呈现多束藻丝和不均等的厚壁孢子.本研究分离得到的藻株JM-1001与科马雷克藻属模式种的形态特征相同,都有厚壁孢子,且出现由营养细胞分化形成的异形胞,整个生活史的过程也相似,细胞外无胶被包裹.从生态学上看,模式种参考藻株发现于大西洋巴西沿岸热带雨林的树皮上;本研究藻株发现于我国桂林市阳朔县九马画山(亚热带地区),从山体墙壁上分离下来,与模式种具有相似的生境.16S rRNA基因相似性表明,实验藻株与参考藻株Komarekiella atlantica CCIBt 3481、K. atlantica CCIBt 3552、K. chia TAU-MAC 0117、K. delphini-convector SJR-PJCV1、K. globosa PJ104和K. gloeocapsoidea PJ101的基因序列相似性极高,达到96.8%~99.9%,高于细菌学上属的划分界限,进一步说明实验藻株属于科马雷克藻属;并且实验藻株与模式种K. atlantica CCIBt 3481和K. atlantica CCIBt 3552的序列同源相似性最高,达到99.9%,说明实验藻株属于模式种K. atlantica的藻株.16S rRNA系统发育树分析表明,实验藻株与科马雷克藻属中的模式种参考藻株单独聚在1个分支上,具有很近的亲缘关系;同时,16S-23S ITS二级结构也表明,实验藻株JM-1001的D1-D1′、Box-B和V3螺旋结构也与模式种参考藻株一致.因此,综合形态学特征、生态学、16S rRNA系统发育分析和ITS二级结构的结果,分离自广西壮族自治区桂林市阳朔县九马画山的陆生蓝藻藻株JM-1001属于大西洋科马雷克藻.科马雷克藻属是中国的新记录属,大西洋科马雷克藻Komarekiella atlantica JM-1001是中国的新记录种.
本研究中还发现,在系统进化树中Komarekiella chia与Parakomarekiella sp. 9E-03聚为1个紧密的分支,亲缘关系很近;其与科马雷克藻属的另外4个种处于不同属水平上的分支中.SOARES等[16]在2020年曾提出Parakomarekiella与科马雷克藻属在形态学上非常相似,但是在异形胞大小、厚壁孢子内容物上有较小的区别.并且通过系统发育分析表明Parakomarekiella与Komarekiella分别处于独立的分支中.这与本研究系统发育分析的结果较为一致.因此,科马雷克藻属的分类尽管现在取得了一些较好的进展,但是未来希望能够进一步的修订,使其分类地位更加完善和精确.
本研究中的科马雷克藻属是在中国(桂林市阳朔县九马画山)的首次报道,显示出我国陆生蓝藻的物种多样性.未来希望能够继续应用多相分析方法去发现、分离和鉴定更多的蓝藻,也期待探索不同生境下蓝藻多样性和丰富度信息,为蓝藻分类学和其他生物学的研究提供更多的材料.
参 考 文 献
[1]" CASAMATTA D.The Cyanobacteria:Molecular Biology,Genomics and Evolution[J].Phycologia,2008,47(2):236-237.
[2]WARD D M,CASTENHOLZ R W,MILLER S R.Cyanobacteria in geothermal habitats[M]//Ecology of Cyanobacteria II.Dordrecht:Springer Netherlands,2012:39-63.
[3]ACHTMAN M,WAGNER M.Microbial diversity and the genetic nature of microbial species[J].Nature Reviews Microbiology,2008,6(6):431-440.
[4]DAVYDOV D,VILNET A.Review of the Cyanobacterial Genus Phormidesmis(Leptolyngbyaceae) with the Description of Apatinema gen.nov[J].Diversity,2022,14(9):731.
[5]MARTINS M D,MACHADO-DE-LIMA N M,BRANCO L H Z.Polyphasic approach using multilocus analyses supports the establishment of the new aerophytic cyanobacterial genus Pycnacronema(Coleofasciculaceae,Oscillatoriales)[J].Journal of Phycology,2019,55(1):146-159.
[6]KOMREK J,KATOVSKY′ J,MARE J,et al.Taxonomic classification of cyanoprokaryotes(cyanobacterial genera)2014,using a polyphasic approach[J].Preslia,2014,86(4):295-335.
[7]BOYER S L,JOHANSEN J R,FLECHTNER V R,et al.Phylogeny and genetic variance in terrestrial Microcoleus(Cyanophyceae) species based on sequence analysis of the 16S rRNA gene and associated 16S-23S ITS region1[J].Journal of Phycology,2002,38(6):1222-1235.
[8]ENGENE N,CAMERON COATES R,GERWICK W H.16S rRNA gene heterogeneity in the filamentous marine cyanobacterial genuslyngbya1[J].Journal of Phycology,2010,46(3):591-601.
[9]KIM M,OH H S,PARK S C,et al.Towards a taxonomic coherence between average nucleotide identity and 16S rRNA gene sequence similarity for species demarcation of prokaryotes[J].International Journal of Systematic and Evolutionary Microbiology,2014,64(Pt 2):346-351.
[10]JOHANSEN J R,KOVACIK L,CASAMATTA D A,et al.Utility of 16S-23S ITS sequence and secondary structure for recognition of intrageneric and intergeneric limits within cyanobacterial taxa:Leptolyngbya corticola sp.nov.(Pseudanabaenaceae,cyanobacteria)[J].Nova Hedwigia,2011,92(3/4):283-302.
[11]DEBNATH M,SINGH T,BHADURY P.New records of Cyanobacterial morphotypes with Leptolyngbya indica sp. nov.from terrestrial biofilms of the Lower Gangetic Plain,India[J].Phytotaxa,2017,316(2):101.
[12]SHALYGIN S,SHALYGINA R R,REDKINA V V,et al.Description of Stenomitos kolaenensis and S. hiloensis sp. nov.(Leptolyngbyaceae,Cyanobacteria)with an emendation of the genus[J].Phytotaxa,2020,440(2):108-128.
[13]KOMREK J.A polyphasic approach for the taxonomy of cyanobacteria:principles and applications[J].European Journal of Phycology,2016,51(3):346-353.
[14]KOMREK J.Several problems of the polyphasic approach in the modern cyanobacterial system[J].Hydrobiologia,2018,811(1):7-17.
[15]STRUNECKY′ O,IVANOVA A P,MARE J.An updated classification of cyanobacterial orders and families based on phylogenomic and polyphasic analysis[J].Journal of Phycology,2023,59(1):12-51.
[16]SOARES F,RAMOS V,TROVO J,et al.Parakomarekiella sesnandensis gen. et sp. nov.(Nostocales,Cyanobacteria) isolated from the Old Cathedral of Coimbra,Portugal(UNESCO World Heritage Site)[J].European Journal of Phycology,2021,56(3):301-315.
[17]SCOTTA H G,JOHANSEN J R,PIETRASIAK N,et al.Komarekiella Atlantica gen. et sp. nov.(Nostocaceae,cyanobacteria):a new subaerial taxon from the Atlantic rainforest and Kauai,Hawaii[J].Fottea,2017,17(2):178-190.
[18]PANOU M,GKELIS S.Unravelling unknown cyanobacteria diversity linked with HCN production[J].Molecular Phylogenetics and Evolution,2022,166:107322.
[19]BROWN A O,ROMANIS C S,DVOK P,et al.A new species of cryptic cyanobacteria isolated from the epidermis of a bottlenose dolphin and as a bioaerosol[J].Phycologia,2021,60(6):603-618.
[20]JUNG P,BRUST K,SCHULTZ M,et al.Opening the gap:rare lichens with rare cyanobionts-unexpected cyanobiont diversity in cyanobacterial lichens of the order lichinales[J].Frontiers in Microbiology,2021,12:728378.
[21]RIPPKA R.Isolation and purification of cyanobacteria[J].Methods in Enzymology,1988,167:3-27.
[22]RAO P V L,BHATTACHARYA R,GUPTA S D.Isolation,culture,and toxicity of the Cyanobacterium(blue-green Alga) Microcystis aeruginosa from a freshwater source in India[J].Bulletin of Environmental Contamination and Toxicology,1994,52(6):878-885.
[23]CLARKE J D.Cetyltrimethyl ammonium bromide(CTAB) DNA miniprep for plant DNA isolation[J].Cold Spring Harbor Protocols,2009,2009(3):pdb.prot5177.
[24]EDWARDS U,ROGALL T,BLCKER H,et al.Isolation and direct complete nucleotide determination of entire genes.Characterization of a gene coding for 16S ribosomal RNA[J].Nucleic Acids Research,1989,17(19):7843-7853.
[25]GKELIS S,RAJANIEMI P,VARDAKA E,et al.Limnothrix redekei(Van Goor) Meffert(Cyanobacteria) strains from Lake Kastoria,Greece form a separate phylogenetic group[J].Microbial Ecology,2005,49(1):176-182.
[26]KATOH K,STANDLEY D M.MAFFT multiple sequence alignment software version 7:improvements in performance and usability[J].Molecular Biology and Evolution,2013,30(4):772-780.
[27]KUMAR S,STECHER G,TAMURA K. MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J].Molecular Biology and Evolution,2016,33(7):1870-1874.
[28]SWOFFORD D L.Phylogenetic Analysis Using Parsimony[EB/OL].[2023-08-30].https://bioweb.pasteur.fr/docs/modules/paup/4b10/Cmd_ref_v2.pdf.
[29]KALYAANAMOORTHY S,MINH B Q,WONG T K F,et al.ModelFinder:fast model selection for accurate phylogenetic estimates[J].Nature Methods,2017,14(6):587-589.
[30]REUTER J S,MATHEWS D H.RNAstructure:software for RNA secondary structure prediction and analysis[J].BMC Bioinformatics,2010,11:129.
[31]GARCIA-PICHEL F,WOJCIECHOWSKI M F.The evolution of a capacity to build supra-cellular ropes enabled filamentous cyanobacteria to colonize highly erodible substrates[J].PLoS One,2009,4(11):e7801.
[32]GARCIA-PICHEL F,LOZA V,MARUSENKO Y,et al.Temperature drives the continental-scale distribution of key microbes in topsoil communities[J].Science,2013,340(6140):1574-1577.
[33]PRADO J,HIRAI R Y,SHIMIZU G H,et al.The Nomenclature Section in Shenzhen(China)and the principal changes in the International Code of Nomenclature for Algae,Fungi,and Plants[J].Rodriguesia,2017,68(4):1499-1503.
[34]MACHADO DE LIMA N M,BRANCO L H Z.Biological soil crusts:new Genera and species of Cyanobacteria from Brazilian semi-arid regions[J].Phytotaxa,2020,470(4):263-281.
[35]HROUZEK P,VENTURA S,LUKEOV A,et al.Diversity of soil Nostoc strains:phylogenetic and phenotypic variability[J].Archiv Für Hydrobiologie,2005,117:251-264.
[36]HROUZEK P,LUKEOV A,MARE J,et al.Description of the cyanobacterial genus Desmonostoc gen. nov. including D. muscorum comb. nov. as a distinct,phylogenetically coherent taxon related to the genus Nostoc[J].Fottea,2013,13(2):201-213.
Komarekiella(Nostocale,Cyanobacteria):a terrestrial novel recorded genus from China
Chen Jiaxin1, Xi Yu2, Li Shuheng1, Cai Fangfang1
(1. School of Animal Science and Nutritional Engineering, Wuhan Polytechnic University, Wuhan 430072, China;
2. Ecological Environment Monitoring and Scientific Research Center, Taihu Basin" amp;" East China
Sea Ecological Environment Supervision and Administration Bureau, Ministry of
Ecology and Environment, Shanghai 200125, China)
Abstract: Cyanobacteria, with strong adaptability, living in not only freshwater and marine environments, but also desert, polar, hot springs and other extreme habitats. In this study, a strain of cyanobacteria, JM-1001, was isolated and purified from the soil layer of Jiumahuashan, Guilin City, Guangxi Zhuang Autonomous Region, China. The phylogenetic and taxonomic characteristics of JM-1001 were studied based on"" polyphasic analysis combining morphological, ecological and molecular data. Morphological characteristics of the cyanobacteria strain was very similar to Komarekiella, which with unicellular, filamentous or in colonies, distinct life history, heterocytes and akinete. The phylogenetic analysis of 16S rRNA gene sequence manifested that this strain was clustered in the Komarekiella clade, and JM-1001 and Komarekiella atlantica were closely clustered into a phylogenetic clade with high bootstrap values(98/93/1). The 16S rRNA gene similarity between JM-1001 and K." atlantica is 99.9%, which supports it to be a member of K. atlantica. In addition, the 16S-23S ITS secondary structure of JM-1001 is consistent with that of K. atlantica. All results supported it to be a member of K. atlantica. And the genus Komarekiella is a newly recorded genus in China.
Keywords: cyanobacteria; new recorded genus; phylogenetic; taxonomy
[责任编校 刘洋 赵晓华]

