高光谱结合机器学习鉴别林下参参龄
摘要 建立一种基于高光谱技术结合机器学习快速、无损鉴别市售林下生晒参参龄的方法。 以常见市售林下生晒参为研究对象,采集不同年限的林下生晒参高光谱图像并基于阈值分割方法从感兴趣区域提取光谱数据,然后对数据进行多元散射矫正(MSC)-Savitzky-Golay smoothing(S-G smoothing)-一阶导数(FD)和MSC-S-G smoothing-二阶导数(SD)预处理消除干扰,将预处理后的数据进行3种机器学习,包括支持向量机回归(SVR)、主成分回归(PCR)和偏最小二乘回归(PLSR)。高光谱数据存在冗余性,采用竞争性自适应重加权采样法(CARS)、连续投影算法(SPA)和无信息变量消除算法(UVE)3种算法筛选波段去除无用的波长信息。 CARS-MSC-S-G smoothing-SD-SVR有较低的均方根误差RMSEC(0. 0027),RMSEP(0. 0120),较高的决定系数R2C(0. 9998),R2P(0. 9993),以及相对分析误差RPD(38),竞争性自适应重加权采样法(CARS)结合SVR模型效果最好,能够实现对林下生晒参年限的准确分类。 基于高光谱成像技术结合机器学习能够实现对林下生晒参参龄的快速、无损鉴别。
关键词 林下生晒参;不同年限;高光谱成像;鉴别
中图分类号:O657. 3 文献标识码:A 文章编号:1000-0518(2025)01-0069-09
国家自然科学基金(No. 82073969)、长春市科技局项目(No. 21ZGY10)和吉林省科技发展计划项目(Nos. 20210401108YY,20220401104YY)资助
高光谱成像技术(HSI)[1]是将光谱与图像相结合,获得图像数据并得到图像中每个像素的光谱信息,具有无需前处理、快速、无损和绿色的特点。 高光谱技术被广泛应用于农产品、中药材等的快速鉴别。 如采用高光谱技术能够快速无损的实现对高粱掺假[2]、三七粉质量等级[3]以及柑橘属水果[4]的有效鉴别,准确率可达90%以上。 HSI的发展为人参年限的快速无损鉴定提供了新思路。
人参(Panax ginseng C. A. Mey.)是五加科多年生草本植物的干燥根及根茎[5],历史悠久,具有大补元气、补脾益肺和安神益智的功效[6]。 林下参是在野外环境中生长,通过人工将种子播种于深山中,若干年后采收[7]。 林下生晒参易于储存和运输,目前在业内和商界对于参龄鉴别仍然采用感官性状经验鉴别方法,对于消费者来说,难以根据感官经验对人参的生长年限进行鉴别,容易购买到伪品、赝品。因此,人参参龄的识别对于保护人参品牌和质量、保证人参市场的稳定发展至关重要。 林下参常用薄层鉴别[8]、超高效液相色谱-质谱联用(UHPLC-MS)[9]、气相色谱-质谱联用(GC-MS)[10]、傅里叶变换红外光谱(FT-IR)[11]和核磁共振波谱(NMR)[12]等方法鉴别,但这些方法往往具有前处理过程繁琐、重现性差和对样品造成破坏等缺点。
目前,鲜有林下生晒参快速无损鉴别的报道。 本研究利用HSI结合机器学习拟初步建立对林下生晒参参龄的快速、无损鉴别的方法,为林下生晒参的质量控制、鉴别标准建立提供理论依据。
1 实验部分
1. 1 仪器、试剂和材料
高光谱成像设备是由中国科学院长春光学精密力学和物理研究所自主搭建的,采用Imec快照扫描高光谱成像系统,波长在400~1000 nm之间,空间分辨率为2048×1088像素。 高光谱成像系统结构如图1所示,该成像系统采用线性扫描传感器技术,通过推扫的扫描方法获取物体的反射率数据,利用立方体的x和λ尺寸逐线进行采集。 本章内容使用的高光谱成像系统,波段数为300个,积分时间是3089 μs,帧周期为68 fps,速度为5. 6 mm/s。 ENVI 5. 3是一种高光谱图像处理软件,具备图像增强、校正等功能。
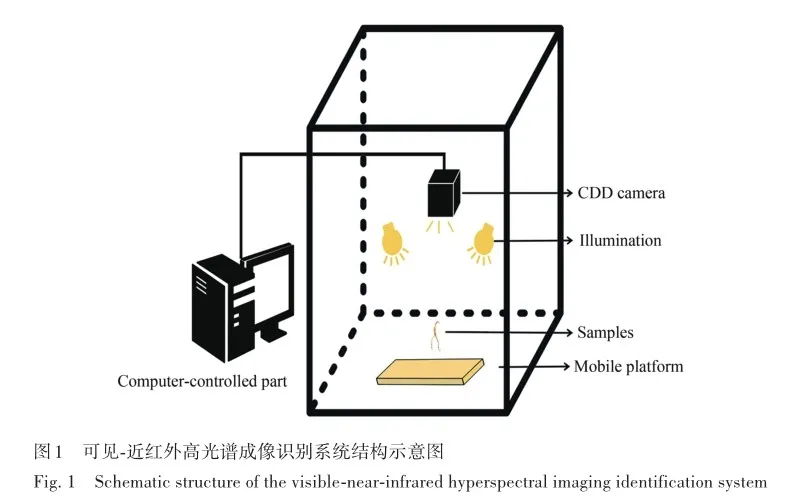
实验所用的306份林下参样本采挖于2023年9月,地点为吉林省集安市车道岭镇,由吉林省人参科学研究院陈长宝研究员鉴定为10、15和20年的二马牙品种林下参。 随后对鲜林下参用蒸馏水进行简单的淋洗和程序干燥,干燥过程如下,晾晒后的林下参置于60~70 ℃的干燥箱干燥2 h,在40~50 ℃下干燥10 h左右,然后35~40 ℃再干燥6 h左右,干燥过程中注意适时排潮,直至林下参最终含水质量分数为13%。 如图2所示,图2A?2C分别为10年、15年和20年干燥后的林下生晒参样本。

1. 2 实验方法
1. 2. 1 高光谱采集及校正

式中, R表示经过校正后的图像,I0表示原始图像,IW表示白板图像,ID表示黑板图像。
1. 2. 2 光谱数据提取及数据预处理
利用ENVI 5. 3软件在每个林下生晒参样品高光谱图像基于阈值分割方法选取一个大小相近的感兴趣区域(ROI),提取每个ROI内的平均光谱作为本实验样本的光谱数据。 高光谱数据包含大量的信息,易受到环境中多种因素的影响[13]。 在建模之前对光谱数据进行预处理可改善环境因素影响、提高模型的准确率[14]。 高光谱预处理包括多元散射校正(MSC)、平滑法和导数法等方法。 本研究采用多元散射矫正(MSC)-Savitzky-Golay smoothing(S-G smoothing)-一阶导数(FD)和多元散射矫正(MSC)-Savitzky-Golay smoothing(S-G smoothing)-二阶导数(SD)2种方法对高光谱数据进行预处理,找到最佳的数据预处理方法。
1. 2. 3 数据集划分
收集了来自同一产地的3个不同生长年限(10、15和20年)的306份人参样本,利用高光谱成像系统共获取306条高光谱数据,将采集到的306条林下生晒参高光谱数据,利用Kennard-stone(K-S)算法按照2∶1的比例划分训练集和预测集。
1. 2. 4 判别模型方法
采用偏最小二乘回归(PLSR)[15]、主成分分析回归(PCR)[16]和支持向量机回归(SVR)[17]3种判别模型对林下生晒参参龄进行判别分析。
PLSR[15]是一种将预测变量简化为一组较小的不相关分量并对这些分量而不是原始数据执行最小二乘回归的技术。 与多元回归不同,PLSR假定预测变量是可变的,从而可以有效地增加预测准确性。根据预测变量,PLS可以更有效地检测不确定性,从而增加其鲁棒性。
PCR[16]以主成分为自变量进行的回归分析。 用主成分分析法对回归模型中的多重共线性进行消除后,将主成分变量作为自变量进行回归分析,然后根据得分系数矩阵将原变量代回得到的新的模型。
SVR[17]将每个样本数据表示为空间中的点,使不同类别的样本点尽可能明显地区分开。 通过将样本的向量映射到高维空间中,寻找最优化区分2类数据的超平面,使各类到超平面的距离最大化,距离越大表示分类误差越小,即使数据集的边缘点到分界超平面的距离最大,称边缘点为支持向量。 通过非线性映射将原始数据空间映射到高维特征空间并在新空间中求取最优化线性分类面。
1. 2. 5 模型的评价指标
模型构建完成后,为了能够直观地评价模型,通过多个统计参数来评价所建模型的稳定性与可靠性。 本研究采用决定系数(R2)[18]、均方根误差 (RMSE)[19]以及相对分析误差(RPD)[20]3种参数作为模型评价指标比较所建模型PLSR、PCR和SVR的稳定性。 R2取值范围从0~1,越接近1,说明模型能够解释观测数据的变化,表示模型对数据的拟合程度好[18]。 R2C代表训练集的决定系数; R2CV代表训练集交叉验证的决定系数; R2P代表预测集的决定系数。 RMSE表示模型预测值与实际观测值之间的差异,用于衡量模型的预测精度[19]。 RMSE的计算结果越小,表示模型的预测精度越高,预测值与实际观测值的差异较小,其中RMSEC是训练集的均方根误差; RMSECV是训练集交叉验证的均方根误差; RMSEP是训练集的均方根误差。 RPD是评估模型性能的一项重要指标[20],其具有明确的评判标准: 当RPD<1. 4时,表明所构建的模型在可靠性方面存在明显不足,难以满足实际应用需求,故而认为该模型不可靠;若1. 4
1. 2. 6 特征波段提取
高光谱数据量大且无关变量多,通过特征波段提取算法可以去除大量繁琐无用的波谱信息,以达到增强模型的预测能力,提高模型的运算速度。 本研究采用竞争性自适应重加权采样法(Competitive adapative reweighted sampling,CARS)[21]、连续投影法(Successive projections algorithm,SPA)[22]和无信息变量消除法(Uninformative variable elimination,UVE)[23]3种筛选特征算法提取特征波段。
CARS是一种结合蒙特卡洛采样与PLS模型回归系数的特征变量选择方法,模仿达尔文理论中的“适者生存”的原则[21]。 CARS算法中,每次通过自适应加权采样(ARS)保留PLS模型中回归系数绝对值权重较大的点作为新的子集,去掉权值较小的点,然后基于新的子集建立PLS模型,经过多次计算,选择PLS模型交互验证均方根误差(RMSECV)最小的子集中的波长作为特征波长。
SPA是前向特征变量选择方法[22]。 SPA利用向量的投影分析,通过将波长投影到其他波长上,比较投影向量大小,以投影向量最大的波长为待选波长,然后基于矫正模型选择最终的特征波长。
UVE能够去除对建模共效率较小的波长变量,选出特征波长变量,被去除的波长变量我们称之为无信息变量[23]。 无信息变量去除算法的建立是基于偏最小二乘(PLS)算法。
1. 2. 7 实验数据处理
使用ENVI 5. 3软件提取每个感兴趣区域内的平均光谱数据,使用The Unscrambler X 10. 4(64-bit)软件对光谱数据进行预处理及建立林下参参龄的判别模型,使用Matlab 2022b软件对光谱数据筛选特征波段。
2 结果与讨论
2. 1 不同年限林下生晒参的光谱曲线
分别对10、15和20年3个年份的林下生晒参高光谱数据求平均值,绘制3个年份的林下生晒参平均光谱曲线图,如图3所示,3个年限的林下生晒参光谱曲线趋势大体相同,但光谱反射率值有明显的差异,其中在500~900 nm之间,20年林下生晒参的反射率值最大,15年次之,10年最小,反射率值与年限呈正相关。

2. 2 全波段林下生晒参高光谱数据预处理结果
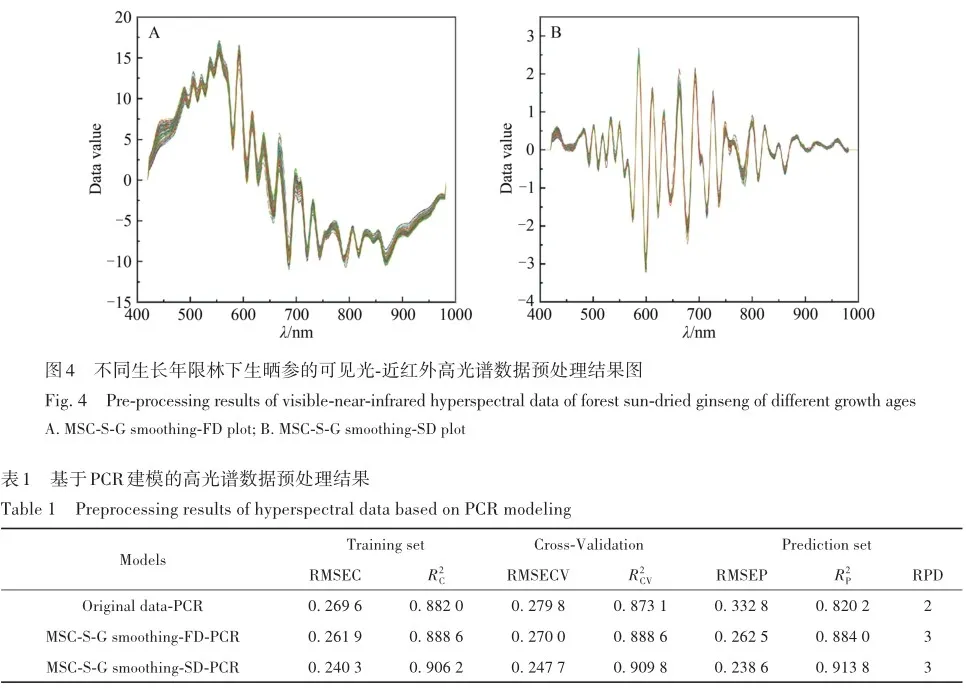
对不同生长年限的林下生晒参高光谱数据进行预处理操作,包括MSC-S-G smoothing-FD以及MSC-S-G smoothing-SD,经预处理后所获得的光谱曲线图如图4所示。 由图4可知,经过预处理后的光谱曲线形状较为复杂,在不同波长处有多个波峰和波谷,这些曲线在整体趋势上有相似之处,但在具体的波峰和波谷位置以及数据值大小上存在差异。
2. 3 基于全波段对林下生晒参数据预处理方式以及参龄判别模型的筛选
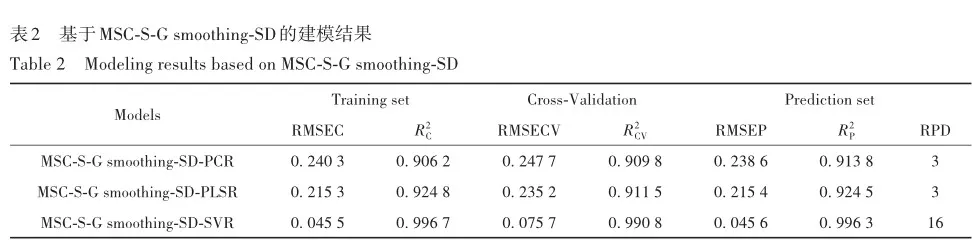
首先,将高光谱原始数据以及预处理后的高光谱数据作为输入变量,构建PCR判别模型,实现对不同生长年限林下参的精准预测,结果见表1。 通过表1可知,在同一判别模型下,预处理方式MSC-S-G smoothing-SD的R2更接近于1,RMSEP更接近于0,意味着林下参预测值和实际值之间的差异非常小,RPD值为3且大于2,说明所建模型可靠。 综上所述,MSC-S-G smoothing-SD方法更适于林下生晒参高光谱数据的预处理,能够实现不同生长年限林下参的预测。
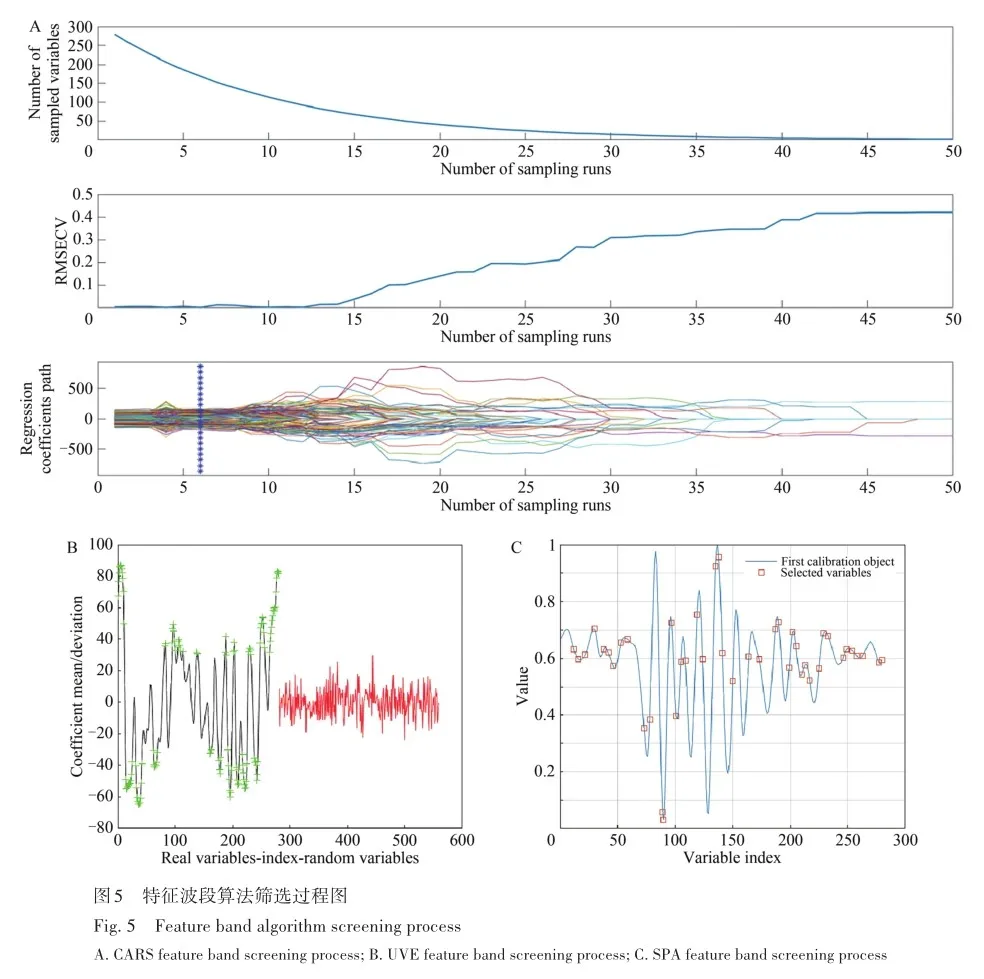
在光谱预处理采用MSC-S-G smoothing-SD方法之后,构建了PCR、PLSR和SVR判别模型,建模结果见表2。 由表2可知,使用SVR判别模型在预测林下生晒参参龄方面表现优异,具有较高的R2值,RPD值以及较低的RMSEP值,表明SVR判别模型在林下生晒参参龄的预测方面表现良好。 相对于PCR与PLSR,SVR适合处理复杂的数据集,在高维数据空间中表现突出,通过核函数将数据映射到更高维的空间,对异常值不太敏感,数据点影响极小[24]。 综合上述分析与研究结果可知,在预测林下生晒参参龄方面,MSC-S-G smoothing-SD与SVR相结合的方法构成了最优组合策略。
2. 4 特征波段筛选结果
基于不同生长年限林下生晒参的Vis-NIR高光谱数据,利用特征波段算法进行特征选择,筛选过程和结果如图5所示。 使用CARS筛选特征波段,设定蒙特卡罗迭代次数为50次,进行5折交叉验证策略以确保筛选的可靠性与稳定性。 经实验发现,当迭代次数为6次时,RMSECV值达到最小,其数值为0. 014,此时成功筛选出169个波段,占全部波段的比例为 56. 3%; 同时,使用SPA算法进行筛选最终确定43个波段,占全波段的 14. 3%; 采用UVE算法筛选出168个波段,占全波段56. 00%。
2. 5 基于特征波段筛选算法对MSC-S-G smoothing-SD-SVR方法的优化
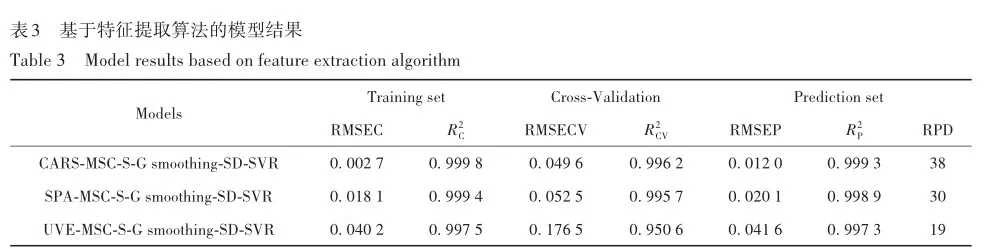
在MSC-S-G smoothing-SD-SVR方法基础上,运用CARS、SPA和UVE 3种特征波段算法筛选特征波段以实现优化,进而构建了CARS-MSC-S-G smoothing-SD-SVR、SPA-MSC-S-G smoothing-SD-SVR、UVE-MSC-S-G smoothing-SD-SVR 3种模型,结果见表3。 由表3可知,CARS-MSC-S-G smoothing-SD-SVR具有较低的均方根误差RMSEC(0. 0091),RMSEP(0. 0015),较高的相关系数R2C(0. 9999),R2P(0. 9999),以及RPD(38),CARS-MSC-S-G smoothing-SD-SVR的RMSEP值和R2值以及RPD值均优于其他2种算法,表明经过CARS算法优化后的模型在林下生晒参参龄预测方面表现良好。 相较于SPA和UVE,CARS筛选特征波段算法有效的筛选了林下生晒参光谱数据变量,提高了模型精确度。 研究结果表明,在林下生晒参参龄预测方面,CARS 算法更契合MSC-S-G smoothing-SD-SVR判别模型的优化需求。 而刘子健等[25]在基于特征波段建立山楂分类模型时,发现SPA算法为最佳特征波段提取方法,与本研究得到的结果不同,这说明对于不同的检测对象,应当选用不同的特征提取方法,而对于林下生晒参样本,CARS相比于SPA特征波长提取效果更好。
与MSC-S-G smoothing-SD-SVR相比,经CARS算法优化后的模型在训练集和预测集均呈现出更为优异的表现,其R2值更高且RMSEP值更低。 综合上述研究结果可知,CARS算法与SVR模型相结合的方式在林下生晒参参龄鉴别方面具有更强的适用性与有效性,能够精准地判定林下生晒参参龄,为林下生晒参参龄鉴别提供了一种可靠且高效的方法。
3 结 论
本研究开发了一种基于高光谱成像技术结合机器学习的快速、无损鉴别市售林下生晒参参龄的方法。 研究以10、15和20年的林下生晒参为对象,通过高光谱成像获取光谱数据,并利用多种预处理方法消除干扰。 利用竞争性自适应重加权采样法(CARS)、连续投影算法(SPA)和无信息变量消除算法(UVE)筛选出特征波段,最终结合支持向量机回归(SVR)模型实现对林下生晒参参龄的准确分类。 结果表明,CARS结合SVR模型在参龄鉴别中表现最佳。本研究的创新之处在于将高光谱成像技术与机器学习结合,实现了林下生晒参参龄的快速、无损鉴别,为人参的质量控制和市场监管提供了新的方法。 与传统的鉴别方法相比,基于高光谱成像技术的方法具有无需前处理、快速、无损和绿色环保的优点。 特别是CARS-SVR模型的应用,显著提高了参龄预测的准确性和效率。 在实验过程中,通过对高光谱数据的预处理和特征波段的筛选,有效地减少了数据冗余,提升了模型的预测能力。 然而,本研究也存在一些局限性,例如样本量适中,未来可以扩大样本量并引入更多年限的人参样本进行验证。 此外,环境因素对高光谱数据的影响也是需要进一步研究的问题,未来可以结合更多的数据校正和处理方法,以进一步提高模型的鲁棒性和泛化能力。 总体而言,本研究为林下生晒参参龄的鉴别提供了一个可行的解决方案,具有重要的应用前景和研究价值。
参考文献
[1]PAN Y, ZHANG H, CHEN Y, et al. Applications of hyperspectral imaging technology combined with machine learning in quality control of traditional Chinese medicine from the perspective of artificial intelligence: a review[J]. Crit Rev Anal Chem, 2023, 29: 1-15.
[2]BAI Z, HU X, TIAN J, et al. Rapid and nondestructive detection of sorghum adulteration using optimization algorithms and hyperspectral imaging[J]. Food Chem, 2020, 30(331): 127-290.
[3]姚坤杉, 孙俊, 陈晨, 等. 基于高光谱技术的三七不同部位粉末的无损鉴别[J]. 光谱学与光谱分析, 2023, 43(7):2027-2031. YAO K S, SUN J, CHEN C, et al. Nondestructive identification of powder from different parts of Panax pseudoginseng based on hyperspectral technique[J]. Spectrosc Spectral Anal, 2023, 43(7): 2027-2031.
[4]高梁喨. 基于高光谱技术的柑橘品质检测研究[D]. 雅安: 四川农业大学, 2023. GAO L L. Research on citrus quality detection based on hyperspectral technology[D]. Ya′an: Sichuan Agricultural University,2023.
[5]DE OLIVEIRA ZANUSO B, DE OLIVEIRA DOS SANTOS A R, MIOLA V F B, et al. Panax ginseng and aging related disorders: a systematic review[J]. Exp Gerontol, 2022, 161: 111731.
[6]LEE I S, KANG K S, KIM S Y. Panax ginseng pharmacopuncture: current status of the research and future challenges[J]. Biomolecules, 2019, 10(1): 33.
[7]余江锋, 罗吉, 黄亿健, 等. 吉林省不同种类人参UPLC-PDA指纹图谱及化学模式识别研究[J]. 中药材, 2019, 42(4): 822-827.YU J F, LUO J, HUANG Y J, et al. UPLC-PDA fingerprinting and chemical pattern recognition of different species of ginseng in Jilin Province[J]. Chin Mater Med, 2019, 42(4): 822-827.
[8]肖霞, 曹俊凯. 人参当归颗粒的薄层鉴别[J]. 中国实用医药, 2012, 7(9): 249-250. XIA X, CAO J K. Thin-layer identification of ginseng and angelica granules[J]. Chin Practical Med, 2012, 7(9):249-250.
[9]ZHU H, LIN H, TAN J, et al. UPLC-QTOF/MS-based nontargeted metabolomic analysis of mountain-and garden-cultivated ginseng of different ages in northeast China[J]. Molecules, 2018, 24(1): 33.
[10]ZHANG L, WANG P, LI S, et al. Differentiation of mountain- and garden-cultivated ginseng with different growth years using HS-SPME-GC-MS coupled with chemometrics[J]. Molecules, 2023, 28(5): 2016.
[11]LEE B J, KIM H Y, LIM S R, et al. Discrimination and prediction of cultivation age and parts of Panax ginseng by Fourier-transform infrared spectroscopy combined with multivariate statistical analysis[J]. PLoS One, 2017, 12(10):e 0186664.
[12]YANG S, SHIN Y S, HYUN S H, et al. NMR-based metabolic profiling and differentiation of ginseng roots according to cultivation ages[J]. J Pharm Biomed Anal, 2012, 58: 19-26.
[13]刘秋安, 徐芳芳, 张欣, 等. 基于近红外光谱技术和分类与回归树算法建立天舒片崩解时间预测模型[J]. 中草药,2021, 52(16): 4837-4843. LIU Q A, XU F F, ZHANG X, et al. Establishment of a disintegration time prediction model for Tianshu tablets based on near-infrared spectroscopy and classification and regression tree algorithms[J]. Chin Herbal Med, 2021, 52(16): 4837-4843.
[14]LI X, WEI Y, XU J, et al. SSC and pH for sweet assessment and maturity classification of harvested cherry fruit based on NIR hyperspectral imaging technology[J]. Postharvest Biol Technol, 2018, 143: 112-118.
[15]ABDI H, WILLIAMS L J. Partial least squares methods: partial least squares correlation and partial least square regression[J]. Methods Mol Biol, 2013, 930: 549-579.
[16]HEMMATEENEJAD B, MIRI R, ELYASI M. A segmented principal component analysis--regression approach to QSAR study of peptides[J]. J Theor Biol, 2012, 305: 37-44.
[17]MCKEARNAN S B, VOCK D M, MARAI G E, et al. Feature selection for support vector regression using a genetic algorithm[J]. Biostatistics, 2023, 24(2): 295-308.
[18]傅晨, 王开军. 决定系数与相关系数相关系数辅助的LASSO回归[J]. 福建师范大学学报(自然科学版), 2024,40(2): 57-63, 89. FU C, WANG K J. LASSO regression assisted by coefficient of determination and correlation coefficient[J]. J Fujian Norm Univ (Nat Sci Ed), 2024, 40(2): 57-63, 89.
[19]徐萍, 米琪, 罗文秀, 等. 滇重楼皂苷近红外光谱快速定量分析模型建立[J/OL]. 中成药, 1-7[2024-05-20]. XU P, MI Q, LUO W X, et al. Modeling of near-infrared spectroscopy for rapid quantitative analysis of Dianthus annuus saponins[J/OL]. Proprietary Chin Med, 1-7[2024-05-20].
[20]陈露萍, 徐芳芳, 张欣, 等. 基于偏最小二乘法建立大株红景天片素片硬度近红外光谱预测模型[J]. 中草药,2023, 54(8): 2446-2452. CHEN L P, XU F F, ZHANG X, et al. Establishment of a near-infrared spectral prediction model for the hardness of Rhodiola rosea tablets based on the partial least squares method[J]. Chin Herbal Med, 2023, 54(8): 2446-2452.
[21]LI H, LIANG Y, XU Q, et al. Key wavelengths screening using competitive adaptive reweighted sampling method for multivariate calibration[J]. Anal Chim Acta, 2009, 648(1): 77-84.
[22]ZHU H, CHU B, ZHANG C, et al. Hyperspectral imaging for presymptomatic detection of tobacco disease with successive projections algorithm and machine-learning classifiers[J]. Sci Rep, 2017, 7(1): 4125.
[23]HAN Q, WU H, CAI C, et al. An ensemble of Monte Carlo uninformative variable elimination for wavelength selection[J]. Anal Chim Acta, 2008, 612(2): 121-125.
[24]YU H, LU J, ZHANG G. Continuous support vector regression for nonstationary streaming data[J]. IEEE Trans Cybern,2022, 52(5): 3592-3605.
[25]刘子健, 顾佳盛, 周聪, 等. 基于高光谱成像技术的山楂产地判别研究[J]. 食品工业科技, 2024, 45(10): 282-291. LIU Z J, GU J S, ZHOU C, et al. Research on the origin discrimination of hawthorn based on hyperspectral imaging technology[J]. Food Ind Sci Technol, 2024, 45(10): 282-291.
Identification of Age of Forest Sun-Dried Ginseng Based on Hyperspectral Imaging Technology
HAN Lu-Sheng1, WANG Yu-Dan1, LI Yu-Hang2, LI Gen-Yue1, SONG Xin-Hong1, YANG Kai-Li1,TAN Xin2*, WANG En-Peng1*
1(Jilin Ginseng Academy, Changchun University of Chinese Medicine, Changchun 130117, China)2(Changchun Institute of Optics, Fine Mechanics and Physics, Chinese Academy of Sciences,Changchun 130033, China)
Abstract The study establishes a method for rapid and non-destructive identification of the age of commercially available forest sun-dried ginseng based on hyperspectral technology combined with machine learning. Using common commercially available forest sun-dried ginseng as the research object, hyperspectral images of ginseng of different ages were first collected, and spectral data were extracted from regions of interest using a threshold segmentation method. The data were then preprocessed using Multiplicative Scatter Correction (MSC)-SavitzkyGolay smoothing (S-G smoothing)-First Derivative (FD) and MSC-S-G smoothing-Second Derivative (SD) to eliminate interference. Three machine learning models, including Support Vector Machine Regression (SVR),Principal Component Regression (PCR), and Partial Least Squares Regression (PLSR), were applied to the preprocessed data. Due to the redundancy in hyperspectral data, three algorithms—Competitive Adaptive Reweighted Sampling (CARS), Successive Projections Algorithm (SPA), and Uninformative Variable Elimination (UVE) were used to select bands and remove redundant wavelength information. The CARS-MSCS-G smoothing-SD-SVR model showed the best performance, with lower Root Mean Square Errors (RMSEC:0. 0027, RMSEP: 0. 0120), higher Correlation Coefficients (R2C: 0. 9998, R2P: 0. 9993), and RPD (38). This model effectively achieved accurate classification of the age of forest sun-dried ginseng. Combining hyperspectral imaging technology with machine learning enables rapid and non-destructive identification of the age of forest sun-dried ginseng.
Keywords Forest sun-dried ginseng; Different ages; Hyperspectralimaging; Identification
Received 2024?07?03; Accepted 2024?12?20
Supported by the National Natural Science Foundation of China (No. 82073969), the Science and Technology Bureau Project of Changchun (No. 21ZGY10) and the Science and Technology Development Program Project of Jilin Province (Nos. 20210401108YY,20220401104YY)

