鹅掌楸LcPIN1a基因的克隆及其对植株生长发育的影响
摘要:【目的】PIN-FORMED(PIN)属于生长素转运蛋白,能够介导生长素的极性运输,在植物的生长和发育过程中扮演着关键角色。本研究旨在解析鹅掌楸PIN1基因对植物生长和发育的影响。【方法】通过蛋白序列同源比对、系统发育树构建以及蛋白结构域预测的方法,鉴定鹅掌楸中PIN1的同源蛋白LcPIN1s。然后,通过转录组数据解析LcPIN1s基因的组织特异性表达情况,并通过实时荧光定量逆转录PCR(RT-qPCR)方法研究LcPIN1s在不同苗龄再生植株的根、茎和叶组织中的表达动态。此外,通过转录组数据解析LcPIN1s基因在低温、高温以及干旱胁迫条件下的时序动态表达,并进一步利用RT-qPCR方法研究LcPIN1s响应干旱胁迫与内源ABA合成之间的关联。最后,通过克隆鹅掌楸叶片中高表达的PIN1同源基因LcPIN1a,构建由CaMV35S启动子驱动的过表达载体(35S:LcPIN1a),通过异源转化拟南芥(Arabidopsis thaliana)和同源转化杂种鹅掌楸(Liriodendron × sinoamericanum),筛选并获得的异源过表达(LcPIN1a-HO)和同源过表达(LcPIN1a-OE)阳性再生植株,分别进行生长和发育性状的测定和分析。【结果】通过生物信息学方法在鹅掌楸基因组中成功鉴定到了3个PIN1同源蛋白,分别命名为LcPIN1a、LcPIN1b和LcPIN1c。组织特异性表达分析显示,LcPIN1a主要在叶片中高表达,而LcPIN1b和LcPIN1c主要在茎和根以及成年后的雌蕊中高表达。另外,鹅掌楸3个PIN1同源基因均受到低温(4 ℃)和干旱(质量分数15% PEG6000)处理的诱导而表现出先上调后下调的表达模式,但在高温(40 ℃)胁迫下则会急剧下调表达。利用聚乙二醇(PEG)、脱落酸(ABA)以及ABA合成抑制剂氟啶酮(Flu)处理,发现LcPIN1s在响应干旱诱导时表现出不同的模式,即LcPIN1a不依赖于内源ABA的合成,而LcPIN1b和LcPIN1c则依赖于内源ABA的合成。最后,通过对拟南芥异源过表达株系(LcPIN1a-HO)再生植株的生长性状统计分析发现,其在根长和株高方面均显著低于野生型,同时雄蕊数发生显著变异,从野生型的6枚雄蕊变为以5枚为主。在杂交鹅掌楸过表达株系(LcPIN1a-OE)中,其体胚成苗率显著降低,并且成苗后的再生植株在根长和株高方面均显著低于野生型,同时根系结构发生明显变化,主根不明显。【结论】鹅掌楸PIN1蛋白在植株的营养和生殖生长方面发挥重要作用,过量表达不利于植株的正常生长和发育。
关键词:鹅掌楸;生长素转运蛋白;PIN1基因;生长发育
中图分类号:S718;S68"""nbsp;"" 文献标志码:A开放科学(资源服务)标识码(OSID):
文章编号:1000-2006(2024)06-0051-11
Cloning of the" Liriodendron chinense LcPIN1a genes and its effect on plant growth and development
HAO Zhaodong,MA Xiaoxiao,WANG Dandan,LU Ye,SHI Jisen,CHEN Jinhui*
(State Key Laboratory of Tree Genetics and Breeding, Co-Innovation Center for Sustainable Forestry in Southern China, College of Forestry and Grassland, Nanjing Forestry University, Nanjing 210037, China)
Abstract: 【Objective】This study aimed to explore the role of the auxin transporter PIN1 in plant growth and development in Liriodendron chinense.【Method】Three PIN1 homologous proteins were" identified in the Liriodendron genome using bioinformatic methods, and expression pattern analyses of the three LcPIN1s genes were performed in different tissues and response to various abiotic stresses. An overexpression vector driven by the CaMV35S promoter was then constructed and transformed into Arabidopsis and Liriodendron×sinoamericanum, followed by phenotypic determination of growth and developmental traits in the transgenic positive lines.【Result】Three PIN1 homologous proteins were identified in the Liriodendron genome, named LcPIN1a, LcPIN1b" and LcPIN1c. Expression pattern analyses showed that LcPIN1a was mainly expressed in leaves, while LcPIN1b and LcPIN1c were primarily expressed in roots and stems and stigmas when the plantlets transitioned into reproductive growth. In addition, all three LcPIN1 genes transcriptionally responded to drought stress, with LcPIN1b and LcPIN1c showing dependence on the biosynthesis of endogenous ABA, while LcPIN1a does not. Root length and plant height were significantly reduced in LcPIN1a-heterologous overexpression (LcPIN1a-HO) lines compared to wild-type Arabidopsis. The number of stamens was predominantly five in LcPIN1a-HO lines, whereas wild-type Arabidopsis typically contained six stamens. The regeneration of plantlets in LcPIN1a-overexpressing (LcPIN1a-OE) Liriodendron×sinoamericanum" was significantly reduced compared to wild-type plants. In addition, the root length and plant height of LcPIN1a-OE regenerated seedlings were" significantly lower than those of the wild type. The root structure of LcPIN1a-OE plants was significantly changed, with the taproot being less distinct.【Conclusion】The PIN1 proteins of L. chinense play a crucial role in vegetative and reproductive growth. Overexpression of LcPIN1 genes can be detrimental to normal plant growth and development.
Keywords:Liriodendron chinense; auxin transporter; PIN1 gene; growth and development
生长素(auxin)是一类植物激素,在植物生长发育的各个方面均发挥重要作用[1]。由生长素参与的生物学过程主要受到3个层面的调控,即生长素代谢(包括合成与失活)、运输以及信号转导[2]。其中,生长素的极性运输作用于植物细胞间生长素浓度梯度的建立,从而调控生长素的作用[3]。生长素转运蛋白主要包括PIN-FORMED(PIN)、ATP 结合盒B家族(ABCB)以及AUXIN1/LIKE AUXIN1(AUX1/LAX)[4]。其中,PIN蛋白主要作用于细胞内生长素的外排,通过其自身在细胞内的极性定位影响植物组织器官中生长素极大或极小值点的建立,进而调控由生长素介导的植物器官模式建成和向性运动[5-7]。
拟南芥(Arabidopsis thaliana)中存在8个PIN蛋白成员,分别为AtPIN 1—8,其中AtPIN1是第一个被鉴定的生长素外排载体,参与维管组织的发育和侧生器官的形成[8]。随后,越来越多的研究显示拟南芥中PIN家族成员广泛参与植物生长和发育的各个方面。比如,AtPIN1和AtPIN7参与胚胎发生中顶—基轴的建立[9],AtPIN2和AtPIN3参与根的向重力性响应[10-11],AtPIN4参与根的模式建成[7],AtPIN5和AtPIN8一起作用于生长素内稳态和花粉发育[12],AtPIN6介导的生长素转运和内稳态作用于侧根和不定根器官发生[13]。
在所有拟南芥PIN基因中,AtPIN1突变后的表型缺陷最为严重,整个花序茎只有茎,没有茎生叶和花器官,形态就像一个大头针(pin),这也是pin-formed1(pin1)名字的由来[14]。后续有关AtPIN1的研究显示,该基因广泛参与维管组织发育[8]、叶脉模式建成[15]、向光和向重力性[16]、早期胚胎发生[9]等多个生长发育过程。然而,在除拟南芥以外的大多数植物中,PIN1基因一般存在多个同源拷贝,相互之间存在功能分化。比如,水稻(Oryza sativa)中存在4个OsPIN1基因(OsPIN1a—1d),其中OsPIN1a/b主要作用于根、茎和花序的发育,OsPIN1c/d则主要在稻穗形成中发挥功能[17]。
鹅掌楸属(Liriodendron)是木兰科(Magnoliaceae)单属植物,包含2种,即鹅掌楸(L. chinense)和北美鹅掌楸(L. tulipifera)。鹅掌楸天然分布于东亚地区[18],而北美鹅掌楸天然分布于北美东部地区[19],二者之间分化1 000万~1 600万年[20],组成了一对东亚-北美东部洲际间隔分布的姊妹树种。鹅掌楸属种间杂交可育,且杂交种具有生长快、抗性强、材性优等特点,是重要的速生造林和园林景观树种[21-22]。Chen等[23]于2019年成功破译了鹅掌楸全基因组,并在杂交鹅掌楸中建立了经由体胚发生途径的高效离体再生体系及遗传转化体系[24-25],为鹅掌楸属植物重要基因挖掘和基因功能鉴定提供了参考。
有关鹅掌楸PIN基因鉴定和功能研究还处于起步阶段。Hu等[26]基于鹅掌楸基因组鉴定了LcPIN基因家族,发现该家族包含11个成员,并通过转录组和荧光实时定量发现LcPIN3和LcPIN6a可能作用于雄蕊和花瓣发育,LcPIN2可能作用于根伸长,以及LcPIN5和LcPIN8可能作用于非生物胁迫响应,但缺乏直接证据。Li等[27]发现鹅掌楸LcPIN3主要在鹅掌楸根尖中表达,并通过在拟南芥中异源过表达发现其能够促进根的伸长,并能够回补atpin1在根长生长和花器官形成方面的缺陷。
作为最早被发现的PIN家族成员,有关PIN1在植物生长和发育中的功能报道最多,其在单双子叶植物广泛参与组织器官的发育和形态建成,但LcPIN1在鹅掌楸生长发育中的作用还不清楚。本研究通过对鹅掌楸LcPIN1基因的生物信息学鉴定、表达模式分析,以及在拟南芥和鹅掌楸中的过表达分析,初步揭示其对于植物生长发育的影响。
1 材料与方法
1.1 试验材料
鹅掌楸叶片采自于南京林业大学林木遗传育种全国重点实验室,保存于南京林业大学下蜀林场(119°13′E,32°7′N)的鹅掌楸庐山种源材料。杂种鹅掌楸(Liriodendron×sinoamericanum)3个月瓶苗和2年生大苗均为同一实验室分别保存于南京林业大学新庄校区和白马校区的材料。拟南芥为同一实验室保存于南京林业大学新庄校区的哥伦比亚型。
1.2 LcPIN1基因的生物信息学鉴定
鹅掌楸参考基因组序列从国家基因库(https://www.cngb.org)下载,以从TAIR数据库检索获得的AtPIN1序列作为提交序列[28],利用blastP搜索鹅掌楸蛋白库[29],筛选参数E期望值(Evalue)小于0.000 1,一致性大于50%。然后将筛选获得的序列作为提交序列,利用blastP搜索拟南芥蛋白库,最佳比对为AtPIN1的蛋白作为鹅掌楸PIN1候选蛋白。
利用同样的鉴定方法,从Phytozome数据库中鉴定小立碗藓(Physcomitrium patens)、毛果杨(Populus trichocarpa)、葡萄(Vitis vinifera)、水稻(Oryza sativa)、高粱(Sorghum bicolor)、玉米(Zea mays)、无油樟(Amborella trichopoda)等物种的PIN1同源蛋白[30]。利用Clustal-omega软件进行多序列比对[31],然后利用RAxML软件进行系统发育树的构建[32],最后分别使用TBtools和iTOL进行多序列比对和系统发育树的可视化[33-34]。另外,利用Protter软件(http://wlab.ethz.ch/protter/start/)在线预测鹅掌楸PIN1同源蛋白的跨膜结构[35]。
1.3 LcPIN1基因的表达模式分析
3月龄瓶苗和2年生大苗杂交鹅掌楸的根、茎和叶片于现场采集后立即用液氮速冻,然后保存于实验室-80 ℃冰箱中。另外,3月龄瓶苗用于质量分数20%的PEG6000、50 mg/L的ABA以及20%的PEG和50 μmol/L氟啶酮组合处理,分别于处理前及处理后12和24 h收集植株材料,液氮速冻后保存于-80 ℃冰箱中。
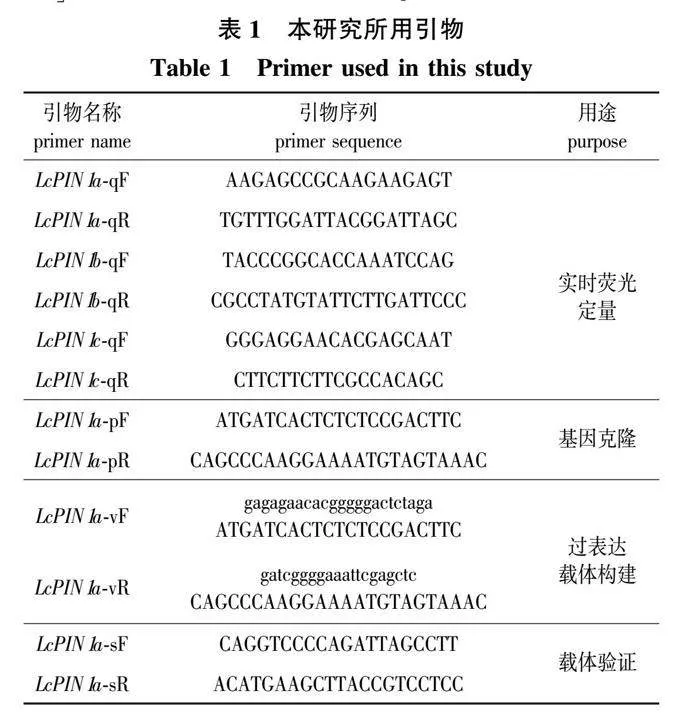
总RNA提取和cDNA合成分别使用FastPure Plant Total RNA Isolation Kit和HiScript III 1st Strand cDNA Synthesis Kit试剂盒(南京诺唯赞生物科技股份有限公司)。实时荧光定量逆转录PCR(Real-time reverse transcription PCR,RT-qPCR)实验的引物见表1。LcPIN1基因的RT-qPCR使用AceQ qPCR SYBR Green Master Mix试剂盒(南京诺唯赞生物科技股份有限公司),内参基因选择α-tubulin和18S RNA[36],相对表达量计算使用2-ΔΔCt方法[37]。所有RT-qPCR实验均为3次生物学重复、3次技术重复,即每个样本共9次重复。
1.4 LcPIN1a基因的克隆
利用CTAB法提取鹅掌楸叶片DNA[38],根据鉴定到的LcPIN1a基因序列设计引物(表1),合成引物后使用高保真DNA聚合酶Phanta Max Super-Fidelity DNA Polymerase(南京诺唯赞生物科技股份有限公司)进行克隆,最后使用普通琼脂糖凝胶DNA回收试剂盒[天根生化科技(北京)有限公司]进行目的片段的切胶回收。
1.5 LcPIN1a基因过表达载体构建
首先,将克隆到的LcPIN1a cDNA片段连接中间载体pClone007 blunt vector(南京擎科生物科技有限公司)。然后,转化大肠杆菌DH5α感受态细胞[天根生化科技(北京)有限公司],挑单菌落进行测序验证。对测序验证正确的菌落进行扩大培养,并利用质粒小提取试剂盒[天根生化科技(北京)有限公司]提取质粒DNA。设计同源重组引物(表1),从含目的片段的中间载体中扩增目的片段,后利用ClonExpress Ⅱ One Step Cloning Kit试剂盒(南京诺唯赞生物科技股份有限公司)进行同源重组,将LcPIN1a构建到CaMV35S启动子介导的植物表达载体pBI121上,获得 35S:LcPIN1a载体。
1.6 LcPIN1a基因在拟南芥中的过表达
将经过测序验证正确的 35S:LcPIN1a质粒转入农杆菌GV3101菌株(上海唯地生物技术有限公司),采用花序侵染法转化拟南芥[39],获得转基因T1代种子。对T1代种子萌发出的拟南芥幼苗进行PCR验证,并收集转基因阳性植株的T2代种子。进一步对T2代种子进行播种,根据孟德尔遗传定律,收集纯合的T3代种子,播种后用于生长和生殖相关性状的表型测量和统计分析。
1.7 LcPIN1a基因在杂交鹅掌楸中的过表达
将经过测序验证正确的35S:LcPIN1a质粒转入农杆菌EHA105菌株(上海唯地生物技术有限公司),以杂交鹅掌楸胚性愈伤为受体材料,进行稳定遗传转化[25]。在抗生素筛选并获得胚性愈伤后,进行PCR验证,对验证阳性的愈伤进行体胚诱导,获得成熟子叶胚[24]。将获得的成熟子叶胚接种到基本培养基上,于2月后统计成苗率,后测量生长相关性状并进行统计分析。
2 结果与分析
2.1 LcPIN1基因的鉴定与生物信息分析
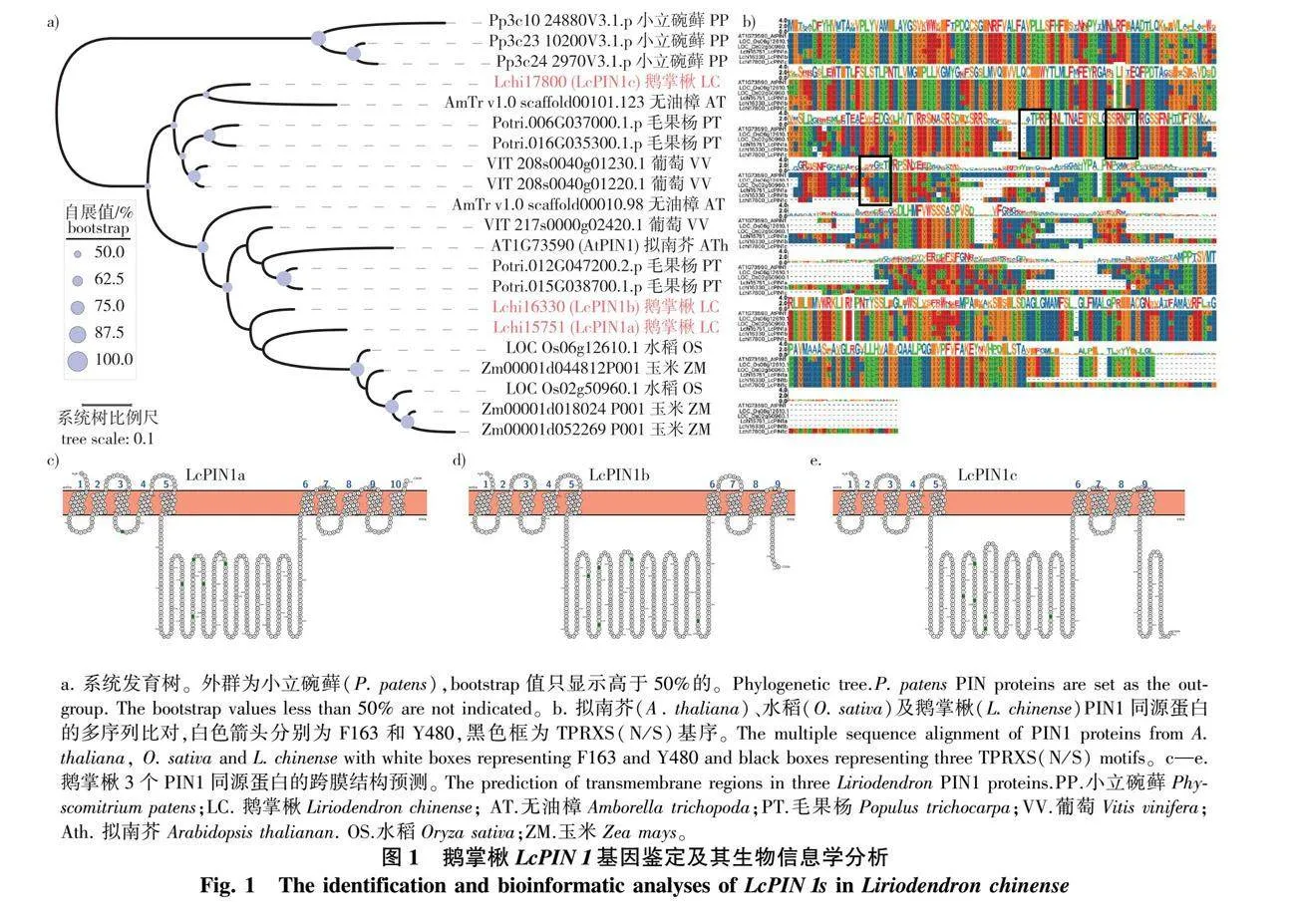
为鉴定鹅掌楸中的LcPIN1基因,利用拟南芥中的AtPIN1(AT1G73590)蛋白序列检索鹅掌楸基因组[23],共鉴定到5条相似性序列(Evaluelt;0.000 1,一致性大于50%)。进一步将这5条相似性序列比对到拟南芥基因组上[28],发现其中2条(Lchi05137和Lchi05662)的最佳比对分别为AtPIN2(AT5G57090)和AtPIN3(AT1G70940),剩余3条(Lchi15751、Lchi16330和Lchi17800)的最佳比对均为AtPIN1。
为了进一步明确上述鉴定的3条候选基因是否确定为LcPIN1同源基因及其系统发育关系,利用上述方法鉴定了小立碗藓、毛果杨、葡萄、水稻、高粱、玉米、无油樟等植物种中的PIN1同源蛋白,并与拟南芥和鹅掌楸PIN1同源蛋白一起,以小立碗藓PINs蛋白为外群构建了系统发育树(图1a)。结果显示,鹅掌楸中鉴定到的3条候选序列的确能够与其他物种的PIN1同源蛋白聚为一支,分别命名为LcPIN1a(Lchi15751)、LcPIN1b(Lchi16330)和LcPIN1c(Lchi17800)。根据系统发育树,PIN1基因在被子植物共同的祖先发生过1次基因复制事件,而后形成了两个进化大支。其中,鹅掌楸LcPIN1a、LcPIN1b与拟南芥AtPIN1同属一个进化支,而LcPIN1c则分属另外一支(图1a)。
进一步通过对鹅掌楸、拟南芥及水稻PIN1同源蛋白的多序列比对分析,发现PIN1蛋白的C端和N端较为保守,而中间区段则变异程度较大(图1b)。已有研究显示,PIN1蛋白为质膜定位蛋白,其N端和C端均为疏水结构域,分别含多个跨膜区[40]。通过对鹅掌楸PIN1同源蛋白的跨膜结构分析显示,LcPIN1a在N端和C端分别含有5个跨膜区,而LcPIN1b和LcPIN1c在C端的跨膜区为4个,比N端少1个(图1c)。
此外,研究表明PIN1蛋白的疏水结构域中包含多个作用于其质膜定位及其介导的生长素转运的关键信号。其中,PIN1蛋白N端165号位的苯丙氨酸(F163)和C端480号位的酪氨酸(Y480)作用于与衔接蛋白复合物亚基μ-adaptin结合,从而作用于其蛋白运输和质膜定位[41-42]。根据多序列比对结果,这2个氨基酸残基位点在鹅掌楸3个PIN1同源蛋白中较为保守(图1b)。
另外,PIN1蛋白疏水结构域还存在3个演化上保守的基序TPRXS(N/S),其中丝氨酸残基的磷酸化对PIN1介导的生长素极性运输至关重要[43]。根据多序列比对结果,鹅掌楸PIN1同源蛋白在这3个基序上的氨基酸较为保守,除了LcPIN1c蛋白的第3个基序由TPRXSN变为SPRQSN,但丝氨酸残基并未发生突变(图1b)。
2.2 LcPIN1基因的组织特异性表达
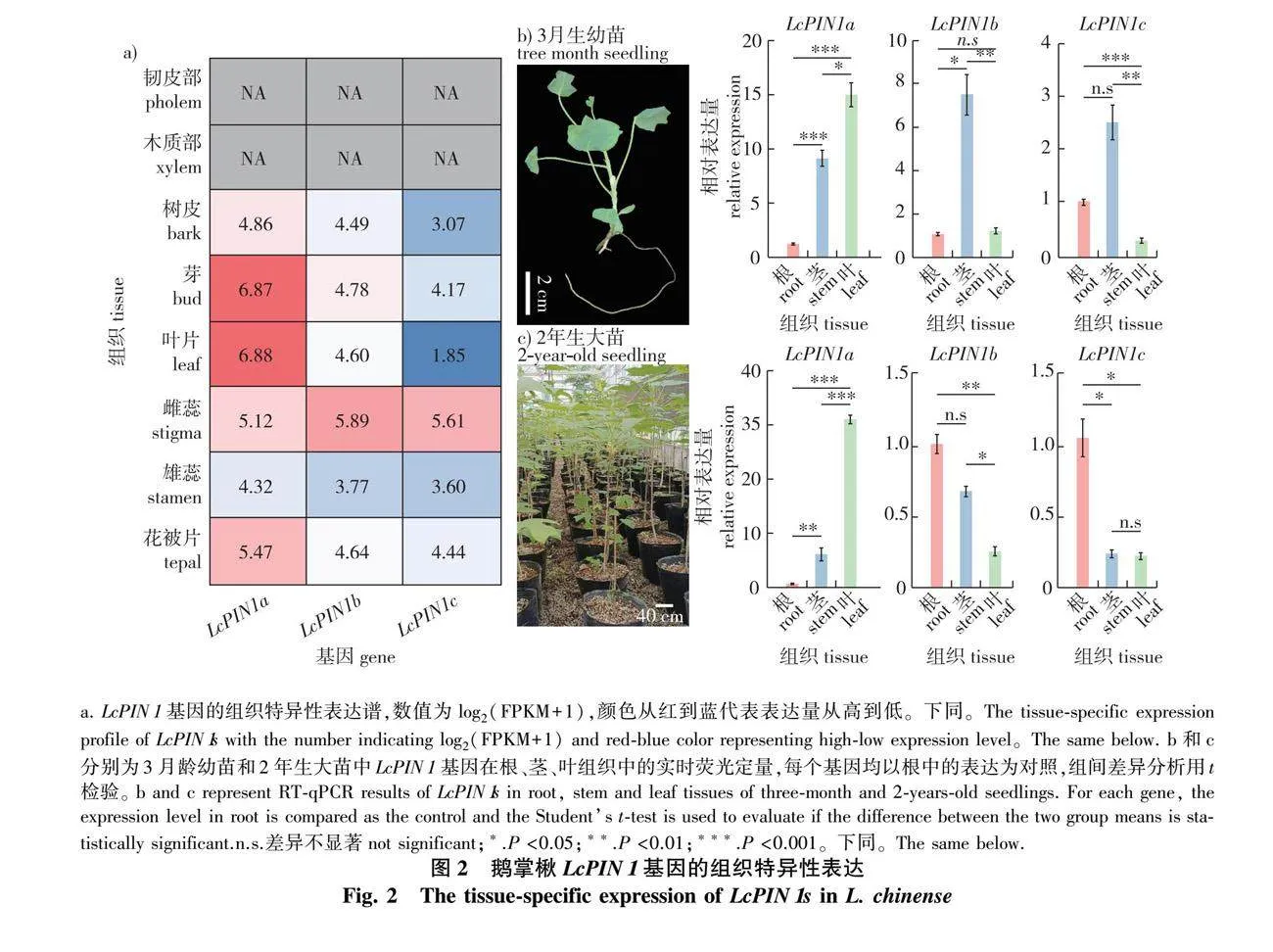
为了初步明确鹅掌楸LcPIN1基因在植物生长和发育中的作用,开展了组织特异性表达模式分析。成年鹅掌楸植株的不同组织转录组数据显示,LcPIN1a在鹅掌楸叶片和芽中表达量较高,而LcPIN1b和LcPIN1c均相对在雌蕊中表达量较高(图2a)。为了进一步明确LcPIN1基因的组织特异性表达,选取3月龄幼苗和2年生大苗的根、茎、叶组织进行RT-qPCR实验,结果显示LcPIN1a均在叶片中高表达,而LcPIN1b和LcPIN1c表达情况较为复杂,2月龄时在茎中表达较高,而在2年生时则在根中表达相对较高(图2b、2c)。这些结果表明,鹅掌楸LcPIN1不同旁系同源基因之间出现了明显的功能分化,其中LcPIN1a在整个生长周期中主要作用于叶片的发育,而LcPIN1b和LcPIN1c则在营养生长时期主要作用于茎和根的发育,而后在生殖生长时期主要作用于雌蕊的发育。
2.3 LcPIN1基因在非生物胁迫下的表达
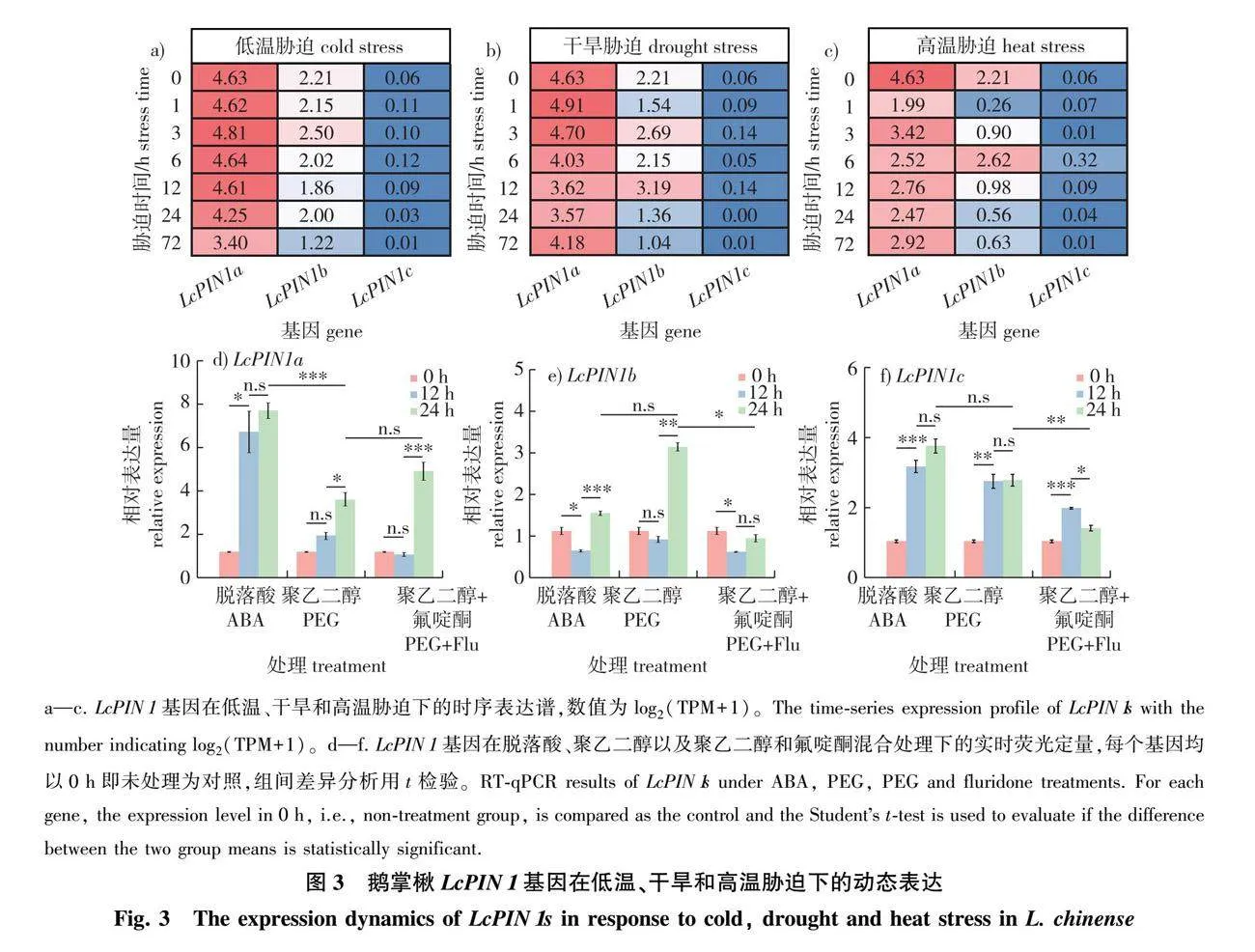
为了进一步解析鹅掌楸LcPIN1基因的功能,利用已有的转录组数据,定量了其在干旱(15% PEG6000)、高温(40 ℃)和低温(4 ℃)胁迫下的表达动态变化(图3a—3c)。结果显示,LcPIN1a基因的表达水平较高,LcPIN1b基因次之,LcPIN1c基因表达水平最低,该结果可能是由于该系列转录组的样本取自于胁迫处理后的叶片组织[44-45],与上述组织特异性表达的结果一致(图2)。在不同非生物胁迫之间,LcPIN1基因对于低温和干旱胁迫的转录响应较为一致,整体趋势为先上调再下调(图3a、3b),表明其可能在早期胁迫响应过程中发挥功能。相反,在高温胁迫下,LcPIN1基因的表达水平会迅速降低,随后短期内有一定的恢复,但随后也会迅速降低(图3c)。
为了验证非生物胁迫下LcPIN1基因的转录响应以及进一步解析其响应机制,利用PEG、ABA以及PEG和ABA合成抑制剂氟啶酮混合处理并进行RT-qPCR实验(图3d—3f)。结果显示,LcPIN1基因均对ABA处理敏感,尤其是LcPIN1a和LcPIN1c基因,在处理12 h后表达倍数增加了3~7倍(图3d、3f)。另外,PEG处理条件下,LcPIN1基因的表达均有不同程度的上调,在处理24 h后,表达倍数增加约3倍(图3d—3f)。在PEG和氟啶酮处理条件下,LcPIN1a基因的表达与PEG处理并无显著差异,表明PEG诱导的LcPIN1a基因表达上调并不依赖于内源的ABA合成(图3d)。与之相反,LcPIN1b和LcPIN1c基因在PEG和氟啶酮处理条件下要比单独的PEG处理表达水平显著降低,表明PEG诱导的LcPIN1b和LcPIN1c基因表达上调依赖于内源的ABA合成(图3e、3f)。
综上,这些研究结果显示了LcPIN1基因能够在短期内响应非生物胁迫而转录上调,但是响应机制可能在不同旁系同源基因之间存在差异,进而参与鹅掌楸的非生物胁迫响应。
2.4 LcPIN1a基因在拟南芥中的过表达
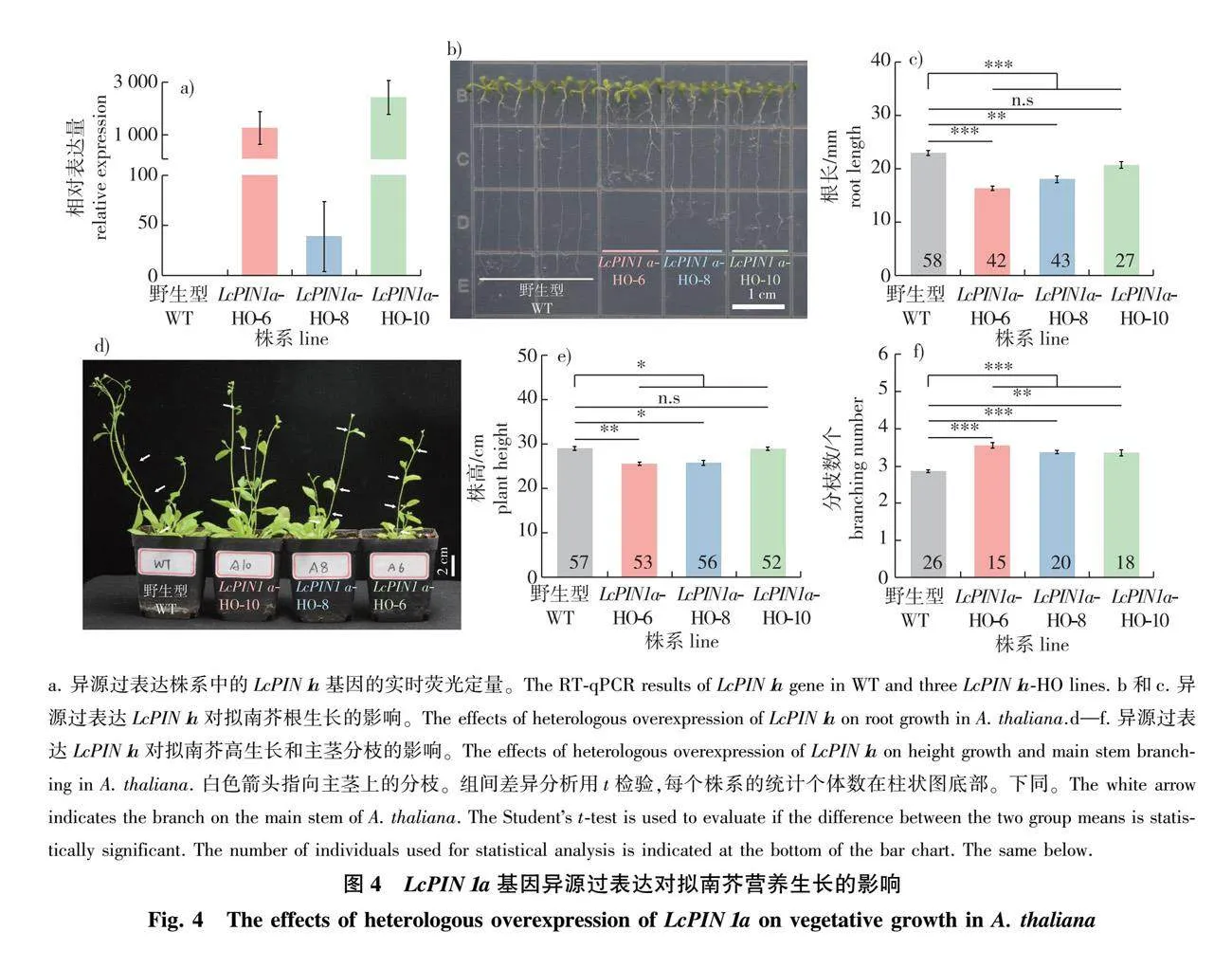
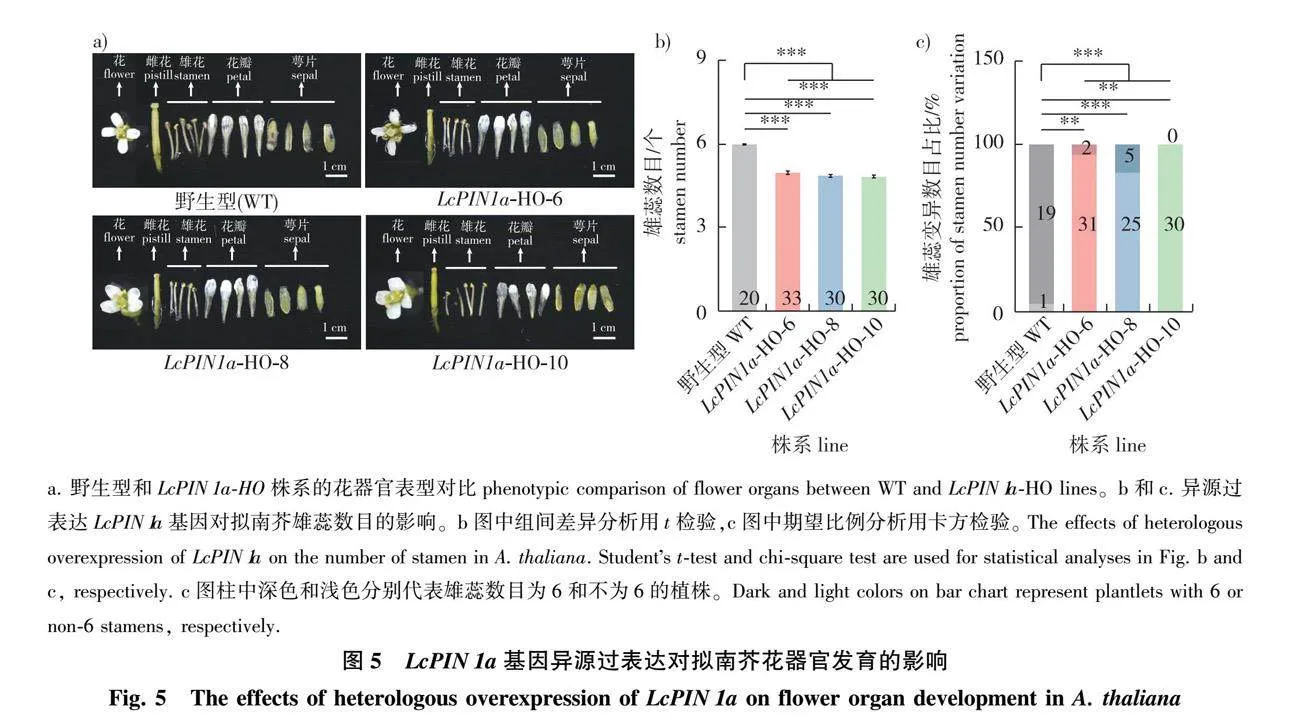
为了进一步解析鹅掌楸LcPIN1基因的功能,选择并克隆在叶片中高表达的LcPIN1a基因,通过构建CaMV35S启动子驱动的过表达载体,利用花序侵染法异源转化拟南芥,成功获得了拟南芥LcPIN1a异源过表达株系(LcPIN1a-HO,图4a)。在1/2 MS培养基上生长7 d后,野生型根长平均为(22.93 ± 7.65) mm,而LcPIN1a-HO株系的根长[平均(18.03 ± 7.81) mm]显著短于野生型(图4b、4c),表明LcPIN1a基因的异源过表达对拟南芥根生长起抑制作用。另外,通过观察拟南芥在营养土中生长第25天的表型,发现LcPIN1a-HO株系平均株高为(26.69 ± 7.95) cm,要显著矮于野生型拟南芥[平均(29.06 ± 7.15) cm,图4d、4e],并且主茎上的分枝数要显著低于野生型(图4d、4f),表明LcPIN1a基因的异源过表达抑制了拟南芥的高生长和分枝。
另外,由于鹅掌楸LcPIN1基因于生殖生长期间在雌蕊中高表达,因此进一步观察了LcPIN1a-HO株系在生殖生长时期花器官发育的表型(图5)。结果发现,与野生型正常6个雄蕊(占比95%)相比,LcPIN1a-HO株系的雄蕊数目发生了明显的变异,4和5个雄蕊的植株最多,分别占比24%和63%,6个雄蕊的植株仅占比8%(图5b、5c)。但是,LcPIN1a-HO株系的雌蕊、花瓣和萼片并未观察到数目或其他表型上的明显变异(图5a)。
2.5 LcPIN1a基因在鹅掌楸中的过表达
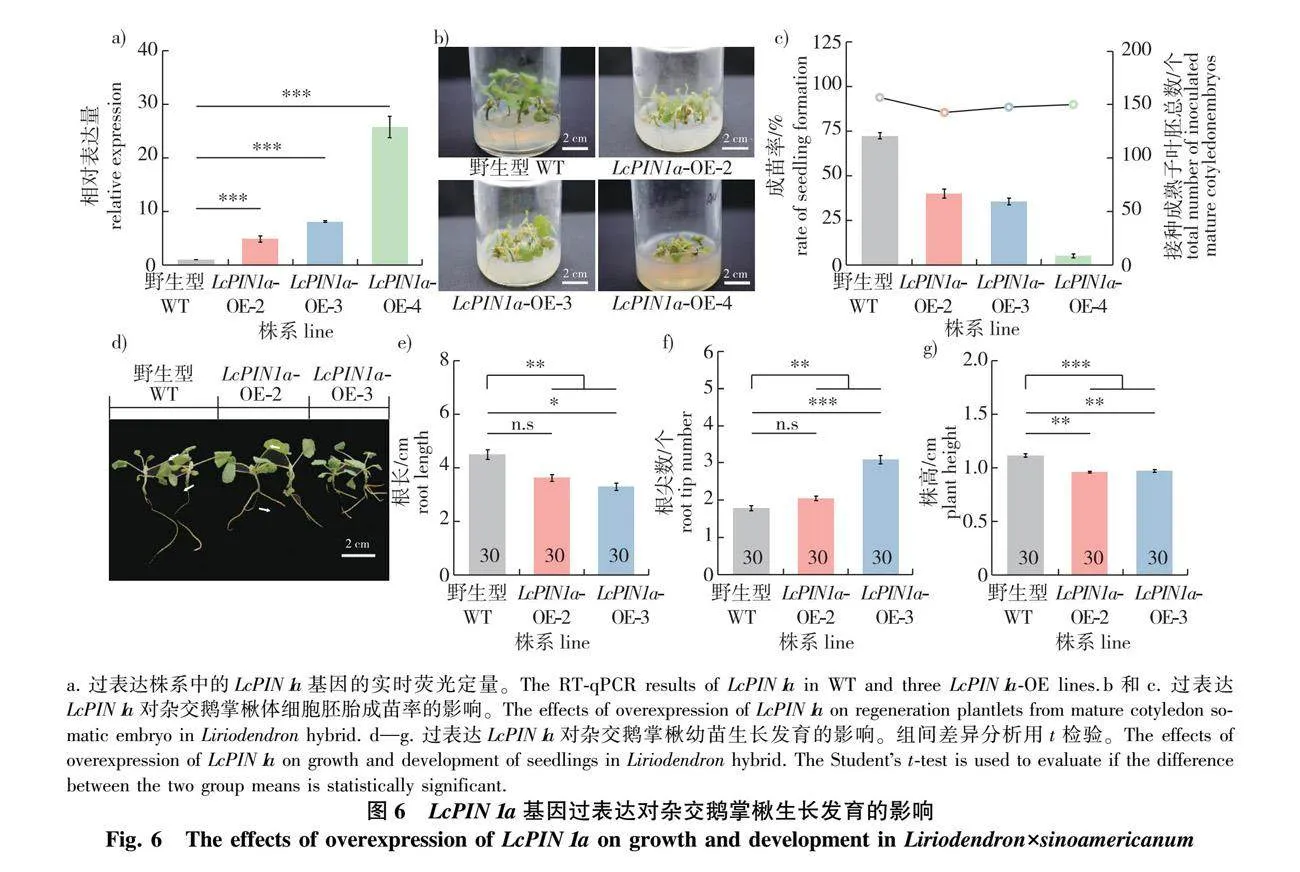
为了进一步解析LcPIN1a基因的功能,以杂交鹅掌楸胚性愈伤为受体材料进行了遗传转化,并通过对阳性愈伤的体胚发生,成功获得了杂交鹅掌楸LcPIN1a过表达株系(LcPIN1a-OE)。其中,LcPIN1a-OE株系4中LcPIN1a基因的表达倍数最高,达到了25倍左右(图6a)。以转基因阳性愈伤为材料进行体胚发生,在获得成熟子叶胚后接种到基本培养基中,结果发现2月后,LcPIN1a-OE-4株系仅能再生出7株幼苗,再生植株转化率低至5%(图6b、6c)。因此,LcPIN1a-OE-4株系未被纳入后续的表型分析。
通过对再生植株生长性状的测量与统计分析,发现LcPIN1a基因的过表达导致了杂交鹅掌楸在根长和株高方面都显著低于野生型(图6e、6g)。同时,LcPIN1a-OE株系的主根不明显,能够从根茎连接处形成多个“不定根”,其平均根尖数约为2.5个,而野生型为1.7个(图6d、6f)。综上,LcPIN1a基因的过表达对杂交鹅掌楸地上和地下器官的生长均起抑制作用,同时会影响其根系结构。
3 讨 论
植物正常的生长发育是一个复杂而又精密的过程,其同时受到外源环境因素和内源激素信号的调控。通常来说,温度、光照、水分、重力等外源环境因子需要通过影响生长素、细胞分裂素等内源激素的含量和信号转导来调控植物生长和发育,从而实现可塑性的生长发育。其中,生长素在植物响应环境的可塑性生长发育方面发挥着至关重要的作用[46]。作为生长素外排载体,PIN蛋白活性受到包括生长素、细胞分裂素、赤霉素、脱落酸、茉莉酸、水杨酸、油菜素内酯以及独脚金内酯在内的多种激素途径的调节,从而在不同的生长发育过程中介导生长素的极值点和浓度梯度的建立,最终参与植物生长发育的调控[4]。因此,PIN介导的生长素极性运输是一个整合内源性和外源性信号的中心枢纽。
鹅掌楸LcPIN1基因存在3个旁系同源基因,其中LcPIN1a主要在叶片中高表达,而LcPIN1b和LcPIN1c在幼苗时期主要在茎中高表达,而在2年生大苗中主要在根中高表达,这些结果表明,LcPIN1的3个旁系同源基因之间至少存在由于表达模式的不同而导致的功能分化。进一步的干旱胁迫响应进一步说明了LcPIN1s之间的表达模式变异,即LcPIN1a不依赖于ABA途径转录响应干旱处理,而LcPIN1b/c依赖转录响应干旱处理依赖于内源ABA的生物合成。该结果表明鹅掌楸不同LcPIN1基因拷贝之间存在着明显的转录分化,进而会导致其在生物学功能上存在一定的分化。类似的结果同样存在于水稻的4个LcPIN1旁系同源基因之间,其中OsPIN1a和OsPIN1b主要参与水稻根和茎的发育,而OsPIN1c和OsPIN1d主要参与稻穗的形成[17]。这些结果表明,通过基因复制后的表达模式变异,进而调控不同的生物学过程,可能是不同植物种采取的保守演化策略,以确保生长和发育的稳健性[47]。
另外,在PIN1介导的植物非生物胁迫响应研究方面,最近有研究报道AtPIN1能够与细胞分裂素转运蛋白AZG1互作,从而介导拟南芥根在盐胁迫下的根系结构塑造[48]。同时,AtPIN1还能够在干旱和盐胁迫条件下调控植物表皮细胞的发育,从而介导植株在不同环境下的发育稳健性和可塑性[49]。综合鹅掌楸中LcPIN1s对干旱胁迫的转录响应结果,暗示LcPIN1s可能在鹅掌楸耐旱性中发挥重要调控作用,为鹅掌楸的抗逆分子育种提供了一个候选的靶基因,相关结果有待进一步的实验验证。
在植物正常生长和发育方面,无论是在拟南芥中,还是在杂交鹅掌楸中,LcPIN1a的过表达均导致了株高和根长的显著降低。该结果与在拟南芥中过表达AtPIN1在根长方面的表型类似,即导致根长显著变短[50]。但是,在水稻中过表达OsPIN1仅会导致株高显著变矮,而主根根长却明显增加[51]。在拟南芥中,AtPIN1过表达会导致根分生组织中积累更多的生长素[52],进而抑制根的伸长生长[53]。水稻中有关OsPIN1过表达增加根长的分子机制还不清楚,但是鹅掌楸LcPIN1似乎与拟南芥AtPIN1在根长生长方面的功能更为相似。
另外,虽然OsPIN1过表达并不影响水稻的不定根数量,但是OsPIN1干扰表达明显降低了其不定根的数量[50]。同时,在柳枝稷(Panicum virgatum)中,PvPIN1过表达能够明显促进不定根的形成[54]。此外,在苹果(Malus domesticu)插条生根过程中,MdPIN1在不定根形成早期被诱导高表达[55]。结合LcPIN1a过表达导致杂交鹅掌楸主根不明显,出现多个“不定根”的表型,推测PIN1可能参与了由生长素介导的植物不定根形成[56]。相关研究结果表明LcPIN1可以作为潜在候选基因,用作未来对鹅掌楸根系生长发育的进一步研究以及根系结构的定向改造。
综上,本研究通过生物信息学分析、表达模式鉴定,以及基因功能验证,初步鉴定了鹅掌楸PIN1同源基因在植物生长和发育中的作用,发现其广泛参与了植物地上和地下器官的发育过程,并且过量表达不利于植株的正常生长和发育。
参考文献(reference):
[1]ZHAO Y D.Auxin biosynthesis and its role in plant development[J].Annu Rev Plant Biol,2010,61:49-64.DOI: 10.1146/annurev-arplant-042809-112308.
[2]FUKUI K,HAYASHI K I.Manipulation and sensing of auxin metabolism,transport and signaling[J].Plant Cell Physiol,2018,59(8):1500-1510.DOI: 10.1093/pcp/pcy076.
[3]NARAMOTO S.Polar transport in plants mediated by membrane transporters:focus on mechanisms of polar auxin transport[J].Curr Opin Plant Biol,2017,40:8-14.DOI: 10.1016/j.pbi.2017.06.012.
[4]TAN S T,LUSCHNIG C,FRIML J.Pho-view of auxin:reversible protein phosphorylation in auxin biosynthesis,transport and signaling[J].Mol Plant,2021,14(1):151-165.DOI: 10.1016/j.molp.2020.11.004.
[5]ADAMOWSKI M,FRIML J.PIN-dependent auxin transport:action,regulation,and evolution[J].Plant Cell,2015,27(1):20-32.DOI: 10.1105/tpc.114.134874.
[6]BLILOU I,XU J,WILDWATER M,et al.The PIN auxin efflux facilitator network controls growth and patterning in Arabidopsis roots[J].Nature,2005,433(7021):39-44.DOI: 10.1038/nature03184.
[7]FRIML J,BENKOV E,BLILOU I,et al.AtPIN4 mediates sink-driven auxin gradients and root patterning in Arabidopsis[J].Cell,2002,108(5):661-673.DOI: 10.1016/s0092-8674(02)00656-6.
[8]GLWEILER L,GUAN C,MLLER A,et al.Regulation of polar auxin transport by AtPIN1 in Arabidopsis vascular tissue[J].Science,1998,282(5397):2226-2230.DOI: 10.1126/science.282.5397.2226.
[9]FRIML J,VIETEN A,SAUER M,et al.Efflux-dependent auxin gradients establish the apical-basal axis of Arabidopsis[J].Nature,2003,426(6963):147-153.DOI: 10.1038/nature02085.
[10]MLLER A,GUAN C,GLWEILER L,et al.AtPIN2 defines a locus of Arabidopsis for root gravitropism control[J].EMBO J,1998,17(23):6903-6911.DOI: 10.1093/emboj/17.23.6903.
[11]FRIML J,WISNIEWSKA J,BENKOV E,et al.Lateral relocation of auxin efflux regulator PIN3 mediates tropism in Arabidopsis[J].Nature,2002,415(6873):806-809.DOI: 10.1038/415806a.
[12]DING Z J,WANG B J,MORENO I,et al.ER-localized auxin transporter PIN8 regulates auxin homeostasis and male gametophyte development in Arabidopsis[J].Nat Commun,2012,3:941.DOI: 10.1038/ncomms1941.
[13]SIMON S,SKU。PA P,VIAENE T,et al.PIN6 auxin transporter at endoplasmic reticulum and plasma membrane mediates auxin homeostasis and organogenesis in Arabidopsis[J].New Phytol,2016,211(1):65-74.DOI: 10.1111/nph.14019.
[14]OKADA K,UEDA J,KOMAKI M K,et al.Requirement of the auxin polar transport system in early stages of Arabidopsis floral bud formation[J].Plant Cell,1991,3(7):677-684.DOI: 10.1105/tpc.3.7.677.
[15]GOVINDARAJU P,VERNA C,ZHU T B,et al.Vein patterning by tissue-specific auxin transport[J].Development,2020,147(13):dev187666.DOI: 10.1242/dev.187666.
[16]NOH B,BANDYOPADHYAY A,PEER W A,et al.Enhanced gravi-and phototropism in plant mdr mutants mislocalizing the auxin efflux protein PIN1[J].Nature,2003,423(6943):999-1002.DOI: 10.1038/nature01716.
[17]LI Y,ZHU J S,WU L L,et al.Functional divergence of PIN1 paralogous genes in rice[J].Plant Cell Physiol,2019,60(12):2720-2732.DOI: 10.1093/pcp/pcz159.
[18]郝日明,贺善安,汤诗杰,等.鹅掌楸在中国的自然分布及其特点[J].植物资源与环境,1995,4(1):1-6.HAO R M,HE S A,TANG S J,et al.Geographical distribution of Liridendron chinense in China and its significance[J].J Plant Resour Environ,1995,4(1):1-6.
[19]PARKS C R,MILLER N G,WENDEL J F,et al.Genetic divergence within the genus Liriodendron (Magnoliaceae)[J].Ann Mo Bot Gard,1983,70(4):658.DOI: 10.2307/2398983.
[20]PARKS C R,WENDEL J F.Molecular divergence between Asian and north American species of Liriodendron (Magnoliaceae) with implications for interpretation of fossil floras[J].Am J Bot,1990,77(10):1243.DOI: 10.2307/2444585.
[21]李火根,施季森.杂交鹅掌楸良种选育与种苗繁育[J].林业科技开发,2009,23(3):1-5.LI H G,SHI J S.Breeding and propagation of Liriodendron hybrids[J].J For Eng,2009,23(3):1-5.DOI: 10.3969/j.issn.1000-8101.2009.03.001.
[21]李火根,施季森.杂交鹅掌楸良种选育与种苗繁育[J].林业科技开发,2009(3):1-5.LI H G,SHI J S.Breeding and propagation of Liriodendron hybrids[J].China For Sci Technol,2009(3):1-5. DOI:10.3969/j.issn.1000-8101.2009.03.001.
[21]李火根,施季森.杂交鹅掌楸良种选育与种苗繁育[J].林业科技开发,2009,23(3):1-5.LI H G,SHI J S.Breeding and propagation of Liriodendron hybrids[J].China For Sci Technol,2009,23(3):1-5. DOI:10.3969/j.issn.1000-8101.2009.03.001.
[22]向其柏,王章荣.杂交马褂木的新名称:亚美马褂木[J].南京林业大学学报(自然科学版),2012,36(2):1-2.SHANG C B,WANG Z R.A new scientific name of hybrid Liriodendron: L. sino americanum[J].J Nanjing For Univ (Nat Sci Ed),2012,36(2):1-2.DOI: 10.3969/j.issn.1000-2006.2012.02.001.
[23]CHEN J H,HAO Z D,GUANG X M,et al.Liriodendron genome sheds light on angiosperm phylogeny and species-pair differentiation[J].Nat Plants,2019,5(1):18-25.DOI: 10.1038/s41477-018-0323-6.
[24]陈金慧,施季森,诸葛强,等.杂交鹅掌楸体细胞胚胎发生研究[J].林业科学,2003,39(4):49-53,177.CHEN J H,SHI J S,ZHUGE Q,et al.Studies on the somatic embryogenesis of Liriodendron hybrids (L. chinense × L. tulipifera)[J].Sci Silvae Sin,2003,39(4):49-53,177.DOI: 10.3321/j.issn:1001-7488.2003.04.008.
[25]LI M P,WANG D,LONG X F,et al.Agrobacterium-mediated genetic transformation of embryogenic callus in a Liriodendron hybrid (L.chinense × L.tulipifera)[J].Front Plant Sci,2022,13:802128.DOI: 10.3389/fpls.2022.802128.
[26]HU L F,WANG P K,LONG X F,et al.The PIN gene family in relic plant L.chinense:genome-wide identification and gene expression profiling in different organizations and abiotic stress responses[J].Plant Physiol Biochem,2021,162:634-646.DOI: 10.1016/j.plaphy.2021.03.030.
[27]LI R,PAN Y,HU L F,et al.PIN3 from Liriodendron may function in inflorescence development and root elongation[J].Forests,2022,13(4):568.DOI: 10.3390/f13040568.
[28]BERARDINI T Z,REISER L,LI D H,et al.The Arabidopsis information resource:making and mining the “gold standard” annotated reference plant genome[J].Genesis,2015,53(8):474-485.DOI: 10.1002/dvg.22877.
[29]JOHNSON M,ZARETSKAYA I,RAYTSELIS Y,et al.NCBI BLAST:a better web interface[J].Nucleic Acids Res,2008,36(Web Server issue):5-9.DOI: 10.1093/nar/gkn201.
[30]GOODSTEIN D M,SHU S Q,HOWSON R,et al.Phytozome:a comparative platform for green plant genomics[J].Nucleic Acids Res,2012,40(Database issue):D1178-D1186.DOI: 10.1093/nar/gkr944.
[31]SIEVERS F,HIGGINS D G.Clustal Omega for making accurate alignments of many protein sequences[J].Protein Sci,2018,27(1):135-145.DOI: 10.1002/pro.3290.
[32]TOGKOUSIDIS A,KOZLOV O M,HAAG J,et al.Adaptive RAxML-NG:accelerating phylogenetic inference under maximum likelihood using dataset difficulty[J].Mol Biol Evol,2023,40(10):msad227.DOI: 10.1093/molbev/msad227.
[33]CHEN C J,WU Y,LI J W,et al.TBtools-Ⅱ:a “one for all,all for one” bioinformatics platform for biological big-data mining[J].Mol Plant,2023,16(11):1733-1742.DOI: 10.1016/j.molp.2023.09.010.
[34]LETUNIC I,BORK P.Interactive tree of life (iTOL) v5:an online tool for phylogenetic tree display and annotation[J].Nucleic Acids Res,2021,49(W1):293-296.DOI: 10.1093/nar/gkab301.
[35]OMASITS U,AHRENS C H,MLLER S,et al.Protter:interactive protein feature visualization and integration with experimental proteomic data[J].Bioinformatics,2014,30(6):884-886.DOI: 10.1093/bioinformatics/btt607.
[36]LI T T,YUAN W G,QIU S,et al.Selection of reference genes for gene expression analysis in Liriodendron hybrids’ somatic embryogenesis and germinative tissues[J].Sci Rep,2021,11(1):4957.DOI: 10.1038/s41598-021-84518-w.
[37]LIVAK K J,SCHMITTGEN T D.Analysis of relative gene expression data using real-time quantitative PCR and the 2 (-Delta Delta C(T)) Method[J].Methods,2001,25(4):402-408.DOI: 10.1006/meth.2001.1262.
[38]MAVRODIEV E V,DERVINIS C,WHITTEN W M,et al.A new,simple,highly scalable,and efficient protocol for genomic DNA extraction from diverse plant taxa[J].Appl Plant Sci,2021,9(3):e11413.DOI: 10.1002/aps3.11413.
[39]CLOUGH S J,BENT A F.Floral dip:a simplified method for Agrobacterium-mediated transformation of Arabidopsis thaliana[J].Plant J,1998,16(6):735-743.DOI: 10.1046/j.1365-313x.1998.00343.x.
[40]KRECEK P,SKUPA P,LIBUS J,et al.The PIN-FORMED (PIN) protein family of auxin transporters[J].Genome Biol,2009,10(12):249.DOI: 10.1186/gb-2009-10-12-249.
[41]SANCHO-ANDRS G,SORIANO-ORTEGA E,GAO C J,et al.Sorting motifs involved in the trafficking and localization of the PIN1 auxin efflux carrier[J].Plant Physiol,2016,171(3):1965-1982.DOI: 10.1104/pp.16.00373.
[42]MARCOTE M J,SANCHO-ANDRS G,SORIANO-ORTEGA E,et al.Sorting signals for PIN1 trafficking and localization[J].Plant Signal Behav,2016,11(8):e1212801.DOI: 10.1080/15592324.2016.1212801.
[43]HUANG F,ZAGO M K,ABAS L,et al.Phosphorylation of conserved PIN motifs directs Arabidopsis PIN1 polarity and auxin transport[J].Plant Cell,2010,22(4):1129-1142.DOI: 10.1105/tpc.109.072678.
[44]WU W H,ZHU S,XU L,et al.Genome-wide identification of the Liriodendron chinense WRKY gene family and its diverse roles in response to multiple abiotic stress[J].BMC Plant Biol,2022,22(1):25.DOI: 10.1186/s12870-021-03371-1.
[45]WU W H,ZHU S,ZHU L M,et al.Characterization of the Liriodendron chinense MYB gene family and its role in abiotic stress response[J].Front Plant Sci,2021,12:641280.DOI: 10.3389/fpls.2021.641280.
[46]MROUE S,SIMEUNOVIC A,ROBERT H S.Auxin production as an integrator of environmental cues for developmental growth regulation[J].J Exp Bot,2018,69(2):201-212.DOI: 10.1093/jxb/erx259.
[47]GU Z L,STEINMETZ L M,GU X,et al.Role of duplicate genes in genetic robustness against 1 mutations[J].Nature,2003,421(6918):63-66.DOI: 10.1038/nature01198.
[48]TESSI T M,MAURINO V G,SHAHRIARI M,et al.AZG1 is a cytokinin transporter that interacts with auxin transporter PIN1 and regulates the root stress response[J].New Phytol,2023,238(5):1924-1941.DOI: 10.1111/nph.18879.
[49]BAWA G,LIU Z X,WU R,et al.PIN1 regulates epidermal cells development under drought and salt stress using single-cell analysis[J].Front Plant Sci,2022,13:1043204.DOI: 10.3389/fpls.2022.1043204.
[50]ZHANG J,VANNESTE S,BREWER P B,et al.Inositol trisphosphate-induced Ca2+ signaling modulates auxin transport and PIN polarity[J].Dev Cell,2011,20(6):855-866.DOI: 10.1016/j.devcel.2011.05.013.
[51]XU M,ZHU L,SHOU H X,et al.A PIN1 family gene,OsPIN1,involved in auxin-dependent adventitious root emergence and tillering in rice[J].Plant Cell Physiol,2005,46(10):1674-1681.DOI: 10.1093/pcp/pci183.
[52]MRAVEC J,KUBES M,BIELACH A,et al.Interaction of PIN and PGP transport mechanisms in auxin distribution-dependent development[J].Development,2008,135(20):3345-3354.DOI: 10.1242/dev.021071.
[53]LI K,KAMIYA T,FUJIWARA T.Differential roles of PIN1 and PIN2 in root meristem maintenance under low-B conditions in Arabidopsis thaliana[J].Plant Cell Physiol,2015,56(6):1205-1214.DOI: 10.1093/pcp/pcv047.
[54]XU K J,SUN F L,WANG Y F,et al.The PIN1 family gene PvPIN1 is involved in auxin-dependent root emergence and tillering in switchgrass[J].Genet Mol Biol,2016,39(1):62-72.DOI: 10.1590/1678-4685-GMB-2014-0300.
[55]GUAN L,LI Y J,HUANG K H,et al.Auxin regulation and MdPIN expression during adventitious root initiation in apple cuttings[J].Hortic Res,2020,7(1):143.DOI: 10.1038/s41438-020-00364-3.
[56]ROTH O,YECHEZKEL S,SERERO O,et al.Slow release of a synthetic auxin induces formation of adventitious roots in recalcitrant woody plants[J].Nat Biotechnol,2024.DOI: 10.1038/s41587-023-02065-3.
(责任编辑 吴祝华)

