小麦DBB基因家族的生物信息学与表达分析


摘要:为了解小麦DBB转录因子基因家族成员的基本特征,分析其在基因结构、蛋白结构、系统发育进化树、染色体定位、不同物种间的进化情况,并对小麦DBB基因家族成员在小麦不同组织中的表达水平进行分析。基于小麦中国春的基因组数据,共鉴定得到29个DBB基因家族成员,它们不均匀地分布在小麦15条染色体上;系统发育进化树将小麦DBB蛋白分为4个亚族,其中拟南芥DBB基因家族成员在聚类中显示出明显的特异性,与其他物种具有较远的进化关系;motif1位于小麦DBB蛋白的保守结构域,大部分TaDBB基因含有2~3个外显子,最多为5个外显子;小麦DBB基因的启动子区域具有丰富的调控植物生长发育、响应激素以及非生物胁迫的顺式作用元件,暗示TaDBB基因在调控小麦生长发育和胁迫响应中发挥着多种潜在作用;小麦DBB基因在基因组内的进化主要是通过基因片段复制完成的,且同源基因间的片段复制在进化过程中发挥着主导作用;不同物种间的进化结果表明,小麦DBB基因与玉米具有更近的亲缘关系;转录组数据的基因表达谱分析显示,TaDBB1、TaDBB15和TaDBB21基因的表达具有明显的组织特异性。该结果将为后续解析DBB基因在小麦中的生物学功能奠定基础。
关键词:小麦;DBB基因家族;生物信息学;表达分析
中图分类号:S512.103;S330 文献标志码:A
文章编号:1002-1302(2024)18-0059-09
收稿日期:2024-03-11
基金项目:国家重点研发计划(编号:2017YFD0301101、2023YFD2301502);河南省科技攻关项目(编号:232102110018)。
作者简介:连少英(1985—),女,河南滑县人,硕士,助理研究员,主要从事植物保护与小麦遗传育种研究。E-mail:lianshaoying@126.com。
通信作者:李 磊,博士,研究员,主要从事小麦遗传育种研究,E-mail:lilei@henau.edu.cn;倪永静,硕士,副研究员,主要从事植物保护与小麦遗传育种研究,E-mail:nyj317@163.com。
转录因子(transcription factors,TFs) 可以激活或抑制植物体内靶基因的转录,在调控基因表达方面发挥着重要作用[1]。目前,小麦至少包括56个转录因子类基因家族[2]。锌指蛋白是一类具有B-box结构域锌指结构的转录因子家族,该家族成员按照结构域的数量和种类可以分为不同的亚家族[3]。其中,含有B-box 基序的蛋白称为 BBX,DBB(double B-Box)家族是锌指蛋白的一个亚家族,DBB蛋白的突出特点是N端具有2个或以上的B-Box结构域,且C端缺乏CCT结构域[4]。
在拟南芥中共鉴定得到32个BBX蛋白成员,其中8个成员为DBB蛋白[5]。随后,越来越多的证据表明,植物BBX蛋白不仅参与植物的生长发育过程,在叶片发育、根的生长、果实发育、开花诱导等多种生理生化过程和非生物胁迫反应中发挥着关键作用[6]。CONSTANS (CO)/AtBBX1是首个被研究的拟南芥BBX基因,该基因通过触发Flowering Locus T (FT)基因的表达来调控开花时。在长日照条件下,co突变体显著延迟开花时间,而在长日照和短日照条件下,co过表达的转基因植物都提前开花[7]。其他的BBX基因如BBX4、BBX7和BBX32,被发现调控开花时间[8],至少有10个BBX基因已被确定为早期光形态发生的调节因子[9]。BBX蛋白也参与非生物应激反应和激素信号网络。例如,BBX18和BBX23是热形态形成的调节因子,BBX18和BBX23的缺陷突变导致热响应性下胚轴伸长减少[10]。在拟南芥中,BBX24最初是作为一种耐盐蛋白(STO)分离出来的,可增加酵母细胞的耐盐活性[9]。在拟南芥中过表达BBX24可促进高盐度条件下根系的生长[11]。BBX19是DBB蛋白的一种,在拟南芥中被鉴定为光形态发生的负调节因子,在植物激素脱落酸(ABA)的抑制作用和光的促进作用之间具有重要的连接作用。
随着越来越多植物基因组测序的完成,不同植物的DBB基因家族成员也逐渐被鉴定出来,包括水稻[12]、玉米[5]、白菜[11]、葡萄[13]、橡胶树[14]等。小麦作为一种重要粮食作物,鉴定和研究小麦DBB基因家族成员具有重要的意义。本研究,系统分析了小麦DBB基因家族成员的结构特征、系统发育关系、染色体位置和基因进化过程中的共线性关系,同时进一步探讨了其在不同小麦组织中的表达模式,研究结果可为进一步深入分析小麦DBB基因家族的功能提供参考。
1 材料与方法
1.1 试验时间与地点
本试验于2024年2月在河南省商丘市农林科学院内完成。
1.2 小麦DBB基因家族成员鉴定
利用已公开的小麦基因组版本IWGSC refseqv1.1 (http://plants.ensembl.org/)的基因组序列和蛋白序列对小麦DBB基因家族成员进行鉴定。下载已报道的拟南芥DBB蛋白序列,使用BLASTP在小麦数据库中进行检索。同时,从Pfam数据库(http://pfam.xfam.org/)下载DBB基因对应的隐马尔可夫模型PF00643,使用HMMER 3.3[15](http://www.hmmer.org/)搜索DBB基因家族成员,以e值≤1×10-5作为筛选阈值。
考虑到小麦基因组中每个基因可能有多个转录本,采用最长转录本对应的氨基酸序列来鉴定DBB基因家族成员。所有候选基因通过CDD (https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)和SMART程序(http://smart.embl-heidelberg.de/)进一步检测,以证实B-Box蛋白保守结构域的存在。
1.3 小麦DBB基因家族成员序列分析
利用在线程序Gene Structure Display Server (http://www.gsds.cbi.pku.edu.cn)确定并绘制小麦DBB基因家族成员的外显子-内含子结构。利用蛋白质序列分析的MEME在线程序(https://meme-suite.org/meme/tools/meme)鉴定小麦DBB基因家族成员中的保守基序,MEME参数设置为:motif发现模式为经典模式,位点分布为Zero or One Occurrence Per Sequence (zoops),motif发现的最大数目为5,其他参数为默认值。
1.4 小麦DBB基因家族成员的系统发育分析与分类
采用MEGA 7.0(http://www.megasoftware.net/)软件的Neighbor-Joining (NJ)方法绘制小麦、水稻、玉米和拟南芥共4种植物DBB基因家族成员的系统发育树,进化树构建选择泊松分布模型、双端比较,bootstrap设置为1 000次。
1.5 小麦DBB基因启动子顺式作用元件分析
从植物基因组数据库 (http://plants.ensembl.org/)检索各小麦DBB基因编码区上游2 000 bp的启动子序列。使用PlantCARE的在线程序(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)进行DBB基因顺式作用元件的预测[16]。
1.6 小麦DBB基因家族成员染色体分布
基于小麦基因组数据库中的物理位置信息,使用Circos软件[17]将所有DBB基因家族成员定位到小麦染色体上。采用多重共线性扫描工具(Multiple Collinearity Scan toolkit,MCScanX)分析基因重复事件,参数默认[18]。
1.7 小麦DBB基因家族成员的比较基因组分析
使用共线性绘图软件Multiple Systeny Plot (https://github.com/CJ-Chen/TBtools)绘制分别绘制小麦-拟南芥、小麦-玉米、小麦-水稻基因组之间的DBB基因家族成员共线图,进行比较基因组分析。
1.8 小麦DBB基因家族成员在小麦各器官组织中的表达分析
原始基因表达数据从小麦表达浏览器(http://www.wheat-expression.com/)下载。对来自六倍体春小麦品种Azhurnaya的测序数据进行分析,数据来自根、茎、旗叶、幼穗、雄蕊、雌蕊、籽粒等7个重要的小麦组织或器官。基因表达水平由每百万转录本(TPM)值估计,并以log2转换的规范化TPM表示,热图的可视化由TBtools软件绘制[19]。
2 结果与分析
2.1 小麦DBB基因家族成员的鉴定结果
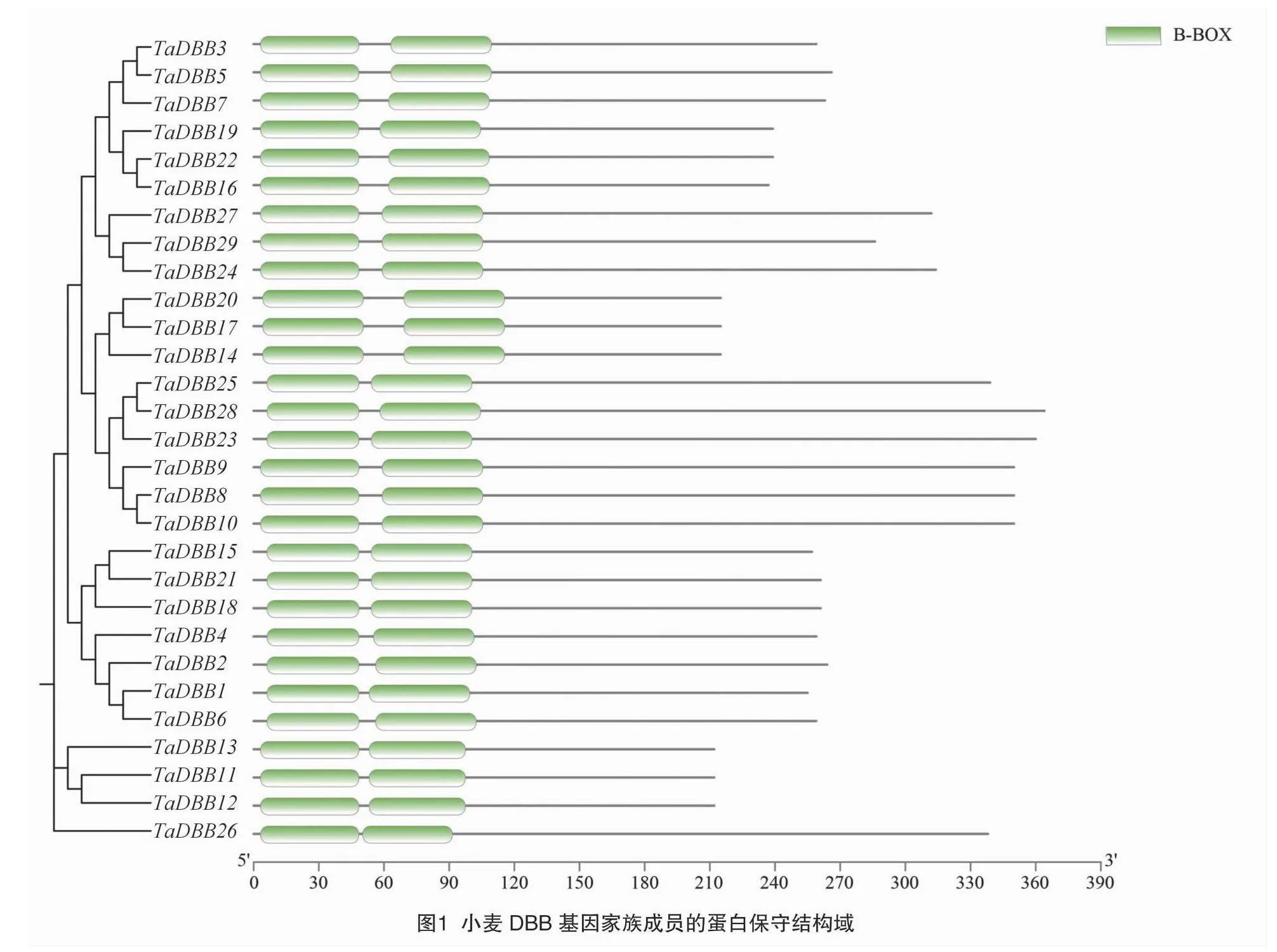
利用隐马尔可夫(HMM)模型,在小麦基因组中共获得29个DBB基因家族成员,对这些蛋白序列进一步验证B-Box结构域的存在,发现这29个DBB蛋白序列在N端都含有2个B-Box蛋白保守结构域(图1)。因此,可以判定小麦DBB基因家族共有29个成员。
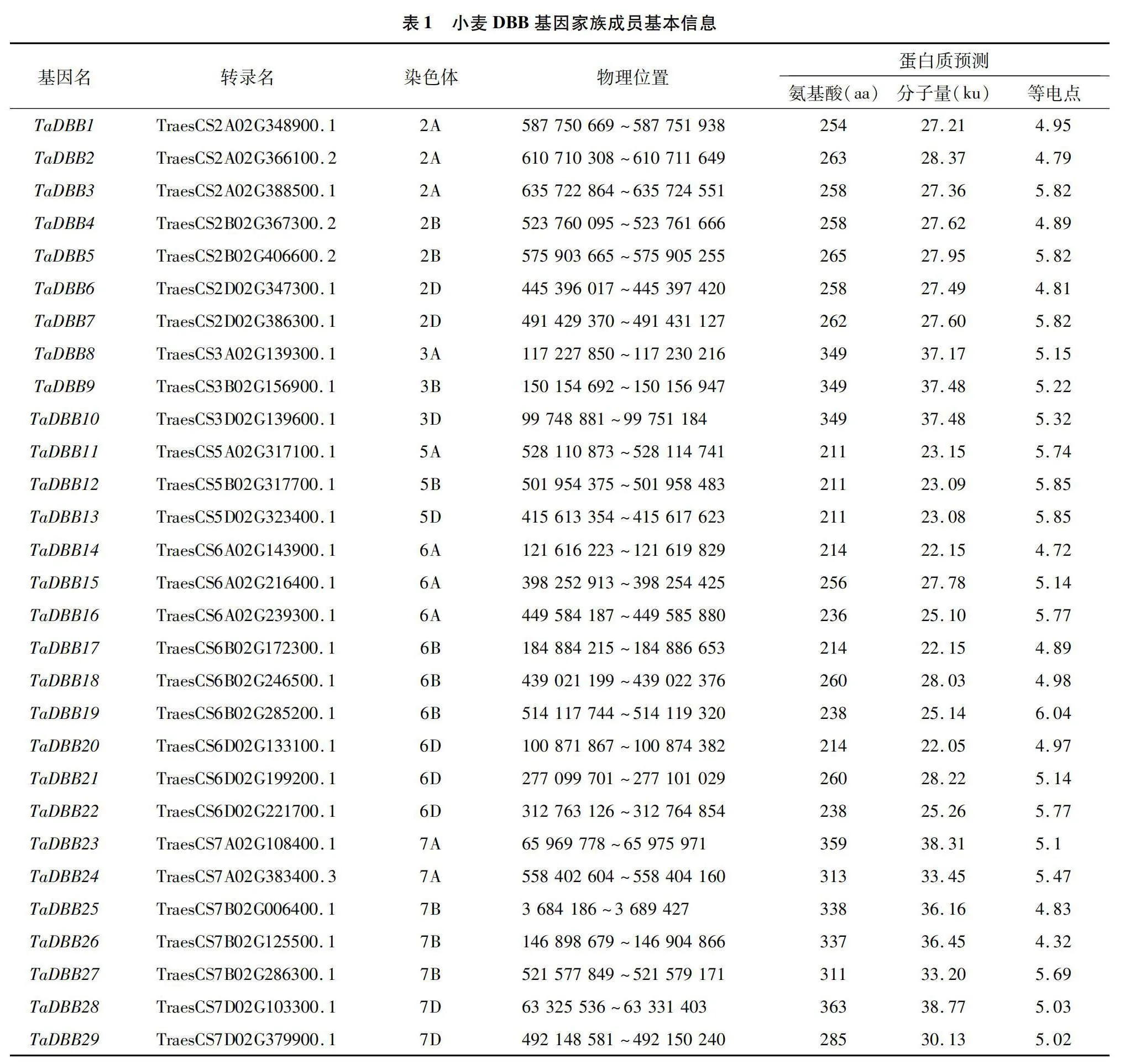
根据小麦DBB基因家族的29个成员在染色体上的位置信息,对其进行命名为TaDBB1~TaDBB29(表1),每个基因对应唯一的转录本。29个TaDBB基因不均匀地分布在除1号染色体(包括1A、1B和1D)和4号染色体(包括4A、4B和4D)的15条染色体上。对TaDBB蛋白进一步分析发现,TaDBB蛋白序列的氨基酸长度范围为211~363 aa,分子量为22.05~38.77 ku,等电点(pI)为4.32~6.04,且全部为酸性蛋白(pI<7.5),表明小麦DBB蛋白在理化性质上存在差异,暗示其基因功能的多样性,表明这些成员可能在小麦生长发育过程中发挥着不同的调控作用。
2.2 小麦DBB基因家族成员的系统发育分析
为研究小麦DBB基因家族成员的系统发育,将小麦DBB基因家族成员与拟南芥、玉米、水稻等3个物种进行聚类分析(图2),29个DBB蛋白分为Group1、Group2、Group3和Group4共4个亚族,拟南芥仅存在于Group1、Group2和Group3这3个亚族中,且拟南芥DBB显示出其聚类的特异性,与其他物种进化关系较远。此外,聚类的4个亚族中均存在小麦、水稻和玉米的DBB基因家族成员,显示小麦、水稻和玉米的DBB基因之间具有较为紧密的聚类关系,推测其功能相似。
2.3 小麦DBB蛋白保守基序和基因结构分析
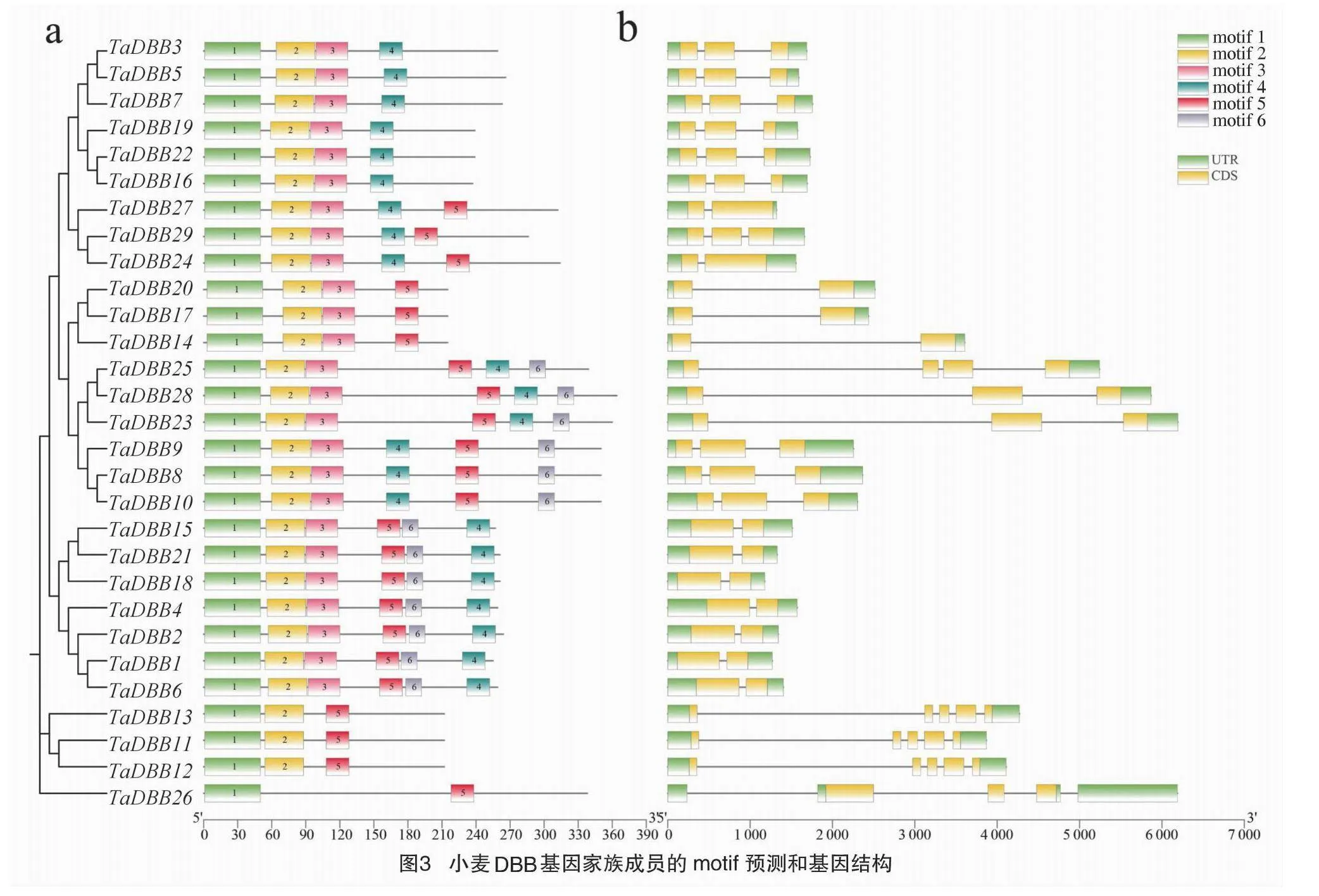
小麦DBB基因家族成员的蛋白保守基序表现出一定的差异,其中motif1在29个蛋白序列中均有分布,因此推测motif1位于小麦DBB蛋白的保守结构域。motif2在除TaDBB26蛋白外的其他28个TaDBB蛋白序列中都有分布,且主要分布在蛋白序列的N端,motif3存在于除TaDBB11、TaDBB12、TaDBB13、TaDBB26的其他25个TaDBB蛋白序列中,其他的motif4存在于除TaDBB3、TaDBB5、TaDBB7、TaDBB16、TaDBB19和TaDBB22的其他23个TaDBB蛋白序列中。此外,motif5和motif6在不同的DBB蛋白序列中也表现出差异(图3)。
小麦DBB基因家族成员的基因结构存在一定的差异(图3),具体表现在外显子和内含子数量上的差异,其中TaDBB11、TaDBB12和TaDBB13分别含有5个外显子,具有最多的外显子;大部分TaDBB基因含有2~3个外显子,基因聚类发现,每个聚类的基因簇具有相似的基因结构,比如TaDBB3、TaDBB5和TaDBB7为一簇;TaDBB16、TaDBB19和TaDBB22为一簇;TaDBB23、TaDBB25和TaDBB28为一簇等,推测其基因功能相似。根据蛋白保守基序和基因结构的分析结果,在一定程度上反映出不同DBB基因之间的差异性,以及同源基因蛋白功能的相似性。
2.4 小麦DBB基因家族成员顺式作用元件分析
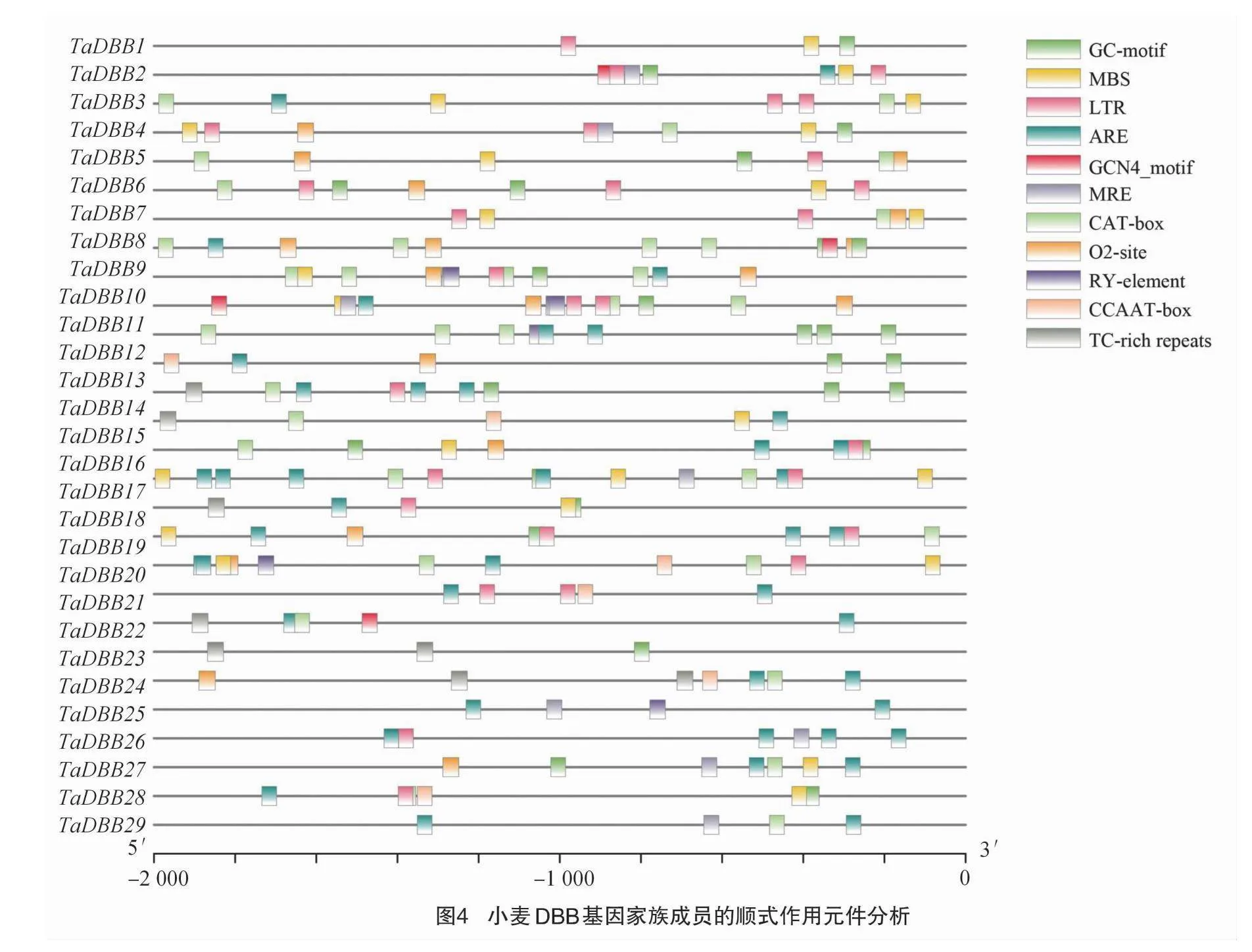
小麦DBB基因的启动子具有丰富的顺式作用元件(图4),其中与植物生长发育相关的顺式作用元件主要包括ARE、CAT-box、GC-motif、GCN4-motif、O2-site、RY-element、TGA-element;与胁迫相关的顺式作用元件CCAAT-Box、LTR、MBS、MRE、TC-rich repeats,每个DBB基因都同时包含1个或多个与植物生长发育相关或与胁迫相关的顺式作用元件,这些顺式作用元件表明TaDBB基因在调控小麦生长发育和胁迫响应中发挥着多种潜在作用。
2.5 小麦DBB基因家族成员的染色体定位和共线性分析
小麦DBB基因家族成员不均匀地分布在小麦15条染色体上(除1号和4号染色体),其中6号染色体上具有最多的TaDBB基因数量,共9个;3号和5号染色体各含有3个TaDBB基因。基因在染色体上的数量信息可以在一定程度上反映出其进化时间的长短。
通过共线性分析进一步探索DBB基因家族成员在小麦基因组内的进化机制,在小麦基因组内共发现了40个同源基因对出现片段复制(TaDBB1∶4、TaDBB 3∶5、TaDBB 1∶6等)(图5),共线性关系主要以片段复制的形式存在与同源染色体间,未发现有串联复制事件的发生。如2A、2B和2D之间存在6个共线性基因对,3A、3B和3D之间存在3个共线性基因对,5A、5B和5D之间存在3个共线性基因对,6A、6B和6D之间存在9个共线性基因对,7A、7B和7D之间存在6个共线性基因对;值得一提的是,在不同染色体间(2号染色体和6号染色体)也存在大量的共线性基因对,共13个共线性基因对,表明DBB基因在2号染色体和6号染色体经历了更长的进化时间,TaDBB基因在小麦基因组内的进化主要是通过基因片段复制完成的,且同源基因间的片段复制在进化过程中发挥着主导作用。
2.6 小麦DBB基因家族成员的比较基因组分析
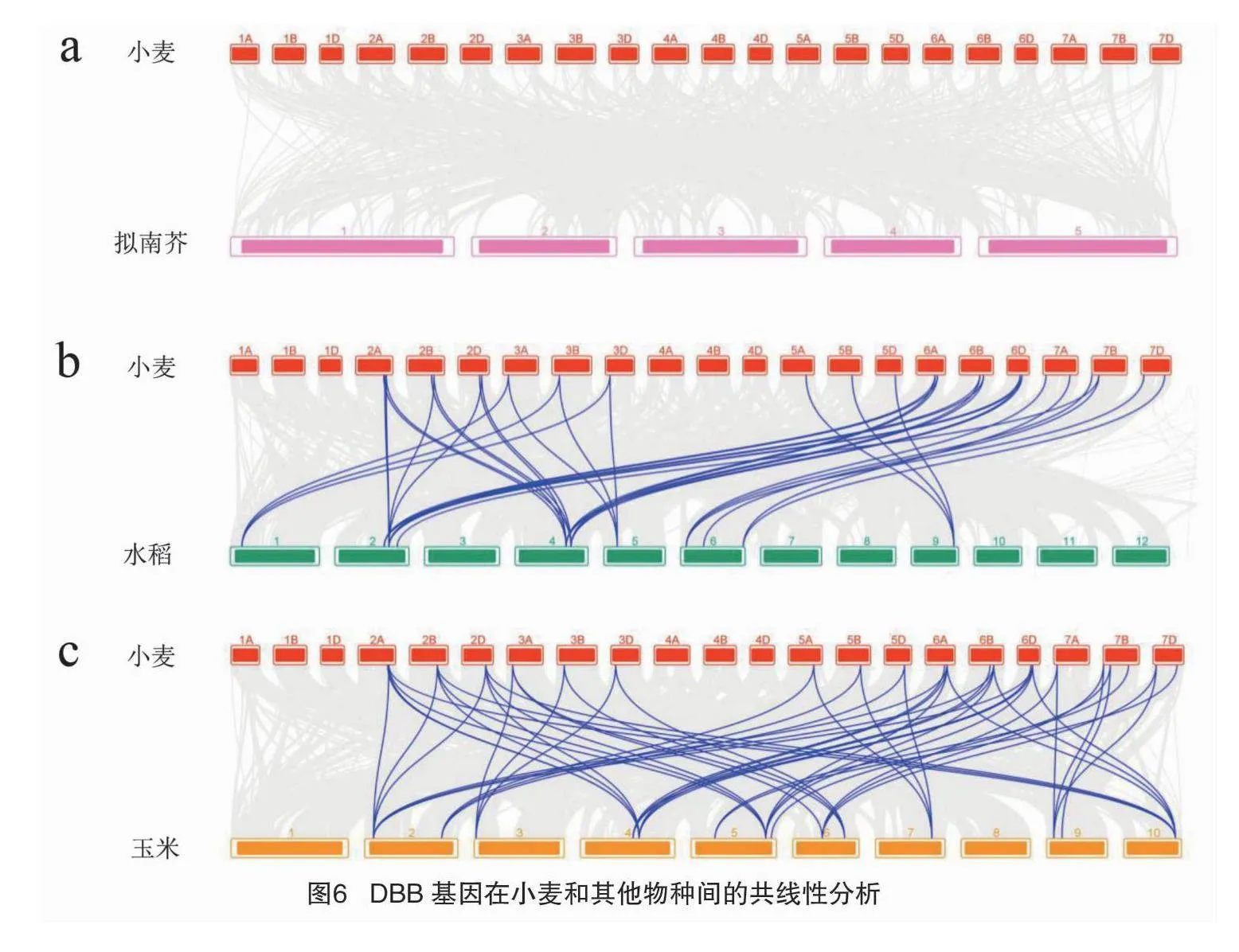
对小麦和其他3个代表性植物(包括十字花科模式植物拟南芥、水稻和玉米)进行不同基因组间的共线性分析(图6)。结果发现,小麦和拟南芥的基因组中不存在DBB基因的共线性关系;小麦和水稻的基因组间存在38个DBB基因的共线性关系;小麦和玉米的基因组中存在51个DBB基因的共线性关系,小麦和玉米基因组拥有的共线性基因对数目最多。表明DBB基因在双子叶植物中拥有更快的进化速度,且相比水稻,DBB基因在小麦和玉米之间具有更近的亲缘关系。
2.7 TaDBB基因在不同组织中的表达模式
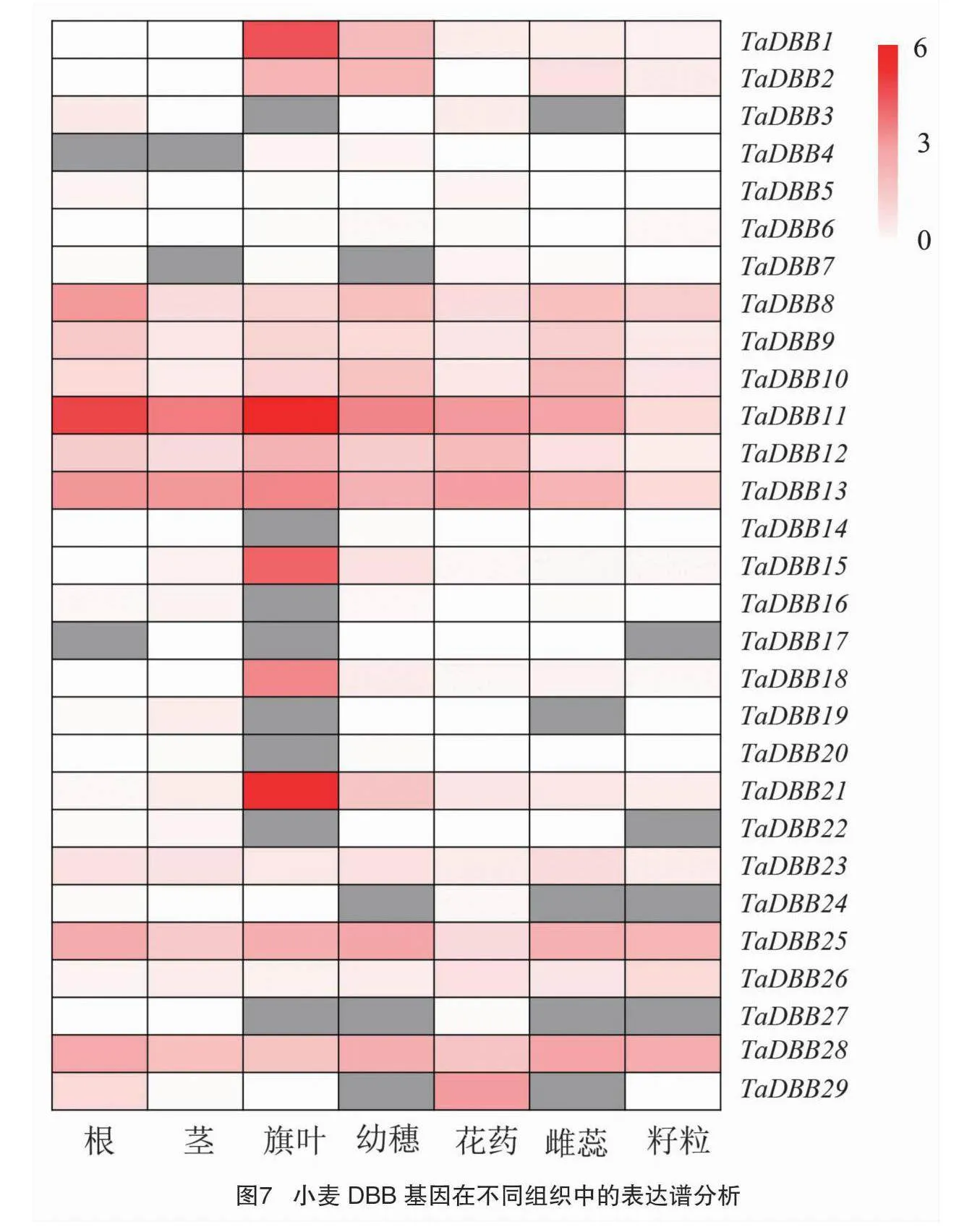
对29个TaDBB基因家族成员的表达模式进行分析,结果(图7)表明,TaDBB基因在不同的植物组织中表达模式具有明显差异。总体来看,小麦DBB家族基因主要在小麦叶片的生长发育中具有较高的表达水平,大部分基因在小麦各个组织的表达水平接近,一般表现出基因表达水平较低或者不表达,如TaDBB5、TaDBB6、TaDBB8、TaDBB9和TaDBB10,推测这些基因在小麦的生长发育过程中发挥着较为基础的作用以保障小麦正常的生长发育。部分基因的表达水平表现出明显的组织特异性,如TaDBB1、TaDBB15和TaDBB21在小麦的旗叶中表达水平显著较高。
3 讨论与结论
本研究中鉴定得到29个小麦DBB基因家族成员,将小麦与拟南芥、水稻、玉米的DBB基因进行系统发育分析,所有DBB基因可划分为4个亚类(图2)。同一亚家族中大多数DBB基因具有相似的外显子/内含子结构,具有相似结构的GRF基因具有相似的进化历史和功能,在一定程度上反映了植物DBB基因的进化关系[20]。在每个分支中,拟南芥的DBB基因与其他物种的DBB基因都表现出更远的亲缘关系,而单子叶(小麦、水稻和玉米)的DBB基因则聚集在一起,表明单子叶和双子叶分化后DBB基因是分开进化的,这一发现与报道的结果[21]一致,基因的串联复制事件和片段复制事件在基因家族成员的扩增中起着至关重要的作用。
本研究对小麦DBB基因家族的串联复制和片段复制进行分析后发现,在小麦基因组中,发现存在大量片段复制的基因对,未发现串联复制。结果表明,小麦DBB基因家族的扩增主要是通过片段重复事件进行的。此外,在其他的小麦基因家族中也发现有类似的基因家族进化的潜在机制[22]。通过分析DBB基因在不同组织或部位的表达模式可以预测其基因功能的多样性,研究发现,DBB基因不仅具有不同的基因结构,不同的基因表达模式也有很大的差异,但基因的表达模式与启动子密切相关[23]。到目前为止,DBB基因的功能研究大多集中在拟南芥中进行,大多数TaDBBs也具有典型的组织特异性表达谱,表明它们在小麦发育中的不同功能。值得一提的是,有研究表明,DBB基因在拟南芥中与光信号转导途径有关,本研究发现,TaDBB1、TaDBB15和TaDBB21在小麦的旗叶中表达水平显著较高,具有明显的组织特异性,该发现可以为研究小麦的叶片发育提供重要参考。
参考文献:
[1]Gong W,Shen Y P,Ma L G,et al. Genome-wide ORFeome cloning and analysis of Arabidopsis transcription factor genes[J]. Plant Physiology,2004,135(2):773-782.
[2]Jin J P,Tian F,Yang D C,et al. PlantTFDB 4.0:toward a central hub for transcription factors and regulatory interactions in plants[J]. Nucleic Acids Research,2017,45(D1):D1040-D1045.
[3]Kumagai T,Ito S,Nakamichi N,et al. The common function of a novel subfamily of B-box zinc finger proteins with reference to circadian-associated events in Arabidopsis thaliana[J]. Bioscience,Biotechnology,and Biochemistry,2008,72(6):1539-1549.
[4]Khanna R,Kronmiller B,Maszle D R,et al. The Arabidopsis B-box zinc finger family[J]. The Plant Cell,2009,21(11):3416-3420.
[5]LiW L,Wang J C,Sun Q,et al. Expression analysis of genes encoding double B-box zinc finger proteins in maize[J]. Functional & Integrative Genomics,2017,17(6):653-666.
[6]Ding L,Wang S,Song Z T,et al. Two B-box domain proteins,BBX18 and BBX23,interact with ELF3 and regulate thermomorphogenesis in Arabidopsis[J]. Cell Reports,2018,25(7):1718-1728.e4.
[7]Tripathi P,Carvallo M,Hamilton E E,et al. Arabidopsis B-BOX32 interacts with CONSTANS-LIKE3 to regulate flowering[J]. Proceedings of the National Academy of Sciences of the United States of America,2017,114(1):172-177.
[8]Wei H R,Wang P P,Chen J Q,et al. Genome-wide identification and analysis of B-BOX gene family in grapevine reveal its potential functions in berry development[J]. BMC Plant Biology,2020,20(1):72.
[9]Lippuner V,Cyert M S,Gasser C S. Two classes of plant cDNA clones differentially complement yeast calcineurin mutants and increase salt tolerance of wild-type yeast[J]. The Journal of Biological Chemistry,1996,271(22):12859-12866.
[10]Nagaoka S,Takano T. Salt tolerance‐related protein STO binds to a Myb transcription factor homologue and confers salt tolerance in Arabidopsis[J]. Journal of Experimental Botany,2003,54(391):2231-2237.
[11]陈 雨,邓九州,吴晓宇,等. 白菜DBB基因家族的鉴定与表达分析[J]. 山东农业大学学报(自然科学版),2021,52(2):174-181.
[12]郝爱平,韩雪杨,薛巨坤,等. 水稻DBB基因家族的生物信息学分析 [J]. 西南农业学报,2023,36(10):2135-2143.
[13]Zhang X,Zhang L,Ji M,et al. Genone wide idenification and expresion analysis of the B-box transcription factor gene family in grapevine (Vitis vinifera L.)[J]. BMC Genomics,2021,22(1):221.
[14]张鸿韬,杨玉海,肖小虎,等. 巴西橡胶树HbDBB家族基因的鉴定与表达分析[J]. 分子植物育种,2022,20(13):4314-4322.
[15]Finn R D,Clements J,Eddy S R. HMMER web server:interactive sequence similarity searching[J]. Nucleic Acids Research,2011,39(S2):W29-W37.
[16]Lescot M,Déhais P,Thijs G,et al. PlantCARE,a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences[J]. Nucleic Acids Research,2002,30(1):325-327.
[17]KrzywinskiM,Schein J,Birol I,et al. Circos:an information aesthetic for comparative genomics[J]. Genome Research,2009,19(9):1639-1645.
[18]Wang Y P,Tang H B,Debarry J D,et al. MCScanX:a toolkit for detection and evolutionary analysis of gene synteny and collinearity[J]. Nucleic Acids Research,2012,40(7):e49.
[19]Chen C J,Chen H,Zhang Y,et al. TBtools:an integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant,2020,13(8):1194-1202.
[20]Roy S W,Penny D. On the incidence of intron loss and gain in paralogous gene families[J]. Molecular Biology and Evolution,2007,24(8):1579-1581.
[21]Huang J Y,Zhao X B,Weng X Y,et al. The rice B-box zinc finger gene family:genomic identification,characterization,expression profiling and diurnal analysis[J]. PLoS One,2012,7(10):e48242.
[22]Li J C,Jiang Y M,Zhang J,et al. Key auxin response factor (ARF) genes constraining wheat tillering of mutant dmc[J]. PeerJ,2021,9:e12221.
[23]Cao S H,Kumimoto R W,Gnesutta N,et al. A distal ccaat/nuclear factor y complex promotes chromatin looping at the flowering locus t promoter and regulates the timing of flowering in Arabidopsis[J]. The Plant Cell,2014,26(3):1009-1017.

