复合侵染云南西番莲的两种菜豆金色花叶病毒属病毒的分子鉴定及序列分析






摘要西番莲Passiflora caerulea是我国南部省区广泛种植的一种热带果树。本研究于2020年6月从云南省德宏州西番莲种植区采集到4份疑似感染菜豆金色花叶病毒属病毒的西番莲样品,利用PCR扩增、克隆、测序以及生物信息学分析等方法,明确了西番莲样品受菜豆金色花叶病毒属病毒侵染。对4份西番莲样品进行菜豆金色花叶病毒属病毒全基因组扩增,从中共获得5条菜豆金色花叶病毒属病毒全基因组序列,分别命名为YN7292-6、YN7293-12、YN7293-4、YN7294-16和YN7295-25,均具有菜豆金色花叶病毒属单组分病毒的典型结构,编码7个开放阅读框(open reading frame, ORF);其中YN7292-6、YN7293-12、YN7294-16及YN7295-25与一品红曲叶病毒(Euphorbia leaf curl virus, EuLCuV)的不同分离物的核苷酸相似性均高于99.05%,为EuLCuV的不同分离物;而YN7293-4则与分离自江西西番莲样品的广东番木瓜曲叶病毒(papaya leaf curl Guangdong virus, PaLCuGdV)分离物GNPF1(GenBank登录号:MK673114.1)的核苷酸相似性最高,为99.70%,为PaLCuGdV的一个分离物;重组分析发现,2个分离物EuLCuV-YN7293-12和PaLCuGdV-YN7293-4 均为重组病毒。本研究首次在云南省发现EuLCuV和PaLCuGdV这2种菜豆金色花叶病毒属病毒复合侵染西番莲,为有效预防和控制西番莲病毒病害的发生和传播提供理论依据。
关键词广东番木瓜曲叶病毒;一品红曲叶病毒;西番莲;复合侵染;重组
中图分类号:S 432.1文献标识码:ADOI:10.16688/j.zwbh.2023514Molecular identification and sequence analysis of two begomoviruses from
Passiflora caerulea in Yunnan province, ChinaZHONG Jing1,LI Tingting1,ZHAO Liling1,HOU Yue2,YE Suzheng1,XIE Mian2,DING Ming1*(1. Key Laboratory of Agricultural Biotechnology of Yunnan Province, Key Laboratory of the Southwestern Crop
Gene Resources and Germplasm Innovation, Ministry of Agriculture and Rural Affairs, Institute of Biotechnology
and Germplasm Resources, Yunnan Academy of Agricultural Sciences, Kunming650223, China; 2. Agricultural
Technology Extension Center of Mangshi County, Dehong Prefecture, Mangshi678400, China)AbstractPassiflora caerulea is a kind of tropical fruit tree widely planted in the southern provinces of China. The suspected virus-infected P.caerulea has been found in the growing area of Dehong prefecture, Yunnan province. In this study four P.caerulea samples were collected in June 2020 and identified using PCR amplification, cloning, sequencing and bioinformatics analysis. Five full genome sequences, namely YN7292-6, YN7293-12, YN7294-4, YN7294-16 and YN7295-25 were obtained from this four samples, they all have typical monopartite begomoviruses genome structure, encoding seven open reading frames (ORF). The sequences YN7292-6, YN7293-12, YN7294-16 and YN7295-25 were most closed to different isolates of Euphorbia leaf curl virus (EuLCuV) with the similarity higher than 99.05% in total nucleotide sequences. The sequence YN7293-4 were most closely related to isolate GNPF1(GenBank accession no: MK673114.1)of papaya leaf curl Guangdong virus (PaLCuGdV) with the similarity of 99.70% in total nucleotide sequences. Recombinant analysis showed that EuLCuV-YN7293-12 and PaLCuGdV-YN7293-4 were both recombinant begomoviruses. This is first report of EuLCuV and PaLCuGdV synergistically infected passionflower in Yunnan province, which provide a theoretical basis for effective prevention and control of P.caerulea viral diseases.
Key wordspapaya leaf curl Guangdong virus;Euphorbia leaf curl virus;Passiflora caerulea;mixed infection;
recombination双生病毒科Geminiviridae病毒是广泛分布于全球热带及亚热带地区的一类单链环状DNA病毒,对全球植物具有严重的危害。但近年来,双生病毒在温带设施大棚中也时有发生,说明该类病毒的分布范围在不断扩大,推测其危害可能将会日趋严重[12]。根据国际病毒分类委员会(International Committee on Taxonomy of Viruses, ICTV)的分类将双生病毒科分为14个属[3],分别为玉米线条病毒属Mastrevirus、曲顶病毒属Curtovirus、番茄伪曲顶病毒属Topocuvirus、菜豆金色花叶病毒属Begomovirus、甜菜曲顶病毒属Becurtovirus、画眉草病毒属Eragrovirus、芜菁曲顶病毒属Turncurtovirus、葡萄斑点病毒属Grablovirus、美杜莎大戟潜隐病毒属Capulavirus[45]、柑橘褪绿矮缩相关病毒属Citlodavirus、苹果病毒属Maldovirus、桑叶皱叶病毒属Mulcrilevirus、仙人掌病毒属Opunvirus和番茄顶叶卷曲病毒属Topilevirus[6],其中菜豆金色花叶病毒属病毒是种类最多、分布最广和造成危害最严重的一个属,该属病毒主要通过烟粉虱Bemisia tabaci以持久性方式传播,因此又称为粉虱传双生病毒。该属病毒为环状单链DNA病毒,病毒粒子大小约为18 nm×30 nm,主要侵染双子叶植物[78]。根据基因组类型该属病毒分为单组分和双组分病毒,双组分病毒包含了2条大小为2.5~2.8 kb的DNA分子(DNA-A和DNA-B),如菜豆金色花叶病毒(bean golden mosaic virus, BGMV)[9],单组分病毒仅含一条大小约为2.8 kb的DNA分子(DNA-A),但单组分菜豆金色花叶病毒属病毒通常还伴随卫星分子,如中国番茄黄化曲叶病毒(tomato yellow leaf curl China virus, TYLCCNV),通常伴随中国番茄黄化曲叶beta卫星分子(tomato yellow leaf curl China betasatellite, TYLCCNB)[10]。
西番莲Passiflora caerulea是西番莲科Passifloracea西番莲属Passiflora植物,原产于南美洲,现广泛种植于热带和亚热带地区,至今已在我国台湾、广东、海南、福建、贵州、广西、云南、四川等省区大量推广栽种[1112]。西番莲植株用途较广,可作为食用、药用和绿化观赏苗木等[13],由于其果肉和果汁都具有独特的香味也被称为百香果。近年来,西番莲在我国南方种植面积不断扩大,已成为部分区域特色产业中的重要果树。前期病害调查发现,随着云南省西番莲种植规模不断扩大,不同区域西番莲种植田块均有病毒病发生,严重时发病率高达100%,严重影响西番莲的产量、质量和该产业的持续、健康发展,明确西番莲病毒病的病原从而精准防治西番莲病毒病对于西番莲产业发展显得尤为重要。
1993年在波多黎各首次报道了菜豆金色花叶病毒属病毒侵染西番莲,通过田间症状描述为西番莲叶斑驳病[14],后经鉴定为麻风树花叶病毒(Jatropha mosaic virus, JMV)[15];2003年Novaes等在巴西分离到了西番莲小叶花叶病毒(passionflower flower little leaf mosaic virus, PLLMV)[16];2017年Vaca-Vaca等在哥伦比亚从西番莲中分离并命名了西番莲叶片皱缩病毒(passionflower fruit leaf distortion virus, PLDV)[17];国内台湾省已报道侵染西番莲的菜豆金色花叶病毒属病毒有2个种,分别为一品红曲叶病毒(Euphorbia leaf curl virus, EuLCuV)和广东番木瓜曲叶病毒(papaya leaf curl Guangdong virus, PaLCuGdV)[18];随后在广西、福建等地也发现这2种菜豆金色花叶病毒属病毒可侵染西番莲植株[1920]。本研究对云南省德宏州疑似感染双生病毒的西番莲植株进行病毒种类鉴定,并通过分子生物信息学相关分析方法进行病毒序列进化及重组分析,以期为该区域西番莲病毒病害防控提供理论依据。
1材料和方法
1.1材料
1.1.1样品来源
样品采集:2020年10月从云南省德宏州芒市西番莲种植区采集西番莲疑似感染病毒的样品4株,将样品编号并装入带标记的自封袋中(图1),置于4℃冰箱中保存备用。
1.1.2试剂和仪器
试剂:Taq Plus DNA聚合酶购自上海申能博彩生物科技有限公司;dNTPs(2.5 μmol/L)、DNA Ladder Marker购自TaKaRa公司;KOD FX DNA聚合酶购自日本东洋纺;AxyPrep DNA凝胶回收试剂盒购自美国Axygen;琼脂糖购自德国BioFroxx;pEASY-Blunt Zero Cloning Kit购自北京全式金生物技术有限公司;pGEM-T Easy购自美国Promega公司;T4 DNA连接酶购自赛默飞世尔科技(中国)有限公司;TempliPhiTM Kit购自GE Healthcare Life Sciences公司。
仪器:5418R型高速台式离心机,Eppendof;C1000 Touch Thermal Cycler,BIO-RAD;DK-8D型数显恒温水浴锅,江苏金怡仪器科技有限公司;Tissuelyser-192型全自动样品快速研磨仪、MK2000-2E型干式恒温器,上海净信实业发展有限公司;SKP-02.420型电热恒温培养箱,黄石市恒丰医疗器械有限公司;THZ-C-1全温振荡器、琼脂糖水平电泳仪、SIM-F140AY6制冰机,SANYO。
1.2试验方法
1.2.1总DNA的提取
西番莲样品总DNA的提取采用CTAB法[21],提取的DNA用超纯水溶解后于-20℃冰箱保存备用。
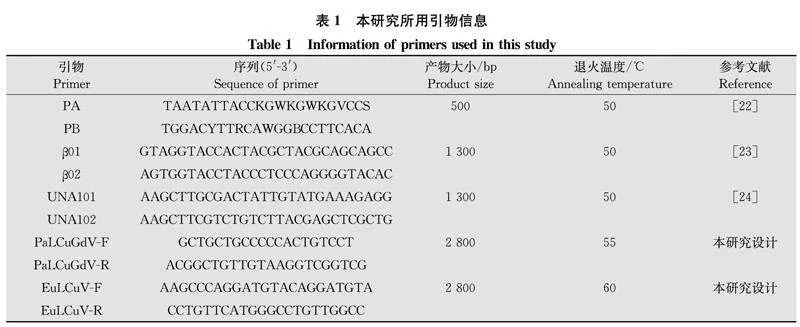
1.2.2西番莲样品中菜豆金色花叶病毒属病毒检测用菜豆金色花叶病毒属病毒的通用引物PA/PB(表1)[22]对4份西番莲样品进行PCR检测,同时用beta卫星分子和alpha卫星分子通用引物β01/β02[23]和UNA101/UNA102[24](表1)对4份西番莲样品进行PCR检测,以含有菜豆金色花叶病毒属病毒全基因组的重组质粒为阳性对照,以健康西番莲样品为阴性对照。
PCR反应体系:10×PCR 反应缓冲液(含 Mg2+)5 μL,dNTPs(2.5 μmol/L)4 μL,F/R 引物(20 μmol/L)各 1 μL,Taq Plus DNA 聚合酶(5 U/μL)1 μL,模板DNA 0.8 μL,加超纯水定容至50 μL。PCR扩增条件:94℃预变性 2 min;94℃变性45 s,50℃退火45 s,72℃延伸60 s(PA/PB)或90 s(β01/β02和UNA101/UNA102),32个循环;72℃延伸10 min。将PCR产物于1.0%琼脂糖凝胶中进行电泳检测,利用AxyPrep DNA凝胶回收试剂盒进行纯化回收目标条带,将回收产物与pGEM-T Easy载体连接,热激法转化到大肠杆菌DH5α中后进行涂板,倒置过夜培养,挑取阳性克隆,送华大基因测序。
1.2.3西番莲样品中菜豆金色花叶病毒属病毒滚环扩增利用TempliPhiTM Kit对样品DNA进行滚环扩增(rolling circle amplification, RCA),RCA可对环状DNA进行扩增,实现靶核酸信号的放大。具体步骤:在0.2 mL离心管中加入1 μL植物总DNA样品和5 μL样品缓冲液,95℃变性3 min后于冰上放置10 min,再向离心管中加入0.2 μL酶和5 μL反应缓冲液,在30℃条件下反应18 h,65℃灭活10 min,4℃ 10 min,反应结束后于-20℃保存备用。
1.2.4西番莲样品中菜豆金色花叶病毒属病毒全基因组扩增根据PA/PB引物扩增、克隆及测序后所得序列设计病毒全长引物PaLCuGdV-F/-R和EuLCuV-F/-R(表1)分别对PaLCuGdV和EuLCuV的全基因组序列进行扩增,分别以含有PaLCuGdV和EuLCuV全基因组的重组质粒为阳性对照,以健康西番莲样品为阴性对照。PCR扩增体系:2×PCR 反应缓冲液(含 Mg2+)25 μL,dNTPs(2 mmol/L)10 μL,F/R引物(20 μmol/L)各1 μL,KOD FX DNA 聚合酶(1 U/μL)1 μL,RCA 产物 0.5 μL,加超纯水定容至50 μL。PCR扩增条件:98℃预变性2 min;98℃变性10 s,49℃退火30 s,68℃延伸 3 min,32个循环;68℃延伸10 min。PCR产物于1.0%琼脂糖凝胶中进行电泳检测,利用AxyPrep DNA凝胶回收试剂盒纯化回收目标条带,利用pEASY-Blunt Zero Cloning Kit与载体进行连接,42℃热激转化到大肠杆菌DH5α中后进行涂板,倒置培养12 h,挑取阳性克隆,送华大基因测序。
1.2.5数据分析
首先,通过NCBI中的BLAST(https:∥blast.ncbi.nlm.nih.gov/Blast.)进行基因序列相似性初步比较,其次,采用DNAStar Lasergene V 7.1(Madison,Wis.,USA)软件进一步比较基因序列相似性,并寻找基因组序列中的开放阅读框;使用MEGA6中的邻近法(neighbor-joining)构建系统进化树[25],进化树的可信度使用1 000次自导复制验证;采用Recombination Detection Program(RDP)V.4.56[26]的默认设置进行序列的重组分析。
2结果与分析
2.1西番莲病样中菜豆金色花叶病毒属病毒检测用菜豆金色花叶病毒属病毒通用引物PA/PB(表1)分别对4个西番莲样品YN7292、YN7293、YN7294和YN7295进行PCR检测,结果4个样品均扩增到片段长度约为500 bp的目的条带(图2)。通过测序获得4条序列,分别命名为YN7292-1、YN7293-3、YN7294-2和YN7295-5,将所获得的核苷酸序列进行BLAST比对后发现,YN7293-3与广东番木瓜曲叶病毒各分离物的核苷酸序列相似性在82.91%~95.22%之间,YN7292-1、YN7294-2和YN7295-5与一品红曲叶病毒各分离物的核苷酸序列相似性在83.1%~97.6%之间。因此,推测西番莲样品YN7292、YN7293、YN7294和YN7295均受菜豆金色花叶病毒属病毒侵染。
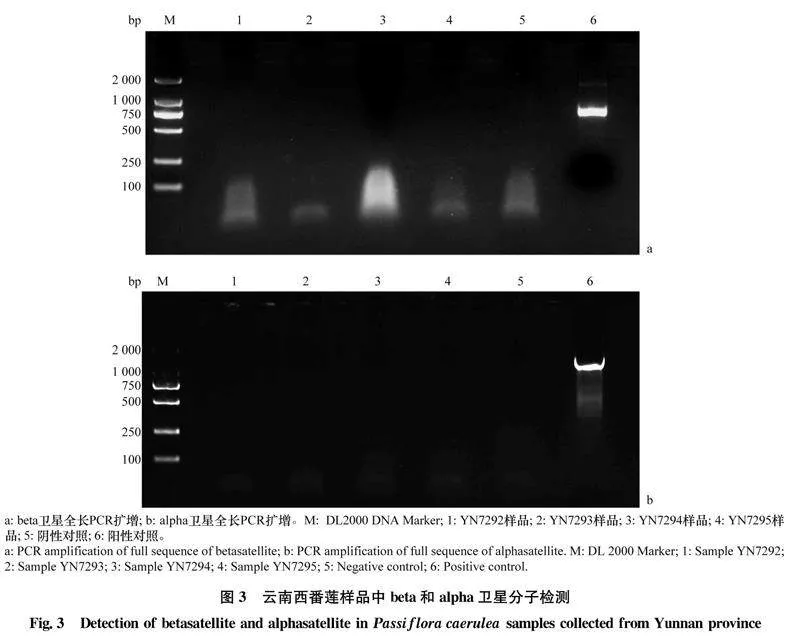
2.2西番莲病样中beta和alpha卫星分子检测应用菜豆金色花叶病毒属病毒伴随性卫星分子beta和alpha特异性引物β01/β02和UNA101/UNA102(表1)对西番莲样品YN7292、YN7293、YN7294和YN7295进行PCR检测,结果发现,4个西番莲样品中均未检测到beta和alpha卫星分子(图3)。2.3西番莲病样中菜豆金色花叶病毒属病毒全基因组结构分析用一品红曲叶病毒全长特异引物EuLCuV-F/-R对YN7292、YN7293、YN7294和YN7295进行扩增,4个样品均扩增到片段长度约为2 800 bp的目标条带(图4a)。通过回收、克隆及测序,共获得4条序列,分别命名为YN7292-6、YN7293-12、YN7294-16以及YN7295-25。应用广东番木瓜曲叶病毒全长特异引物PaLCuGdV-F/-R对4个西番莲样品进行扩增,结果仅在YN7293中扩增到片段长度约为2 800 bp的目标条带(图4b)。通过回收、克隆及测序获得1条序列,命名为YN7293-4。
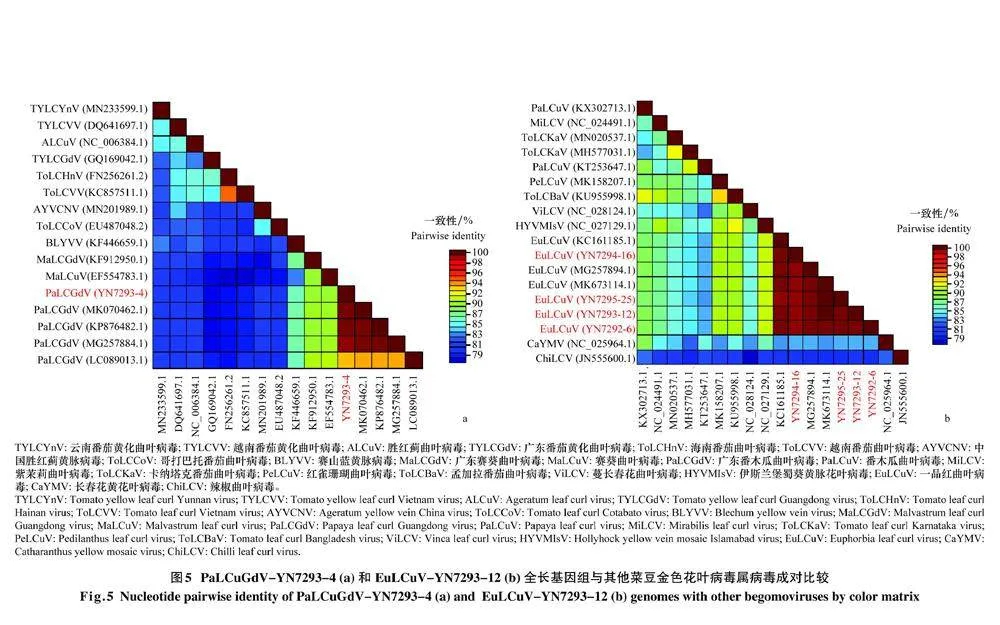
通过SDT比对发现,YN7292-6、YN7293-12以及YN7295-25与江西西番莲EuLCuV分离物GNPF1(GenBank登录号:MK673114.1)核苷酸序列相似性最高,在99.05%~99.78%之间;YN7294-16与中国台湾西番莲EuLCuV分离物PF1(GenBank登录号:KC161185.1)的核苷酸序列相似性最高,为99.42%;而YN7293-4则与韩国西番莲PaLCuGdV分离物JN17-PF10(GenBank登录号:MK070462.1)的核苷酸序列相似性最高,为99.70%(图5)。根据ICTV对菜豆金色花叶病毒属病毒的分类标准,病毒全基因组DNA-A核苷酸序列相似性大于91%则为同一种病毒,小于91%则为不同种病毒[3],因此,西番莲中获得的YN7292-6、YN7293-12、YN7294-16以及YN7295-25 4条序列均为EuLCuV的不同分离物,分别命名为EuLCuV-YN7292-6、EuLCuV-YN7293-12、EuLCuV-YN7294-16、EuLCuV-YN7295-25;而YN7293-4则为PaLCuGdV的一个分离物,命名为PaLCuGdV-YN7293-4(图5)。
本研究所获得的菜豆金色花叶病毒属病毒不同分离物全基因组序列均具有典型的单组分菜豆金色花叶病毒属病毒基因组结构,全基因组共编码7个开放阅读框,正义链编码V1和V2蛋白,负义链编码C1、C2、C3、C4和C5蛋白,以及一个包含菜豆金色花叶病毒属病毒复制和转录必需的各种元件的基因间隔区(intergenic region, IR)。将所获序列在NCBI上进行登录注释,获得的登录号见表2。
2.4西番莲病样中菜豆金色花叶病毒属病毒全基因组相似性比较在西番莲样品YN7292、YN7294和YN7295中仅检测出一品红曲叶病毒,而在YN7293样品中同时检测出一品红曲叶病毒和广东番木瓜曲叶病毒,为2种病毒复合侵染,故选取YN7293样品的PaLCuGdV-YN7293-4和EuLCuV-YN7293-12序列进行相似性分析。
PaLCuGdV-YN7293-4分离物编码区和非编码区与PaLCuGdV不同分离物相似性较高,其中全长基因组核苷酸序列与韩国西番莲PaLCuGdV分离物JN17-PF10(GenBank登录号:MK070462.1)的相似性最高(99.7%),IR区和V1与中国台湾西番莲PaLCuGdV分离物BXG4(GenBank登录号:KP876482.1)的相似性最高(99.4%和99.5%),V2、C1、C2、C3和C4与韩国西番莲PaLCuGdV分离物JN17-PF10(GenBank登录号:MK070462.1)的相似性最高(表3)。
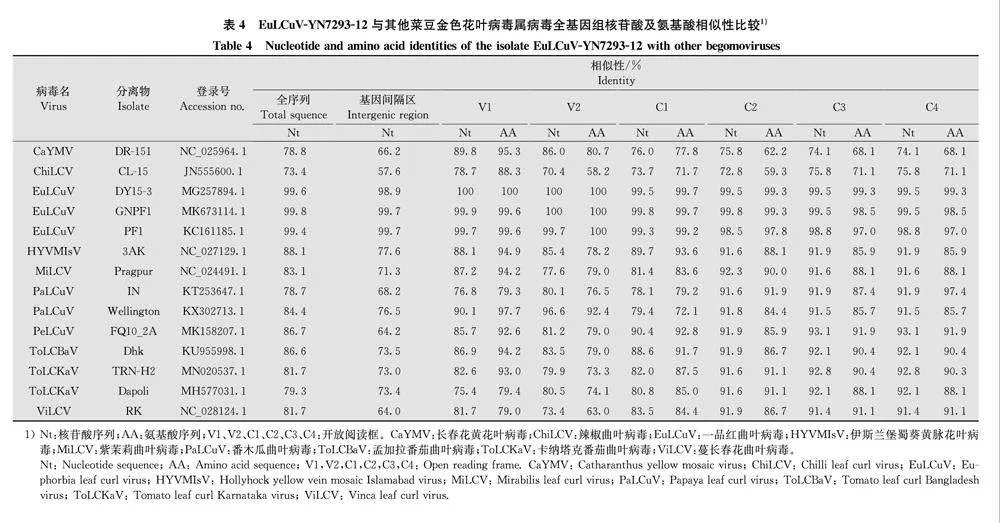
EuLCuV-YN7293-12分离物编码区和非编码区与EuLCuV不同分离物相似性较高,其中全长基因组核苷酸序列与江西EuLCuV分离物GNPF1(GenBank登录号:MK673114.1)的相似性最高(99.8%);IR区与江西EuLCuV分离物GNPF1和中国台湾EuLCuV分离物PF1(GenBank登录号:KC161185.1)的相似性均最高(均为99.7%);V1和V2与韩国西番莲EuLCuV分离物DY15-3(GenBank登录号:MG257894.1)的相似性最高(均为100%);C1、C2、C3和C4均分别与PF1和DY15-3的核苷酸相似性最高(99.5%以上)(表4)。
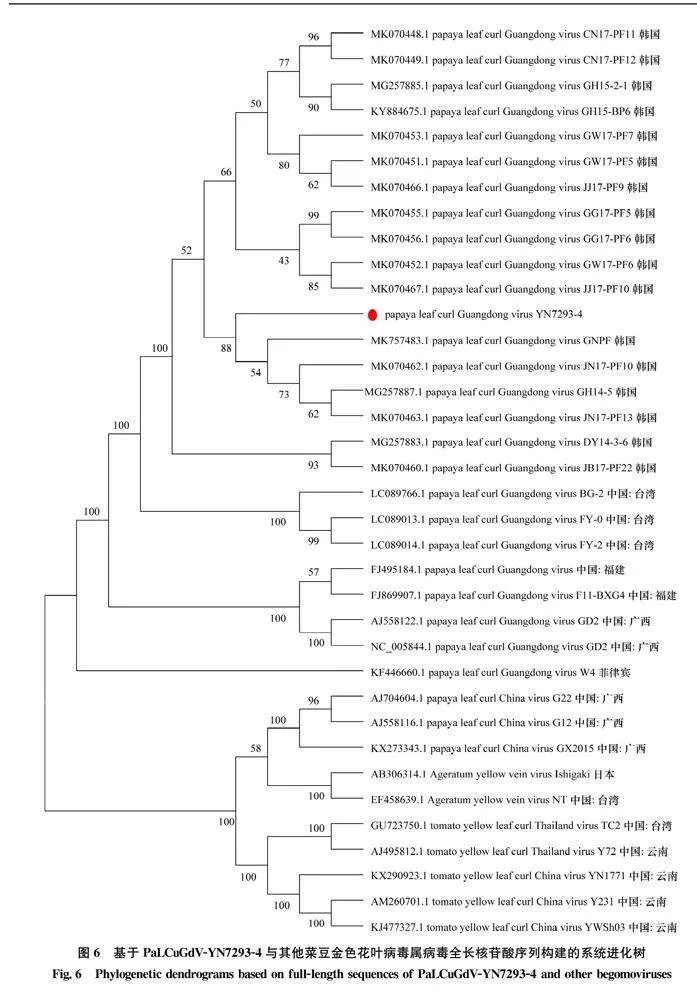
2.5西番莲病样中菜豆金色花叶病毒属病毒系统进化分析将PaLCuGdV-YN7293-4分离物与其他菜豆金色花叶病毒属病毒不同分离物进行系统进化分析发现,PaLCuGdV-YN7293-4与PaLCuGdV的不同分离物聚为一个大的分支,其中又与韩国PaLCuGdV分离物形成一个相对独立的进化分支,表明云南西番莲病样中分离到的PaLCuGdV分离物与韩国分离物亲缘关系较近,而与中国或其他国家的PaLCuGdV不同分离物亲缘关系较远(图6),同时也表明PaLCuGdV的进化与地理来源具有明显相关关系。
将EuLCuV-YN7293-12分离物与其他菜豆金色花叶病毒属病毒进行系统进化分析发现,EuLCuV-YN7293-12与EuLCuV的不同分离物聚为一个大的分支,与江西西番莲分离物GNPF1(GenBank登录号:MK673114.1)和韩国不同分离物聚于一个小的分支,表明EuLCuV-YN7293-12与中国分离物和部分韩国分离物亲缘关系较近,同时也表明EuLCuV的进化与地理来源无明显关系(图7)。
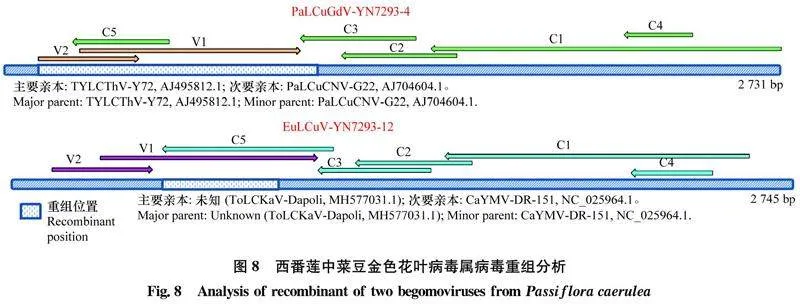
2.6西番莲中菜豆金色花叶病毒属病毒重组分析
用RDP软件中的9种重组检测方法对PaLCuGdV-YN7293-4和EuLCuV-YN7293-12两个分离物进行重组分析表明,2个分离物均存在重组事件。
EuLCuV-YN7293-12发生重组的位置位在523-946 nt之间,位于病毒基因组的V1区;其主要亲本未知,但极有可能为ToLCKaV-Dapoli(GenBank登录号:MH577031.1),次要亲本为CaYMV-DR-151(GenBank登录号:NC_025964.1)(RDP值:5.862×10-8,GENECONV值:1.647×10-9,BootScan值:7.422×10-8,MaxChi值:2.547×10-5,Chimaera值:1.994×10-6,SiScan值:7.607×10-20,3Seq值:4.035×10-2)(图8)。
PaLCuGdV-YN7293-4发生重组的位置位在90-1 116 nt之间,位于病毒基因组的V1和V2区;其主要亲本为TYLCThV-Y72(GenBank登录号:AJ495812),次要亲本为PaLCuCNV-G22(GenBank登录号:AJ704604)(RDP值:9.559×10-3,BootScan值:3.213×10-4,MaxChi值:2.171×10-5,Chimaera值:1.556×10-5,SiScan值:1.309×10-12,3Seq值:6.264×10-3)(图8)。
3结论与讨论
近年来,随着国内西番莲种植规模和范围的不断扩大,西番莲病毒病在全国呈现不断蔓延趋势,广西、广东、贵州、福建、台湾等西番莲种植区均有西番莲病毒病的报道[1819, 2728]。本研究对采集自云南省德宏州的4个西番莲样品进行病毒种类鉴定。通过PCR扩增、克隆及测序获得了5条菜豆金色花叶病毒属病毒全长序列,通过序列分析后发现,侵染云南省德宏州西番莲的菜豆金色花叶病毒属病毒分别为图6基于PaLCuGdV-YN7293-4与其他菜豆金色花叶病毒属病毒全长核苷酸序列构建的系统进化树
广东番木瓜曲叶病毒和一品红曲叶病毒,其中西番莲样品YN7293为2种病毒复合侵染,并进一步分析了2种病毒全基因组结构特征和进化重组关系。
一品红曲叶病毒和广东番木瓜曲叶病毒均属于菜豆金色花叶病毒属病毒,其中一品红曲叶病毒于2004年首次在广西报道[29],之后我国台湾和山东也相继在西番莲和一品红上发现一品红曲叶病毒[18,30],该病毒除在我国发生外,目前仅在韩国有报道[31]。一品红曲叶病毒可以侵染的寄主植物主要有西番莲(GenBank登录号:KT259282.1)、一品红Euphorbia pulcherrima[30]、胜红蓟Ageratum conyzoides(GenBank登录号:AJ811911.1)等。广东番木瓜曲叶病毒仅在中国的鸡蛋果Passiflora edulis[18]、韩国的灯笼椒[32]和菲律宾的赛山蓝Ruellia blechum(GenBank登录号:KF446660.1)有报道,在GenBank上有记录的寄主还包括番木瓜(NC_005844.1)、洋桔梗Eustoma grandiflorum(LC089766.1)、西番莲(KP876482.1)、烟草Nicotiana tabacum(FJ869907.1)和一品红(FJ495184.1)。与番茄黄化曲叶病毒(tomato yellow leaf curl virus, TYLCV)相比,一品红曲叶病毒和广东番木瓜曲叶病毒报道的寄主范围并不广泛,下一步可对这2种病毒的寄主范围进行鉴定。
本研究在云南省德宏州芒市的西番莲样品中检测到EuLCuV和PaLCuGdV,获得的PaLCuGdV分离物与韩国的不同分离物相似性较高,系统进化关系也较近;而EuLCuV的进化关系较为复杂,没有明显的地理分布特征,但云南西番莲EuLCuV分离物除了与我国EuLCuV广州分离物聚于一个小的分支,还与很多韩国EuLCuV分离物聚于一个大的分支,亲缘关系也较近,因此云南西番莲病样中2种病毒的分离物均有可能来源于韩国。
重组是双生病毒科病毒进化的主要动力,重组后的病毒不但能够扩大其寄主范围还能使寄主症状加重,从而造成严重的经济损失[3336]。如印度木薯曲叶病是由斯里兰卡木薯花叶病毒(Sri Lankan cassava mosaic virus, SLCMV)和印度木薯花叶病毒(Indian cassava mosaic virus, ICMV)为主的菜豆金色花叶病毒属不同病毒引起的病害,而不同病毒的复合侵染使当地不同种病毒之间出现重组并产生了致病性更强的毒株,从而对当地木薯造成更为严重的危害[36];而在西班牙,重组病毒马拉加番茄黄化曲叶病毒(tomato yellow leaf curl Málaga virus)不仅在茄科植物番茄中检测出,还能在豆科植物菜豆中检测出,甚至检出率更高,说明该病毒重组后寄主范围在扩大[37]。本研究对4个西番莲样品进行病毒全序列扩增时发现,YN7293样品是由PaLCuGdV和EuLCuV 2种病毒复合侵染的,并且通过重组分析发现,2个分离物均不同程度存在重组事件。我国广西、福建以及台湾等地也发现了西番莲样品中PaLCuGdV和EuLCuV复合侵染,因此需警惕两种病毒在西番莲中复合侵染后产生病毒基因组的交换等变异导致产生致病性更强的病毒种类或株系,从而造成更为严重的危害。针对西番莲菜豆金色花叶病毒属病害发生危害范围及程度不断增加,应加强西番莲不同种植区种苗繁殖、调运、媒介昆虫的防治及种植过程中菜豆金色花叶病毒属病毒的监测,为科学防控西番莲病毒病害提供理论基础。
参考文献
[1]SALEHZADEH M, AFSHARIFAR A, DEHGHANPOUR F S, et al. The first report of the chilli leaf curl virus and its beta satellite from bell peppers and tomatoes from the central provinces of Iran [J]. Iranian Journal of Plant Pathology, 2022, 57(4): 337341.
[2]杜江, 张丽, 崔丽艳, 等. 侵染河北土贝母的中国南瓜曲叶病毒分子特征分析[J]. 植物病理学报, 2023, 53(3): 539545.
[3]FIALLO-OLIVE E, LETT J M, MARITIN D P, et al. ICTV virus taxonomy profile: Geminiviridae 2021 [J/OL]. Journal of General Virology, 2021, 102(12): 001696. DOI: 10.1099/jgv.0.001696.
[4]VARSANI A, NAVAS-CASTILLO J, MORIONES E, et al. Establishment of three new genera in the family Geminiviridae: Becurtovirus, Eragrovirus and Turncurtovirus [J]. Archives of Virology, 2014, 159(8): 21932203.
[5]ZERBINI F M, BRIDDON R W, IDRIS A, et al. ICTV virus taxonomy profile: Geminiviridae [J]. Journal of General Virology, 2017, 98(2): 131133.
[6]ROUMAGNAC P, LETT J M, FIALLO-OLIVE E, et al. Establishment of five new genera in the family Geminiviridae: Citlodavirus, Maldovirus, Mulcrilevirus, Opunvirus, and Topilevirus [J]. Archives of virology, 2022, 167(2):695710.
[7]ROJAS M R, HAGEN C, LUCAS W J, et al. Exploiting chinks in the plant’s armor: evolution and emergence of geminiviruses [J]. Annual Review of Phytopathology, 2005, 43(1): 361394.
[8]HARRISON B D, BARKER H, BOOK K R, et al. Plant viruses with circular single-stranded DNA [J]. Nature, 1977, 270(5639): 760762.
[9]XAVIER C A D, GODINHO M T, MAR T B, et al. Evolutionary dynamics of bipartite begomoviruses revealed by complete genome analysis [J]. Molecular Ecology, 2021, 30(15): 37473767.
[10]DING M, LUO Y Q, DONG J H, et al. First report of tomato yellow leaf curl China virus with DNA β infecting Datura stramonium in China [J]. Australasian Plant Disease Notes, 2007, 2(1): 63.
[11]吴均秀. 浅谈西番莲及其发展前景[J]. 广西热作科技, 1994, 50(1): 4346.
[12]王秀荣, 段安安, 许玉兰. 国内西番莲引种栽培现状及改良思路[J]. 西南林学院学报, 2003, 23(2): 8891.
[13]李莉萍. 西番莲综合开发利用研究进展[J].安徽农业科学, 2012, 40(28): 1384013843.
[14]BROWN J K, BIRD J, FLETCHER D C. First report of Passiflora leaf mottle disease caused by a whitefly-transmitted geminivirus in Puerto Rico [J]. Plant Disease, 1993, 77(12): 1264.
[15]BROWN J K, BIRD J. Introduction of an exotic whitefly (Bemisia) vector facilitates secondary spread of Jatropha mosaic virus, a geminivirus previously vectored exclusively by the Jatropha biotype [M]. United Kingdom: Intercept Publications, Wimborne, 1996: 351353.
[16]NOVAES Q S, FREITAS-ASTUA J V, YUKI A, et al. Partial characterization of a bipartite begomovirus infecting yellow passion flower in Brazil [J]. Plant Pathology, 2003, 52(5): 648654.
[17]VACA-VACA J C, CARRASCO-LOZANO E C, LPEZ-LPEZ K. Molecular identification of a new begomovirus infecting yellow passion fruit (Passiflora caerulea) in Colombia [J]. Archives of Virology, 2017, 162: 573576.
[18]CHENG Y H, DENG T C, CHEN C C, et al. First report of Euphorbia leaf curl virus and papaya leaf curl Guangdong virus on passion fruit in Taiwan [J]. Plant Disease, 2014, 98(12): 1746.
[19]谢丽雪, 张小艳, 郑姗, 等. 福建西番莲上广东番木瓜曲叶病毒的分子鉴定[J]. 东南园艺, 2019, 7(5): 1922.
[20]罗金水, 周知恩, 王隆燊, 等. 西番莲病毒病的发生与防控[J]. 东南园艺, 2019, 7(6): 3640.
[21]DOYLE J J,DOYLE J L. A rapid DNA isolation procedure for small quantities of fresh leaf tissue [J]. Phytochemistry Bulletin, 1987, 19: 1115.
[22]DENG D, MCGRATH P F, ROBINSON D J, et al. Detection and differentiation of whitefly-transmitted geminiviruses in plants and vector insects by the polymerase chain-reaction with degenerate primers [J]. Annals of Applied Biology, 1994, 125(2): 327336.
[23]BRIDDON R W, BULL S E, MANSOOR S, et al. Universal primers for the PCR-mediated amplification of DNA beta-a molecule associated with some monopartite begomoviruses [J]. Molecular Biotechnology, 2002, 20(3): 315318.
[24]BULL S E, BRIDDON R W, MARKHAM P G. Universal primers for the PCR-mediated amplification of DNA 1: a satellite-like molecule associated with begomovirus-DNA β complexes [J]. Molecular Biotechnology, 2003, 23(1): 8386.
[25]TAMURA K, STECHER G, PETERSON D, et al. MEGA6: Molecular evolutionary genetics analysis 3ab39e81c3d461a6f488b89815fb4980version 6.0 [J]. Molecular Biology and Evolution, 2013, 30(12): 27252729.
[26]MARTIN D, RYBICKI E. RDP: detection of recombination amongst aligned sequences [J]. Bioinformatics, 2000, 16(6): 562563.
[27]BROWN J K, ZERBINI F M, NAVAS-CASTILLO J, et al. Revision of Begomovirus taxonomy based on pairwise sequence comparisons [J]. Archives of Virology, 2015, 160(6): 15931619.
[28]严佳文, 袁启凤, 解璞, 等. 利用小RNA测序技术检测贵州西番莲病毒[J]. 热带作物学报, 2019, 40(8): 15771584.
[29]MA X Y, CAI J H, LI G X, et al. Molecular characterization of a distinct begomovirus infecting Euphorbia pulcherrima in China [J]. Journal of Phytopathology, 2004, 152(4): 215218.
[30]李刚, 赵黎明, 高颖, 等. 侵染一品红的一品红曲叶病毒山东分离物的全基因组克隆和序列分析[J]. 植物保护, 2014, 40(3): 133137.
[31]KIL E J, SEO H, BYUN H S, et al. First report of Euphorbia leaf curl virus in passion fruits in South Korea and its natural occurrence in papaya [J]. Plant Disease, 2016, 100(4): 865.
[32]KIM M, HONG S B, KIM J, et al. First report of papaya leaf curl Guangdong virus in bell pepper in Korea [J]. Plant Disease, 2018, 102(10): 2046.
[33]HANLEY-BOWDOIN L, SETTLAGE S B, OROZCO B M, et al. Geminiviruses: models for plant DNA replication, transcription, and cell cycle regulation [J]. Critical Reviews in Plant Sciences, 1999, 18(1): 71106.
[34]PADIDAM M, SAWYER S, FAUQUET C M. Possible emergence of new geminiviruses by frequent recombination [J]. Virology, 1999, 265(2): 218225.
[35]LEFEUVRE P, MORIONES E. Recombination as a motor of host switches and virus emergence: geminiviruses as case studies [J]. Current Opinion in Virology, 2015, 10: 1419.
[36]ROTHENSTEIN D, HAIBLE D, DASGUPTA I, et al. Biodiversity and recombination of cassava-infecting begomoviruses from southern India [J]. Archives of Virology, 2006, 151(1): 5569.
[37]GARCA-ANDRS S, ACCOTTO G P, NAVAS-CASTILLO J, et al. Founder effect, plant host, and recombination shape the emergent population of begomoviruses that cause the tomato yellow leaf curl disease in the Mediterranean basin [J]. Virology, 2007, 359(2): 302312.
(责任编辑:杨明丽)

