栎属麻栎亚组与冬青栎组的质体捕获历史研究




摘 要: 栎属(Quercus)麻栎亚组(subsect. Campylolepides)包括麻栎(Q. acutissima)、栓皮栎(Q. variabilis)及小叶栎(Q. chenii)3个物种,是栎属土耳其栎组(section Cerris)的东亚分支,前人已经对麻栎亚组或组内物种的物种形成和谱系地理做过深入研究,也发现了土耳其栎组(section Cerris)曾与冬青栎组(section Ilex)发生过古基因渐渗并导致质体捕获,但目前麻栎亚组与冬青栎组质体的具体进化历史仍不清楚。该研究对15个冬青栎组的样品进行了基因组浅层测序,并整合先前发表的麻栎亚组及其近缘类群共计325个重测序数据,其中麻栎亚组3物种19居群276个体,利用这些数据进行质体基因组组装和分析。结果表明:(1)麻栎亚组3个物种间存在共享单倍型,但整个麻栎亚组的质体单倍型基本构成一个单系分支,嵌套在华中至四川凉山州一带冬青栎组物种组成的分支中。(2)麻栎亚组物种中一个来自辽东半岛的孑遗麻栎单倍型与冬青栎组物种万山栎(Q. pseudosetulosa)聚为一个进化枝。(3)两次质体捕获事件均发生在中新世中期,在此之后麻栎亚组与冬青栎组未发生过质体捕获,推测麻栎亚组跟冬青栎组物种目前已经形成近乎完全的生殖隔离。该研究表明冬青栎组和麻栎亚组在质体基因组的演化历史中经历了古老的质体捕获事件,并最终逐步形成了相对独立的演化路径。
关键词: 栎属, 麻栎亚组, 土耳其栎组, 冬青栎组, 质体捕获, 质体基因组渐渗
中图分类号: Q943
文献标识码: A
文章编号: 1000-3142(2024)09-1721-11
Plastid capture history of subsect. Campylolepides
and section Ilex (Fagaceae: Quercus)
Abstract: Quercus subsect. Campylolepides contains three species: Q. acutissima, Q. variabilis and Q. chenii, which is the East Asian clade of Quercus section Cerris. Species formation and phylogeography of whole subsection or species within the subsection have been studied in detail. Ite1MpHJ7UsfGb+UNpNjWNohLGffYrwjAApELEIlCd8NQ= has been also found that section Cerris had an ancient gene introgression with section Ilex, which led to the plastid capture. However, the specific evolutionary history of plastids in subsect. Campylolepides and section Ilex remains unclear. Our study performed low-coverage whole genome sequencing on 15 samples from section Ilex and integrated previously published data from subsect. Campylolepides and its relatives, resulting in a total of 325 resequencing data, of which 276 individuals were from three subsect. Campylolepides species and 19 populations. We assembled 325 plastids to perform phylogeographic analysis. The results were as follows: (1) There were shared haplotypes among three species of subsect. Campylolepides, but the plastid haplotypes of the entire subsect. Campylolepides form a monophyletic branch nested within section Ilex species which are distributed from central China to Liangshan, Sichuan. (2) A relictual haplotype of subsect. Campylolepides from the Liaodong Peninsula clustered with Q. pseudosetulosa, a specie of the section Ilex. (3) Both plastid capture events occurred in the middle Miocene, after which no plastid capture occurred between subsect. Campylolepides and section Ilex. It is hypothesized that subsect. Campylolepides and section Ilex have formed almost complete reproductive isolation. The study demonstrates that section Ilex and subsect. Campylolepides have undergone ancient plastid capture events in their evolution history, ultimately lead to the gradual formation of relatively independent evolutionary paths.
Key words: Quercus, subsect. Campylolepides, section Cerris, section Ilex, plastid capture, plastid genome introgression
“核质不一致”现象在系统发育研究中经常遇到,即质体基因组与核基因组构建的系统发育树的拓扑结构不一致,通常由不完全谱系分选(Meleshko et al., 2021),杂交或基因渐渗造成(Stull et al., 2023)。质体捕获(plastid capture)是种间杂交导致的特殊现象。当不同物种杂交后,杂交种与一个亲本物种重复回交会导致质体基因组从一个物种融入到另一个物种中,最终导致细胞核与细胞质来自不同的物种(Rieseberg & Soltis, 1991;Kleinkopf et al., 2019)。遗传模型研究发现,如果细胞质替代在种子生产中提供优势,质体捕获便容易发生(Tsitrone et al., 2003)。大量的实例研究,如矾根属(Heuchera)(Liu et al., 2020)、柳属(Salix)(Percy et al., 2014)、杨属(Populus)(Liu et al., 2017)等证明质体捕获普遍发生于被子植物类群中。在部分类群中仅仅依靠质体基因去构建系统发育关系会导致物种关系误判(Rieseberg & Soltis, 1991);而在个别类群中,近缘物种的质体基因通常与地理位置相关而非物种亲缘关系,这一现象在栎属(Quercus)物种中极为普遍并有大量的相关研究(Simeone et al., 2016;Yang et al., 2016;Tekpinar et al., 2021;Yang et al., 2021)。
麻栎(Quercus acutissima)、栓皮栎(Q. variabilis)和小叶栎(Q. chenii)原产地为东亚,在国内栎属分类系统中,这3种栎属物种因形态相近被划分为一组(周浙昆,1992);在最近的栎属系统发育框架中(Hipp et al., 2020),3个物种共同组成了栎属土耳其栎组(Quercus section Cerris)的东亚分支——Camus(1936)定义的麻栎亚组(subsect. CG6lTlCyffq8boDR4PEE8Yg==ampylolepides)。麻栎和栓皮栎是东亚暖温带阔叶林重要成分之一(李垚等,2014);而小叶栎分布地仅限于华东地区(李垚等,2016)。麻栎亚组物种作为东亚暖温带阔叶林主要成分,国内外已有大量学者对其杂交渐渗、物种形成开展大量研究。麻栎亚组内物种杂交渐渗现象频繁,在麻栎和栓皮栎中相似生境的居群可能会共享适应性渐渗片段,而适应性渐渗位点倾向于分布在重组率受抑制的区域(Fu et al., 2022);麻栎亚组物种的编码区普遍存在大量正选择和负选择,而连锁选择进一步驱动遗传分化(Liang et al., 2022)。李垚(2019)对麻栎亚组物种的谱系地理、演化历史做了详尽研究,认为麻栎遗传结构呈现出以第二、第三级阶梯为界的东西分化,小叶栎的遗传结构则受限于华东地区的地形和气候,并且几个物种间广泛共享单倍型,而种间单倍型共享现象则来源于种间渐渗及祖先多态性共享。
在基于简化基因组构建的系统发育关系中,麻栎亚组物种与欧洲土耳其栎物种互为姐妹关系并组成一个单系分支(Hipp et al., 2020)。Zhou等(2022)对整个壳斗科进行了系统发育基因组学研究,发现壳斗科栎属不同组间以及与近缘属类群的质体基因组谱系可能存在大量质体捕获事件。本研究中来自湖北、广西、山东、湖南、江苏、云南的麻栎亚组物种并没有与欧洲的土耳其栎组物种聚为一支,而是与湖北地区的冬青栎组物种铁橡栎(Q. cocciferoides)及匙叶栎(Q. dolicholepis)聚为了姐妹支,因此土耳其栎组东亚物种被确定曾与冬青栎组物种发生质体捕获。Simeone等(2018)对13种土耳其栎组物种谱系地理关系的研究表明,土耳其栎组物种可能在渐新世早期于东北亚起源后扩散至西亚地区,并与当地冬青栎组物种相互接触后产生种间基因交流。
综上所述,前人已经对麻栎亚组或者组内单一物种的谱系地理,麻栎亚组物种间基因组的分化规律以及物种形成机制方面做过大量且深入的研究,但目前对东亚地区麻栎亚组与冬青栎组的质体捕获历史尚不清楚。借助先前研究积累的大量基因组数据,我们将麻栎亚组纳入包含其近缘类群的栎属质体基因组系统发育框架中以探究其与近缘类群间的质体谱系共享及演化历史。本研究重点对位于麻栎亚组分布区内的冬青栎组样品进行了基因组浅层测序,并综合Zhou等(2022)、Fu等(2022)和Liang等(2022)研究的栎属重测序数据进行了质体全基因组组装和注释。对麻栎亚组及其近缘类群的质体基因组进行系统进化分析,以期解决或探讨以下问题:(1)国内麻栎亚组物种跟冬青栎组物种发生过几次质体捕获,在较近的历史时期麻栎亚组是否与栎属其他类群尤其是冬青栎组物种共享近缘的质体基因型;(2)国内麻栎亚组物种与冬青栎组物种进行古基因交流的时间和可能过程。
1 材料与方法
1.1 样品信息
本研究全新测序了15个冬青栎组(section Ilex)的样品(表1),样品选取原则为尽量选择位于麻栎亚组分布区内的样品;并与前人研究中的三部分重测序数据整合,总计325条序列。三部分重测序数据包括Zhou等(2022)研究中的轮叶三棱栎属(Trigonobalanus)2条、栗属(Castanea)2条、锥属(Castanopsis)2条、柯属(Lithocarpus)2条、栎属土耳其栎组(section Cerris)5条、栎属冬青栎组7条、栎属青冈组(section Cyclobalanopsis)8条、水青冈属(Fagus)1条,共计29条序列。整合Fu等(2022)及Liang等(2022)研究所用的样品,选取Fu等(2022)研究中的栎亚属(subgenus Quercus)5条序列及7个麻栎样品居群(BWL、GSM、LCX、TCX、TMS、ZHX、ZJS)、9个麻栎、栓皮栎同域分布居群(DWX、JJL、KUM、KYS、LFS、LSK、MPX、PWX、TBS);选取Liang等(2022)研究中2个栓皮栎、小叶栎同域分布居群(HYX、JJX)及1个麻栎、栓皮栎、小叶栎同域居群(LYG);共计19个居群,276个个体(表2)。
1.2 质体基因组组装
本研究测序的样品为硅胶干燥的叶片,通过CATB法提取总DNA,使用Covaris超声波破碎仪打断,成功构建350 bp小片段文库后,使用Illumina高通量测序平台进行测序。样品测序数据量为2 G,整个测序过程由北京诺禾致源股份有限公司完成。本研究测序样品与其余三部分已经发表的重测序数据组成本研究全部样品数据集,重测序数据使用SRA-TOOL下载,Bioproject号为PRJNA773751、PRJNA763710、PRJNA769460。用GetOrganelle v1.6.2(Jin et al., 2020)组装,K为105、121,R为15。在NCBI中BLAST获得相似度最高的序列为小叶栎叶绿体基因组(NC_039428),并以该序列为参考序列,在PGA中对本研究所有的质体基因组进行注释(Qu et al., 2019)。在Geneious v9.0.2中进行检查和补充注释,共获得了325条麻栎、栓皮栎及其近缘类群质体全基因组序列,蛋白编码序列(coding sequence,CDS)由Geneious中Export Annotations工具提取并串联后用于后续分析。
1.3 数据分析
1.3.1 单倍型共享 在DnaSP v6.0中确定276个麻栎亚组样品的单倍型数目和分布(Rozas et al., 2017),使用RAxML v8.0构建单倍型的最大似然树(maximum likelihood,ML)(Stamatakis,2014),并在ArcMap v10.4.1中绘制单倍型地理分布图。
1.3.2 系统发育关系及分化时间 对获得的276条麻栎亚组物种质体序列使用MAFFT v7.490比对后(Katoh & Standley, 2013),在DnaSP v6.0中进行滑窗分析(Rozas et al., 2017),窗口大小为600,步长为200。构建系统发育树时,对276条麻栎亚组序列进行缩减,每个单倍型中选取不同物种和来自不同居群的序列, 共获得75条麻栎亚组质体基因组序列,与其近缘类群49条共同组成系统发育框架。编码序列进行提取,将76个共有蛋白编码基因进行串联后,首先使用MAFFT v7.490软件对序列进行比对(Katoh & Standley, 2013),其次使用trimAl v1.4(Capella-Gutidrrez et al., 2009)对序列进行修剪,然后使用RAxML v8.0构建系统发育树,最后在ITOL中修饰并呈现(Letunic' & Bork, 2021)。
利用BEAST 2软件包对麻栎亚组物种及其近缘类群的分化时间进行推断(Bouckaert et al., 2014),使用jModelTest 2计算碱基替换模型(Darriba et al., 2012),采用Yule Model模型,宽松分子钟,进化速率为1.0。采用两个化石校准点对分化时间进行校正,第一个化石点为壳斗科最早的大化石记录(Grímsson et al., 2016),用于校准外类群水青冈属与内类群的分化时间;第二个化石点为锥属最早化石记录(Wilf et al., 2019),用于校准锥属与栗属的分化时间。独立运行3次MCMC模拟,每次代数为100 000 000,每1 000代取样1次,在TRACER v1.7.1中查看其ESS是否大于200(Rambaut et al., 2018),如果ESS>200,则说明结果收敛。使用TREEANNOTATOR v2.6.0软件构建最大可信树,前30%作为burn-in舍弃。
2 结果与分析
2.1 麻栎亚组不同物种单倍型共享分析
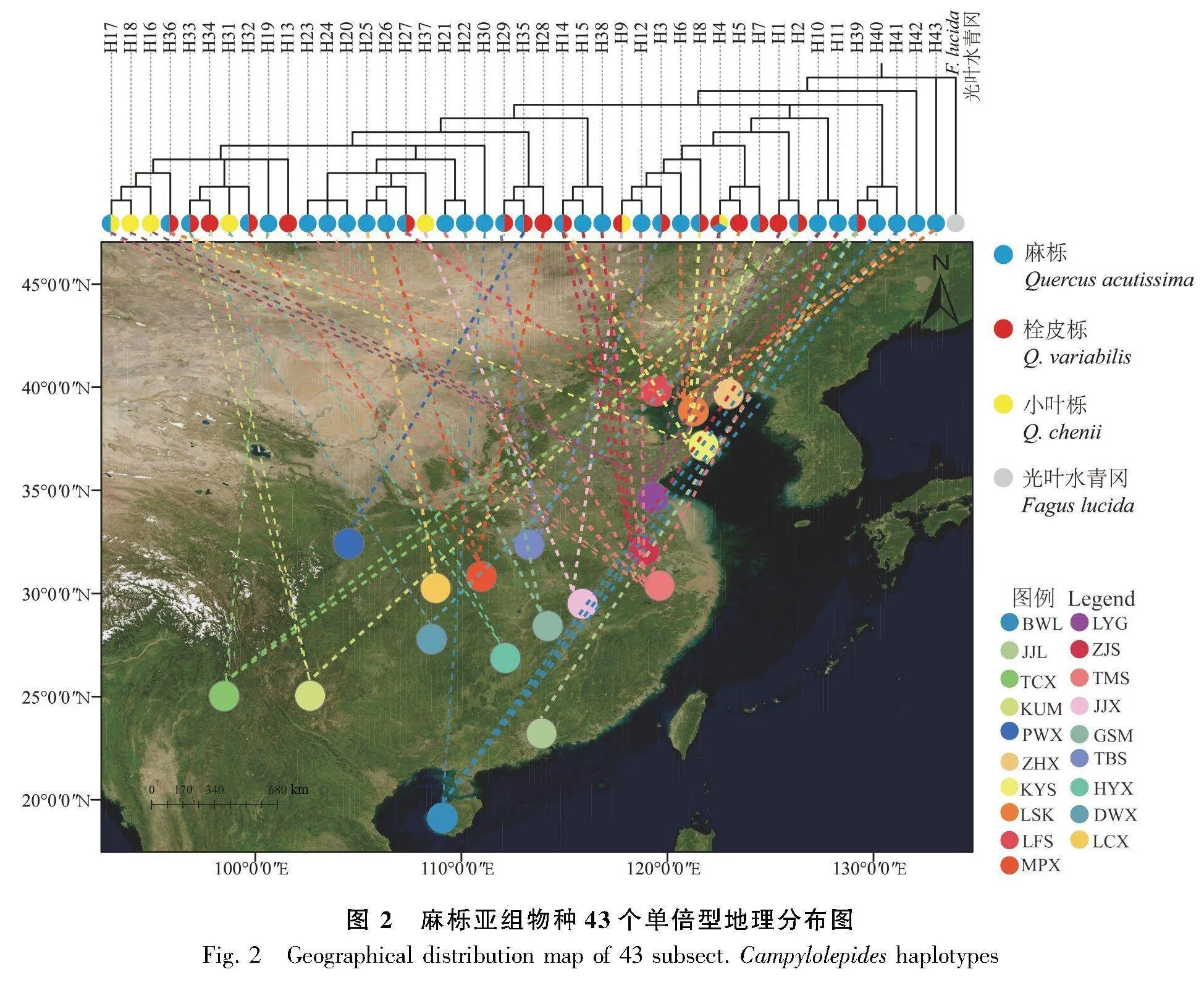
从国内麻栎亚组物种276个个体获得了77个共有蛋白编码基因,串联后长度为67 863 bp,共检测出43个单倍型。3个物种间存在单倍型共享现象(图1),12个单倍型为麻栎、栓皮栎共享,H14为麻栎和小叶栎共享,H19为栓皮栎和小叶栎共享,H4为3个物种共享单倍型。共检测到28个独享单倍型,其中19个为麻栎独享,5个为栓皮栎独享,4个为小叶栎独享。由图2可知,15个麻栎亚组物种共享单倍型中有7个分布在多个不同居群,主要涉及东北、华东、西南、华南地区11个居群的混合现象;而28个物种独享单倍型分布则更倾向于单个居群,仅4个物种独享单倍型出现在不同居群。
2.2 质体基因组特征及其系统发育关系
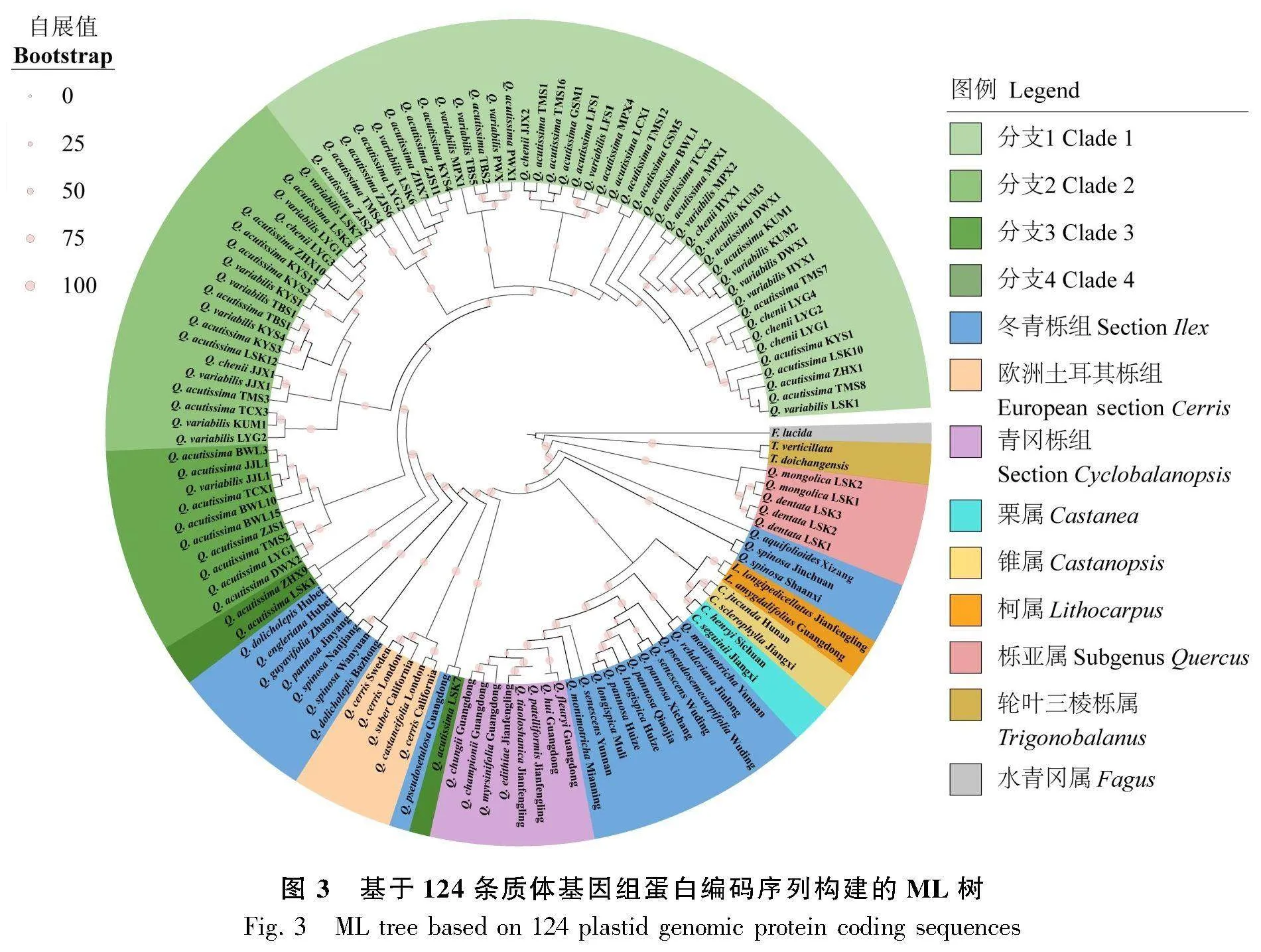
276条麻栎亚组质体全基因组总长范围为160 806~161 311 bp,G+C含量为37.8%,对276条序列进行共线性分析发现其无基因重排现象,对质体全基因组进行滑窗分析,高变异区段集中在rbcL-accD、ccsA-ndhD、ycf1、trnK(UUU)-rps16区段,对276条全基因组序列比对后识别到846个有效变异位点,其中309个位于蛋白编码序列。基于蛋白编码基因序列的系统发育树(图3)中,国内麻栎亚组物种基本全部聚在一支上,仅LSK居群的一个麻栎样品与冬青栎组物种万山栎(Q. pseudosetulosa)聚在一支。麻栎亚组物种先是与湖北地区的匙叶栎、巴东栎(Q. engleriana)组成一个分支,然后与四川西南部的帽斗栎(Q. guyavifolia)、黄背栎(Q. pannosa)及四川东北边缘的刺叶栎(Q. spinosa)、匙叶栎组成一个大分支,再后来这一大分支又与欧洲土耳其栎组物种组成的分支互为姐妹支,其中包含欧洲地区的土耳其栎(Q. cerris)、欧洲栓皮栎(Q. suber)、栗叶栎(Q. castaneifolia)。根据麻栎亚组物种主体分支的内部聚集情况,总体上可将其划分为4个小分支:分支1涵盖了18个居群的43个体;分支2涵盖了10个居群的19个体;分支3由7个居群的10个体组成;分支4由2个居群的2个麻栎个体组成。
2.3 种群分歧时间
在BEAST推断的系统发育树(图4)中,冬青栎组物种与土耳其栎组物种的系统发育关系与ML树并不一致,在BEAST构建的系统发育中,欧洲土耳其栎组物种与四川东北部的冬青栎组物种聚为一支。而麻栎亚组分支内部拓扑关系与ML法构建的系统发育树大体一致,麻栎亚组物种4个主要分支中,最近共同祖先时间分别为分支1(8.37 Ma)、分支2(8.98 Ma)、分支3(9.76 Ma);分支1、分支2、分支3形成的分支与分支4的最近共同祖先时间是12.70 Ma。整个分布区的麻栎亚组物种与湖北、四川西南部地区的冬青栎组物种最近共同祖先时间为18.41 Ma;欧洲土耳其栎组物种与四川东北边缘的冬青栎物种最近共同祖先时间为18.61 Ma;旅顺口地区的麻栎(单倍型H43)与广东地区的万山栎最近共同祖先时间为14.42 Ma。东亚土耳其栎组-冬青栎组分支与欧洲土耳其栎组物种最近共同祖先时间为19.98 Ma。麻栎亚组物种中,首先分化出来的是辽东半岛旅顺口(LSK)居群的H43单倍型,然后是旅顺口(LSK)与庄河(ZHX)居群共享单倍型H42(分支4),最后在相近时间里分支1、分支2、分支3相继分化出来。
3 讨论与结论
3.1 麻栎亚组3个物种的单倍型共享
对国内麻栎亚组3个物种276个个体的质体单倍型研究表明,麻栎亚组3个物种间单倍型存在共享。就数量性状而言,共享单倍型数量为15,出现频率较高,共计172个个体,占全部个体的62.32%;而独享单倍型数量为28,出现频率较低,104个个体仅占全部个体的37.68%。Li等(2022)的研究发现麻栎亚组3个物种存在广泛的单倍型共享,并推测3个麻栎亚组物种曾共享一个祖先质体基因库,在现代物种形成之前可能已经分化为不同谱系,麻栎亚组内单倍型广泛共享主要来源于过去发生和正在进行的渐渗/杂交以及祖先多态性的保留。本研究由于使用质体基因组数据,在单倍型解析上较之前的研究有了更高的分辨率,并且发现物种间共享单倍型大多数在同一地点或临近地点共享,这一现象表明物种间的质体共享主要是由近期同域杂交导致的质体捕获造成,因为谱系筛选是一个随机过程,不太可能形成如此显著的同域物种间质体共享现象。
值得一提的是,东北地区检测到了出现在不同进化支上多种不同类型的单倍型,表明该区域可能是麻栎亚组一个长期稳定的冰期避难所。前人对山杨(Hou et al., 2018)、蒙古栎(Zeng et al., 2015)、五味子(Ye et al., 2019)等大量谱系地理研究也表明,东北地区在末次冰期是长期稳定的森林植物避难所。辽东半岛以及渤海地区可以容许麻栎亚组物种能够在冰期分布区收缩和冰期后扩张期间稳定地在东北地区定殖。而华中、华东地区则是单倍型共享发生最频繁的区域,Li等(2021)的研究就表明70%的麻栎亚组杂交个体均来自此地。
3.2 麻栎亚组与东亚冬青叶栎组的质体捕获
核基因组数据构建的系统发育表明整个土耳其栎组构成一个单系类群(Hipp et al., 2020),东亚分支和欧洲分支的分歧时间约在早渐新世(30 Ma);而在质体基因组构建的系统发育树及分化时间中,麻栎亚组首先与东亚的冬青栎组物种组成东亚土耳其栎-冬青栎(Cerris-Ilex)分支,而且在东亚Cerris-IlLjFais5WhTde7e0QhsJpFtkXSOmZ3yXH71CYZ7dmGBU=ex分支内,麻栎亚组并没有与整个东亚的冬青栎组组成姐妹分支,而是仅与湖北的巴东栎和匙叶栎组成姐妹分支,其他多个早出的冬青栎组分支是由四川东部和凉山地区的冬青栎组物种构成。整个东亚Cerris-Ilex分支与Cerris欧洲分支开始分化的时间为中新世早期(19.98 Ma),与Zhou等(2022)研究中质体基因组推断的时间比较接近。相比核基因组推断的土耳其栎组东亚分支与欧洲分支的间断时间,质体基因组推断的时间晚了将近10 Ma,说明其与冬青栎组物种接触并产生基因交流的时间要晚于土耳其栎组东亚和欧洲分支地理分化的时间。土耳其栎组东亚分支与欧洲分支在地理分布上呈间断分布,与喜马拉雅隆升、古地中海退却(孙航,2002)造成的铁筷子属(Helleborus)(Sun et al., 2001)、绿绒蒿属(Meconopsis)(庄璇,1981)、马甲子属(Paliurus)(Chen et al., 2017)等类群的经典东亚-地中海间断分布模式不同,土耳其栎组的间断分布很可能是由亚洲北部温带落叶森林植被消失所造成(Denk et al., 2023)。由此,我们推测麻栎亚组的祖先物种很可能是由于北部温带落叶森林植被消失导致瓶颈效应,分布区被迫南迁且种群相对较小,与广泛分布在亚热带区域的冬青栎组物种接触并发生杂交,从而得以捕获冬青栎组的质体。
土耳其栎组与冬青栎组关系密切,曾发生过多次质体捕获事件,如土耳其栎组东亚分支与欧洲分支中均发现了与冬青栎组物种杂交的“痕迹”(Simeone et al., 2016)。东亚分支内,本研究在旅顺口(LSK)居群中发现了一个与冬青栎组物种万山栎聚为一支的单倍型(H43),这一单倍型表明土耳其栎组物种和冬青栎组物种可能发生过不止一次质体捕获。但是,由于万山栎是万山群岛的一个孑遗物种,在地理上跟东北地区的麻栎亚组物种没有直接接触,因此此次质体捕获很可能来自其他与万山栎共享相同质体类型的冬青栎组物种,如乌冈栎等;尽管目前东北地区并没有冬青栎组的分布,但地质历史上国内冬青栎组的历史地理分布可能更为靠北。
现存麻栎亚组的质体主要是与冬青栎组其中一次捕获的结果,这一捕获事件发生时间为早渐新世至中新世之间,即核基因推断的土耳其栎组东亚分支与欧洲分支间断时间之后,质体基因推断的麻栎亚组与冬青栎组质体单倍型分化的时间之前,现代麻栎亚组与冬青栎组发生质体捕获的地理位置很可能是我国的东北至华中地区一带。麻栎亚组在与冬青栎组物种发生杂交和质体捕获后,逐步与冬青栎组物种形成生殖隔离并扩散至东亚各地。麻栎亚组近期的种间渐渗主要发生在亚组内3个物种之间。麻栎亚组物种于中新世晚期分化为4个地理规律不强的分支。这一时期全球气候的持续变冷(Guo et al., 2008)及东亚季风气候加强(Sun & Wang, 2005)造成了南北植被频繁变迁,使得麻栎亚组不同谱系间多次接触,麻栎亚组的遗传谱系也逐渐失去地理规律。第四纪冰期使得该类群的分布区进一步片段化,如东北地区就存在独立的避难所,先前的研究也表明华中及华东地区的山地存在麻栎亚组3个物种共同的避难所(Li et al., 2021),而间冰期局部扩张现象及不同避难所间的迁徙事件加剧了居群间单倍型共享。
本研究表明,麻栎亚组3个物种间存在质体基因组共享。土耳其栎组与冬青栎组曾发生多次质体捕获事件,但麻栎亚组现今的质体主要来自其中一次捕获事件。麻栎亚组物种与冬青栎组物种接触并捕获其质体发生在麻栎亚组物种现代谱系形成之前,时间限于早渐新世与中新世中期之间。在中新世中期以后2个类群未再发生过质体置换,推测2个类群已经形成了极强的生殖隔离。
参考文献:
BOUCKAERT RR, HELED J, KHNERT D, et al., 2014. BEAST 2: A software platform for bayesian evolutionary analysis [J]. PLoS Comput Biol, 10(4): e1003537.
CAMUS A, 1936. Les chênes: Monographie du genre Quercus et monographie du genre Lithocarpus [M]. Paris: Encyclopédie conomique de Sylviculture.
CAPELLA-GUTIRREZ S, SILLA-MARTNEZ JM, GABALDN T, 2009. trimal: a tool for automated alignment strimming in large scale phylogenetic analyses [J]. Bioinformatics, 25(15): 1972-1973.
CHEN YS, MESEGUER AS, GODEFROID M, et al., 2017. Out-of-India dispersal of Paliurus (Rhamnaceae) indicated by combined molecular phylogenetic and fossil evidence [J]. Taxon, 66(1): 78-90.
DARRIBA D, TABOADA GL, DOALLO R, et al., 2012. jModelTest 2: more models, new heuristics and parallel computing [J]. Nat Methods, 9(8): 772.
DENK T, GRIMM GW, HIPP AL, et al., 2023. Niche evolution in a northern temperate tree lineage: biogeographical legacies in cork oaks (Quercus section Cerris) [J]. Ann Bot, 131(5): 769-787.
FU RR, ZHU Y, LIU Y, et al., 2022. Genome-wide analyses of introgression between two sympatric Asian oak species [J]. Nat Ecol Evol, 6(7): 924-935.
GRMSSON F, GRIMM GW, ZETTER R, et al., 2016. Cretaceous and paleogene Fagaceae from North America and Greenland: evidence for a Late Cretaceous split between Fagus and the remaining Fagaceae [J]. Acta Palaeobot, 56(2): 247-305.
GUO ZT, SUN BN, ZHANG ZS, et al., 2008. A major reorganization of Asian climate by the early Miocene [J]. Clim Past, 4(3): 153-174.
HIPP AL, MANOS PS, HAHN M, et al., 2020. Genomic landscape of the global oak phylogeny [J]. New Phytol, 226(4): 1198-1212.
HOU Z, WANG ZS, YE ZY, et al., 2018. Phylogeographic analyses of a widely distributed Populus davidiana: Further evidence for the existence of glacial refugia of cool-temperate deciduous trees in northern East Asia [J]. Ecol Evol, 8(24): 13014-13026.
JIN JJ, YU WB, YANG JB, et al., 2020. GetOrganelle: a fast and versatile toolkit for accurate de novo assembly of organelle genomes [J]. Genome Biol, 21(1): 241.
KATOH K, STANDLEY DM, 2013. MAFFT multiple sequence alignment software version 7: improvements in performance and usability [J]. Mol Biol Evol, 30(4): 772-780.
KLEINKOPF JA, ROBERTS WR, WAGNER WL, et al., 2019. Diversification of Hawaiian Cyrtandra (Gesneriaceae) under the influence of incomplete lineage sorting and hybridization [J]. J Syst Evol, 57(6): 561-578.
LETUNIC' I, BORK P, 2021. Interactive Tree Of Life (iTOL) v5: an online tool for phylogenetic tree display and annotation [J]. Nucl Acid Res, 49(W1): W293-W296.
LI Y, 2019. Genetic structure and evolutionary history of Chinese oak species in Quercus section Cerris [D]. Nanjing: Nanjing Forestry University: 183. [李垚, 2019. 中国栎属麻栎组树种的遗传结构与演化历史 [D]. 南京: 南京林业大学: 183.]
LI Y, WANG L, ZHANG XW, et al., 2022. Extensive sharing of chloroplast haplotypes among East Asian Cerris oaks: The imprints of shared ancestral polymorphism and introgression [J]. Ecol Evol, 12(8): e9142.
LI Y, ZHANG XW, FANG YM, 2014. Predicting the impact of global warming on the geographical distribution pattern of Quercus variabilis in China [J]. Chin J Appl Ecol, 25(12): 3381-3389. [李垚, 张兴旺, 方炎明, 2014. 气候变暖对中国栓皮栎地理分布格局影响的预测 [J]. 应用生态学报, 25(12): 3381-3389.]
LI Y, ZHANG XW, FANG YM, 2016. Responses of the distribution pattern of Quercus chenii to climate change following the Last Glacial Maximum [J]. Chin J Plant Ecol, 40(11): 1164-1178. [李垚, 张兴旺, 方炎明, 2016. 小叶栎分布格局对末次盛冰期以来气候变化的响应 [J]. 植物生态学报, 40(11): 1164-1178.]
LI Y, ZHANG XW, WANG L, et al., 2021. Influence of pliocene and pleistocene climates on hybridization patterns between two closely related oak species in China [J]. Ann Bot, 129(2): 231-245.
LIANG YP, SHI Y, YUAN S, et al., 2022. Linked selection shapes the landscape of genomic variation in three oak species [J]. New Phytol, 233(1): 555-568.
LIU L, DU YX, FOLK RA, et al., 2020. Plastome evolution in Saxifragaceae and multiple plastid capture events involving Heuchera and Tiarella [J]. Front Plant Sci, 11: 361.
LIU X, WANG Z, SHAO W, et al., 2017. Phylogenetic and taxonomic status analyses of the abaso section from multiple nuclear genes and plastid fragments reveal new insights into the North Americaorigin of Populus (Salicaceae) [J]. Front Plant Sci, 7: 2022.
MELESHKO O, MARTIN MD, KORNELIUSSEN TS, et al., 2021. Extensive genome-wide phylogenetic discordance is due to incomplete lineage sorting and not ongoing introgression in a rapidly radiated bryophyte genus [J]. Mol Biol Evol, 38(7): 2750-2766.
PERCY DM, ARGUS GW, CRONK QCB, et al., 2014. Understanding the spectacular failure of DNA barcoding in willows (Salix): Does this result from a trans-specific selective sweep? [J]. Mol Ecol, 23(19): 4737-4756.
QU XJ, MOORE MJ, LI DZ, et al., 2019. PGA: a software package for rapid, accurate, and flexible batch annotation of plastomes [J]. Plant Methods,15: 50.
RAMBAUT A, DRUMMOND AJ, XIE D, et al., 2018. Posterior summarization in Bayesian phylogenetics using tracer 1.7 [J]. Syst Biol, 67(5): 901-904.
RIESEBERG LH, SOLTIS DE, 1991. Phylogenetic consequences of cytoplasmic gene flow in plants [J]. Am J Bot, 5(1): 65-84.
ROZAS J, FERRER-MATA A, SNCHEZ-DELBARRIO JC, et al., 2017. DnaSP 6: DNA sequence polymorphism analysis of large data sets [J]. Mol Biol Evol, 34(12): 3299-3302.
SIMEONE MC, CARDONI S, PIREDDA R, et al., 2018. Comparative systematics and phylogeography of Quercus section Cerris in western Eurasia: inferences from plastid and nuclear DNA variation [J]. PeerJ, 6: e5793.
SIMEONE MC, GRIMM GW, PAPINI A, et al., 2016. Plastome data reveal multiple geographic origins of Quercus Group Ilex [J]. PeerJ, 4: e1897.
STAMATAKIS A, 2014. RAxML version 8: a tool for phylogenetic analysis and post-analysis of large phylogenies [J]. Bioinformatics, 30(9): 1312-1313.
STULL GW, PHAM KK, SOLTIS PS, et al., 2023. Deep reticulation: the long legacy of hybridization in vascular plant evolution [J]. Plant J, 114(4): 743-766.
SUN H, 2002. Tethys tetreat and Himalayas-Hengduanshan Mountains uplift and their significance on the origin and development of the Sino-Himalayan elements and alpine flora [J]. Acta Bot Yunnan, 24(3): 273-288. [孙航, 2002. 古地中海退却与喜马拉雅-横断山的隆起在中国喜马拉雅成分及高山植物区系的形成与发展上的意义 [J]. 云南植物研究, 24(3): 273-288.]
SUN H, MCLEWIN W, FAY MF, 2001. Molecular phylogeny of Helleborus (Ranunculaceae), with an emphasis on the East Asian-Mediterranean disjunction [J]. Taxon, 50(4): 1001-1018.
SUN XJ, WANG PX, 2005. How old is the Asian monsoon system?— Palaeobotanical records from China [J]. Palaeogeogr Palaeoclimatol Palaeoecol, 222(3/4): 181-222.
TEKPINAR AD, AKTA瘙塁C, KANSU , et al., 2021. Phylogeography and phylogeny of genus Quercus L. (Fagaceae) in Turkey implied by variations of trnT(UGU)-L(UAA)-F (GAA) chloroplast DNA region [J]. Tree Genet Genom, 17: 1-18.
TSITRONE A, KIRKPATRICK M, LEVIN DA, 2003. A model for chloroplast capture [J]. Evolution, 57(8): 1776-1782.
WILF P, NIXON KC, GANDOLFO MA, et al., 2019. Eocene Fagaceae from Patagonia and Gondwanan legacy in Asian rainforests [J]. Science, 364(6444): eaaw5139.
YANG J, DI XY, MENG X, et al., 2016. Phylogeography and evolution of two closely related oak species (Quercus) from north and northeast China [J]. Tree Genet Genom, 12(89): 1-14.
YANG YY, QU XJ, ZHANG R, et al., 2021. Plastid phylogenomic analyses of Fagales reveal signatures of conflict and ancient chloroplast capture [J]. Mol Phylogenet Evol, 163: 107232.
YE JW, ZHANG ZK, WANG HF, et al., 2019. Phylogeography of Schisandra chinensis (Magnoliaceae) reveal multiple refugia with ample gene flow in Northeast China [J]. Front Plant Sci, 10: 199.
ZENG YF, WANG WT, LIAO WJ, et al., 2015. Multiple glacial refugia for cool-temperate deciduous trees in northern East Asia: the Mongolian oak as a case study [J]. Mol Ecol, 24(22): 5676-5691.
ZHOU BF, YUAN S, CROWL AA, et al., 2022. Phylogenomic analyses highlight innovation and introgression in the continental radiations of Fagaceae across the Northern Hemisphere [J]. Nat Commun, 13(1): 1320.
ZHOU ZK, 1992. Origion, phylogeny and dispersal of Quercus from China [J]. Acta Bot Yunnan, 14(3): 227-236. [周浙昆, 1992. 中国栎属的起源演化及其扩散 [J]. 云南植物研究, 14(3): 227-236.]
ZHUANG X, 1981. The systematic evolution and the geographical distribution of Meconopsis Vig [J]. Acta Bot Yunnan, 3(2): 139-146. [庄璇, 1981. 绿绒蒿属的系统演化及地理分布 [J]. 云南植物研究, 3(2): 139-146.]

