红松天然种群遗传多样性分析及核心种质构建

摘要:【目的】红松(Pinus koraiensis)是分布在中国东北地区的珍贵树种,由于近百年的人类干扰,其自然种群分布数量及范围逐渐减少。探究红松的遗传多样性和构建天然红松的核心种质,可为有效保存、管理和利用红松种质资源提供科学依据。【方法】以东北地区黑龙江省鹤北、五营、小北湖和鸡西,以及吉林省露水河5个保存现状良好的红松种群为研究对象,采用表型数据和分子标记相结合的方法,进行其核心种质构建。【结果】分子和表型方差分析结果均表明:红松天然种群的遗传变异主要来源于个体间,分别占总变异的96%和72.84%。鸡西种群与其他种群有着较远的遗传距离,平均遗传分化指数(Fst)为0.026 8,同时具有较高的遗传多样性,Shannon指数和表型多样性指数分别为1.111和2.00。结构分析表明5个红松天然种群没有明显亚群结构。不同林龄红松种群的遗传多样性没有明显变化,且较小林龄的种群不存在杂合缺失和近亲繁殖的现象,针叶性状与地理因素存在广泛的相关性,造成不同红松种群的表型分化。【结论】以分子和表型标记共同构建30%取样比例的红松核心种质,Shannon指数与表型多样性指数分别为1.076、2.018,能够较好地代表红松天然种群的遗传现状,也能更好地促进红松种质资源管理、保护和利用;根据红松遗传结构特征,建议重点从原地保护方面开展对红松天然种质的保护,以促进红松天然种群的生态恢复、种质保护及利用。
关键词:红松;天然种群;遗传多样性;遗传结构;核心种质;林木育种;乡土树种
中图分类号:S791.247;S722"""" 文献标志码:A开放科学(资源服务)标识码(OSID):
文章编号:1000-2006(2024)05-0069-12
Analysis of genetic diversity and construction of core collections of Korean pine (Pinus koraiensis) natural population
YAN Pingyu, ZHANG Lei, WANG Jiaxing, FENG Kele, WANG Haohao,ZHANG Hanguo
(State Key Laboratory of Tree Genetics and Breeding, Northeast Forestry University, Harbin" 150040, China)
Abstract:【Objective】 Korean pine (Pinus koraiensis) is a valuable tree species that is distributed throughout northeastern China. Over the past century, human interference has led to a gradual decrease in the number of individuals and distribution" of its natural population. Assessing the genetic diversity and building a core collection of natural Korean pine could provide a scientific basis for the effective conservation, management, and utilization of Korean pine germplasm resources. 【Method】 A total of five well-preserved natural populations of Korean pine in Hebei, Wuying, Xiaobeihu" and Jixi in Heilongjiang Province and Lushuihe in Jilin Province in northeast China were studied. A combination of phenotypic data and molecular markers was used to construct the core collection. 【Result】 Molecular and phenotypic ANOVA results showed that the genetic variation of Korean pine natural populations mainly originated from inter-individual differences, which accounted for 96% and 72.84% of the total variation, respectively. The Jixi population was genetically distant from other populations, with an average Fst of 0.026 8. It also had a high genetic diversity, with Shannon and phenotypic" diversity index values of 1.111 and 2.00, respectively. The population structure analysis showed that the five Korean pine natural populations had no obvious subpopulation structure. There were no significant changes in the genetic diversity of Korean pine populations among the different forest ages. Additionally, in the younger forest there was no evidence of heterozygous deletions or inbreeding. There was a broad correlation between needle traits and geographic factors, resulting in the phenotypic differentiation of Korean pine populations. 【Conclusion】 The Shannon and phenotypic" diversity indexes of the core collection constructed by combining molecular and phenotypic markers with a 30% sampling ratio were 1.076 and 2.018, respectively, which was representative of the genetic status of Korean pine populations. This information can be used to better manage the germplasm resources of Korean pine and promote its protection and use. The genetic structure characteristics indicated a need to focus on in situ protection of the natural germplasm and" to promote ecological recovery, germplasm protection, and use of Korean pine.
Keywords:Korean pine(Pinus koreciensis); natural populations; genetic diversity; genetic structure; core collection; forest breeding; native trees
红松(Pinus koraiensis)天然分布范围主要在我国东北地区长白山及其北部的张广才岭、老爷岭、完达山和小兴安岭[1],在俄罗斯、朝鲜和日本也有分布[2],既是优良的用材树种,也是珍贵的食用干果和油料树种[3]。由于红松生长缓慢且童期较长,在自然条件下更新缓慢,一旦被破坏就很难恢复[4]。红松天然林资源较少,已被列为国家重点二级保护野生植物[2]。因此,评估红松的遗传多样性以促进对现有天然红松种质资源的有效管理和保护十分必要。
对物种多样性进行全方位评价和遗传背景调查是保护野外种群的重要环节[5]。群体遗传多样性研究是保护遗传学的基础,种质资源保护是其核心[6]。对物种遗传多样性的研究可对物种现有的生存方式及状态进行有效评估[7]。目前除了传统的形态学标记,分子标记技术在多样性分析方面已被广泛应用。如张巍等[8]用10个ISSR标记对10个不同红松天然林种源进行多样性分析结果表明,小兴安岭林区红松种群的遗传多样性水平较高,各种源遗传分化大且与地理分布相关;童茜坪等[9]利用11条具有多态性的ISSR引物进行红松种子园单株遗传多样性分析,证明了红松种子园单株具有丰富的遗传多样性。
核心种质资源(core collection)指保存种质资源的一个核心子集,是采用一定方法从保存的种质资源中进行抽取,以最少的遗传资源样本量来最大限度地代表整个资源群体的遗传多样性和分布范围,而未列入核心种质的其他资源材料则作为保留样本予以保存[10-11]。因此核心种质可以作为种质资源研究和利用的切入点,从而提高整个种质库的管理和利用水平。一些学者也对植物核心种质构建的方法进行了分析和探讨,如多样性指数法的系统抽样法、分组法和最小距离逐步抽样法[12-14]。
由于分子标记克服了表型性状易受环境影响的缺点,从而提高了所构建核心种质的有效性,目前分子标记已成为构建植物核心种质的有效研究手段,被广泛应用于树木的核心种质资源库构建中。如Boccacci等[15]使用10个简单重复序列(SSR)标记,建立了代表欧榛(Corylus avellana)遗传多样性的核心种质库;Yang等[16]采用20个SSR标记对来自中国8个地点304株马尾松(P. massoniana)的遗传多样性进行了评价,构建了104个个体的核心库。然而在构建核心种质资源库的过程中,植物的表型也不应被忽视,因为表型变异是受遗传因素和环境因素共同影响,是同种植物适应不同环境条件的结果,在适应和进化上具有重要意义。因此,也有不少研究将分子标记和表型性状结合来进行核心种质库的构建,如Duan等[17]测量了中国南方6个省份的杉木(Cunninghamia lanceolata)10个生长和材性性状,并使用SSR标记评估该种质资源的遗传变异,建立了一个能够代表整个种质的核心库。Guo等[18]收集了1 054份刺槐(Robinia pseudoacacia)种质,利用48个SSR标记、13个表型性状和3个生理性状对种质多样性进行了评价,构建了一个刺槐核心种质库。基于分子标记与表型性状共同构建核心种质库在松树中也有研究,如Wang等[19]利用780个样本的表型特征和SRAP标记的综合数据,构建了云南松(P. yunnanensis)的核心库。然而以上研究主要基于已收集或栽培驯化的种质进行评价及核心库的构建,通过构建核心种质的方法促进天然群体种质保护和利用的应用较少,依据遗传多样性水平的高低结合表型遗传变异大小来构建红松核心种质(库)以促进天然种质资源保护的研究也鲜见报道。
近几十年间,由于过度砍伐导致大量红松遗传资源丢失,天然种群结构及规模被严重破坏。因此,调查现有天然红松种群并进行遗传分析,探究核心种质构建技术,以最少的种质材料代表原始种质资源的全部或大多数,将为当前有效收集、评价和利用红松种质资源带来便利,同时也为红松天然种群恢复提供科学依据。
1 材料与方法
1.1 试验材料
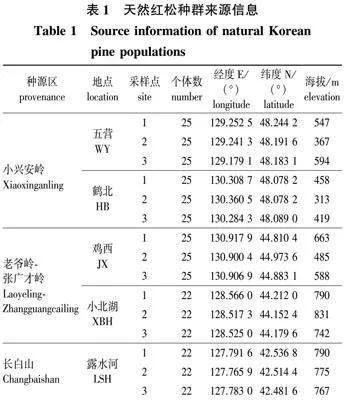
参考夏德安等[20]关于红松种源的区划,选择了黑龙江省鹤北、五营、小北湖和鸡西以及吉林省露水河5个保存现状良好的红松种群。每个种群设置3个采样点,采样点相距8 km以上,记录采样点坐标及海拔(表1)。每个采样点随机选择22或25株中壮龄天然红松,单株间距离50 m以上,于2022年7月初采集1~2年生针叶,并测量所选择红松单株胸径。
1.2 试验方法
使用植物基因组提取试剂盒(天根DP-320,中国北京)对红松样本总DNA进行提取,使用质量分数1%琼脂糖凝胶检测DNA的完整性,并用MicroDrop 3000(Bio-DL,中国上海)检测DNA浓度与质量。将每个DNA样本的质量浓度稀释至10 ng/μL,-20 ℃保存用于SSR扩增。
从已发表的松科SSR引物中选择火炬松(P. taeda)[21-22]、美国白松(P. albicaulis)[23]、大别山五针松(P. dabeshanensis)[24]、华山松(P. armandii)[25]、红松(P. koraiensis)[3,26-27]、马尾松(P. massoniana)[28]和毛枝五针松(P. wangii)[29]7个物种总计142对SSR引物,由生工生物工程(中国上海)股份有限公司合成,随机选取10个红松DNA样本筛选多态性引物。PCR体系体积为20 μL,包含2U TaqDNA聚合酶、400 μmol/L dNTP、2.0 μL 10倍缓冲液、正反向引物各0.5 μmol/L和10 ng DNA。PCR程序如下:94 ℃ 3 min,35个循环;94 ℃ 30 s,退火温度Tm下30 s,72 ℃延伸15 s;72 ℃下最终延伸7 min。PCR产物通过7%非变性PAGE进行检测,并通过银染观察最终筛选出11对具有明显多态性且可稳定重复的SSR标记,在避光条件下用上述相同反应体系进行PCR,PCR产物由生工生物工程(上海)股份有限公司在ABI3730遗传分析仪上进行检测,用Data Collection 3.0程序(ABI 3730遗传分析仪自带)收集原始二倍体基因型数据。
每株选择3束2年生红松针叶,用直尺及游标卡尺分别测量针叶长、针叶宽,计算叶长宽比;用万分之一天平测量鲜质量,65 ℃烘干至质量恒定后,测量针叶干质量并计算含水量;以丙酮-乙醇法[30]测定叶绿素a、叶绿素b及总叶绿素含量。
1.3 数据处理
1.3.1 遗传分析
将DNA多态性信息处理为数据矩阵,利用软件Data Formater将数据矩阵转换成各种格式,以供进一步分析[31]。利用软件PowerMarker 3.25计算引物多态性信息含量(PIC)[32],运用GenAIEX6.51b2计算等位基因数(Na)、有效等位基因数(Ne)、Shannon指数(I)、观测杂合度(Ho)、期望杂合度(He)、基因流(Nm)、无偏期望杂合度(Hue)、固定指数(F)和遗传分化指数(Fst)[33],进行分子方差分析以剖分遗传变异来源,以及进行主坐标(PCoA)分析,生成种群间Nei遗传距离及基因流矩阵,利用MEGA11生成红松种质聚类图[34]。
利用Structure 2.3.4对遗传结构进行分析,群体数(K)设置1~10,每个K值模拟运算10次,将不作数迭代和不作数迭代后的MCMC均设为10万次;运算结果导入Structure Harvester网站(https://taylor0.biology.ucla.edu/structureHarvester/),根据Evanno 等[35]的方法选择最佳K值。
根据红松种群地点坐标,通过以下公式计算种群间地理距离:
d=R×arcos[cos(Y1)×cos(Y2)×cos(X1-X2)+sin(Y1)×sin(Y2)]。(1)
式中:d为地点间距离,km;R为地球半径6 371.0 km;X1、X2、Y1、Y2分别为两个地点坐标弧度,弧度为坐标×π/180;用SPSS 27.0检测5个天然种群间地理距离和遗传距离之间的相关性。
对于天然红松单株来说,常用生长锥钻取木芯或伐木得到个体年龄,但对树体破坏性较大,鉴于此,本研究采用胸径大小代替年龄结构分析其动态特征,计算公式[36]如下:
t=e(+321.980 15)69.879 16。(2)
式中:t为估算林龄等级;为平均胸径。
1.3.2 针叶性状分析
用SPSS 27.0对红松天然种群针叶表型性状的平均值()、标准差(δ)、方差和变异系数进行计算,并进行表型多样性指数、表型相关性分析,以及主成分和聚类分析。胸径及针叶性状被划分为10个级别,数值lt;(-2σ)为第1级别,数值≥(+2σ)为第10级别,其中每0.5σ划分为1个级别。
针叶表型多样性指数采用Shannon-Weaver多样性指数(H′)[37-38],计算公式如下:
H′=-∑Pi(lnPi)。(3)
式中:H′为某一性状的多样性程度;Pi为某一性状第i个级别的种质份数占总种质材料的百分比。
采用Lindgren等效纬度[39]的概念,其计算公式如下:
Le=L+(H-300)/a。(4)
式中:Le为等效纬度;L表示纬度;H表示海拔。当H≥300 m时,a=140;当H<300 m时,a=200。
1.3.3 天然红松核心库的构建与评价
计算个体间表型性状欧氏距离和分子标记遗传距离,并参考徐海明[40]的方法对表型和分子遗传距离进行整合,基于以上3种距离按照UMPGA方法进行聚类,分别按照10%、20%、30%、40%和50%的比例,利用逐步聚类取样法进行取样;分别对各集合的遗传参数和表型多样性指数进行计算,并按照Hu等[41]的方法,计算均值差异百分率(MD)、方差差异百分率(VD)、极差符合率(CR)、变异系数(CV)和变异系数变化率(VR)来评价核心种质的代表性。
基于核心种质研究结果,分别利用针叶表型和分子标记为第1、第2主成分因子得分绘制保留种质和核心种质的主坐标分布图。
2 结果与分析
2.1 天然红松种群遗传分析
2.1.1 遗传多样性分析
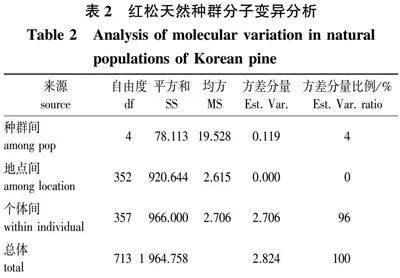
为确定5个天然种群的变异特征,分子变异分析结果表明(表2)其遗传变异主要存在于个体间,占总变异的96%,种群间遗传变异的发生率仅为4%,这表明种群间的遗传分化小,而种群内个体间的遗传变异大。
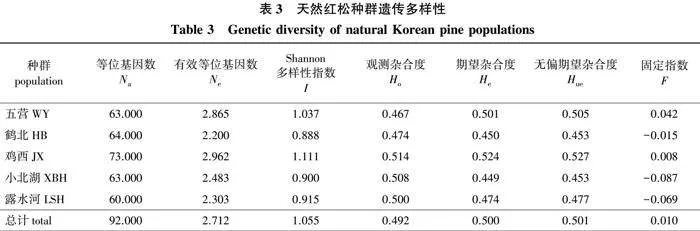
为了评估5个红松种群的遗传多样性和遗传分化程度,对其进行遗传多样性分析,结果表明,红松种群整体表现出较高的遗传多样性,Shannon多样性指数为1.055(表3)。5个种群遗传多样性水平差异不大,其中鸡西种群遗传多样性最高,等位基因和有效等位基因最多,分别为73.000和2.962,Shannon多样性指数、观测杂合度、期望杂合度和无偏期望杂合度也最高,分别为1.111、0.514、0.524和0.527;鹤北种群Shannon多样性指数最低,为0.888,有效等位基因也最少,为2.200;观测杂合度最低的为五营种群。整体的固定指数(F)为0.010,说明红松天然种群整体上杂合度适中,各种群固定指数从-0.087(小北湖)至0.042(五营)不等,显示出五营种群存在轻微的杂合缺失和近亲繁殖。
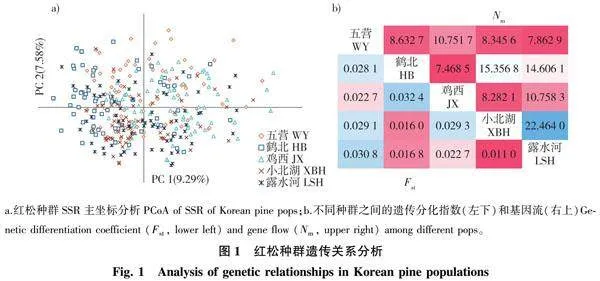
对5个种群的个体进行主坐标分析结果(图1a)显示,轴1解释了9.29%的变异,轴2解释了7.58%的变异,说明以上分子标记各自具有较高的独立性。图中种群间具有高度的分布重叠,不存在明显的遗传分化。鸡西种群分布范围较大,部分个体显示出相对独立性,鹤北和小北湖种群部分个体分布相对聚集,且鹤北种群大多分布在坐标轴左侧,表明鹤北种群内遗传多样性相对较低,这与Shannon多样性指数估算结果相似。5个天然红松种群间的遗传分化指数Fst值从0.011 0~0.032 4不等(平均为0.024),种群间基因流Nm值在7.468 5~22.464 0之间变动(平均为11.453),说明种群间遗传分化小且存在高频率的基因流(图1b)。红松种群的Nei遗传距离和地理距离间Person相关系数为0.135(P=0.704),说明红松种群遗传距离与其地理距离之间没有显著的相关性。
2.1.2 遗传结构分析
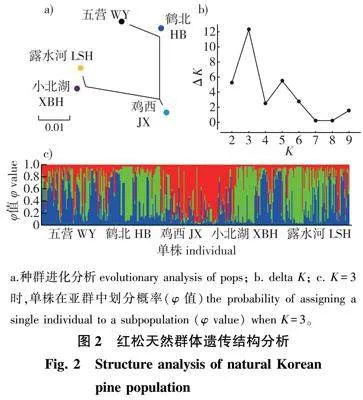
为进一步探索种群间遗传关系,通过种群间的Nei遗传距离进行种群进化分析,结果(图2a)显示,五营种群和鹤北种群为同一进化分支,露水河种群与小北湖种群为同一进化分支且遗传距离最短,鸡西种群位于两个遗传簇的中间位置,这与以上分析结果一致,且说明了鸡西种群与其他种群相对特殊的遗传关系。对所有参试材料进行遗传结构分析(图2b、2c),K=1~10中,最佳K值为3,说明遗传变异来源于3个基因池,而且都是混合的,没有明显的遗传结构,说明红松天然种群不存在明显的结构分化。
2.1.3 不同龄级遗传分析
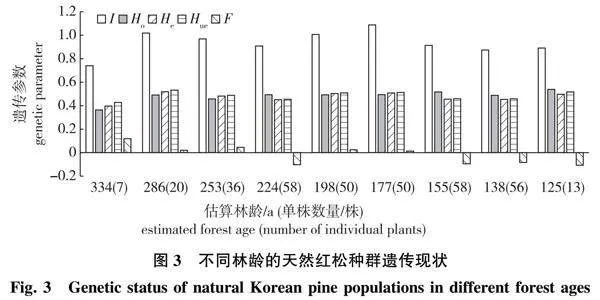
以胸径调查结果代替不同年龄等级来分析红松种群时间分布上的动态,结果(图3)显示:5个红松种群共分为林龄由大到小的9个种群,估算平均林龄最大种群为334 a,最小为125 a。整体上看,各个林龄种群的Shannon指数和杂合度没有明显变化。值得注意的是,年龄较小的3个种群与其他种群相比,固定指数均小于0,说明相对于林龄较大的种群,较小林龄的红松种群在自然更替的过程中不存在杂合缺失以及近亲繁殖的现象,尤其是林龄最小的种群9,虽然仅有13个单株,但仍保持了较高的多样性以及杂合度。
2.2 红松种群针叶性状分析
2.2.1 红松种群针叶性状表型变异和多样性分析
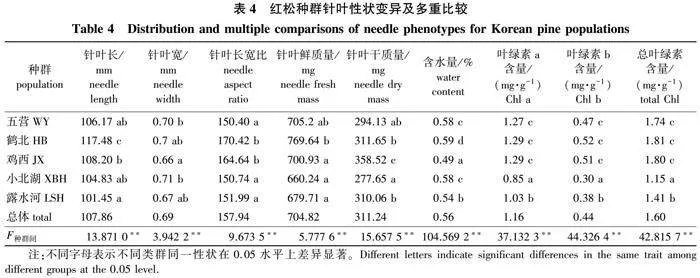
对不同红松天然种群间针叶性状分析(表4)表明,9个针叶指标在不同种群间均存在极显著差异(Plt;0.01),各性状来源于种群间和种群内的方差分量百分比平均值分别为27.16%和72.84%,说明针叶性状变异主要来源于种群内,其中针叶宽性状尤为明显,其种群内方差分量占比达95.73%,仅针叶含水量(质量分数)的方差分量种群间(61.08%)大于种群内(38.92%),因为含水量指标可能更多受种群所处地点空气及土壤干湿程度所影响。另外,鹤北种群的针叶表型性状数值较大,小北湖和露水河种群数值最小,这可能与所处纬度及海拔等地理因素有关。
针叶性状变异及多样性分析结果(表5)表明,各性状平均变异系数最小的是鹤北种群(16.51%),最大的是小北湖种群(21.32%),是唯一高于整体变异水平(20.59%)的种群。各种群表型平均多样性指数与变异系数结果不同,鸡西种群的平均多样性指数最高(2.00),露水河种群的平均多样性指数最低(1.97),均小于总体的平均多样性指数(2.04)。各种群表型遗传变异大小及表型多样性指数除受遗传控制外,还受立地条件及人为干扰程度影响,较大的遗传变异反映出该种群较宽的遗传背景外,也反映出适宜的立地条件以及较少的人为干扰。
2.2.2 红松种群针叶性状主坐标分析
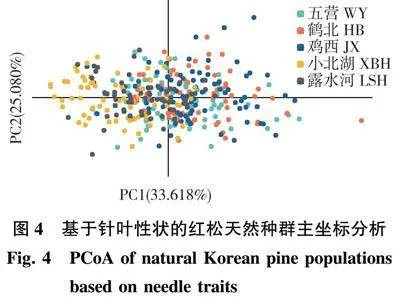
基于红松种群针叶性状的主成分分析,提取前两个主成分因子得分,能够分别解释总方差的33.618%和25.080%。绘制5个种群的主坐标分布图(图4),结果显示,各种群内个体分布在坐标的中心具有高度重合,种群间具有各自相应分布范围,小北湖种群和露水河种群主要分布在纵坐标左侧,相对露水河种群,小北湖种群分布范围更大,五营种群主要分布在中间范围,鹤北种群和鸡西种群高度重合,主要分布在中心范围及纵坐标右侧,鸡西种群有着最大的分布范围,这与基于分子标记的红松种群主坐标分析结果一致。
2.2.3 红松种群针叶性状相关分析
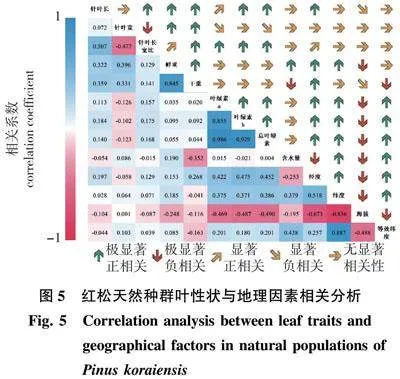
红松种群针叶性状与所处地理气候因子的相关性分析(图5)表明:红松针叶性状与地理因素存在明显的相关性,其中经度与针叶长、鲜质量、干质量、叶绿素含量存在极显著的正相关,与针叶长宽比存在显著正相关,而与含水量显著负相关;纬度与针叶鲜质量、叶绿素、含水量的相关性极显著;海拔与针叶性状主要为负相关,与针叶长、针叶干质量显著负相关,与针叶鲜质量、叶绿素含量以及含水量极显著负相关。红松针叶性状间也存在明显相关关系:针叶长与针叶鲜质量、针叶干质量以及叶绿素含量存在极显著的正相关;针叶宽与针叶鲜质量、干质量极显著正相关,与叶绿素含量显著负相关;针叶长宽比与针叶干质量及叶绿素含量极显著正相关。
2.3 红松核心种质资源构建
2.3.1 不同抽样比例和抽样策略的核心库比较
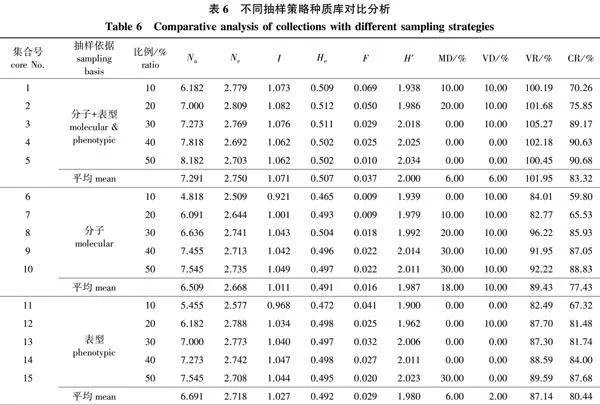
按照逐步聚类取样法分别以10%、20%、30%、40%、50%的取样比例重复10次抽样,在3种取样数据的基础上进行核心种质库的构建。比较不同取样比例构建的核心种质遗传差异发现,等位基因数、表型多样性指数和极差符合率随着取样比例的增大而增大,有效等位基因数、期望杂合度、Shannon多样性指数和变异系数变化率均在取样比例为30%时达到最大值,观测杂合度在30%、40%、50%的取样比例时差别不大,分别为0.485、0.484、0.486,均值差异百分率和方差差异百分率则无明显规律,且分别在50%和20%的取样比例下表现出最大值。基于核心种质库应以最少样本量最大限度地代表整个种群的原则,在红松核心种质的构建中,30%的取样比例最为适宜。在构建红松核心种质库时,依据不同类型的取样数据得到的结果存在较大差异(表6)。
依据分子标记及针叶表型共同构建的5个集合整体表现最佳,5个集合的平均等位基因数、有效等位基因数、Shannon多样性指数和期望杂合度均最大,分别为7.291、2.750、1.071和0.507,同时具有最大的表型多样性指数(2.00)、变异系数变化率(101.95%)、极差符合率(83.32%)和最小的均值差异百分率(6.00%)。平均等位基因数、有效等位基因数、Shannon多样性指数、期望杂合度和极差符合率最低的是依据分子标记构建的5个集合,分别为6.509、2.668、1.011、0.491和77.43%,但其均值差异百分率和方差差异百分率也最大(18.00%和10.00%);基于针叶表型构建的5个集合多样性指数最低(1.980),因此在构建红松核心种质时,应当结合分子标记和表型性状数据进行分析。基于分子标记及表型性状且以30%取样比例构建的集合3不仅能够较好地还原原始种质的均值和极差,提高变异系数,还具有较高的Shannon多样性指数和表型多样性指数及最大的观测杂合度,最终确定集合3为天然红松种群的核心种质。
2.3.2 核心种质的确认
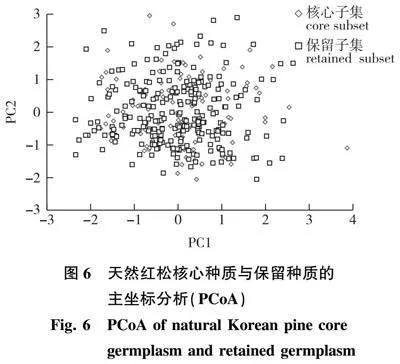
为了进一步了解核心种质资源代表性,基于针叶表型和分子标记分别为第1、第2主成分因子得分绘制原始种质和核心种质主坐标分布图,结果(图6)表明:核心种质与原始种质的分布范围相当且在原始种质中均匀分布,表明核心种质库去除了冗余,在针叶表型和遗传多样性方面均有良好的代表性。
对核心种质在各种群中的分布情况进行分析,结果显示,在本研究初步构建的核心种质中,鸡西种群的种质份数占比最多,为39.25%,五营种群的种质占比最小,仅为5.61%,鹤北、鸡西、小北湖种群核心种质占比均高于原始种质,表明3个种群在红松天然种群中具有更重要的代表性。从材料来源地点种源区(表1)看,来自长白山种源区的核心种质占比最少,为12.15%,占比最高的为老爷岭-张广才岭种源区(59.81%),也是唯一高于原始种质的种源区,小兴安岭种源区的核心种质占比为28.04%,远低于其原始种质的42.02%。
3 讨 论
基于SSR标记的结果显示,本研究中红松的遗传变异主要来源于天然种群内个体间,占96%,种群间变异仅占4%,表明红松不同种群之间遗传分化很小(Fst=0.024),这与冯富娟等[42]研究认为种源的多样性主要来源于种源内部的结果一致,这一现象应与红松为异花风媒授粉植物以及具有较长生命周期有关。
鸡西种群遗传多样性水平最高(Ne=2.962,I=1.111,Ho=0.514),鹤北种群遗传多样性水平最低(Ne=2.200,I=0.888,Ho=0.474),其中五营种群的有效等位基因数(2.865)和Shannon指数(1.037)大于张巍等[8]的研究结果(1.079 5和0.879 7),这可能是由于样本量大小不同。除五营种群外,其他种群都不存在近亲繁殖现象,整个红松天然种群具有合理的交配体系。红松为演替的顶极群落中的优势树种,分布地域广,寿命长达300~400 a,这都是红松具有较高遗传多样性的生物学基础。遗传多样性和遗传结构研究是制定物种保护措施的前提条件,也是科学合理地利用种质资源和改进遗传特性的理论基础[43]。聚类分析与主坐标分析结果得到了相似的结论,也证明了个体间的遗传变异远远大于种群间,说明红松种群间没有阻止基因流动的严重障碍,形成了较广泛的基因交流。对思茅松(P. kesiya var. langbianensis)相关研究也有类似发现[44]。现阶段种群演替在空间上的差异可以反映种群在时间上的变化,不同时期红松的遗传多样性略有波动,但整体上看波动不大,基本呈现稳定的趋势,这与邵丹等[45]的研究结果相似。值得注意的是,与较早的种群相比,较近时间内的红松种群不存在杂合缺失、过量纯和的现象,说明各时期的天然种群变化属于自然演替过程,遗传多样性基本稳定,更新种群不存在近亲繁殖现象。
通过对红松天然分布的5个种群针叶表型性状的研究发现,红松针叶表型性状种群内变异占据了更主要的位置(占比72.84%),这与白皮松(P. bungeana)相关研究结果类似[46],变异主要来自针叶宽、针叶鲜质量和针叶长宽比,但各性状在种群间仍存在极显著的差异。大量研究表明,物种的表型变异程度往往与其分布范围的大小呈正相关[47-49],这在本研究中也得到了证实,红松分布范围较小且分布零散,难以形成单一纯林或群落,所以其表型变异较小,平均变异系数为20.59%,变异幅度为4.48%~36.94%。在实地调查过程中发现,与其他种群相比,鹤北种群分布相对较为集中,故其针叶表型性状的变异系数较低,针叶表型多样性指数也得到了类似的结果。正因为红松分布区较小,分布区内的气候条件差异不大,所以种群间变异也较小。一般来说,异花授粉植物的交配系统可以促进种群间基因交流、增加有效种群大小、减少基因漂变对遗传结构的影响。红松是雌雄同株的风媒异花授粉植物,这在大大提升繁衍后代概率的同时也促进了个体间的基因交流。红松在适应不同环境的过程中产生了表型的变异,本研究中各性状在不同种群间也具有极显著的差异,如小兴安岭种源区的五营与鹤北种群具有较大的针叶长、含水量和叶绿素含量,而最南部长白山种源区的露水河种群针叶长和针叶鲜质量均最小;对针叶性状进行主坐标分析可得到相同的结果。针叶表型与地理因素的相关分析也验证了这一结果,针叶长、针叶鲜质量、针叶干质量、叶绿素含量和叶含水量等的性状与其地点的纬度、海拔和等效纬度之间广泛存在显著甚至极显著的相关性。
保护一个物种全部种质资源的遗传多样性既困难又昂贵,因此应该构建具有最小重复性和最大遗传多样性的核心子集,以代表一个物种的整个基因库[50]。目前核心种质库的构建主要依据表型性状或分子标记手段,如陈存等[38,51]分别利用表型性状和SSR分析标记技术进行美洲黑杨核心种质库的构建,结果认为应结合表型分析进一步利用分子标记的方法构建核心种质库,从而更有效地保存种质资源。徐益等[52]也认为同时结合种质的表型和遗传信息特征的聚类分析结果对其进行分组,构建的核心种质更具有代表性。Belaj等[53]认为木本植物的取样比例应为10.00%~45.00%。本研究分别利用表型性状、分子标记数据以及两者结合,利用逐步聚类取样方法分别按照10%、20%、30%、40%、50%的取样比例进行红松核心种质库的构建,共得到15个不同的集合。目前,主要通过与原始种质表型性状均值差异百分率(MD)、方差差异数(VD)、极差符合率(CR)、变异系数符合率(VR)以及等位基因数(Na)、Shannon多样性指数(I)、观测杂合度(Ho)之间的差异比较,评价核心种质库的代表性,MD越小,VD、CR和VD越大,核心种质库的代表性和表型多样性越好[54]。基于以上原则,结合表型和分子的方法在30%取样比例时最能有效代表原始种质,其有效等位基因数、Shannon多样性指数和观测杂合度分别为2.769、1.076和0.497,表型多样性指数、均值差异数、方差差异数、极差符合率和变异系数符合率分别为2.018、0.00%、10.00%、105.27%和89.17%,最终确定为红松核心种质资源库。通过比较核心种质和原始种质的主坐标对核心种质库的代表性进行确认,在原始种质中均匀分布的核心种质最大限度地还原了原始种质的多样性。在各种群的种质分布比例上,鸡西种群、小北湖种群和鹤北种群比原始种质占比更大,表明上述3个种群的变异程度较大,五营种群变异程度较小。对比SSR多样性与表型变异,在评价整体与各种群遗传水平时可以得出一致的结果,但在种群间表型的变异远大于SSR,这说明SSR标记在反应基因组水平上的变异存在一定的局限性,同时表型性状由于受到环境的影响而表现出大于基因水平多样性的变异,通过将二者结合的手段可以克服以上方法所存在的缺点。
由于人类的过度采伐和生产活动造成天然红松林分布区缩小及生态条件恶化,导致红松遗传资源急剧减少,因此有必要加强对天然红松的保护和积极的人为扩展,促进红松种质遗传多样性恢复到其应有水平,为保护种群稳定存在创造有利的遗传基础。一般而言,原地保存和异地保存是植物种群保护的两种主要方式[55],针对现有种群进行就地保护是最有效的保护措施[56],异地保存通常针对群体间存在分化且遗传多样性低的群体[46],原地保存通常针对群体间无明显的遗传分化且遗传多样性高的群体[55,57]。作为东北地区典型的珍贵树种,应进一步加强对红松种质资源的开发利用,尽早选育以实现经济价值为目的的优良种质,在实现生态价值的同时利用红松种质创造更多社会价值及经济价值。面对红松未来育种需要,本研究所收集种质及所构建的核心种质将利用更多技术手段,如高通量测序技术、表型组学技术等进行评估,同时对红松种质的结实、材性等经济性状进行继续观测,以满足未来育种目标的需要。红松核心种质库的建立也能更好地促进红松种质资源管理、保护和利用,为有效保存、管理和利用红松种质资源提供科学依据。
根据本研究结果,红松的保护策略应从原地保存方面重点开展,对树龄较大或性状表现优异的单株进行挂牌保护,还可以在全分布区建立长期观测种源的试验基地[58]。另外也应在异地保存方面开展部分工作,如尽量采集不同种源地的种质资源,建立红松种质资源圃进行异地保存,最大程度保存遗传多样性的同时也可有效增加红松对环境的适应能力和适应范围,有利于对红松适应性、抗逆性及经济性等各方面进行深入研究。
致谢:
本研究得到冯可乐、王佳兴同学在采样过程中的协助,王浩浩同学在数据处理过程中的协助,张含国老师和张磊老师在试验设计以及文章撰写过程中的指导。
参考文献(reference):
[1]马建路,庄丽文,陈动,等.红松的地理分布[J].东北林业大学学报,1992,20(5):40-48.MA J L,ZHUANG L W,CHEN D, et al.Geographic distribution of Pinus koraiensis in the world[J].J Northeast For Univ,1992,20(5):40-48.
[2]YU D P,ZHOU L,ZHOU W M,et al.Forest management in northeast China:history,problems,and challenges[J].Environ Manage,2011,48(6):1122-1135.DOI: 10.1007/s00267-011-9633-4.
[3]张振,张含国,莫迟,等.红松转录组SSR分析及EST-SSR标记开发[J].林业科学,2015,51(8):114-120.ZHANG Z,ZHANG H G,MO C,et al.Transcriptome sequencing analysis and development of EST-SSR markers for Pinus koraiensis[J].Sci Silvae Sin,2015,51(8):114-120.DOI: 10.11707/j.1001-7488.20150815.
[4]FENG F J,HAN S J,WANG H M.Genetic diversity and genetic differentiation of natural Pinus koraiensis population[J].J For Res,2006,17(1):21-24.DOI: 10.1007/s11676-006-0005-5.
[5]王欢利,严灵君,黄犀,等.南京椴群体遗传多样性和遗传结构分析[J].南京林业大学学报(自然科学版),2023,47(1):145-153.WANG H L,YAN L J,HUANG X,et al.Genetic diversity and genetic structure of Tilia miqueliana population[J].J Nanjing For Univ (Nat Sci Ed),2023,47(1):145-153. DOI: 10.12302/j.issn.1000-2006.202110049.
[6]顾万春.森林遗传资源学概论[M].北京:中国科学技术出版社,1998.GU W C.Introduction of forest genetic resource science[M].Beijing:China Science and Technology Press,1998.
[7]尚占环,姚爱兴.生物遗传多样性研究方法及其保护措施[J].宁夏农学院学报,2002,23(1):66-69.SHANG Z H,YAO A X.Research means of genetic diversity and its protective measures[J].J Ningxia Agric Coll,2002,23(1):66-69. DOI: 10.3969/j.issn.1673-0747.2002.01.021.
[8]张巍,王清君,郭兴.红松不同种源的遗传多样性分析[J].森林工程,2017,33(2):17-21.ZHANG W,WANG Q J,GUO X.Study on the genetic diversity of Pinus koraiensis in different provenances[J].For Eng,2017,33(2):17-21.DOI: 10.16270/j.cnki.slgc.2017.02.004.
[9]童茜坪,剡丽梅,张磊,等.红松种子园单株ISSR-PCR遗传多样性分析[J].林业科技,2020,45(2):17-20.TONG Q P,YAN" L M,ZHANG L,et al.Genetic diversity analysis on indivitual of Pinus koraiensis in seed orchard based on ISSR-PCR[J].For Sci Technol,2020,45(2):17-20.DOI: 10.19750/j.cnki.1001-9499.2020.02.005.
[10]FRANKEL O H,BROWN A H D.Current plant genetic resources: a critical appraisal[M].Londen: Oxford amp; IBH Publishing Co,1984:3-13.
[11]VASCONCELOS E S,CRUZ C D,BHERING L L,et al.Strategies for sampling and establishment of core collections[J].Pesquisa Agropecuaria Brasileira,2007,42:507-514.DOI:10.1590/S0100-204X2007000400008.
[12]李自超,张洪亮,曾亚文,等.云南地方稻种资源核心种质取样方案研究[J].中国农业科学,2000,33(5):1-7.LI Z C,ZHANG H L,ZENG Y W,et al.Study on sampling schemes of core collection of local varieties of rice in Yunnan,China[J].Sci Agric Sin,2000,33(5):1-7.DOI: 10.3321/j.issn:0578-1752.2000.05.001.
[13]赵冰,张启翔.中国蜡梅种质资源核心种质的初步构建[J].北京林业大学学报,2007,29(S1):16-21.ZHAO B,ZHANG Q X.Preliminary construction of core germplasm of Chimonanthus praecox germplasm resources in China[J].J Beijing For Univ,2007,29(S1):16-21.DOI: CNKI: SUN: BJLY.0.2007-S1-005.
[14]LIU M,HU X,WANG X,et al.Constructing a core collection of the medicinal plant Angelica biserrata using genetic and metabolic data[J].Front Plant Sci,2020,11:600249.DOI: 10.3389/fpls.2020.600249.
[15]BOCCACCI P,ARAMINI M,ORDIDGE M,et al.Comparison of selection methods for the establishment of a core collection using SSR markers for hazelnut (Corylus avellana L.) accessions from European germplasm repositories[J].Tree Genet Genomes,2021,17(6):48.DOI: 10.1007/s11295-021-01526-7.
[16]YANG H B,LIU Q H,ZHANG R,et al.Genetic diversity of second generation-parental germplasm of Masson pine revealed by SSR markers and establishment of a core germplasm collection[J].Scand J For Res,2021,36(7/8):524-531.DOI: 10.1080/02827581.2021.1981432.
[17]DUAN H J,CAO S,ZHENG H Q,et al.Genetic characterization of Chinese fir from six provinces in southern China and construction of a core collection[J].Sci Rep,2017,7(1):13814.DOI: 10.1038/s41598-017-13219-0.
[18]GUO Q,LIU J,LI J K,et al.Genetic diversity and core collection extraction of Robinia pseudoacacia L.germplasm resources based on phenotype,physiology,and genotyping markers[J].Ind Crops Prod,2022,178:114627.DOI: 10.1016/j.indcrop.2022.114627.
[19]WANG X L,CAO Z L,GAO C J,et al.Strategy for the construction of a core collection for Pinus yunnanensis Franch.to optimize timber based on combined phenotype and molecular marker data[J].Genet Resour Crop Evol,2021,68(8):3219-3240.DOI: 10.1007/s10722-021-01182-9.
[20]夏德安,杨书文,杨传平,等.红松种源试验研究(Ⅰ):种源的初步区划[J].东北林业大学学报,1991,19(S2):122-128.XIA D A,YANG S W,YANG C P,et al.Experimental study on provenance of Korean pine (Ⅰ): preliminary division of provenance[J].J Northeast For Univ,1991,19(S2):122-128.
[21]LIEWLAKSANEEYANAWIN C,RITLAND C E,EL-KASSABY Y A,et al.Single-copy,species-transferable microsatellite markers developed from loblolly pine ESTs[J].Theor Appl Genet,2004,109(2):361-369.DOI: 10.1007/s00122-004-1635-7.
[22]ECHT C S,SAHA S,DEEMER D L,et al.Microsatellite DNA in genomic survey sequences and UniGenes of loblolly pine[J].Tree Genet Genomes,2011,7(4):773-780.DOI: 10.1007/s11295-011-0373-7.
[23]LEA M V,SYRING J,JENNINGS T,et al.Development of nuclear microsatellite loci for Pinus albicaulis Engelm.(Pinaceae),a conifer of conservation concern[J].PLoS One,2018,13(10):e0205423.DOI: 10.1371/journal.pone.0205423.
[24]XIANG X Y,ZHANG Z X,WANG Z G,et al.Transcriptome sequencing and development of EST-SSR markers in Pinus dabeshanensis,an endangered conifer endemic to China[J].Mol Breed,2015,35(8):158.DOI: 10.1007/s11032-015-0351-0.
[25]DONG W L,WANG R N,YAN X H,et al.Characterization of polymorphic microsatellite markers in Pinus armandii (Pinaceae),an endemic conifer species to China[J].Appl Plant Sci,2016,4(10):apps.1600072.DOI: 10.3732/apps.1600072.
[26]YU J H,CHEN C M,TANG Z H,et al.Isolation and characterization of 13 novel polymorphic microsatellite markers for Pinus koraiensis (Pinaceae)[J].Am J Bot,2012,99(10):e421-e424.DOI: 10.3732/ajb.1200145.
[27]LI X,LIU X T,WEI J T,et al.Development and transferability of EST-SSR markers for Pinus koraiensis from cold-stressed transcriptome through illumina sequencing[J].Genes,2020,11(5):500.DOI: 10.3390/genes11050500.
[28]倪州献,白天道,蔡恒,等.马尾松基因组SSR标记在松属其他树种中的通用性分析[J].分子植物育种,2015,13(12):2811-2817.NI Z X,BAI T D,CAI H,et al.The transferability of Pinus massoniana SSR in other Pinus species[J].Mol Plant Breed,2015,13(12):2811-2817.DOI: 10.13271/j.mpb.013.002811.
[29]DOU J J,ZHOU R C,TANG A J,et al.Development and characterization of nine microsatellites for an endangered tree,Pinus wangii (Pinaceae)[J].Appl Plant Sci,2013,1(2):apps.1200134.DOI: 10.3732/apps.1200134.
[30]何启平,陈莹.校园常见植物叶绿素提取方法比较及其含量测定[J].黑龙江农业科学,2015,38(10):117-120.HE Q P,CHEN Y.Comparision on different extaction techniques about chlorophyll and determanation of chlorophyll content of common plants in campus[J].Heilongjiang Agric Sci, 2015,38(10):117-120.DOI: 10.11942/j.issn1002-2767.2015.10.0117.
[31]樊文强,盖红梅,孙鑫,等.SSR数据格式转换软件DataFormater[J].分子植物育种,2016,14(1):265-270.FAN W Q,GAI H M,SUN X,et al.Data formater,a software for SSR data formatting to develop population genetics analysis[J].Mol Plant Breed,2016,14(1):265-270.DOI: 10.13271/j.mpb.014.000265.
[32]LIU K J,MUSE S V.Power marker:an integrated analysis environment for genetic marker analysis[J].Bioinformatics,2005,21(9):2128-2129.DOI: 10.1093/bioinformatics/bti282.
[33]PEAKALL R,SMOUSE P E.GenAlEx 6.5:genetic analysis in Excel.Population genetic software for teaching and research:an update[J].Bioinformatics,2012,28(19):2537-2539.DOI: 10.1093/bioinformatics/bts460.
[34]TAMURA K,PETERSON D,PETERSON N,et al.MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Mol Biol Evol,2011,28(10):2731-2739.DOI: 10.1093/molbev/msr121.
[35]EVANNO G,REGNAUT S,GOUDET J.Detecting the number of clusters of individuals using the software STRUCTURE:a simulation study[J].Mol Ecol,2005,14(8):2611-2620.DOI: 10.1111/j.1365-294X.2005.02553.x.
[36]徐存宝,刘滨凡,刘维斌.天然红松林结构规律的探讨[J].林业科技,1991,16(4):17-19.XU C B,LIU B F,LIU W B.Discussion on structural law of natural Korean pine forest[J].For Sci Technol,1991,16(4):17-19.
[37]郭文丽,李义良,赵奋成,等.湿加松无性系表型遗传多样性研究[J].植物研究,2019,39(2):259-266.GUO W L,LI Y L,ZHAO F C,et al.Phenotypic genetic diversity of Pinus elliottii × P.caribaea Morelet var. hondurensis clones[J].Bull Bot Res,2019,39(2):259-266.DOI: 10.7525/j.issn.1673-5102.2019.02.012.
[38]陈存,丁昌俊,黄秦军,等.美洲黑杨表型核心种质库构建[J].林业科学研究,2021,34(2):1-11.CHEN C,DING C J,HUANG Q J,et al.Construction of phenotypic core collection of Populus deltoides[J].For Res,2021,34(2):1-11.DOI: 10.13275/j.cnki.lykxyj.2021.02.001.
[39]贾丙瑞,周广胜,刘永志,等.中国天然林凋落物量的空间分布及其影响因子分析[J] .中国科学:生命科学,2016,46(11):1304-1311. JIA B R,ZHOU G S,LIU Y Z,et al.Spatial pattern and environmental controls of annual litterfall production in natural forest ecosystems in China [J] .Sci Sin Vit,2016,46(11):1304-1311. DOI:10.1360/N052015-00319.
[40]徐海明.种质资源核心库构建方法的研究及其应用[D].杭州:浙江大学,2005.XU H M.Study on methods of constructing core collection of germplasm and their applications in core construction[D].Hangzhou:Zhejiang University,2005.
[41]HU J,ZHU J,XU H M.Methods of constructing core collections by stepwise clustering with three sampling strategies based on the genotypic values of crops[J].Theor Appl Genet,2000,101(1):264-268.DOI: 10.1007/s001220051478.
[42]冯富娟,隋心,张冬东.不同种源红松遗传多样性的研究[J].林业科技,2008,33(1):1-4.FENG F J,SUI X,ZHANG D D.Studies on the genetic diversity of Pinus koraiensis in different provenance[J].For Sci Technol,2008,33(1):1-4.DOI: 10.3969/j.issn.1001-9499.2008.01.001.
[43]张亚红,贾会霞,王志彬,等.滇杨种群遗传多样性与遗传结构[J].生物多样性,2019,27(4):355-365.ZHANG Y H,JIA H X,WANG Z B,et al.Genetic diversity and population structure of Populus yunnanensis[J].Biodivers Sci,2019,27(4):355-365.DOI: 10.17520/biods.2019016.
[44]CHEN S Y,ZHAO W S,WANG J.Genetic diversity and genetic differentiation of natural populations of Pinus kesiya var. langbinanensis[J].J For Res,2002,13(4):273-276.DOI: 10.1007/BF02860090.
[45]邵丹,裴赢,张恒庆.凉水国家自然保护区天然红松种群遗传多样性在时间尺度上变化的cpSSR分析[J].植物研究,2007,27(4):473-477.SHAO D,PEI Y,ZHANG H Q.cpSSR analysis of variation of genetic diversity in temporal dimension of natural population of Pinus koraiensis in Liangshui National Nature Reserve[J].Bull Bot Res,2007,27(4):473-477.DOI: 10.3969/j.issn.1673-5102.2007.04.020.
[46]李斌,顾万春,卢宝明.白皮松天然群体种实性状表型多样性研究[J].生物多样性,2002,10(2):181-188.LI B,GU W C,LU B M.A study on phenotypic diversity of seeds and cones characteristics in Pinus bungeana[J].Biodivers Sci,2002,10(2):181-188.DOI: 10.3321/j.issn:1005-0094.2002.02.008.
[47]LOVELESS M D,HAMRICK J L.Ecological determinants of genetic structure in plant populations[J].Annu Rev Ecol Syst,1984,15:65-95.DOI: 10.1146/annurev.es.15.110184.000433.
[48]BROWN A H D.Plant population genetics,breeding,and genetic resources[M].Sunderland Mass:Sinauer Associates,1990.
[49]FRANKHAM" R,BALLOU" J D,BRISCOE" D A,et al.Introduction to conservation genetics:the broader context:population viability analysis (PVA)[M].Cambridge:Cambridge University Press,2002.
[50]陈向向,盖中帅,翟军团,等.中国西北地区天然胡杨群体遗传多样性及核心保护单元的构建[J].生物多样性,2021,29(12):1638-1649.CHEN X X,GAI Z S,ZHAI J T,et al.Genetic diversity and construction of core conservation units of the natural populations of Populus euphratica in northwest China[J].Biodivers Sci,2021,29(12):1638-1649.DOI: 10.17520/biods.2021249.
[51]陈存,丁昌俊,张静,等.美洲黑杨群体结构分析及核心种质库构建[J].林业科学,2020,56(9):67-76.CHEN C,DING C J,ZHANG J,et al.Population structure analysis and core collection construction of Populus deltoides[J].Sci Silvae Sin,2020,56(9):67-76.DOI: 10.11707/j.1001-7488.20200908.
[52]徐益,张列梅,郭艳春,等.黄麻核心种质的遴选[J].作物学报,2019,45(11):1672-1681.XU Y,ZHANG L M,GUO Y C, et al.Core collection screening of a germplasm population in jute(Corchorus spp.)[J].Acta Agron Sin,2019,45(11):1672-1681.DOI: 10.3724/SP.J.1006.2019.94008.
[53]BELAJ A,DEL CARMEN DOMINGUEZ-GARCA M,ATIENZA S G,et al.Developing a core collection of olive (Olea europaea L.) based on molecular markers (DArTs,SSRs,SNPs) and agronomic traits[J].Tree Genet Genomes,2012,8(2):365-378.DOI: 10.1007/s11295-011-0447-6.
[54]ZHANG Y X,ZHANG X R,CHE Z,et al.Genetic diversity assessment of sesame core collection in China by phenotype and molecular markers and extraction of a mini-core collection[J].BMC Genet,2012,13:102.DOI: 10.1186/1471-2156-13-102.
[55]DZIALUK A,CHYBICKI I,GOUT R,et al.No reduction in genetic diversity of Swiss stone pine (Pinus cembra L.) in Tatra Mountains despite high fragmentation and small population size[J].Conserv Genet,2014,15(6):1433-1445.DOI: 10.1007/s10592-014-0628-6.
[56]武星彤,陈璐,王敏求,等.丹霞梧桐群体遗传结构及其遗传分化[J].生物多样性,2018,26(11):1168-1179.WU X T,CHEN L,WANG M Q,et al.Population structure and genetic divergence in Firmiana danxiaensis[J].Biodivers Sci,2018,26(11):1168-1179.DOI: 10.17520/biods.2018223.
[57]TIJERINO A,KORPELAINEN H.Molecular characterization of Nicaraguan Pinus tecunumanii Schw.ex Eguiluz et Perry populations for in situ conservation[J].Trees,2014,28(4):1249-1253.DOI: 10.1007/s00468-014-1005-2.
[58]吕锋,解孝满,韩彪,等.基于SSR标记的麻栎天然群体遗传多样性分析[J].南京林业大学学报(自然科学版),2022,46(3):109-116.LYU F,XIE X M,HAN B,et al.Genetic diversity analyses of Quercus acutissima based on SSR markers[J].J Nanjing For Univ (Nat Sci Ed),2022,46(3):109-116.DOI: 10.12302/j.issn.1000-2006.202101025.
(责任编辑 郑琰燚)
基金项目:中央高校基本科研业务费专项资金项目 (2572022AW13);林木遗传育种全国重点实验室(东北林业大学)创新项目(2022A02)。
第一作者:闫平玉(1321830425@qq.com),博士生。
*通信作者:张含国(hanguozhang1@sina.com),教授。

