基于SSR分子标记的睡莲遗传多样性分析


摘要:【目的】睡莲(Nymphaea spp.)是重要的水生花卉。随着睡莲产业的迅速发展,因引种管理不规范,众多新种质引进后未能及时有效辨析其遗传背景,在投入生产使用时常出现种苗命名混乱、亲本来源不清晰等问题,不利于睡莲种质资源的创新与利用。开发睡莲全基因组水平上的SSR分子标记,用于该属植物的亲缘关系鉴定和遗传多样性分析,为睡莲种质资源的保护与育种提供理论依据。【方法】以公布的睡莲全基因组序列为参考,采用微卫星识别工具(MISA)分析睡莲14条染色体基因序列的SSR位点,用Primer 3.0设计150对SSR引物;选取5份原种/品种对150对引物进行PCR扩增;分别采用琼脂糖和变性聚丙烯酰胺凝胶电泳筛选多态性好的SSR引物;对筛选的SSR引物进行荧光引物(FAM、HEX)合成,然后对147份睡莲样品进行PCR扩增;采用毛细管电泳检测扩增产物,利用GeneMarker进行原始数据读取,并将每个样品在各个等位基因位点的片段大小统计成0/1矩阵,采用Popgenen、NTSYS进行遗传多样性指数计算、聚类分析和主成分分析。【结果】由琼脂糖凝胶和聚丙烯酰胺凝胶电泳结果可知,筛选出11对扩增条带清晰、多态性高的SSR引物;筛选引物被用于147份睡莲种质的遗传多样性分析,扩增产物的毛细管电泳检测结果显示,共检测出307个等位基因位点,经生物信息学软件计算11对引物的等位基因数变幅为4~7,平均值5.36;多态性指数(PIC)变幅为0.46~0.60,平均值0.53;有效等位基因数(Ne)为1.042 8~1.117 5,平均值1.071 8;Neis基因多样性指数(H)为0.038 0~0.086 2,平均值0.056 2;Shannon多样性信息指数(I)为0.085 6~0.163 8,平均值为0.114 4。聚类分析表明147份睡莲属植物的遗传相似系数为0.781 8~0.993 5,平均值为0.899 2;在遗传相似系数0.879 0时将147份睡莲属植物划分为6个类群。PCoA分析表明,147份睡莲样品的主坐标分析结果与形态学分类结果分类相似,第1~3主坐标分别占总遗传变异的16.54%、8.35%和5.43%,共占总遗传变异的30.32%,基于供试种质在主坐标三维图的发散状态可知,第1主坐标与睡莲种质的香气形成和雄蕊发育变异有关,第2主坐标与种质的开花时间和环境适应性相关,而第3主坐标主导花型变异。【结论】SSR荧光标记作为一种以DNA多态性为检测目标的遗传多样分析手段,已广泛应用于植物学系统分类和遗传多样性研究。本研究筛选出的11对SSR引物多态性较好,能有效鉴定出147份睡莲种质的亲缘关系,在系统分类上可将147份种质划分为6大一级分支,分类结果与形态学分类结果相似。筛选的11对SSR引物能够应用于睡莲属植物的遗传多样性分析和品种鉴定。SSR标记结果可为睡莲属植物的种质收集保存、种质创新和保护开发提供科学依据。
关键词:睡莲;SSR分子标记;遗传多样性;毛细管电泳
中图分类号:S718;S682.32"""" 文献标志码:A开放科学(资源服务)标识码(OSID):
文章编号:1000-2006(2024)05-0057-12
Analyzing the genetic diversity" of" Nymphaea" spp." based on SSR markers
MAO Liyan1, LI Huimin1, LONG Lingyun1*, HUANG Qiuwei1, TANG Yuwei1, YU Yanping1, HUANG Xinyi1, TAN Xiaohui1, NONG Xiaohui2, ZHU Tianlong3, LU Zushuang1
(1. Guangxi Subtropical Crops Research Institute, Guangxi Key laboratory of Quality and Safty Control for Subtropical Fruits, Nanning""" 530001, China; 2. Guangxi Pingguo Hualian Science and Technology Research Institute, Baise""" 531499, China;3. Hainan Fodu Lianyuan Ecological Agriculture Co., Ltd.,Haikou"" 570100, China)
Abstract: 【Objective】Nymphaea spp. (waterlily) are important aquatic flowers. The rapid" development of the waterlily industry has led to challenges in identifying"" genetic backgrounds of newly introduced germplasms in a timely manner" due to inadequate introduction management practices. This has resulted in issues such as mislabeling of seedlings and unclear parentage, hampering the effective utilization and innovation of waterlily germplasm resources. This study focuses on developing genome-wide SSR markers for conducting phylogenetic and genetic diversity analyses of waterlily species. These markers are intended to serve as a theoretical foundation for conserving and breeding waterlily germplasm resources.【Method】Based on the published complete genome sequence of waterlily, the SSR loci in 14 chromosome genes were analyzed using the micro satellite identification" (MISA) tool. Subsequently, 150 pairs of SSR primers were designed with the assistance of Primer 3.0 software. Five native germplasms were chosen for PCR amplification utilizing the 150 primer pairs. Following PCR, SSR primers demonstrating high polymorphism were identified through agarose and denaturing polyacrylamide gel electrophoresis. The selected SSR primers were synthesized with fluorescent primers (FAM and HEX) before amplifying 147 waterlily samples via PCR. The resulting products were then assessed using capillary electrophoresis, and the raw data were analyzed using GeneMarker software. Fragment sizes at each allele site for every sample were compiled into a 0/1 matrix. Genetic diversity indices, cluster analysis, and principal component analysis were computed using Popgenen and NTSYS software toos.【Result】11 pairs of SSR primers exhibiting distinct bands and high polymorphism were carefully chosen following the analysis of agarose gel and polyacrylamide gel electrophoresis results. These selected primers were employed to evaluate the genetic diversity among 147 waterlilies. Capillary electrophoresis revealed the presence of 307 alleles. The polymorphism index (PIC) ranged from 0.46 to 0.60, average at 0.53. The effective allele number (Ne) rangedfrom 1.042 8 to 1.117 5, with an average of 1.071 8. Neis gene diversity index (H) ranged from 0.038 0 to 0.086 2, averaging at 0.056 2. The Shannon diversity information index (I) ranged from 0.085 6 to 0.163 8, averaging at 0.114 4. Cluster analysis indicated genetic similarity coefficients ranging" from 0.781 8 to 0.993 5 among the 147 waterlilies, with an average coefficiency of 0.899 2. These waterlilies were classified into six groups based on a genetic" similarity coefficient was 0.879 0. Principal coordinates analysis (PCoA) displayed results closely aligned with traditional morphological classification for the 147 waterlilies. The first, second and third principal coordinates accounted for 16.54%, 8.35% and 5.43% of the total genetic variation, respectively, comprising 30.32% altogether. The first coordinate was" correlated with aroma formation and stamen development, the second with flowering time and environmental adaptability, and the third with flower types.【Conclusion】SSR fluorescent markers, aimed at DNA polymorphism analysis, have been extensively utilized in the botanical system classification and genetic diversity research. In this study, 11 pairs of SSR primers displaying substantial polymorphism were chosen, demonstrating efficacy in distinguishing the genetic relationships among 147 waterlilies. These waterlilies were systematically categorized into six primary branches, with classification outcomes mirroring traditional morphology categorizations. The selected 11 pairs of SSR primers have potential utility in analyzing genetic diversity and identifying waterlily species. The SSR markers findings stand poised to offer a robust scientific foundation for germplasm collection, preservation, innovation, and the development of waterlily species.
Keywords:Nymphaea spp. (waterlily);SSR molecular marker;genetic diversity;capillary electrophoresis
睡莲是睡莲科(Nymphaeaceae)睡莲属(Nymphaea)多年生浮叶草本水生植物,主要分布于热带、亚热带地区,少数延伸至温带地区,全球现有种/变种50多个、园艺品种1 000多个,我国现引种驯化、保存的原种有30多个,经多年杂交培育的国内睡莲园艺品种已达400多个[1]。睡莲是兼具食、赏、药多用途的水生花卉,可用于园林景观建设、湿地水体绿化和净化[2],亦可作为重要原料用于化妆品、香皂等日化产品、食品和医药生产[3-4],具有较好的市场发展前景。睡莲属植物生长迅速、易于繁殖、花色多样、花香浓郁,且基因组较小、位于现存被子植物系统发育树基部,是理解被子植物起源演化的关键一环,开展睡莲种质资源遗传多样性分析对研究睡莲的起源、演化和资源开发利用具有重要意义[3,5-6]。
睡莲属植物按照植物学和生态学分类分为5个亚属,分别为:南非睡莲亚属(Subgen. Castalia 或 Nymphaea)、缺柱亚属(Subgen. Anecphya)、短柱亚属(Subgen. Brachyceras)、带柱亚属(Subgen. Lotos)和棒柱亚属(Subgen. Hydrocallis),与生态型对应分别为耐寒睡莲亚属、澳洲睡莲亚属、广热带睡莲亚属、古热带睡莲亚属和新热带睡莲亚属,后4者又被统称为热带睡莲,其中古热带睡莲亚属和新热带睡莲亚属均为夜开型睡莲[1,7]。虽然已有较多基于形态学[8-9]、同工酶种类[10]、代谢物种类[11]等手段探究睡莲属内种间系统发育关系的研究报道,但这些研究因睡莲属种间杂交现象频繁、表型形态差异较大、同工酶或代谢物信息量普遍不足、研究取样不完整的原因,导致该属内近缘种或杂种之间的关系仍不能明确,制约了睡莲属内种间系统发育、品种鉴定和良种选育研究工作的顺利开展。因此,采用不同技术手段,多角度分析睡莲属植物的遗传多样性,明确各亚属物种间亲缘关系,有助于推动解决上述科学问题。
分子标记是一种以DNA多态性为检测目标的遗传多样分析手段,具有不依赖表型性状、稳定性好、标记明确和重复性好等特点,可弥补表型、同工酶等方法无法有效鉴定近似品种的缺点和不足[12],在睡莲属种质资源中已有应用。Péter等[13]借助ISSR分子标记明确了N. ‘Panama Pacific’、N. caerulea等6种/品种睡莲的起源进化关系;Dkhar等[14]分别采用表型性状、RAPD和ITS标记等手段,对不同地域来源的7个睡莲种质进行遗传多样性分析,提出部分原种的起源可能是远缘杂交而来的推论;Qian等[15]从睡莲核基因组和叶绿体基因组中筛选出ITS、trnT-trnF、rpl16等3个标记片段,有效鉴定了158个野生睡莲种质的亚属分类;苏群等[16]通过对46份睡莲种质进行ISSR分子标记分析,研究结果显示所选种质在亚属水平上的亲缘关系分类与植物学分类相近。目前,国内对睡莲种质资源的原种、园艺品种收集和保存工作基本完成,育种家们开展了大量的亚属内和跨亚属的杂交育种工作,先后选育出近200个新品种,种质资源的丰富度进一步加大,如何准确鉴定它们的亲缘关系与进化演变是一个重要课题。本研究基于已完成的睡莲全基因组测序数据设计和筛选SSR分子标记引物,并对广西壮族自治区亚热带作物研究所于海南、广西、浙江、江苏、云南等省/自治区收集和保存的睡莲种质资源进行遗传多样性分析,明确睡莲属原种、园艺品种及其杂交后代种质的亲缘关系,以期为睡莲种质资源的保护利用和遗传育种提供参考。
1 材料与方法
1.1 试验材料
147份睡莲种质样品分别采自广西壮族自治区亚热带作物研究所睡莲种质资源圃、广西平果华莲科技研究所和海南佛渡莲源生态农业有限公司。于2020年8月晴朗天气采集样品,样品编号及名称详见表1,包括112份睡莲种/品种及35份杂交后代。每种样品均采集水下卷曲或漂浮最内圈叶片,采集叶片后用自来水清洗杂质,擦干表面水分放入自封袋,经硅胶干燥后进行后续试验。
1.2 试验方法
1.2.1 DNA提取
采用CTAB法[17]提取睡莲基因组DNA,并往提取体系中加入磁珠,沉降DNA的乙醇体积分数为80%,配以磁力架进行改良。采用琼脂糖电泳和NanoDrop 2000检测 DNA 的质量和浓度,并统一稀释DNA质量浓度至50 ng/μL,-20 ℃保存备用。
1.2.2 引物设计及引物筛选
根据福建农林大学已发表的睡莲基因组[6]数据,采用微卫星识别(MISA)软件(http://pgrc. ipk-gatersleben. de/misa/)搜索睡莲14条染色体序列基因组的SSR位点,设置SSR检索标准:检索重复单元为 2、3、4、5、6 bp,重复长度≥12 bp;二核苷酸最小重复次数≥6 次(总长度不少于 12 个碱基),三至六核苷酸最小重复次数≥5 次(总长度不少于 15、20、25 和 30 bp)。采用 Primer 3.0 引物批量设计程序对二至六核苷酸重复类型以及复合的 SSR 位点两端序列设计特异性引物,目的产物大小为100~350 bp,经华大基因合成后,分别从5个亚属的种/品种中各选1个样品[分别为:蓝星睡莲(N. colorata)、‘白巨’睡莲(N. gigantea‘Albert de Lestang’)、鲁吉娜睡莲(N. rudgeana)、印度红睡莲(N. rubra)、白睡莲(N. alba)]进行PCR扩增。PCR反应体系(20.0 μL)为ddH2O 14.8 μL,dNTP 0.4 μL,Buffer 2 μL。正反向引物各0.3 μL(2×10-5 mol/L),DNA模板2 μL,Taq 酶0.2 μL。反应程序为94" ℃预变性5 min;94 ℃变性30 s,60~50" ℃(每个循环降低1" ℃,10个循环后50" ℃ 25个循环)复性35 s,72" ℃延伸40 s,共 35个循环;最终72" ℃延伸3 min。扩增产物采用质量分数1.5%琼脂糖电泳进行初步检测,从中选取扩增质量好的SSR引物扩增产物进行进一步的质量分数6%变性聚丙烯酰胺凝胶电泳检测。
1.2.3 荧光引物PCR扩增及毛细管电泳检测
将筛选得到的扩增质量好、多态性高的引物在华大基因有限公司进行荧光引物(FAM、HEX)合成后,进行147份睡莲样品的PCR扩增,扩增体系与反应程序和引物筛选实验一致。扩增产物采用ABI 3730X测序仪(赛默飞世尔,美国)进行毛细管电泳检测。
1.3 数据分析
采用GeneMarker 2.2.0进行原始数据分析,得到初步处理的SSR分子标记数据后,以1个引物为1个等位基因位点,将每个样品在各个等位基因位点的片段大小统计成0/1矩阵,根据0/1矩阵采用Popgene 1.32进行遗传多样性指标计算[平均等位基因数(Na),有效等位基因数(Ne),Neis基因多样性指数(H),Shannons信息指数(I),多态性指数PIC]。采用NTSYS 2.0软件计算遗传相似系数,采用UPGMA法进行聚类分析,采用Mxcomp程序进行遗传相似系数的主坐标分析(PCoA),构建2、3维空间的散点分布图以检验聚类结果。
2 结果与分析
2.1 SSR引物筛选与扩增多态性
2.1.1 SSR引物筛选结果
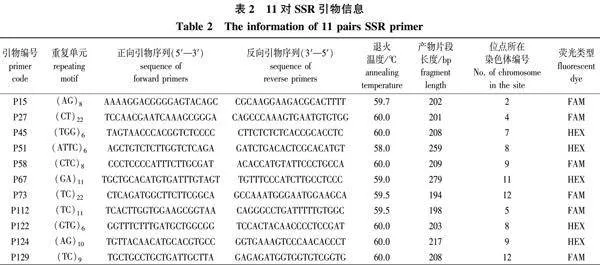
本研究共选取均匀分布于14条染色体的150对SSR引物,对5个种/品种睡莲进行PCR扩增,采用15 g/L的琼脂糖凝胶进行初步筛选,从中筛选出19对扩增质量好的引物(编号依次为P9、P10、P15、P23、P27、P45、P51、P54、P55、P58、P60、P67、P68、P73、P112、P122、P124、P129、P134),采用筛选出的19对引物的扩增产物进一步进行60 g/L的变性聚丙烯酰胺凝胶电泳检测(图1),从中筛选出了11对主带明显、杂带少、背景清晰、扩增条带多、多态性好、条带差异明显的引物,分别位于2、4、5、7、8、9、11、12号染色体上,11对引物的扩增产物大小为194~279 bp(表2)。对筛选出的11对引物进行荧光标记后进行后续147份样品的扩增,标记类型如表2所示。
P65—P79. 引物编号 the No. of primers;35、97、125、128、119. 样品编号 the No. of samples。
2.1.2 扩增多态性分析
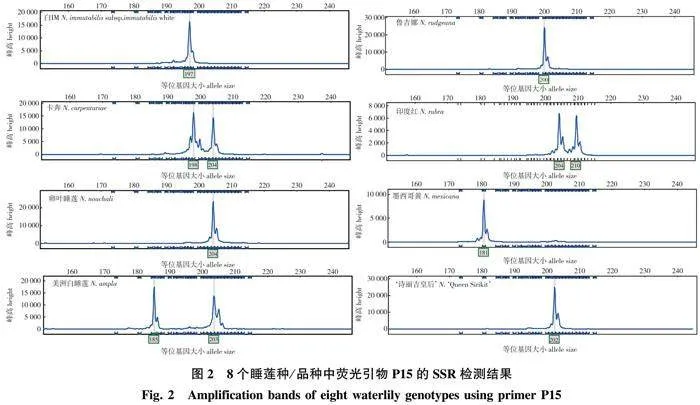
利用11对引物的毛细管电泳结果峰图(其中引物P15对8份随机睡莲样品的毛细管电泳结果峰图见图2),构建147份样品的0/1矩阵,根据0/1矩阵计算遗传多样性指数(表3)可知,11对引物在147个样品中共扩增出307个多态性等位基因位点。软件分析可知,Na变幅为4~7,平均值为5.36,引物P124位点最多为7个,P15和P45位点最少为4个;Ne变幅为1.042 8~1.117 5,平均值为1.071 8;H变幅为0.038 0~0.086 2,平均值为0.056 2;I变幅为0.085 6~0.163 8,平均值为0.114 4;PIC变幅为0.46~0.60,平均值为0.53,其中7 个位点(P15、P27、P45、P67、P112、P124、P129)具有高多态性(PIC>0.50),其余4个位点(P51、P58、P73、P122)具有中等多态性PIC为(0.25,0.50][18]。11对引物中P112的多态性最好,其Ne、H、I、PIC均为11对引物中最高,分别为1.117 5、0.086 2、0.163 8、0.60(表3)。
2.2 聚类分析
2.2.1 147份睡莲样品的聚类结果分析
依据11对SSR引物的多态性构建0/1矩阵计算147份样品的遗传相似系数,据此进行UPGMA法聚类,聚类结果如图3所示。经计算,147份样品的遗传相似系数值范围为0.781 8~0.993 5,平均为0.899 2;其中‘赤子心’(7)和‘锦黄’(82)之间、杂交后代10和11之间的遗传相似系数最大,为0.993 5,亲缘关系最近;‘白巨’(97)和‘科曼契’(110)之间的遗传相似系数最小,为0.781 8,亲缘关系最远。
在遗传相似系数为0.879 0时将147份睡莲样品聚为6个分支,其中越南延药(143)(Ⅱ)和‘科罗拉多’(70)(Ⅲ)分别单独聚为1支。Anecphya亚属的6个种、2个品种和Anecphya亚属×Brachyceras亚属的2个品种遗传相似系数为0.820 8~0.964 2,Anecphya亚属×Brachyceras亚属2个品种遗传相似系数为0.915 3,与Anecphya亚属之间的遗传相似系数为0.820 8~0.902 3,被聚为第Ⅳ[(粉巨(95)、‘卡奔’(96)、‘白巨’(97)]、Ⅴ分支(3个原种:紫IM(100)、白IM(103)和变色澳洲(141),2个品种红IM(99)和‘流苏’(104),Anecphya亚属×Brachyceras亚属2个品种:‘凯斯隆’(101)和‘Tuonta’(102)。Brachyceras亚属的17个原种、53个品种、28个杂交后代(遗传相似系数为0.846 9~0.993 5),1—147. 样品编号 the No. of 147 waterlilies;Ⅰ—Ⅵ. 147份睡莲的聚类编号 the cluster No. of 147 waterlilies;ⅰ、ⅱ. Ⅰ分支的2个聚类编号" the cluster No. of Ⅰ;A—E. ⅰ类的5个聚类编号 the cluster No. of ⅰ;a—c. ⅱ类的聚类编号 the cluster No. of ⅱ;b1、b2. b小支的聚类编号 the cluster No. of b。
全部的Lotos亚属(遗传相似系数为0.912 1~0.990 2)、 Hydrocallis亚属(遗传相似系数为0.899 0~0.941 4)和Nymphaea亚属3个原种、9个品种、Nymphaea亚属×Brachyceras亚属3个品种(遗传相似系数为0.931 6~0.954 4,与Nymphaea亚属的遗传相似系数为0.840 4~0.941 4)、Brachyceras亚属×Anecphya亚属的1个杂交后代(与Brachyceras亚属的遗传相似系数为0.876 2~0.947 9)聚为第Ⅰ分支。Nymphaea亚属1个原种[白睡莲(119)]、4个品种聚为第Ⅵ分支。其中Nymphaea亚属4个原种、14个品种遗传相似系数为0.820 8~0.960 9。
在遗传相似系数0.885 0时将第Ⅰ分支进一步分为两大类:第ⅰ大类由除越南延药(143)外的Brachyceras亚属、Lotos亚属、Hydrocallis亚属及Brachyceras亚属×Anecphya亚属的1个杂交后代组成,第ⅱ大类由除Ⅲ、Ⅵ分支的Nymphaea亚属和Nymphaea亚属×Brachyceras亚属的品种组成。第ⅰ大类在遗传相似系数0.914 0时分为5个小类,其中‘仲克尼’(80)(C)、增殖睡莲(87)(D)及美洲白睡莲(147)(B)分别单独聚为1个小类;Hydrocallis亚属的3个原种和Brachyceras亚属的15个原种、53个品种、28个杂交后代和Brachyceras亚属×Anecphya亚属的1个杂交后聚为第A小类(在遗传相似系数为0.914 5时,Hydrocallis亚属的3个原种聚为1个小分支;Brachyceras亚属的15个原种、53个品种、28个杂交后代,Brachyceras亚属×Anecphya亚属的1个杂交后代聚为另一个小分支),全部的Lotos亚属聚为第E小类。第ⅱ大类在遗传相似系数0.91时分为3个小类,其中第a小类由Nymphaea亚属的原种墨西哥黄(114)及品种‘万维莎’(105)、‘黄西瓜’(109)、‘黄乔伊’(120)组成,第b小类由Nymphaea亚属6个品种和Nymphaea亚属×Brachyceras亚属的3个品种组成(在遗传相似系数为0.93时由Nymphaea亚属6个品种聚为b1小支,Nymphaea亚属×Brachyceras亚属的3个杂交种聚为b2小支),第c小类由原种小白子午莲(116)和雪白睡莲(117)组成。
2.2.2 35份原种和35份杂交后代的聚类分析
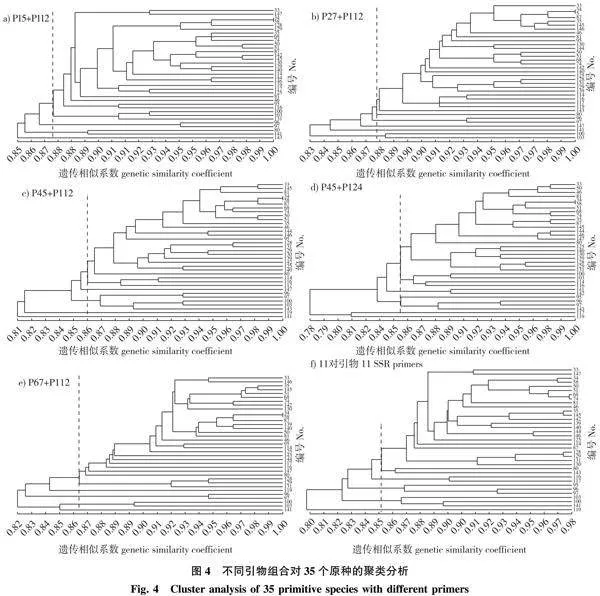
11对引物中未发现能将35个原种完全分开的引物,将11对引物进行两两组合对35个原种进行聚类分析,结果(图4)可知,共获得5个能够将35个原种完全分开的引物组合。根据5个引物组合及11对引物分别在35个原种中产生的多态性位点计算得到遗传相似系数分别是引物P15+P112为0.785 7~1.000 0,P27+P112为0.779 7~1.000 0,P45+P112为0.7091~1.000 0,P45+P124为0.708 3~1.000 0,P67+P112为0.728 8~1.000 0;11对引物为0.751 0~0.979 3。分别在遗传相似系数0.882 0、0.877 5、0.886 0、0.854 5、0.864 0、0.852 5时将其聚为5个分支。5个引物组合和11对引物聚类一致的样品有:33(红叶美洲白睡莲)、34(卵叶睡莲)、35(蓝星睡莲)、46(海南延药)、50(非洲延药)、51(‘蓝蟹爪’)、68(‘粉色好望角’)、74(蓝色好望角)、81(小花)、87(增殖睡莲)、114(墨西哥黄),96(卡奔)、97(‘白巨’),100(紫IM)、103(白IM),125(鲁吉娜)、128(印度红)、129(‘泰国粉’)、130(喀麦隆),139(小雪夜)、140(秘鲁睡莲),
144(印度延药)、145(优雅睡莲)、146(埃及蓝),不同引物对组合的数据聚类方式的聚类结果与形态学分类存在差异,如114(墨西哥黄)、116(小白子午莲)、117(雪白睡莲)、119(白睡莲)在形态学上均属于Nymphaea亚属,87(增殖睡莲)、125(鲁吉娜)、139(小雪夜)、140(秘鲁睡莲)在形态学上均属于Hydrocallis亚属,在本研究中并未能将其很好地聚在一起。
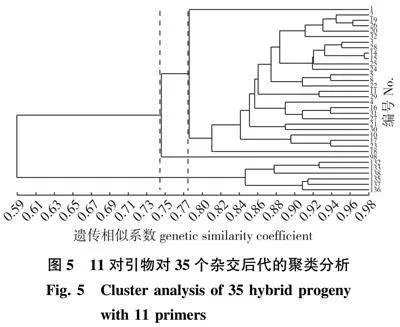
11对引物中不论是单独或者两两组合均不能将35份杂交后代分开(图5),35个杂交后代中的遗传相似系数为0.544 3~0.962 0,分别于0.741 5和0.767 5时将35个杂交后代聚为2类(Brachyceras亚属的28个杂交后代和Brachyceras亚属×Anecphya亚属的1个杂交后代聚为1类,Lotos亚属的6个杂交后代聚为1类)和3类(Brachyceras亚属的28个杂交后代聚为1小支,Brachyceras亚属×Anecphya亚属的1个杂交后代单独聚为1小支,Lotos亚属的6个杂交后代聚为1小支),其聚类结果与实际的杂交结果一致。
2.3 主坐标分析
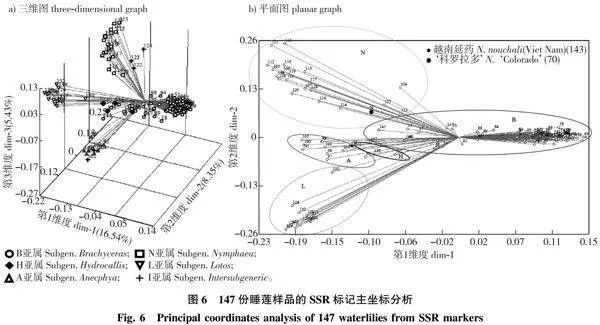
为更好地反映147份睡莲样品的亲缘关系,基于遗传相似矩阵进行了主坐标分析,形成147份睡莲样品的位置分布图(图6),位置相近表示亲缘关系密切,位置距离远表示亲缘关系远[19]。经计算,1~3主坐标分别占比总遗传变异的16.54%、8.35%、5.43%,共占比总遗传变异的30.32%。由图6可知,Brachyceras亚属的多数园艺品种和原种均向轴1的右侧发散,只有少数原种略向左发散(如美洲白睡莲、优雅睡莲等),而Brachyceras亚属是睡莲属中独具芳香和三体雄蕊[7]的睡莲类型,因此推测轴1可能与睡莲种质的香气形成和雄蕊发育变异有关;夜开型的Lotos亚属和Hydrocallis亚属向轴2的下方发散,其他类型睡莲在轴2均向上发散,且Nymphaea亚属睡莲发散得最远,因此轴2可能与睡莲种质的开花时间和环境适应性相关;轴3向下方发散的种质均为Anecphya亚属,其花型与荷花类似,其他亚属均向上发散,因此推测轴3主导着睡莲花型变异。
借助主坐标分析三维图(图6a)可知,147份睡莲样品的主坐标分析结果与传统形态学分类相似。Anecphya、Nymphaea和Lotos亚属种质均各自偏向于同一方向的空间维度上。绝大多数的Brachyceras亚属亦偏向于同一方向的空间维度上,仅有越南延药(143)等少数Brachyceras亚属种质零散分布。Intersubgeneric亚属是跨亚属品种质,其中‘诗丽吉皇后’(122)、‘暹罗粉’1(123)和‘暹罗紫’1(124)偏向于其Nymphaea亚属杂交母本所处的空间维度,‘凯斯隆’(101)和N.‘Tuonta’(102) 偏向于其Anecphya亚属杂交母本所处的空间维度,这与分子聚类结果相同。进一步结合分子聚类结果可知,第Ⅱ分支的延药睡莲处偏向于三维空间中间平面,与第Ⅰ分支中的第Ⅰ分类的白蓝星(142)和优雅睡莲(145)更加接近,第Ⅲ分支的‘科罗拉多’也处于三维空间近中间的平面,但更加偏向于与第1分支Ⅱ分支的墨西哥黄(114)和小白子午莲(116),这表明在亲缘关系上延药睡莲与第Ⅰ分支的第Ⅰ分类种质更近,‘科罗拉多’与第Ⅰ分支的Ⅱ分类种质的亲缘关系更近。
在平面图中,分子聚类结果中的第I分支包含的Lotos亚属种质位于平面的左下角,Anecphya亚属及两个Anecphya亚属为母本的跨亚属杂交种质,位于近水平中线偏左下的位置。Nymphaea亚属及3个Nymphaea亚属为母本的跨亚属都偏向于左上角。大部分的Brachyceras亚属均位于水平线及两侧偏右位置,Hydrocallis亚属种质与Anecphya亚属种质在二维图的偏向性相似。
3 讨 论
3.1 睡莲SSR分子标记引物筛选及遗传多样性
遗传多样性研究对种质资源的保护、遗传改良、杂交育种以及创新利用具有重要意义[20],SSR标记具备扩增谱带少、多态性高、操作简单、易于识别和统计、可快速鉴定品种等优点[17,21],广泛应用于多种作物的遗传多样性分析。其中,SSR荧光标记比普通SSR标记方法的鉴定效率更高、结果更准确[22],已被应用于睡莲属种质资源的遗传多样性研究,如苏群等[23]利用16对引物在240个睡莲品种中检测到205个等位基因;Parveen等[24]利用57对SSR引物在92个N. nouchali个体中检测到277个等位基因。本研究采用荧光标记多态性引物对147份睡莲样品的遗传多样性进行分析,筛选到11对SSR引物,共扩增出307个多态性等位基因位点,这11对引物分布于睡莲全基因组的8条染色体上,大多为2碱基重复引物。研究表明,物种的SSR位点多态性区别在其重复单元的重复次数不同,2碱基重复频率最高,更容易获得较高的SSR多态性[17],表明本研究所筛选的SSR引物可较多获得睡莲属植物的遗传多态性信息。
本研究选用的睡莲种质样品以人工选育的园艺品种(77份)和杂交后代(35份)为主,原种仅为35份。前人研究表明,高强度的人工选择会降低群体遗传多样性[25-27],这可能是造成本研究结果中PIC、Ne、H和I值低于苏群等[23]和Parveen等[24]开展的睡莲种质SSR标记分析结果的重要原因。但试验所选样品来源于不同地域,材料来源较为复杂,加之绝大多数的睡莲为异花授粉,群体拥有较高的杂合度[23],故本研究筛选的11对引物亦能获得丰富的多态性,可用于鉴定睡莲种质的亲缘关系。
3.2 睡莲样品的聚类分析
当前睡莲种质资源的亚属及种间的分类尚未明确,杂交育种工作常面临着依据传统分类学筛选的亲本之间亲缘关系不清晰、杂交不结实或败育率高等问题[16,28-29],SSR标记聚类结果有助于了解睡莲种质之间的亲缘关系,可为睡莲育种亲本的选择提供科学参考。研究表明,物种种质间的遗传相似系数值越大,说明亲缘关系越近;反之,亲缘关系越远,杂交成功率越低,但获得杂种优势的可能性越大[23-24]。本研究通过分析147份睡莲种质亲缘关系发现,第Ⅰ分支分类与传统的形态学分类结果相似,第ⅰ大类由Brachyceras、Lotos和Hydrocallis亚属组成,第ⅱ类由部分Nymphaea亚属组成,其中Brachyceras、Lotos和Hydrocallis亚属按生态学分类归属于热带睡莲,表明特定生境可能导致这3个亚属物种拥有相似的分子水平进化趋势。第Ⅰ分支第ⅰ大类中作为睡莲属仅有的具备花胎生生殖特性的原种增殖睡莲(87),和唯一具备高重瓣特性的原种仲克尼(80)各单独划分为1个小类,表明这2个原种与其他热带睡莲在遗传特性上存在较大差异,亲缘关系较远,推测这一现象可能与这2个原种的独特遗传性状有关。第ⅱ大类中a小类的样品花瓣均有黄色,而b小类的样品花瓣颜色多为粉、红、紫, c小类的两个原生花瓣均为白色。张旻桓等[27]的研究显示,花色相近或相同的牡丹(Paeonia suffruticosa)品种遗传距离较近,亲缘关系与花色存在一定相关性,推测睡莲种质的种间亲缘关系远近亦与花色基因的进化存在关联。
5份跨亚属品种和1个跨亚属杂交后代的聚类结果均与母本材料聚在一起,这与其表型高度一致,表明在睡莲的杂交育种中,杂交后代更偏向于母本的表型,因此在实际杂交育种工作中需根据杂交目的选择合适的母本以便快速达到杂交目的。
已报道的基于睡莲种质表观形态或同工酶种类聚类的遗传多样性研究均显示,供试Nymphaea亚属种质均聚类为一个分支[7,9-10,16,30-31]。本研究供试的Nymphaea亚属种质中,‘科罗拉多’单独聚为第Ⅲ分支,白睡莲(原种)和4个园艺品种‘马哈巴颂’‘科曼契’‘佛琴娜丽丝’‘霍兰迪亚’聚类为第Ⅵ分支,其余的Nymphaea亚属种质则划归第Ⅰ分支类群。这一结果与苏群等[23]对240份睡莲种质的SSR标记结果一致,推测可能是Nymphaea亚属内部具有丰富的遗传变异,仅以形态学进行分类具有一定局限性,而筛选的SSR引物丰富度不够、覆盖面不广也是影响试验结果的重要因素。本研究供试的Lotos亚属种质遗传相似系数均在0.9以上,在第Ⅰ分支的第Ⅰ类中更接近于进化树的基部,表明该亚属进化地位比Brachyceras亚属种质原始,这一结果符合黄国振等[7]关于睡莲属的形态分类学观点。
主坐标分析(PCoA)与UPGMA均能对种质样品进行聚类分析,前者从不同方向、不同层面更加直观地显示各种质间的关系,而后者对密切关注的种质个体具有较高的分辨率,两种聚类分析方法结合使用,可以更加全面地解释聚类结果[32]。赵亚军等[33]的研究显示,油菜(Brassica napus)的23个自育品种(A群)和39个其他主栽品种(B群)聚类为3类分支,自育品种‘青油14’在分子聚类上位于第Ⅲ类,但无法区分它与聚类结果的第Ⅰ、Ⅱ类种质的亲缘关系远近,借助主坐标分析发现‘青油14’位于二维图自育品种(A群)内、三维图的A群右角,最终确定它与A群遗传关系较近,与其他主栽品种(B群)远。本研究中依据传统的形态学分类越南延药属于Brachyceras亚属,‘科罗拉多’划归Nymphaea亚属,但分子聚类将越南延药单独划为第Ⅱ分支,‘科罗拉多’则单独形成第Ⅲ分支,基于聚类结果无法有效地分辨两者种质与其他分支类群的亲缘关系远近,而主坐标分析恰好弥补这一缺陷,更加全面地反映了睡莲种质之间的亲缘关系。
本研究筛选得到了11对多态性丰富的引物,可用于鉴定睡莲种质的亲缘关系。通过对147份睡莲属种质遗传多样性的分析,进一步验证了SSR标记的高效性及准确性。147份睡莲种质可划分为6个一级分支,与传统形态学分类结果相似,但在二级和三级分支上如Nymphaea亚属种质的分类与传统形态学分类观点存在一定差异,且少部分品种如越南延药、‘科罗拉多’依据形态学分类无法有效地鉴定其与其他种质的亲缘关系。笔者结合主坐标分析进一步明确了越南延药和‘科罗拉多’与二级分支上的种质亲缘关系远近。依据亲缘关系越远,获得杂种优势的可能性越大的理论[34],两者均是潜在的优良育种亲本材料。进一步研究中应结合形态表型数据,加大引物和样品的筛选量,进行多种分子标记联合分析,以筛选出能更精准、快速鉴定睡莲属种质的引物和标记类型。
参考文献(reference):
[1]李淑娟,尉倩,陈尘,等.中国睡莲属植物育种研究进展[J].植物遗传资源学报,2019,20(4):829-835.LI S J,YU Q,CHEN C,et al.Breeding progress of waterlilies in China[J].J Plant Genet Resour,2019,20(4):829-835.DOI: 10.13430/j.cnki.jpgr.20181209001.
[2]DEVI S A,THONGAM B,HANDIQUE P J.Nymphaea rubra Roxb.ex Andrews cultivated as an ornamental,food and vegetable in the northeastern Region of India[J].Genet Resour Crop Evol,2015,62(2):315-320.DOI: 10.1007/s10722-014-0177-3.
[3]CHEN F,LIU X,YU C W,et al.Water lilies as emerging models for Darwins abominable mystery[J].Hortic Res,2017,4:17051.DOI: 10.1038/hortres.2017.51.
[4]KAMBLE M Y,POULOSE V C,SINGH L J.The genus Nymphaea L.(Family Nymphaeaceae) in Andaman and Nicobar Islands,India[J].J Bombay Nat Hist Soc,2019,116:35.DOI: 10.17087/jbnhs/2019/v116/108147.
[5]GROUP T A P.An update of the Angiosperm Phylogeny Group classification for the orders and families of flowering plants:APG IV[J].Bot J Linn Soc,2016,181(1):1-20.DOI: 10.1111/boj.12385.
[6]ZHANG L S,CHEN F,ZHANG X T,et al.The water lily genome and the early evolution of flowering plants[J].Nature,2020,577(7788):79-84.DOI: 10.1038/s41586-019-1852-5.
[7]黄国振,邓惠勤,李祖修,等.睡莲[M].北京:中国林业出版社,2009:1-10.HUANG G Z,DENG H Q,LI Z X,et al.Nymphaea[M].Beijing:China Forestry Publishing House,2009:1-10.
[8]万里波,王奎玲,刘庆华,等.青岛耐寒睡莲品种数量分类研究[C]// 青岛:中国观赏园艺研究进展(2014),2014:133-139.
[9]苏群,杨亚涵,田敏,等.49份睡莲资源表型多样性分析及综合评价[J].西南农业学报,2019,32(11):2670-2681.SU Q,YANG Y H,TIAN M,et al.Phenotypic diversity analysis and comprehensive evaluation of 49 waterlily resources[J].Southwest China J Agric Sci,2019,32(11):2670-2681.DOI: 10.16213/j.cnki.scjas.2019.11.027.
[10]张海平.部分睡莲属植物的形态多样性及同工酶分析[D].南京:南京农业大学,2008.ZHANG H P.Isozyme analyses and investigation on the morphological diversity of taxa in genus Nymphaea[D].Nanjing:Nanjing Agricultural University,2008.
[11]袁茹玉.不同品种睡莲花挥发物组成及其茶汤功能成分和抗氧化活性评价[D].南京:南京农业大学,2014.YUAN R Y.Studies on the composition of volatiles in different cultivars of water lily and functional component and antioxidant activity evaluation in its tea[D].Nanjing:Nanjing Agricultural University,2014.
[12]刘闵豪,肖兴翠,张春花,等.基于SSR的四川主要香椿种源遗传多样性分析[J].中南林业科技大学学报,2022,42(12):60-67.LIU M H,XIAO X C,ZHANG C H,et al.Genetic diversity of Toona sinensis provenances in Sichuan province revealed by SSR markers[J].J Cent South Univ For Technol,2022,42(12):60-67.DOI: 10.14067/j.cnki.1673-923x.2022.12.007.
[13]PTER P, KINGA K M, ISTVN S, et al. Genetic variability of thermal Nymphaea (Nymphaeaceae) populations based on ISSR markers: implications on" relationships, hybridization, and conservation[J]. Plant Molecular Biology Reporter, 2011, 29: 906-918.
[14]DKHAR J,KUMARIA S,RAO S R,et al.New insights into character evolution,hybridization and diversity of Indian Nymphaea(Nymphaeaceae):evidence
from molecular and morphological data[J].Syst Biodivers,2013,11(1):77-86.DOI: 10.1080/14772000.2013.773949.
[15]QIAN Z H,MUNYWOKI J M,WANG Q F,et al.Molecular identification of African Nymphaea species (water lily) based on ITS,trnT-trnF and rpl16[J].Plants,2022,11(18):2431.DOI: 10.3390/plants11182431.
[16]苏群,杨亚涵,田敏,等.睡莲种质资源遗传多样性分析及DNA指纹图谱构建[J].热带作物学报,2020,41(2):258-266.SU Q,YANG Y H,TIAN M,et al.Genetic diversity analysis and DNA fingerprinting construction of waterlily germplasm resources[J].Chin J Trop Crops,2020,41(2):258-266.DOI: 10.3969/j.issn.1000-2561.2020.02.008.
[17]李志强,吴超,贺熙勇,等.基于SSR标记的澳洲坚果种质资源DNA指纹图谱的构建[J].果树学报,2022,39(11):2028-2035.LI Z Q,WU C,HE X Y,et al.Construction of DNA fingerprint of macadamia germplasm based on SRR markers[J].J Fruit Sci,2022,39(11):2028-2035.DOI: 10.13925/j.cnki.gsxb.20220122.
[18]梁燕,韩传明,孙超,等.基于SSR标记的核桃种质资源遗传多样性与遗传结构分析[J].北方园艺,2022(9):47-54.LIANG Y,HAN C M,SUN C,et al.Genetic diversity and genetic structure analysis of walnut germplasm resources based on SSR markers[J].North Hortic,2022(9):47-54.DOI: 10.11937/bfyy.20214296.
[19]蔡元保,杨祥燕,陈豪军,等.SRAP结合SCoT标记分析番木瓜种质的遗传多样性[J].植物遗传资源学报,2014,15(2):292-298.CAI Y B,YANG X Y,CHEN H J,et al.Genetic diversity analysis of Papaya resources by SRAP and SCoT combination[J].J Plant Genet Resour,2014,15(2):292-298.DOI: 10.13430/j.cnki.jpgr.2014.02.010.
[20]井敏敏,黄炳钰,戴小红,等.基于SSR标记的澳洲坚果种质资源遗传多样性分析[J].热带作物学报,2022,43(2):262-270.JING M M,HUANG B Y,DAI X H,et al.Genetic diversity analysis of Macadamia germplasm resources by SSR markers[J].Chin J Trop Crops,2022,43(2):262-270.DOI: 10.3969/j.issn.1000-2561.2022.02.005.
[21]任雪锋,邓亚博,臧国长,等.基于SSR标记的河南省狗牙根遗传多样性及群体遗传结构分析[J].草业学报,2022,31(3):60-70.REN X F,DENG Y B,ZANG G Z,et al.A SSR marker analysis of genetic diversity and population genetic structure of bermudagrass in Henan Province[J].Acta Prataculturae Sin,2022,31(3):60-70.DOI: 10.11686/cyxb2021196.
[22]郑永胜,张晗,王东建,等.基于荧光检测技术的小麦品种SSR鉴定体系的建立[J].中国农业科学,2014,47(19):3725-3735.ZHENG Y S,ZHANG H,WANG D J,et al.Development of a wheat variety identification system based on fluorescently labeled SSR markers[J].Sci Agric Sin,2014,47(19):3725-3735.DOI: 10.3864/j.issn.0578-1752.2014.19.001.
[23]苏群,王虹妍,卢家仕,等.睡莲的SSR引物对及合成方法和应用:CN113832254A[P].2021-12-24.SU Q,WANG H Y,LU J S, et al.SSR primer pair of Nymphaea tetragona,and synthesis method and application of SSR primer pair:CN113832254A[P].2021-12-24.
[24]PARVEEN S,SINGH N,ADIT A,et al.Contrasting reproductive strategies of two Nymphaea species affect existing natural genetic diversity as assessed by microsatellite markers:implications for conservation and wetlands restoration[J].Front Plant Sci,2022,13:773572.DOI: 10.3389/fpls.2022.773572.
[25]胡永超,马洁,唐建宁,等.不同树龄枸杞古树的遗传多样性研究[J].植物遗传资源学报,2022,23(3):755-767.HU Y C,MA J,TANG J N,et al.Genetic diversity analysis of ancient trees with different ages of Lycium barbarum L[J].J Plant Genet Resour,2022,23(3):755-767.DOI: 10.13430/j.cnki.jpgr.20211105001.
[26]仲小茹,柯叮,黄献峰,等.基于SSR标记的江西省枫香古树遗传多样性评价[J].植物遗传资源学报,2023,24(2):523-531.ZHONG X R,KE D,HUANG X F,et al.Genetic diversity in the ancient Liquidambar formosana hance revealed by simple sequence repeat markers[J].J Plant Genet Resour,2023,24(2):523-531.DOI: 10.13430/j.cnki.jpgr.20220830003.
[27]张旻桓,姚奕平,黄宇,等.基于SSR标记的江南牡丹品种群遗传多样性及亲缘关系[J].中南林业科技大学学报,2023,43(1):164-172.ZHANG" M H,YAO Y P,HUANG Y,et al.Genetic diversity and genetic relationship of Jiangnan Paeonia suffruticosa based on SSR markers[J].J Cent South Univ For Technol,2023,43(1):164-172.DOI: 10.14067/j.cnki.1673-923x.2023.01.017.
[28]孙春青,陶美奇,姚悦梅,等.睡莲生殖器官发育过程中解剖结构的变化[J].植物资源与环境学报,2022,31(1):21-28.SUN C Q,TAO M Q,YAO Y M,et al.Changes of anatomical structure during the development processes of reproductive organs of Nymphaea tetragona[J].J Plant Resour Environ,2022,31(1):21-28.DOI: 10.3969/j.issn.1674-7895.2022.01.04.
[29]孙春青,潘跃平,单延博,等.睡莲品种墨宝自交结实率低的细胞学机理[J].江苏农业学报,2017,33(4):890-894.SUN C Q,PAN Y P,SHAN Y B,et al.Cellular mechanisms of low self-fertility of water lily variety Almost Black[J].Jiangsu J Agric Sci,2017,33(4):890-894.DOI: 10.3969/j.issn.1000-4440.2017.04.025.
[30]黄祥,杨梅花,楚光明,等.耐寒睡莲种质资源分析及观赏性评价[J].分子植物育种,2022,20(4):1348-1357.HUANG X,YANG M H,CHU G M,et al.Analysis of variety resources and evaluation of ornamental value in hardy waterlily[J].Mol Plant Breed,2022,20(4):1348-1357.DOI: 10.13271/j.mpb.020.001348.
[31]潘庆龙,付瑛格,谷佳,等.海南引种睡莲表型多样性分析及评价[J].热带作物学报,2021,42(10):2777-2788.PAN Q L,FU Y G,GU J,et al.Analysis of phenotypic diversity of Nymphaea Linn. in Hainan,China[J].Chin J Trop Crops,2021,42(10):2777-2788.DOI: 10.3969/j.issn.1000-2561.2021.10.005.
[32]张大乐,李锁平,雷进生,等.利用SSR标记对12个啤酒大麦品种的聚类分析和主坐标分析[J].河北农业大学学报,2007,30(3):26-31.ZHANG D L,LI S P,LEI J S,et al.Cluster and principal coordinate analysis among 12 beer barley by SSRs[J].J Agric Univ Hebei,2007,30(3):26-31.DOI: 10.3969/j.issn.1000-1573.2007.03.006.
[33]赵亚军,王灏,穆建新,等.油菜自育与其他主栽品种的遗传多样性和遗传关系分析[J].分子植物育种,2018,16(8):2714-2722.ZHAO Y J,WANG H,MU J X,et al.Genetic diversity and genetic relationship analysis of our own and other leading rapeseed(Brassica napus L.) varieties[J].Mol Plant Breed,2018,16(8):2714-2722.DOI: 10.13271/j.mpb.016.002714.
[34]肖志娟,翟梅枝,王振元,等.微卫星DNA在分析核桃遗传多样性上的应用[J].中南林业科技大学学报,2014,34(2):55-61.XIAO Z J,ZHAI M Z,WANG Z Y,et al.Application of microsatellite DNA on analyzing genetic diversity of Juglans regia[J].J Cent South Univ For Technol,2014,34(2):55-61.DOI: 10.14067/j.cnki.1673-923x.2014.02.006.
(责任编辑 吴祝华)
基金项目:广西科技厅重点研发计划(桂科AB18221054);广西农业科学院基本科研业务专项(桂农科2021YT152)。
第一作者:毛立彦(2634081433@qq.com),高级农艺师。
*通信作者:龙凌云(349542116@qq.com),高级农艺师。

