食药用菌的基因组学研究进展
摘 要:以食药用菌为研究对象,概述了食药用菌的基因组测序现状及基于基因组测序的基因挖掘和改造等方面的应用。据NCBI数据,担子菌基因组测序数据有980条记录;子囊菌基因组测序数据有3850条记录,目前有130余种食药用菌已经完成基因组测序工作。比较基因组学在灵芝、香菇、樟芝等部分真菌中的应用,为研究该物种的遗传多样性、进化和次生代谢物的生物合成奠定了基础。CRISPR/Cas9基因编辑技术可以快速高效地实现食药用菌基因的精准编辑,包括基因敲除、点突变和基因插入等操作。基因组学的研究将有助于改善食药用菌的品质、提高其营养价值,甚至开发新品种,并促进食药用菌产业的可持续发展。
关键词:食药用菌;核基因组;线粒体基因组;基因挖掘;CRISPR/Cas9
中图分类号:S646 文献标志码:A 文章编号:1673-2871(2024)09-001-08
Advances in genomics of edible and medicinal fungi
WANG Fengli1, HAN Chuang1,2, JIAO Lihe1, YUE Xin1, GUO Yan1, DAI Xiaodong1
(1. Institute of Microbiology, Heilongjiang Academy of Sciences, Harbin 150010, Heilongjiang, China; 2. College of Plant Protection, Northeast Agricultural University/Key Laboratory of Agricultural Microbiology of Heilongjiang Province, Harbin 150030, Heilongjiang, China)
Abstract: Here we present a comprehensive overview of the current status of genome sequencing in edible and medicinal fungi, as well as the application of gene mining and modification based on this technology. As of March 2024, NCBI reports a total of 980 basidiomycete genome sequencing records and 3850 ascomycete genome sequencing records. Additionally, numerous studies have successfully completed genome sequencing for over 130 species of edible and medicinal fungi. Comparative genomics has been applied to investigate genetic diversity, evolution, and secondary metabolite biosynthesis in specific fungi such as Ganoderma lucidum, Lentinus edodes, and Antrodia camphorata. Furthermore, CRISPR/Cas9 gene editing technology enables precise manipulation of genes in edible fungi through knockout, insertion, or point mutation operations. These advancements hold great potential for enhancing the quality and nutritional value of edible fungi while facilitating the development of new varieties to promote sustainable growth in the industry.
Key words: Edible and medicinal fungi; Nuclear genome; Mitochondrial genome; Gene mining; CRISPR/Cas9
收稿日期:2024-05-27;修回日期:2024-06-25
基金项目:黑龙江省省属科研院所科研业务费项目(CZKYF2022SW01)
作者简介:王凤利,女,在读硕士研究生,研究方向为食用菌种质资源评价。E-mail:wang7877153@163.com
通信作者:戴肖东,男,研究员,研究方向为食用菌种质资源评价。E-mail:heiweihlj@126.com
食药用菌是具有较高营养价值和药用价值的大型肉质或胶质子实体的高等真菌[1]。对我国食药用菌的认识、开发和利用可以追溯到仰韶文化时期[2],据报道,目前我国的食药用菌种类有近千种,然而被广泛认识和食用的仅有200余种[3]。自1996年首个真核生物-酿酒酵母基因组测序以来[4],随着测序方法的革新与测序成本的下降,真菌基因组学领域的研究取得了长足的发展。与其他真核物种相比,真菌的基因组结构比较简单,在序列注释、基因改造方面都更为方便。当前,对真菌全基因组的研究已由单一物种的全基因组发展到基于群体基因组的多个基因序列,并将两者进行整合研究,有望揭示基因水平转移、正选择、基因扩张和基因收缩等生物学问题。以真菌基因组的研究与发展作为良好的基础应用于相应物种的转录组、蛋白质组、代谢组及功能基因组研究,促进了真菌生物学乃至整个生物学的研究与发展[4]。近年来,我国食药用真菌的分子生物学研究蓬勃发展,从理论到实际的应用,如在食用菌遗传改良[5]、基因组注释[6]、木质素和纤维素合成酶基因[7]、抗病机制[8]、胁迫[9]等相关研究中都取得了前所未有的突破性成果。
1 食药用菌的基因组测序现状
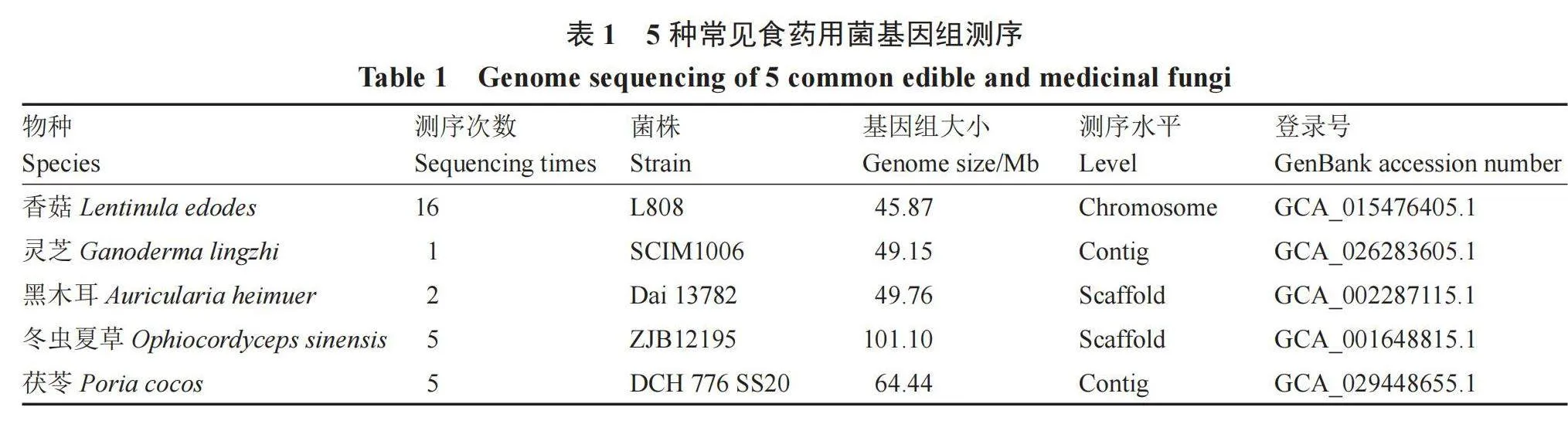
基因组(genome)是指该生物体内的DNA中全部遗传物质的总和[10],一般包括核基因组、线粒体基因组、叶绿体基因组,在食药用菌中只包含核基因组及线粒体基因组。据统计,食药用菌的全基因组测序约有130种[4],对于一些常见食药用菌,如香菇(Lentinula edodes)、灵芝(Ganoderma lucidum)、蛹虫草(Cordyceps militaris)、茯苓(Poria cocos)、黑木耳(Auricularia heimuer)等物种已完成多次基因组测序,其中部分测序水平已经到了染色体水平,对更好地了解食药用菌基因组有重要意义。这些基因组信息的获得使深入的功能分析和系统的比较基因组学研究得以开展,为深入了解其生命规律和调控提供了坚实的理论基础。
1.1 食药用菌核基因组测序
截止2024年3月,据NCBI数据,担子菌基因组测序数据有980条记录;子囊菌基因组测序数据有3850条记录,有研究表明,目前有130余种食药用菌已经完成基因组测序[4],例如裂褶菌(Schizophyllum commune)、香菇、冬虫夏草(Ophiocordyceps sinensis)、灵芝、茯苓、草菇(Volvariella volvacea)、黑木耳、蛹虫草、双孢蘑菇(Agaricus bisporus)、金针菇(Flammulina filiformis)、樟芝(Taiwanofungus camphoratus )、干巴菌(Thelephora ganbajun Zang)等,表1为5种常见食药用菌基因组测序情况。裂褶菌是研究大型真菌遗传发育的模式生物,Ohm等[11]对裂褶菌的基因组(38.5 Mb)进行分析,揭示了其独特的木质素降解机制。裂褶菌是首个完成基因组测序的大型真菌,目前NCBI已有35条裂褶菌基因组数据记录。香菇是世界上第二大菇,该物种的基因组已被多次报道,但Yu等[12]首次结合高通量染色质构象捕获(Hi-C)技术,构建了香菇高质量的染色体水平基因组,大小为45.87 Mb,99%的序列被锚定在10条染色体上,且通过对不同地区野生菌株和商业品种进行群体基因组重测序,在分子水平上确定了香菇的育种历史。冬虫夏草作为传统药物或者保健食品在中国已经有几千年的历史,中国被毛孢(Hirsutella sinensis)是冬虫夏草唯一的正确变形体。为了更深入地了解其生理和药理机制,Jin等[13]对中国被毛孢的基因组进行了测序,首次获得覆盖率>99%的中国被毛孢的基因组(102.72 Mb)。灵芝是一种食药兼用菌,可产生多种生物活性化合物,其药用价值在中国已有2000多年的历史。Wu等[14]首次对灵芝基因组(49.15 Mb)进行了报道,预测编码了13 125个基因。茯苓是中国最重要的传统中药之一,在中国、日本、韩国和其他亚洲国家普遍使用。Li等[15]组装了茯苓第一个高质量的核基因组,包含14条染色体,基因组全长64.44 Mb,Contig N50长度为3.76 Mb,并对菌核的基因组结构、转座子和甲基化、高温适应性和基因家族扩展进行了分析。
1.2 食药用菌线粒体基因组测序
线粒体(mitochondrion,mt)作为食药用菌的第二大基因组[16],其基因组信息主要用于系统发育、分类鉴定、比较基因组学以及线粒体相关疾病的研究。随着科学技术的发展、测序手段逐渐成熟,近年来在真菌线粒体方面的研究成果逐年增多,但是就目前报道情况来看,已完成线粒体基因组测序的真菌与其丰富的物种多样性相对而言,远远不够,且不同真菌线粒体基因组存在较大的差异。
梯棱羊肚菌(Morchella importuna)的线粒体基因组(272.2 kb)是已报道的大型真菌中最大的,包含大量内含子、线粒体非保守开放阅读框和重复序列等[17]。烟色珊瑚菌(Clavaria fumosa)线粒体基因组是目前所报告的担子菌最大的线粒体基因组,Wang等[18]对烟色珊瑚菌的完整线粒体基因组首次进行了测序、组装和比较,长度为256 807 bp。樟芝是中国台湾特有的一种珍贵的药用真菌。Wang等[19]首次对樟芝的线粒体基因组进行了组装,大小为114 922 bp。冬虫夏草菌株1229的完整线粒体基因组被组装为一个157 510 bp的单圆形dsDNA,鉴定出的保守基因包括大和小rRNA亚基、27个tRNA以及15个蛋白编码基因,推测可能是编码RTs和HEs的内含子ncORFs导致了冬虫夏草线粒体基因组的扩增[20]。线粒体基因组的测序在干巴菌、黑木耳、茯苓、草菇、香菇等真菌中也已有报道,多种食药用菌的线粒体基因组均存在大量的基因组扩增、丢失、重排现象,可能是存在大量内含子以及内含子的特殊性导致的,但目前还没有定论。
2 食药用菌的比较基因组学研究
比较基因组学是在基因组测序的基础上,对已知的基因和基因组结构进行比较,来了解基因的功能、表达机制和物种进化[21]。在食药用菌中,比较基因组学一般应用于物种进化、种属间的亲缘关系研究等方面,像灵芝、香菇、樟芝等部分真菌通过对同种或同菌株的不同单核菌株进行测序,通过比较分析,研究该物种的遗传多样性,为了解其生物学、进化和次生代谢物的生物合成奠定了基础。
2.1 比较基因组学在食药用菌核基因组研究中的应用
为了解香菇两个单倍体核基因组结构及其在子实体发育和生长中的相互作用,Gao等[22]利用Illumina、HiFi和Hi-C技术从香菇中分离并组装了SP3和SP30两个单倍体基因组,总长度分别为50.93 Mb和49.80 Mb,虽然这两个基因组都被组装成10条染色体,但SP3和SP30这两个单核体在染色体结构和基因含量方面存在显著差异;经比较基因组分析,菌株尾部基因在这两个基因组中所占比例约为30%。此外,这两个基因组富集了不同的基因和基因家族,包括参与碳水化合物代谢、次级代谢和剪接体形成的基因。其中,SP30菌株的淀粉代谢基因和蔗糖代谢基因显著富集,且SP30菌株在PDA培养基上的生长速度快于SP3菌株。相反,SP3菌株重排基因丰富,可产生风味物质5′-核苷酸。比较基因组分析表明,两个单倍体核可能来自不同的遗传祖先,其中约30%的基因组是独特的或非合性的。在主要交配型位点之外的位置发现了不完整的交配型基因,这表明香菇交配系统可能继续进化。Chen等[23]从两个樟芝双核菌——HC1(橘红色)和SN1(乳白色)中分离出4个单核菌株,对这4个单核体的高质量染色体水平的基因组序列进行了测序、注释和比较分析,为了解该真菌的生物学、进化和次生代谢物的生物合成奠定了基础,证明了樟芝具有四极交配系统,HC1和SN1代表了两个表现出核型变异的物种内分离物。与几种食用菌模式生物相比,樟芝在基因家族和个体基因数量上发生了明显收缩,尤其是在植物、真菌和细菌细胞壁降解酶方面,很好地解释了樟芝在自然情况下罕见以及人工培养困难、耗时,且易受真菌和细菌感染的原因,为深入研究樟芝的遗传操作、子实体改良以及在天然药物生产中的合成生物学应用奠定了基础。
2.2 比较基因组学在食药用菌线粒体基因组研究中的应用
Wang等[18]通过比较烟色珊瑚菌线粒体基因组,发现其线粒体基因组包含最多的内含子和内含子编码开放阅读框(ORF),检测到4个新的内含子类,具有独特的基因排列等特点。基于组合线粒体基因集的76个担子菌的系统发育分析表明,线粒体基因可以作为担子菌进化的有效分子标记。Wang等[19]通过对樟芝线粒体基因组共线性进行分析,发现樟芝线粒体基因组包含一个独特的基因序列,线粒体基因组之间存在大规模的基因重排;内含子的数量和种类在12个多孔菌目属物种中具有显著的变异,证明了在多孔菌目属物种的进化过程中发生了大量的内含子丢失或获得事件。Li等[24]通过对4个灵芝的线粒体基因组进行测序组装,4个线粒体基因组的大小从50 603 bp到73 416 bp不等。灵芝线粒体基因组的大小在不同种类灵芝中存在差异,内含子是影响灵芝线粒体基因组大小变化的主要因素,通过比较分析揭示了灵芝线粒体基因组种内和种间的变异,在内含子类型、非保守基因和基因排列等方面具有丰富的多样性。
3 基于基因组测序对食药用菌基因挖掘
全基因组序列的公布推动了大量相关的基因研究,但是对这些数据中的大量信息还未全部解析,目前阶段还是大量利用生物信息学工具对基因组进行注释,以及蛋白的功能预测,得到一些关于次级代谢产物、活性物质以及木质素降解相关的基因信息,研究主要集中于灵芝、蛹虫草、香菇、金针菇、平菇等具有经济价值的食药用菌。Fernández-fueyo等[25]对平菇基因组的分析表明,在一个木质素降解系统中,多功能过氧化物酶承担了木质素过氧化物酶的作用,研究首次对担子菌中完整的过氧化物酶同工酶进行了表征,揭示了其高热稳定性和pH稳定性差异。Li等[7]对蛹虫草疏水蛋白家族进行了全基因组分析,共鉴定了4个疏水蛋白编码基因,每个疏水酶基因在生命周期和对光照的反应中具有不同的转录模式,具有致病性生活方式的子囊菌真菌比腐生菌更容易获得较多的疏水性编码基因,II类成员之间的变异比I类成员大,只有少数同源蛋白是通过重复进化的。Kim等[26]从金针菇的全基因组序列中鉴定和表征新的金针菇漆酶基因,在15个推测的漆酶基因中,发现4个新漆酶基因含有4个完整的铜结合区(10个组氨酸残基和1个半胱氨酸残基)和4个参与形成二硫桥的半胱氨酸残基,分别为fvlac1、fvlac2、fvLac3和fvlac4。在fvlacc1 (Asn-454)、fvlacc2 (Asn-437和Asn-455)、fvlacc3 (Asn-111和Asn-237)和fvLac4 (Asn-402和Asn-457)的cDNA序列中鉴定出潜在的N-糖基化位点(Asn-Xaa-Ser/Thr),预测这些蛋白的前19~20个氨基酸残基构成信号肽。漆酶活性测定和逆转录聚合酶链式反应分析表明,CuSO4影响这些漆酶基因的诱导和转录水平。灵芝酸(ganoderic acids, GAs)作为灵芝重要的药理活性化合物,是一种三萜类化合物,其生物合成及其调控机制也是研究热点,但直接调控GAs生物合成基因表达的转录因子尚不清楚。Liu等[27]结合基因组学、DAP-seq、RNA-seq、代谢组学等技术以及分子生物学和遗传学方法,确定了转录因子固醇调节元件结合蛋白(SREBP)的可能靶点,包括三萜合成和脂质代谢基因,首次发现SREBP在调节灵芝真菌GAs生物合成中的新未知功能,并证明SREBP通过结合启动子区域,促进GA、麦角甾醇和脂质生物合成相关靶基因的转录,从而增强GAs、麦角甾醇和脂质的代谢能力。
4 基于基因组测序对食药用菌基因的改造情况
随着食药用菌的基因组数据公布,以及新基因的发现及基因功能研究的深入,利用分子手段对食药用菌进行基因改造成为了热点。建立稳定高效的转化体系是分子育种的基础,主要方式包括PEG介导转化法(PMT)、电击法、基因枪法和农杆菌介导法(ATMT)等[28],目前在食药用菌中应用较多的是PMT和ATMT。Chen等[29]利用ATMT技术将质粒转化到蛹虫草,在5-FOA选择性下获得突变子。Tu等[30]通过PMT方法将质粒pU6-ura3-sgRNA1引入到灵芝Cas9菌株和ku70突变体中获得转化子。对食药用菌改造方向主要是利用基因敲除、基因过表达、基因沉默等方法,其中CRISPR/Cas9基因编辑系统是对分子育种的一种重要手段。
4.1 CRISPR/Cas9基因编辑系统
早在1987年,一段间隔短重复序列首次在K12大肠杆菌中被发现[31];直到2002年将其命名为CRISPR,经测序发现,大约有40%的细菌基因组上含有此序列;2012年,法国科学家利用重组Cas9蛋白以及体外转录的crRNA和tracRNA成功实现了体外切割实验;此后华人科学家张峰博士对其进一步优化,并申请专利;2020年,Emmanuelle Charpentier 和 Jennifer A. Doudna由于发现CRISPR/Cas9而获得诺贝尔化学奖[32]。CRISPR/Cas9基因编辑技术,是对靶向基因进行特定修饰的技术。CRISPR/Cas9基因系统由一小段RNA(sgRNA)和一种高效的DNA切割酶(Cas核酸酶)组成,其原理主要是在sgRNA引导下,Cas9蛋白对靶基因位点进行特异性切割,产生DSB,再利用非同源末端连接(NHEJ)或同源重组修复(HDR)进行修复,最终实现目标基因敲除和碱基编辑等基因组遗传修饰[33-34]。
4.2 CRISPR/Cas9基因编辑系统在食药用菌研究中的应用
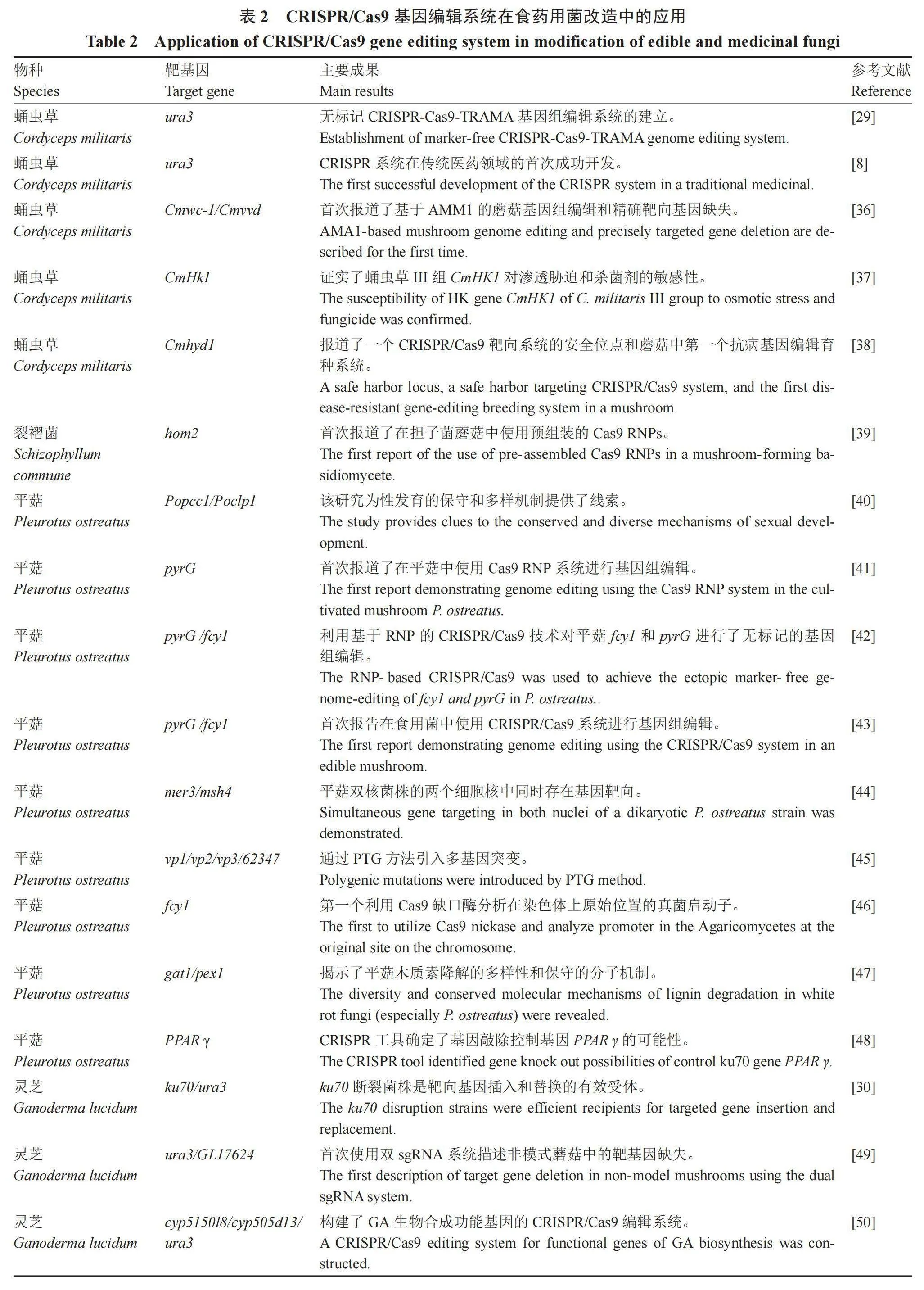
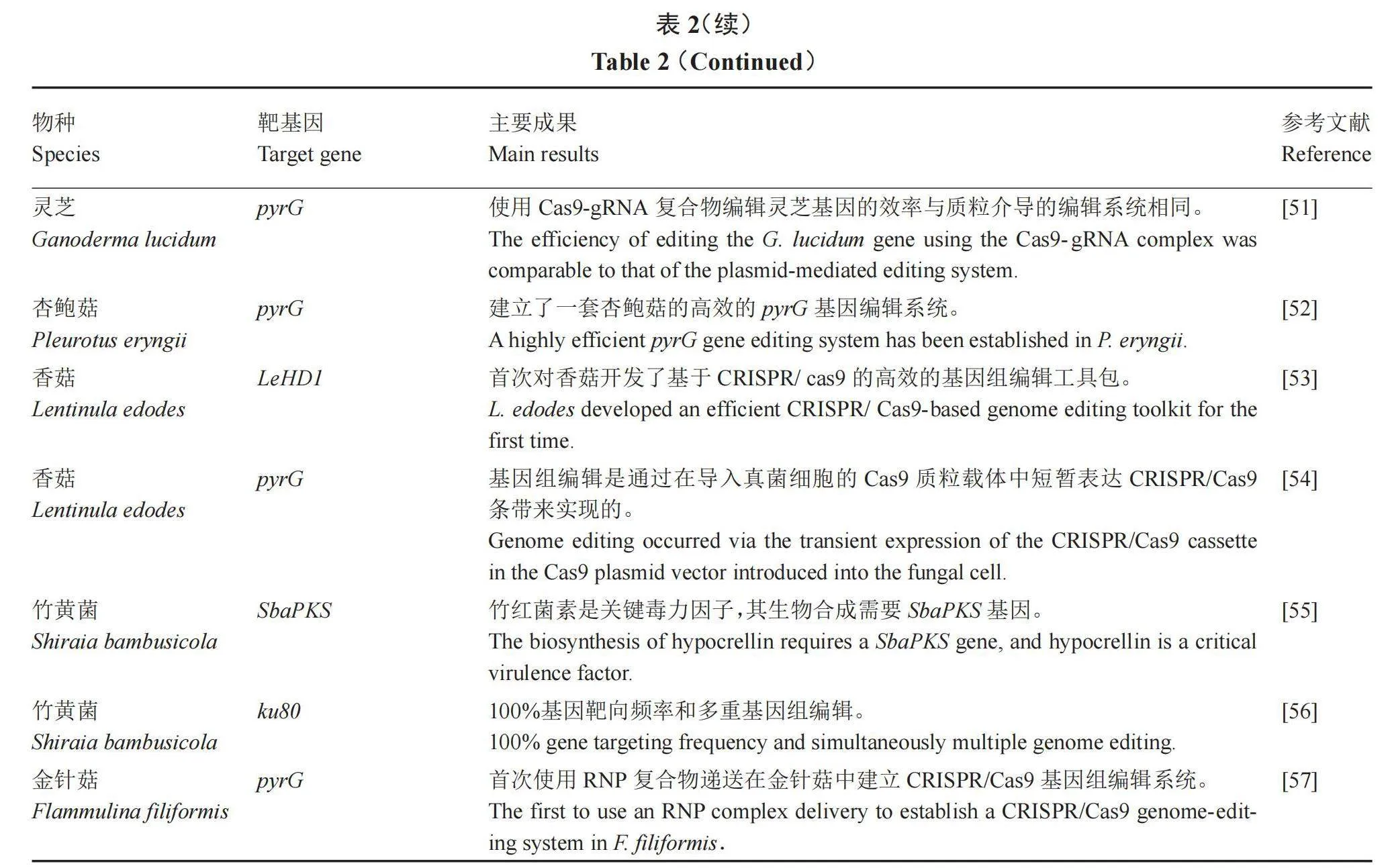
部分食药用菌可产生具有经济价值的天然代谢物,为了获得高产量的三萜、灵芝酸、多糖等代谢产物,通常是利用遗传转化方法对真菌代谢通路进行定向改造。传统的同源重组方法基因敲除效率极低,而且食药用菌多为异核体,获得纯合突变体的概率较低,所以目前研究人员将目光转向了CRISPR/Cas9基因编辑技术。CRISPR/Cas9技术显著提高了基因组编辑的效率,确定适当的靶位点有助于评估编辑效率。在CRISPR/Cas9基因组编辑系统中有3种常见的递送策略:体内Cas9核酸酶和体外sgRNA(体外表达Cas9的底盘与sgRNA)、体内Cas9和sgRNA两者(携带Cas9表达盒和sgRNA盒的全合一质粒),以及体外Cas9和sgRNA两者(RNP复合物)[35]。目前成功进行CRISPR/Cas9改造的食药用菌并不多,并且大多数都是将筛选基因作为靶基因,如灵芝、蛹虫草、平菇等,详见表2。
Liu等[57]开发并优化了一种基于金针菇中体外组装的核糖核蛋白复合物的CRISPR/Cas9基因组编辑方法,并首次将其应用于金针菇,与其他方法相比,该方法避免了使用任何外源DNA,从而在质粒构建时节省了时间和劳动力。Boontawon等[40]将携带Cas9和靶向fcyl和pyrG的不同sgRNA的质粒分别转移到PC 9菌株的原生质体中,产生了分别对5-氟胞嘧啶和5-氟乳清酸表现出抗性的菌株,首次证明在食用菌中使用CRISPR/Cas9系统是可以进行基因编辑的。Chen等[29]在蛹虫草中开发并建立了无标记的CRISPR-Cas9-TRAMA基因组编辑系统,进一步在虫草素和麦角硫因的合成酶中操作编辑,以展示Cas9-TRAMA系统在蛋白质修饰、启动子强度评估和10 kb代谢合成簇缺失中的应用。Meng等[36]首次描述基于AMA1质粒的大型真菌基因组编辑和精确靶向基因缺失,在蛹虫草中构建了基于具有AMAl序列的自主复制质粒的高效CRISPR/Cas9基因组编辑系统,其研究结果将使多个基因的修饰在功能基因组学研究和菌株育种中成为可能。
CRISPR/Cas9基因组编辑系统在大型真菌中的应用较植物、动物发展明显不足,且就目前研究现状来说,CRISPR/Cas9基因组编辑系统在大型真菌中的应用还不完善、成熟,依旧停留在初级摸索阶段。这些问题主要是由于大型真菌的基因组数据没有得到全部解析、基因层面了解不足以及CRISPR/Cas9系统转化效率低下等。
5 展 望
我国拥有极其丰富的食药用菌资源,以前研究者更多的是将关注点放在了食药用菌的分类、栽培、育种等方面,更注重其经济价值。随着组学技术的发展,在基因水平上了解一个物种成为一种趋势。对于食药用菌来说,了解其生长、发育和各种代谢通路产生的代谢物以及研究食药用菌的进化渐渐变成重点。食药用菌全基因组测序可帮助了解食药用菌的基因构成和结构,通过联系性状、特性以及功能,可筛选出该菌与食用、药用价值相关的基因,对推动食药用菌在食品行业的发展,以及为医药产品的开发利用提供理论支持。在食药用菌的基因组应用方面,利用比较基因组方法,不仅为大型真菌生理调节、形态差异、代谢调控等提供有力的理论支持,还可为将来食药用菌的驯化栽培提供参考[58]。深入开展食药用菌基因组研究,挖掘与次生代谢产物合成相关的基因、基因簇,并对其功能进行鉴定,这为高效、可控地生产有效化合物提供了技术手段,也为基因组学与合成生物学的学科交叉奠定了一定的理论基础。真菌基因组的研究与发展作为良好的基础应用于相应物种的转录组、蛋白质组、代谢组及基因组研究,促进了真菌生物学乃至整个生物学的研究和发展。
参考文献
[1] 李才慧,徐爽.生物多样性保护背景下云南省野生食用菌产业可持续发展探讨[J].广西植物,2023,43(8):1446-1456.
[2] 张月,沈雅萍,李利利,等.响应面法优化紫外线处理后双孢菇中维生素D2皂化工艺[J].食品与发酵工业,2019,45(4):181-186.
[3] 张平,周祥斌.粤北林区灵芝林下栽培模式项目可行性分析[J].绿色科技,2019(13):145-146.
[4] 田风华.中国东北元蘑种质资源评价及其三萜合成途径相关基因研究[D].长春:吉林农业大学,2019.
[5] DONG Y T,MIAO R Y,FENG R C,et al.Edible and medicinal fungi breeding techniques,a review:Current status and future prospects[J].Current Research in Food Science,2022,5:2070-2080.
[6] LIANG Y,LU D X,WANG S,et al.Genome assembly and pathway analysis of edible mushroom Agrocybe cylindracea[J].Genomics Proteomics and Bioinformatics,2020,18(3):341-351.
[7] LI X,WANG F,XU Y Y,et al.Cysteine-rich hydrophobin gene family:Genome wide analysis,phylogeny and transcript profiling in Cordyceps militaris[J].International Journal of Molecular Sciences,2021,22(2):643.
[8] CHEN B X,WEI T,YE Z W,et al.Efficient CRISPR-Cas9 gene disruption system in edible-medicinal mushroom Cordyceps militaris[J].Frontiers in Microbiology,2018,9:1157.
[9] DING Q Q,ZHAO H Y,ZHU P L,et al.Genome-wide identification and expression analyses of C2H2 zinc finger transcription factors in Pleurotus ostreatus[J].PeerJ,2022,10:e12654.
[10] 杨能,文绒,管婷婷,等.附子生产利用研究进展及其在西南地区产业发展探讨[J/OL].中成药,[2024-03-27].https://link.cnki.net/urlid/31.1368.R.20240325.1850.004.
[11] OHM R A,DE JONG J F,LUGONES L G,et al.Genome sequence of the model mushroom Schizophyllum commune[J].Nature Biotechnology,2010,28(9):957-963.
[12] YU H L,ZHANG L J,SHANG X D,et al.Chromosomal genome and population genetic analyses to reveal genetic architecture,breeding history and genes related to cadmium accumulation in Lentinula edodes[J].BMC Genomics,2022,23(1):120.
[13] JIN L Q,XU Z W,ZHANG B,et al.Genome sequencing and analysis of fungus Hirsutella sinensis isolated from Ophiocordyceps sinensis[J].AMB Express,2020,10(1):105.
[14] WU Q,LIU H,SHI Y X,et al.Characteristics of the genome,transcriptome and ganoderic acid of the medicinal fungus Ganoderma lingzhi[J].Journal of Fungi,2022,8(12):1257.
[15] LI S J,MENG G L,DONG C H.Homokaryotic high-quality genome assembly of medicinal fungi Wolfiporia hoelen reveals auto-regulation and high-temperature adaption of probable two-speed genome[J].International Journal of Molecular Sciences,2022,23(18):10484.
[16] 敖干.中国蕨麻属(Argentina)植物叶绿体基因组研究[D].呼和浩特:内蒙古师范大学,2021.
[17] LIU W,CAI Y L,ZHANG Q Q,et al.The mitochondrial genome of Morchella importuna(272.2 kb) is the largest among fungi and contains numerous introns,mitochondrial non-conserved open reading frames and repetitive sequences[J].International Journal of Biological Macromolecules,2020,143:373-381.
[18] WANG X,WANG Y J,YAO W,et al.The 256 kb mitochondrial genome of Clavaria fumosa is the largest among phylum Basidiomycota and is rich in introns and intronic ORFs[J].IMA Fungus,2020,11(1):26.
[19] WANG X,JIA L H,WANG M D,et al.The complete mitochondrial genome of medicinal fungus Taiwanofungus camphoratus reveals gene rearrangements and intron dynamics of Polyporales[J].Scientific Reports,2020,10(1):16500.
[20] LI Y,HU X D,YANG R H,et al.Complete mitochondrial genome of the medicinal fungus Ophiocordyceps sinensis[J].Scientific Reports,2015,5:13892.
[21] 毛吉勇,孟赛男,史佳欣,等.比较基因组学研究进展[J/OL].空军军医大学学报,[2024-03-11].https://link.cnki.net/urlid/61.1526.R.20240311.1352.003.
[22] GAO Q,YAN D,SONG S,et al.Haplotype-resolved genome analyses reveal genetically distinct nuclei within a commercial cultivar of Lentinula edodes[J].Journal of Fungi,2022,8(2):167.
[23] CHEN C L,LI W C,CHUANG Y C,et al.Sexual crossing,chromosome-level genome sequences,and comparative genomic analyses for the medicinal mushroom Taiwanofungus camphoratus(syn.Antrodia cinnamomea,Antrodia camphorata)[J].Microbiology Spectrum,2022,10(1):e02032-21.
[24] LI Q,ZHANG T,LI L J,et al.Comparative mitogenomic analysis reveals intraspecific,interspecific variations and genetic diversity of medical fungus Ganoderma[J].Journal of Fungi,2022,8(8):781.
[25] FERNÁNDEZ-FUEYO E,RUIZ-DUEÑAS F J,MARTÍNEZ M J,et al.Ligninolytic peroxidase genes in the oyster mushroom genome:Heterologous expression,molecular structure,catalytic and stability properties,and lignin-degrading ability[J].Biotechnology for Biofuels,2014,7(1):2.
[26] KIM H I,KWON O C,KONG W S,et al.Genome-wide identification and characterization of novel laccase genes in the white-rot fungus Flammulina velutipes[J].Mycobiology,2014,42(4):322-330.
[27] LIU Y N,WU F Y,TIAN R Y,et al.The bHLH-zip transcription factor SREBP regulates triterpenoid and lipid metabolisms in the medicinal fungus Ganoderma lingzhi[J].Communications Biology,2023,6(1):1.
[28] 张春颖.反义PPO转双孢蘑菇的获得及抗褐变特性研究[D].杭州:浙江农林大学,2018.
[29] CHEN B X,XUE L N,WEI T,et al.Multiplex gene precise editing and large DNA fragment deletion by the CRISPR-Cas9-TRAMA system in edible mushroom Cordyceps militaris[J].Microbial Biotechnology,2022,15(12):2982-2991.
[30] TU J L,BAI X Y,XU Y L,et al.Targeted gene insertion and replacement in the Basidiomycete Ganoderma lucidum by inactivation of nonhomologous end joining using CRISPR/Cas9[J].Applied and Environmental Microbiology,2021,87(23):e01510-21.
[31] 鲍艳春,戴伶俐,刘在霞,等.CRISPR/Cas9系统在畜禽遗传改良中研究进展[J].遗传,2024,46(3):219-231.
[32] 汤琦.基因治疗创新进展与热点趋势[J].张江科技评论,2023(5):38-41.
[33] Ran F A,Hsu P D,Wright J,et al.Genome engineering using the CRISPR-Cas9 system[J].Nature Protocols,2013,8:2281-2308.
[34] 曹晓晴,祁显涛,刘昌林,等.编辑ZmCCT10/ZmCCT9/ZmGhd7基因的串联DsRed荧光表达盒的CRISPR/Cas9系统的构建及验证[J].作物学报,2024,50(8):1961-1970.
[35] 乔欢欢,张晴皓,明东.CRISPR/Cas9系统纳米递送及其在肿瘤治疗中应用[J].生物化学与生物物理进展,2021,48(5):570-579.
[36] MENG G L,WANG X P,LIU M Q,et al.Efficient CRISPR/Cas9 system based on autonomously replicating plasmid with an AMA1 sequence and precisely targeted gene deletion in the edible fungus,Cordyceps militaris[J].Microbial Biotechnology,2022,15(10):2594-2606.
[37] CHOI H,PARK S W,OH J,et al.Efficient disruption of CmHk1 using CRISPR/Cas9 ribonucleoprotein delivery in Cordyceps militaris[J].FEMS Microbiology Letters,2023,370:fnad072.
[38] LIU Q,MENG G L,WANG M,et al.Safe-harbor-targeted CRISPR/Cas9 system and Cmhyd1 overexpression enhances disease resistance in Cordyceps militaris[J].Journal of Agricultural and Food Chemistry,2023,71(41):15249-15260.
[39] VONK P J,ESCOBAR N,WÖSTEN H A B,et al.High-throughput targeted gene deletion in the model mushroom Schizophyllum commune using pre-assembled Cas9 ribonucleoproteins[J].Scientific Reports,2019,9:7632.
[40] BOONTAWON T,NAKAZAWA T,HORII M,et al.Functional analyses of Pleurotus ostreatus pcc1 and clp1 using CRISPR/Cas9[J].Fungal Genetics and Biology,2021,154:103599.
[41] BOONTAWON T,NAKAZAWA T,XU H B,et al.Gene targeting using pre-assembled Cas9 ribonucleoprotein and split-marker recombination in Pleurotus ostreatus[J].FEMS Microbiology Letters,2021,368(13):fnab080.
[42] BOONTAWON T,NAKAZAWA T,CHOI Y J,et al.Double-gene targeting with preassembled Cas9 ribonucleoprotein for safe genome editing in the edible mushroom Pleurotus ostreatus[J].FEMS Microbiology Letters,2023,370:fnad015.
[43] BOONTAWON T,NAKAZAWA T,INOUE C,et al.Efficient genome editing with CRISPR/Cas9 in Pleurotus ostreatus[J].AMB Express,2021,11(1):30.
[44] YAMASAKI F,NAKAZAWA T,OH M,et al.Gene targeting of dikaryotic Pleurotus ostreatus nuclei using the CRISPR/Cas9 system[J].FEMS Microbiology Letters,2022,369(1):fnac083.
[45] XU H B,NAKAZAWA T,ZHANG Y F,et al.Introducing multiple-gene mutations in Pleurotus ostreatus using a polycistronic tRNA and CRISPR guide RNA strategy[J].FEMS Microbiology Letters,2022,369(1):fnac102.
[46] NGUYEN D X,NAKAZAWA T,MYO G,et al.A promoter assay system using gene targeting in agaricomycetes Pleurotus ostreatus and Coprinopsis cinerea[J].Journal of Microbiological Methods,2020,179:106053.
[47] NAKAZAWA T,YAMAGUCHI I,ZHANG Y F,et al.Experimental evidence that lignin-modifying enzymes are essential for degrading plant cell wall lignin by Pleurotus ostreatus using CRISPR/Cas9[J].Environmental Microbiology,2023,25(10):1909-1924.
[48] BINDHU J,ARUNAVA D,DAS A.An edible fungi Pleurotus ostreatus inhibits adipogenesis via suppressing expression of PPAR γ and C/EBP α in 3T3-L1 cells:In vitro validation of gene knock out of RNAs in PPAR γ using CRISPR spcas9[J].Biomedicine and Pharmacotherapy,2019,116:109030.
[49] LIU K,SUN B,YOU H,et al.Dual sgRNA-directed gene deletion in basidiomycete Ganoderma lucidum using the CRISPR/Cas9 system[J].Microbial Biotechnology,2020,13(2):386-396.
[50] WANG P A,XIAO H,ZHONG J J.CRISPR-Cas9 assisted functional gene editing in the mushroom Ganoderma lucidum[J].Applied Microbiology and Biotechnology,2020,104(4):1661-1671.
[51] EOM H,CHOI Y J,NANDRE R,et al.The Cas9-gRNA ribonucleoprotein complex-mediated editing of pyrG in Ganoderma lucidum and unexpected insertion of contaminated DNA fragments[J].Scientific Reports,2023,13(1):11133.
[52] WANG T L,YUE S,JIN Y T,et al.Advances allowing feasible pyrG gene editing by a CRISPR-Cas9 system for the edible mushroom Pleurotus eryngii[J].Fungal Genetics and Biology,2021,147:103509.
[53] MOON S,AN J Y,CHOI Y J,et al.Construction of a CRISPR/Cas9-mediated genome editing system in Lentinula edodes[J].Mycobiology,2021,49(6):599-603.
[54] KAMIYA A,UESHIMA H,NISHIDA S,et al.Development of a gene-targeting system using CRISPR/Cas9 and utilization of pyrG as a novel selectable marker in Lentinula edodes[J].FEMS Microbiology Letters,2023,370:fnad042.
[55] DENG H X,GAO R J,LIAO X R,et al.Genome editing in Shiraia bambusicola using CRISPR-Cas9 system[J].Journal of Biotechnology,2017,259:228-234.
[56] DENG H X,LIANG W Y,FAN T P,et al.Modular engineering of Shiraia bambusicola for hypocrellin production through an efficient CRISPR system[J].International Journal of Biological Macromolecules,2020,165:796-803.
[57] LIU J Y,CUI H Y,WANG R J,et al.A simple and efficient CRISPR/Cas9 system using a ribonucleoprotein method for Flammulina filiformis[J].Journal of Fungi,2022,8(10):1000.
[58] 余金凤,周汐,赵春燕,等.食用菌基因组学研究进展[J].蔬菜,2017(3):33-38.

