胰腺导管腺癌免疫相关基因预后风险模型的构建与评估
张昱 任瑞平 万鹏 贺晓兰
摘要:目的 构建整合胰腺导管腺癌(PDAC)分子亚型和免疫相关基因的风险评估模型。方法 以GSE71729数据集(n=145)为训练集,整合PDAC鳞状和非鳞状亚型之间差异表达基因和差异免疫相关基因构建调控网络,筛选发挥主调控鳞状亚型的5个免疫标志基因。基于患者生存信息和免疫标志基因构建整合免疫评分(IIS)模型来预测PDAC患者的临床预后,并在5个验证集(n=758)中检验其预测效能。结果 根据IIS将PDAC患者分为高危组和低危组。在训练集和验证集中,高危组患者的总体生存期均短于低危组(P均<0.001)。多变量Cox回归分析显示IIS是PDAC的独立预后因子(HR=2.16,95%CI=1.50~3.10,P<0.001)。结论 IIS可用于PDAC患者的危险分层,并可能成为PDAC潜在的预后标志物。
关键词:胰腺导管腺癌;免疫相关基因;预后
中图分类号: R735.9 文献标识码: A 文章编号:1000-503X(2024)03-0354-07
DOI:10.3881/j.issn.1000-503X.15736
Construction and Evaluation of a Prognostic Risk Prediction Model of Pancreatic Ductal Adenocarcinoma Based on Immune-Related Genes
ZHANG Yu,REN Ruiping,WAN Peng,HE Xiaolan
Cancer Radiotherapy and Chemotherapy Center,The Affiliated Peoples Hospital of Ningbo University,Ningbo,Zhejiang 315040,China
Corresponding author:ZHANG Yu Tel:0574-87017563,E-mail:zy95589034@163.com
ABSTRACT:Objective To construct a risk prediction model by integrating the molecular subtypes of pancreatic ductal adenocarcinoma (PDAC) and immune-related genes.Methods With GSE71729 data set (n=145) as the training set,the differentially expressed genes and differential immune-related genes between the squamous and non-squamous subtypes of PDAC were integrated to construct a regulatory network,on the basis of which five immune marker genes regulating the squamous subtype were screened out.An integrated immune score (IIS) model was constructed based on patient survival information and immune marker genes to predict the clinical prognosis of PDAC patients,and its predictive performance was tested with 5 validation sets (n=758).Results PDAC patients were assigned into high risk and low risk groups according to the IIS.In both training and validation sets,the overall survival of patients in the high risk group was shorter than that in the low risk group (both P<0.001).The multivariable Cox regression showed that IIS was an independent prognostic factor for PDAC (HR=2.16,95%CI=1.50-3.10,P<0.001).Conclusion IIS can be used for risk stratification of PDAC patients and may become a potential prognostic marker for PDAC.
Key words:pancreatic ductal adenocarcinoma;immune-related gene;prognosis
Acta Acad Med Sin,2024,46(3):354-360
胰腺导管腺癌(pancreatic ductal adenocarcinoma,PDAC)是一种常见的消化道恶性肿瘤,占胰腺癌组织学类型的90%以上,是全球第七大癌症相关死亡原因[1],在我国其患病人数呈逐年上升趋势[2]。尽管近些年PDAC的治疗策略有所改进,但其预后情况仍然十分严峻,5年存活率仅为9%[3]。由于患者个体差异及疾病的异质性,手术和化疗的治疗效果存在差异[4-5]。对PDAC患者进行危险分层,有助于制订精准和个体化的治疗方案,改善患者预后和临床转归。高通量测序技术的不断进步,为揭示基因表达和修饰特性提供了有力支持,从而能够更有效地开发与PDAC预后评估紧密相关的生物标志物[6-8]。根据表达谱聚类结果,PDAC可分为鳞状亚型、胰腺祖细胞亚型、免疫原性亚型和内/外分泌腺异常分化亚型,其中鳞状亚型约占31%,侵袭转移能力强,预后相对较差,中位生存期仅为13.3个月[9]。该亚型的主要分子特征包括TP53和KDM6A基因突变、TP63基因转录网络上调、上皮间质转化基因高表达以及胰腺内胚层细胞命运决定基因的甲基化[10]。识别鳞状亚型的内在分子调控机制有助于PDAC患者的风险评估。此外,研究发现免疫微环境也与PDAC预后密切相关[11-12]。本研究拟通过整合分析鳞状亚型PDAC中的上皮细胞间质转化特征和免疫相关基因的相互关系,利用调控网络推理研究主调控鳞状亚型的免疫标志基因,构建一个风险评估模型,为PDAC患者的精准治疗提供新的思路。
1 资料和方法
1.1 数据来源及处理
本研究纳入6个公共数据集,共903例PDAC患者,其中GSE71729数据集(n=145)作为训练集,PACA-AU(n=242)、PACA-CA(n=193)、TCGA-PAAD(n=146)、GSE85916(n=80)和GSE131050(n=97)作为验证集。采用GEOquery包(V 1.0.7)[13]从基因表达综合(Gene Expression Omnibus,GEO)数据库(https://www.ncbi.nlm.nih.gov/geo/)获取GSE71729、GSE85916和GSE131050数据集的转录谱数据及相应的临床信息。PACA-AU和PACA-CA数据集中标准化的RNA测序数据和临床信息来自国际癌症基因组联盟网站(https://icgc.org/)。采用TCGAbiolinks包(V 2.18.0)[14]从癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库下载TCGA-PAAD数据集的标准化表达谱和相应临床信息。微阵列数据中的探针ID被转换成基因符号,若有多个探针ID对应同一种基因符号,则保留平均表达值最高的探针ID来表示该基因。GSE71729、PACA-AU以及TCGA数据集中的分子分型信息来源于Rashid等[15]的研究。
1.2 整合网络分析
从Immport数据库[16]获取2498个免疫相关基因。采用R软件limma包对PDAC样本进行差异表达分析,以|log2FC|>0.5和校正后P<0.05为阈值筛选鳞状和非鳞状亚型之间差异表达的免疫相关基因。采用RTN包(V 2.10.0)[17]整合差异表达基因并构建调控网络。采用主调控分析计算各免疫基因调控单元中的鳞状亚型特征基因的超几何检验P值,进而得到主调控鳞状亚型的免疫标志基因。
1.3 模型构建和验证
基于筛选的免疫标志基因和患者生存信息,采用多变量Cox比例风险回归构建整合免疫评分(integrating immune score,IIS)模型,模型系数是基因的Cox回归系数,基因值是FPKM。IIS风险赋分公式为:IIS=(0.0068×PLAU)+(0.2584×AQP9)+(-0.3970×COLEC12)+(0.0721×S100A2)+(0.3604×VIM),以上四分位数为阈值将患者分为高危组和低危组。采用Kaplan-Meier生存分析,在5个独立的验证集中对IIS模型进行性能评估。此外,纳入其他临床因素作为协变量,采用单变量和多变量Cox回归分析检验IIS是否为PDAC患者的独立预后因子。
1.4 基因集富集分析
为探究高危组中富集的信号通路,采用R软件HTS包(V 2.3.5)[18]进行基因集富集分析,设定P<0.05为显著性阈值,置换检验次数设置为1000。
1.5 免疫细胞浸润分析
采用CIBERSORT算法表征高危组和低危组患者的肿瘤组织基因表达谱中免疫细胞的浸润丰度。CIBERSORT是一种反卷积算法,可以估算出每个肿瘤组织样本中22种免疫浸润细胞的相对比例。将标准化的表达谱上传至CIBERSORT网站(https://cibersort.stanford.edu/),运行参数设定为1000次随机排列,以LM22作为已知参考集,分析样本中免疫细胞的浸润情况。
1.6 统计学处理
采用R软件limma包(V 3.42.2)[19]进行PDAC鳞状和非鳞状亚型之间的差异分析。采用R软件plotly包对PDAC鳞状和非鳞状亚型之间的差异基因进行主成分分析(principal component analysis,PCA)。采用列线图对多个影响因素进行整合并构建评分,绘制校准曲线评估列线图的预测性能。采用R软件(V 3.6.1)进行统计分析,P<0.05为差异有统计学意义。
2 结果
2.1 免疫标志基因的筛选
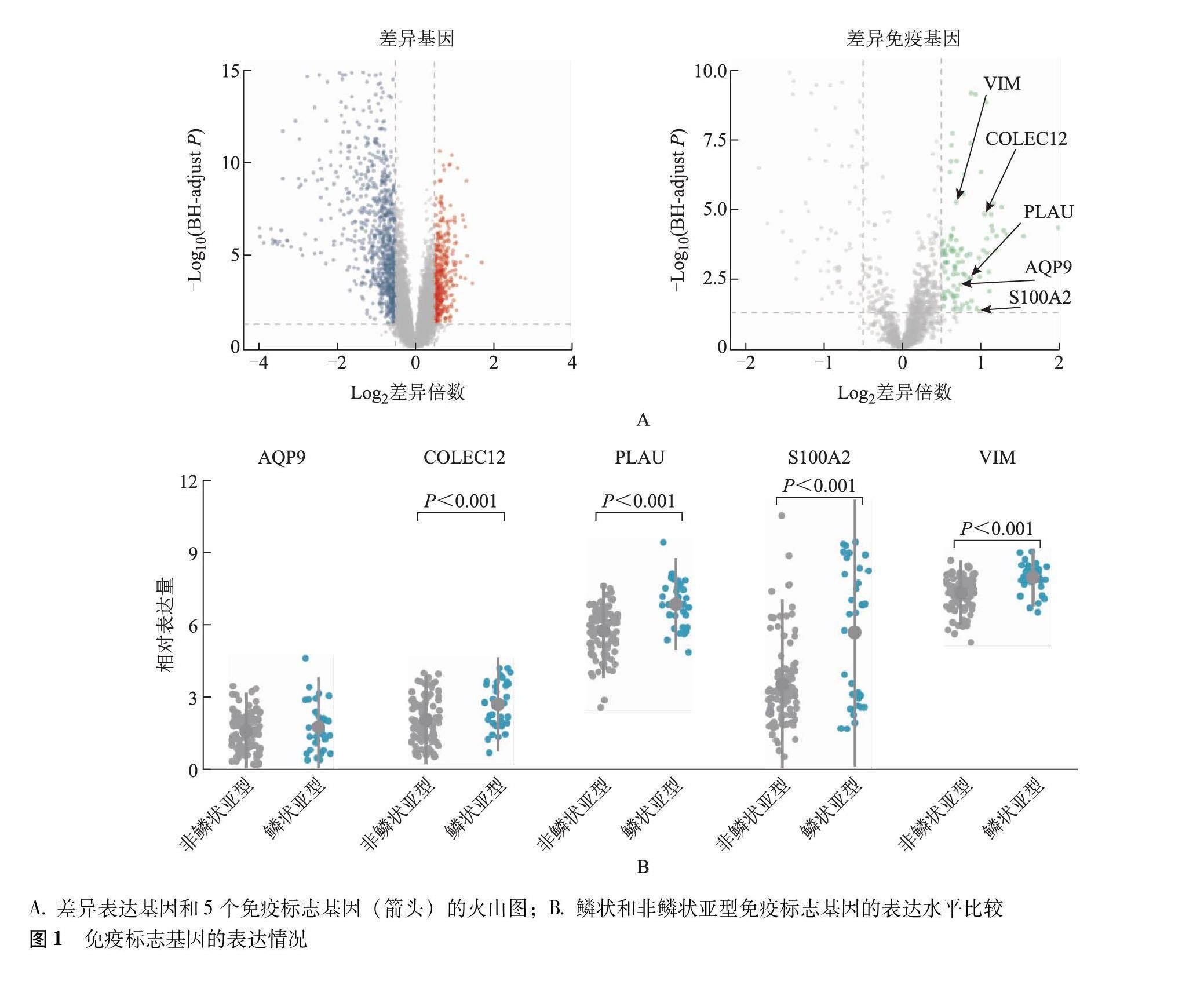
通过差异基因分析从GSE71729数据集中筛选出差异表达的95个免疫相关基因和1269个差异表达基因。PCA法筛选出5个主调控鳞状亚型的免疫标志基因,分别为PLAU(P<0.001)、AQP9(P=0.011)、VIM(P=0.032)、S100A2(P=0.040)和COLEC12基因(P=0.041)(图1A)。在TCGA-PAAD验证集中,与非鳞状亚型比较,鳞状亚型PLAU、VIM、S100A2和COLEC12基因的表达均增高(P均<0.001)(图1B)。
2.2 IIS模型的预测效能
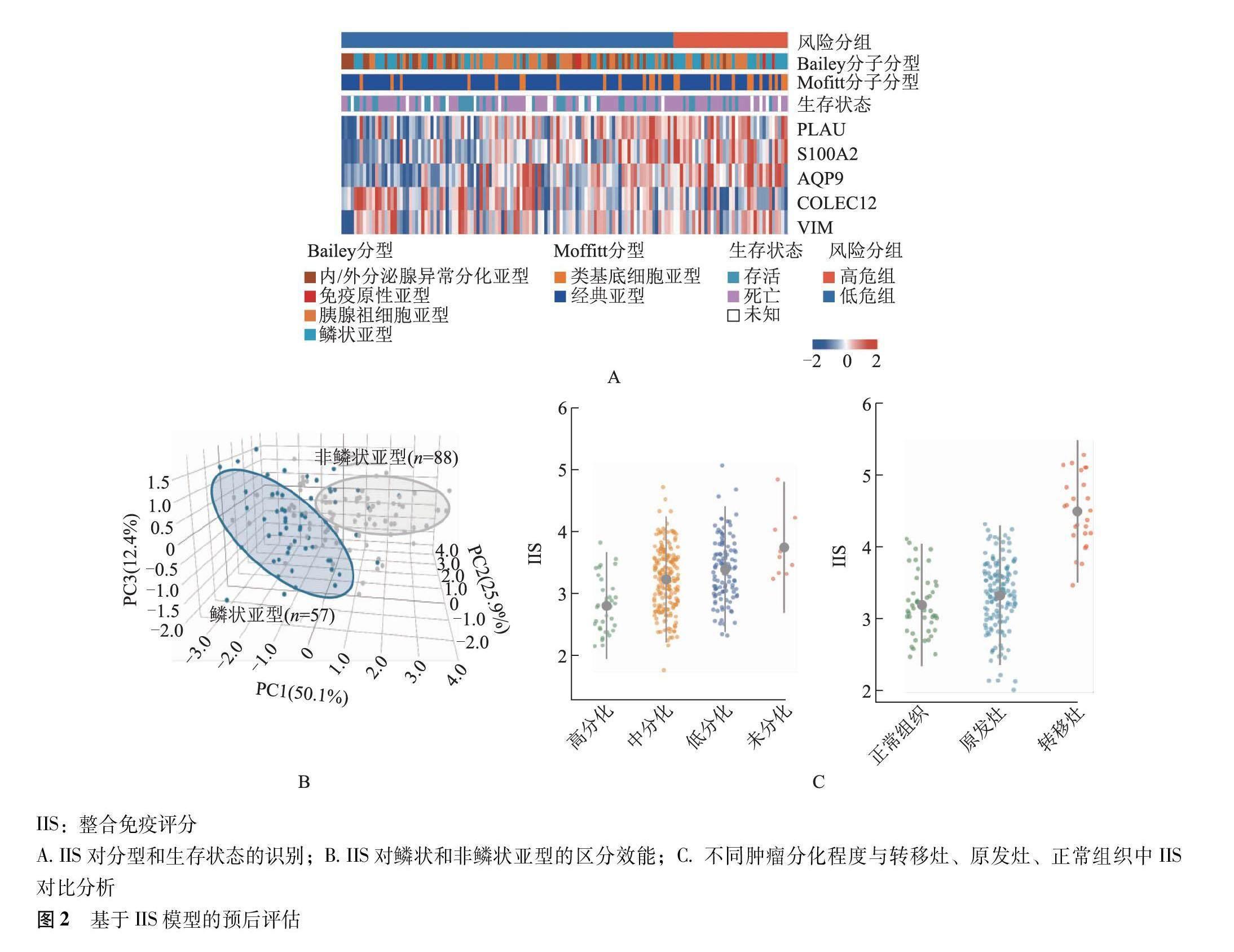
高危组Bailey分型的鳞状亚型(P<0.001)、Moffitt分型的类基底细胞亚型(P<0.001)及死亡患者(P<0.001)的比例高于低危组(图2A)。PCA结果显示,训练集中IIS可以有效地区分鳞状和非鳞状亚型(图2B)。此外,肿瘤分化程度越低IIS越高,且转移灶中的IIS高于原发灶和正常组织(P均<0.001)(图2C)。
2.3 IIS模型验证
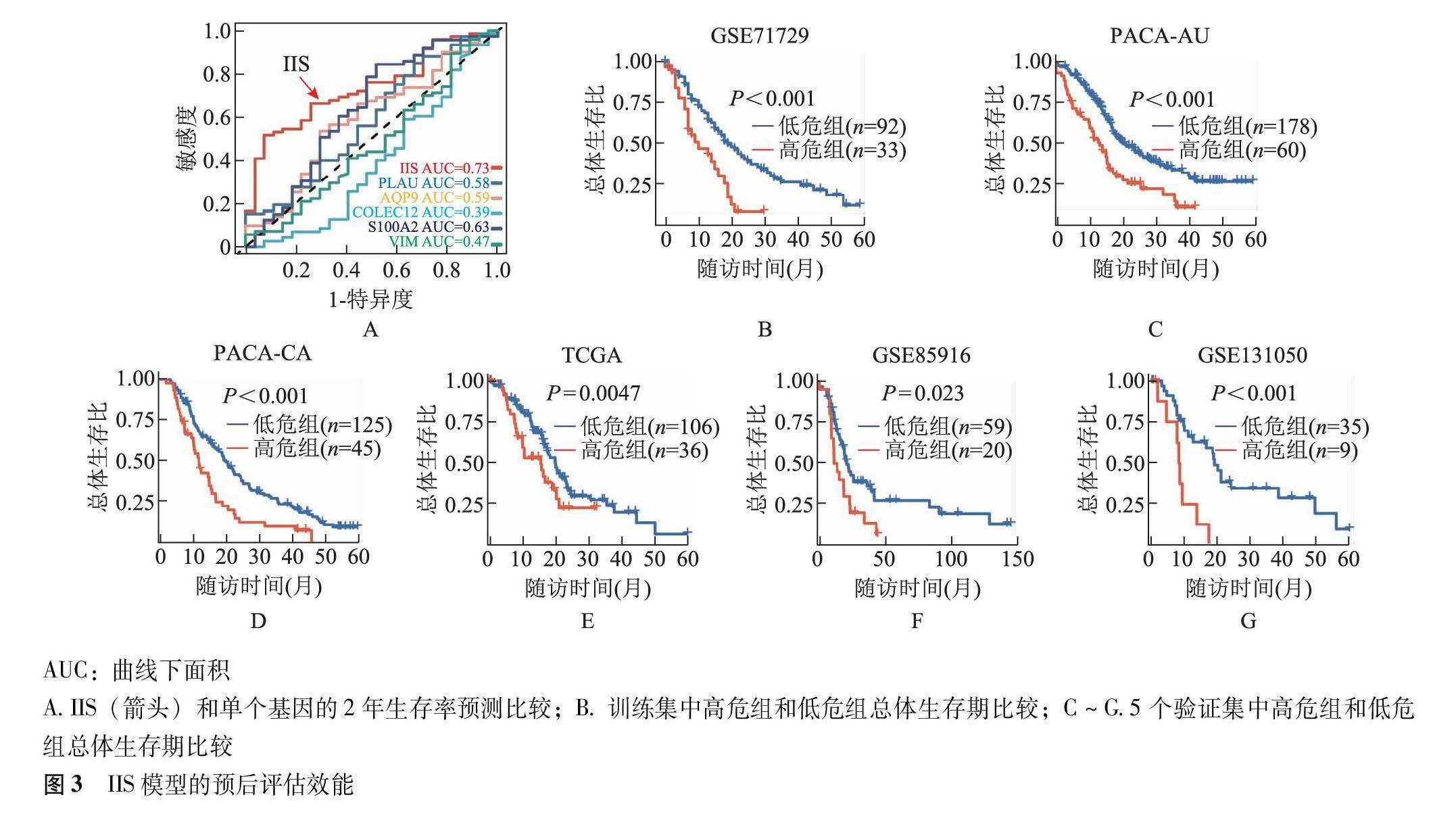
受试者工作特征曲线分析显示,相较于单个免疫标志基因,IIS对患者2年生存时间的预测能力更强,其曲线下面积达到0.73(图3A)。生存分析显示,训练集(HR=2.3,95% CI=1.4~3.8,P<0.001)和PACA-AU(HR=2.0,95% CI=1.4~2.9,P<0.001)、PACA-CA(HR=1.9,95% CI=1.3~2.8,P<0.001)、TCGA-PAAD(HR=1.6,95% CI=1.0~2.7,P<0.001)、GSE85916(HR=1.9,95% CI=1.1~3.4,P<0.001)、GSE131050(HR=4. 95% CI=1.7~10.0,P<0.001)验证集中高危组患者的总体生存期均明显低于低危组(图3B~G)。纳入性别、TNM分期和分化程度等临床因素作为协变量,进行单变量和多变量Cox回归分析,结果显示IIS是PDAC的独立预后因子(HR=2.16,95%CI=1.50~3.10,P<0.001)。
2.4 列线图的预测效能
纳入IIS与年龄、性别、TNM分期和肿瘤分级等变量构建列线图,对患者1年和3年生存率进行预测(图4A);校准曲线显示1年和3年生存率的预测校准曲线趋近标准曲线,说明该模型对PDAC患者预后的预测能力较高(图4B)。
2.5 IIS模型多组学功能分析
基因集富集分析显示,血管生成、上皮间充质转化、胰岛素样生长因子受体、哺乳动物雷帕霉素靶蛋白、血管内皮生长因子、表皮生长因子受体、转化生长因子-β、细胞周期、丝裂原活化蛋白激酶、WNT等相关通路显著富集于高危组,而DNA修复缺陷通路则在高危组中相对下调(图5A)。免疫细胞浸润分析显示,高危组中M0巨噬细胞比例显著增加(P<0.001),低危组中单核细胞和未活化的CD4记忆性T细胞比例显著增加(P<0.00 P=0.009)(图5B)。
3 讨论
PDAC是胰腺癌中最常见的一种类型,其病情隐匿且进展迅速,预后极差。全身化疗是针对进展期和复发PDAC患者最主要的治疗手段,但由于严重的不良反应和患者低耐受性,患者临床收益有限[20]。随着分子生物学的发展以及肿瘤免疫微环境的深入研究,以程序性细胞死亡蛋白1/ 程序性死亡配体1抗体和细胞毒性T淋巴细胞抗原4抑制剂为代表的免疫疗法在肺癌、黑色素瘤、泌尿系统肿瘤中表现出较好的效果,显著改善了患者预后[21]。然而对于PDAC免疫疗法收效甚微,目前的证据表明处于高度免疫抑制状态的肿瘤微环境是阻碍免疫疗法有效性的主要因素。不同的基质和免疫浸润细胞类型与PDAC患者的预后相关[22]。因此,探究肿瘤微环境中的免疫状态对于PDAC整体评估和治疗策略的制订具有重要意义。近年来,癌症分子分型研究揭示了PDAC的分子特征,基于表达谱的特征聚类分析,PDAC被分为鳞状亚型、胰腺祖细胞亚型、免疫原性亚型和内/外分泌腺异常分化亚型,其中鳞状亚型恶性程度最高[9]。本研究筛选了鳞状亚型和非鳞状亚型之间的差异表达基因和差异免疫基因,构建了调控网络,并筛选出主调控鳞状亚型的5个免疫标志基因,分别是PLAU、AQP9、VIM、S100A2和COLEC12基因。这些基因广泛参与免疫信号传导和肿瘤进展。据报道,PLAU在胃癌中呈高表达,其相关基因参与转化生长因子-β、耐药形成等信号通路,免疫细胞浸润状态削弱,提示患者预后不良[23]。AQP9是存在于多种免疫细胞中的一类水甘油通道蛋白,高表达于乳腺癌、结肠癌和肺癌中,与较差预后显著相关,且参与调节CD4+T细胞、CD8+T细胞、中性粒细胞等免疫细胞的浸润[24]。VIM是一种间充质状态标志物,在多种转移性肿瘤中呈高表达[25]。S100A2属于S100蛋白家族,是一类高度保守的EF-手型钙结合蛋白[26],其表达失调与多种癌症的发生发展过程密切相关[27]。Li等[28]研究发现高表达S100A2通过调节糖酵解重编程促进结直肠癌细胞增殖。COLEC12是一种清道夫受体,作为固有免疫系统的模式识别分子,与宿主防御相关的多种功能有关[29],参与白细胞募集和癌症转移[30]。本研究成功构建IIS模型,实现对PDAC患者的危险分层,不同于以往的单基因或多基因标志物,该模型结合免疫系统和转录组水平的分子亚型特征,有效减少了肿瘤异质性的干扰。经IIS危险分层后,生存分析结果发现高危组患者的总体生存期明显低于低危组,且在5个验证集中的表现一致。纳入其他临床因素作为协变量,单变量和多变量Cox回归分析证实IIS是PDAC预后的独立预测因子。
本研究构建了基于公共数据库的PDAC预后模型,尽管纳入了多中心样本并进行了深入分析和验证,但仍存在一些局限性。首先,转录组测序实验操作流程复杂,获取的数据需要依赖专门的计算资源和软件进行处理和解析;其次,不同测序平台数据的标准不一,限制了模型的临床适用性;最后,由于本研究为回顾性分析,样本量有限,统计效力不足。未来工作需结合更多前瞻性数据和分子生物学实验以进一步优化和完善该模型。
综上,本研究结果表明,基于PDAC亚型分类和免疫相关基因构建的IIS模型,在PDAC患者危险分层方面具有一定的应用价值,有助于个体化治疗的实施。
利益冲突 所有作者声明无利益冲突
作者贡献声明 张昱:研究选题设计、数据分析、起草和修改文章;任瑞平:数据收集和整理、数据分析、起草论文;万鹏:数据分析、修订论文学术内容;贺晓兰:研究选题设计、文稿审阅和格式修订
参 考 文 献
[1]Bray F,Ferlay J,Soerjomataram I,et al.Global cancer statistics 2018:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J].CA Cancer J Clin,2018,68(6):394-424.DOI:10.3322/caac.21492.
[2]冯程程,许传志,何杰宇,等.1990-2019年中国20~84岁人群胰腺癌发病趋势分析[J].中华肿瘤防治杂志,2022,29(18):1323-1329.DOI:10.16073/j.cnki.cjcpt.2022.18.04.
[3]DeSantis CE,Ma J,Gaudet MM,et al.Breast cancer statistics,2019[J].CA Cancer J Clin,2019,69(6):438-451.DOI:10.3322/caac.21583.
[4]Springfeld C,Ferrone CR,Katz MHG,et al.Neoadjuvant therapy for pancreatic cancer[J].Nat Rev Clin Oncol,2023,20(5):318-337.DOI:10.1038/s41571-023-00746-1.
[5]Lo W,Zureikat A.Neoadjuvant therapy in pancreatic cancer:a review and update on recent trials[J].Curr Opin Gastroenterol,2022,38(5):521-531.DOI:10.1097/MOG.0000000000000874.
[6]Chen H,Yu Y,Zhou L,et al.Cuproptosis-related LncRNAs signature as biomarker of prognosis and immune infiltration in pancreatic cancer[J].Front Genet,2023,14:1049454.DOI:10.3389/fgene.2023.1049454.
[7]Zhang S,Yang J,Wu H,et al.Establishment of a 7-gene prognostic signature based on oxidative stress genes for predicting chemotherapy resistance in pancreatic cancer[J].Front Pharmacol,2023,14:1091378.DOI:10.3389/fphar.2023.1091378.
[8]Chen K,Wang Q,Liu X,et al.Single cell RNA-seq identifies immune-related prognostic model and key signature-SPP1 in pancreatic ductal adenocarcinoma[J].Genes,2022,13(10):1760.DOI:10.3390/genes13101760.
[9]Collisson EA,Bailey P,Chang DK,et al.Molecular subtypes of pancreatic cancer[J].Nat Rev Microbiol,2019,16(4):207-220.DOI:10.1038/s41575-019-0109-y.
[10]Giordano M,Cacciato Insilla A,Campani D.Molecular subtypes of pancreatic ductal adenocarcinoma[M]//Esposito I,Karamitopoulou-Diamantis E.Pathology of the pancreas.Cham:Springer International Publishing,2022:136-140.DOI:10.1007/978-3-319-28845-1_5533-1.
[11]Falcomatà C,Brthel S,Schneider G,et al.Context-specific determinants of the immunosuppressive tumor microenvironment in pancreatic cancer[J].Cancer Discov,2023,13(2):278-297.DOI:10.1158/2159-8290.CD-22-0876.
[12]Sherman MH,Beatty GL.Tumor microenvironment in pancreatic cancer pathogenesis and therapeutic resistance[J].Annu Rev Pathol,2023,18:123-148.DOI:10.1146/annurev-pathmechdis-031621-024600.
[13]Davis S,Meltzer PS.GEOquery:a bridge between the Gene Expression Omnibus (GEO) and BioConductor[J].Bioinformatics,2007,23(14):1846-1847.DOI:10.1093/bioinformatics/btm254.
[14]Colaprico A,Silva TC,Olsen C,et al.TCGAbiolinks:an R/Bioconductor package for integrative analysis of TCGA data[J].Nucleic Acids Res,2016,44(8):e71.DOI:10.1093/nar/gkv1507.
[15]Rashid NU,Peng XL,Jin C,et al.Purity independent subtyping of tumors (PurIST),a clinically robust,single-sample classifier for tumor subtyping in pancreatic cancer[J].Clin Cancer Res,2020,26(1):82-92.DOI:10.1158/1078-0432.CCR-19-1467.
[16]Bhattacharya S,Andorf S,Gomes L,et al.ImmPort:disseminating data to the public for the future of immunology[J].Immunol Res,2014,58:234-239.DOI:10.1007/s12026-014-8516-1.
[17]Fletcher MNC,Castro MAA,Wang X,et al.Master regulators of FGFR2 signalling and breast cancer risk[J].Nat Commun,2013,4(1):2464.DOI:10.1038/ncomms3464.
[18]Wang X,Terfve C,Rose JC,et al.HTSanalyzeR:an R/Bioconductor package for integrated network analysis of high-throughput screens[J].Bioinformatics,201 27(6):879-880.DOI:10.1093/bioinformatics/btr028.
[19]Ritchie ME,Phipson B,Wu DI,et al.limma powers differential expression analyses for RNA-sequencing and microarray studies[J].Nucleic Acids Res,2015,43(7):e47-e47.DOI:10.1093/nar/gkv007.
[20]Von Hoff DD,Ervin T,Arena FP,et al.Increased survival in pancreatic cancer with nab-paclitaxel plus gemcitabine[J].N Engl J Med,2013,369(18):1691-1703.DOI:10.1056/NEJMoa1304369.
[21]Aroldi F,Zaniboni A.Immunotherapy for pancreatic cancer:present and future[J].Immunotherapy,2017,9(7):607-616.DOI:10.2217/imt-2016-0142.
[22]Mahajan UM,Langhoff E,Goni E,et al.Immune cell and stromal signature associated with progression-free survival of patients with resected pancreatic ductal adenocarcinoma[J].Gastroenterology,2018,155(5):1625-1639.e2.DOI:10.1053/j.gastro.2018.08.009.
[23]Ai C,Zhang J,Lian S,et al.FOXM1 functions collaboratively with PLAU to promote gastric cancer progression[J].J Cancer,2020,11(4):788.DOI:10.7150/jca.37323.
[24]Liu X,Xu Q,Li Z,et al.Integrated analysis identifies AQP9 correlates with immune infiltration and acts as a prognosticator in multiple cancers[J].Sci Rep,2020,10(1):20795.DOI:10.1038/s41598-020-77657-z.
[25]Baumgart E,Cohen MS,Neto BS,et al.Identification and prognostic significance of an epithelial-mesenchymal transition expression profile in human bladder tumors[J].Clin Cancer Res,2007,13(6):1685-1694.DOI:10.1158/1078-0432.CCR-06-2330.
[26]Chen H,Xu C,Liu Z.S100 protein family in human cancer[J].Am J Cancer Res,2014,4(2):89-115.
[27]Wolf S,Haase-Kohn C,Pietzsch J.S100A2 in cancerogenesis:a friend or a foe[J].Amino acids,201 41:849-861.DOI:10.1007/s00726-010-0623-2.
[28]Li C,Chen Q,Zhou Y,et al.S100A2 promotes glycolysis and proliferation via GLUT1 regulation in colorectal cancer[J].FASEB J,2020,34(10):13333-13344.DOI:10.1096/fj.202000555R.
[29]Ma YJ,Hein E,Munthe-Fog L,et al.Soluble collectin-12 (CL-12) is a pattern recognition molecule initiating complement activation via the alternative pathway[J].J Immunol,2015,195(7):3365-3373.DOI:10.4049/jimmunol.1500493.
[30]Elola MT,Capurro MI,Barrio MM,et al.Lewis x antigen mediates adhesion of human breast carcinoma cells to activated endothelium.Possible involvement of the endothelial scavenger receptor C-type lectin[J].Breast Cancer Res Treat,2007,101:161-174.DOI:10.1007/s10549-006-9286-9.
(收稿日期:2023-06-20)

