利用种子红色荧光标记鉴定转基因番茄后代
章力 魏凯 李珊珊 宁宇 路菲菲 王孝宣 国艳梅 刘磊 李鑫 杜永臣 李君明 黄泽军

摘要:转基因技术有助于番茄基因功能研究和遗传改良,其中转基因后代的筛选是一个非常重要但工作量大的环节。本研究基于番茄油体蛋白基因家族序列分析和番茄基因表达数据库SCN-TEA搜索结果,克隆了一个种子特异且高水平表达的油体蛋白基因SIOLEI(Solyc06g034040)。利用无缝克隆的方法将红色荧光蛋白基因TagRFP的编码区序列插入到SIOLE1基因的终止密码子前,形成一个嵌合基因SIOLEl-TagRFP。将嵌合基因插入到pBI121双元载体,构建植物表达载体pSIOLEl-TagRFP,利用农杆菌介导法转化番茄品种‘Money Maker,To代植株的自交种子在荧光显微镜下呈现出明亮红色荧光或无荧光。PCR分子标记进一步验证发现,红色荧光种子萌发的幼苗均存在TagRFP序列,表明在种子阶段检测红色荧光筛选转基因番茄后代的准确率为100%。由此,本研究建立了一种利用SIOLEl-TagRFP嵌台基因,可以通过种子红色荧光可视化分析,简单快速、低成本地鉴定转基因番茄后代的方法。
关键词:番茄;种子;红色荧光蛋白;油体蛋白;转基因鉴定
中图分类号:S641.2:Q781 文献标识号:A 文章编号:1001-4942(2024)04-0001-08
转基因技术以及借助转基因的基因编辑技术,促进了植物基因功能研究,而且有助于改良植物性状和培育优良新品种。植物遗传转化后,转化植株及其后代中外源DNA的检测是一个十分必要的环节,但工作量大、效率有待提高。在植物的遗传转化工作中,常借助选择基因和报告基因筛选转基因植株。以卡那霉素(nptll)和潮霉素(hpt)等抗生素或者EPSPS和Bw等除草剂抗性基因作为选择基因进行筛选,不同植物材料对筛选剂所需的最适浓度差别较大,会出现假阳性结果,甚至有时会影响植株的正常生长。利用cus作为报告基因筛选时,需要对转基因植株组织进行GUS染色检测,会破坏植物组织。
FAST(fluorescence accumulating seed technology)是一种通过种子特异性启动子驱动荧光融合蛋白表达,利用种子荧光快速筛选转基因植株的方法,最早应用于模式植物拟南芥(Arabidopsis thaliana)。荧光蛋白是一类在特定波长下激发强烈荧光的蛋白质,可作为报告基因用于检测基因特异性表达和融合蛋白分子的定位、迁移、构象变化以及分子间的相互作用。红色荧光蛋白(red fluorescem protein,RFP)是从海葵(Discosomasp sp.)中分离出的与绿色荧光蛋白(Ween fluorescent protein,GFP)同源的荧光蛋白,激发波长和发射波长均较长,其中,TagRFP的激发波长和发射波长分别为555 nm和584 nm,其亮度大约是mCherry蛋白的3倍,具有高pH稳定性和荧光寿命长等特点,是目前所报道的相对较亮的单体红色荧光蛋白。FAST技术将荧光蛋白作为可视化筛选标记直接鉴定转基因种子,方法简便且不会对植株产生破坏,能够降低大面积种植的成本,有效提高筛选效率。
油体蛋白(oleosin)是一类特殊的贮藏蛋白,有些油体蛋白在植物种子中特异表达。当外源小分子量蛋白插入到油体蛋白的N端或C端后不会影响其表达和定位,且融合蛋白非常稳定。番茄(Solcumm lycopersicum)是一种具有重要经济价值的蔬菜作物,也是果实发育基础研究的模式植物。转基因技术和基因编辑技术已经大量应用于番茄基础研究和性状改良,然而传统的方法无法在种子阶段区分转基因和非转基因材料,转基因后代鉴定效率比较低。本研究参考FAST技术,利用番茄种子特异且高水平表达的油体蛋白基因StOLEI启动子驱动TagRFP融合蛋白表达,利用种子红色荧光快速有效筛选番茄转基因后代,为后续的基因功能研究和性状改良带来便利。
1材料与方法
1.1植物材料与菌株
番茄品种‘Rio Grande来自美国俄亥俄州立大学Esther van der Knaap实验室,于2020年种植于中国农业科学院蔬菜花卉研究所南区温室。番茄品种‘Money Maker(MM)的种子由中国农业科学院蔬菜花卉研究所番茄遗传育种课题组扩繁保存。大肠杆菌DH5a和农杆菌GV3101购自上海唯地生物技术有限公司。
1.2番茄油体蛋白的鉴定与分析
根据拟南芥油体蛋白研究结果,从TAIR(the arabidopsis information resource)数据库下载17个拟南芥油体蛋白序列。以拟南芥油体蛋白序列为查询序列,采用BLASTP(e-value<1E-5)在茄科基因组数据库SGN(solanaceae genomics network)中检索番茄油体蛋白。从NCBI数据库获得水稻(Oryza sativa,)油体蛋白序列。利用ClustaIW软件进行番茄、拟南芥和水稻油体蛋白多序列比对,使用MEAG-X软件、采用最大似然法(maximum likelihood)构建油体蛋白序列的进化树。利用番茄果实发育成熟高分辨率时空转录图谱数据库SGN-TEA(https://tea.sgn.cornell.edu/)分析油体蛋白基因在成熟期果实组织的表达模式。
1.3TagRFP和番茄油体蛋白基因的克隆
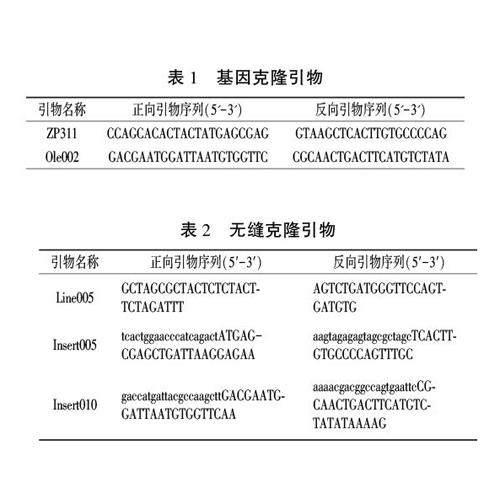
以中国农业科学院蔬菜花卉研究所张金喆老师惠赠的含红色荧光蛋白基因TagRFP编码序列的番茄DNA为模板,使用引物ZP311,利用高保真聚合酶预混液Apex HF FL PCR Master Mix(艾科瑞生物),对TagRFP进行PCR扩增。反应程序为:94℃1min;98℃10 s,60℃15 s,68℃1 min,35个循环。以CTAB法提取的番茄品种Rio Grande幼嫩叶片的基因组DNA为模板,使用引物Ole002扩增番茄油体蛋白基因SlOLEI(Solyc069034040)全长序列。引物序列见表1。
PCR产物经1%琼脂糖凝胶电泳检测后,采用EasyPure@ Quick Gel Extraction Kit(全式金)进行胶回收纯化。TagRFP和StOLEI的回收产物均与Blunt Zero克隆载体(全式金)连接,转化至DH5a感受态细胞,挑选阳性克隆送至生工生物工程(上海)股份有限公司测序。
1.4种子StOLEI-TagRFP红色荧光表达载体的构建
根据研OLEJ克隆载体序列和TagRFP编码序列,采用软件CE Design设计无缝克隆引物(表2)。无缝克隆采用ClonExpressll One Step Ooning Kit(诺唯赞),反应条件为37℃连接30 min。以slOLEI基因克隆载体为模板,使用引物Line005进行反向PCR;以TagRFP克隆载体为模板,使用引物Insen005扩增TagRFP编码序列。分别对PCR产物进行胶回收纯化,然后进行无缝克隆,将TagRFP编码序列插入到SloLEI基因终止密码子前面,构建SlOLEI-TagRFP嵌合基因克隆载体Blunt-OR,嵌合基因序列中仅保留1个终止密码子。使用EcoRI和HindⅢ限制性内切酶对pBI121植物双元表达载体(华越洋)进行双酶切,以克隆载体Blunt-OR为模板,使用引物Insert010扩增SlOLEl-TagRFP嵌合基因片段,胶回收纯化后与pB1121双酶切产物进行无缝克隆,构建表达载体pSIOLEl-TagRFP。
1.5种子SIOLEI-TagRFP红色荧光表达载体的遗传转化
利用冻融法将表达载体pSIOLEl-TagRFP转入农杆菌GV3101,通过农杆菌介导法进行番茄MM子叶的遗传转化。遗传转化方案参照文献[18],将MM的种子消毒后置于1/2MS培养基,待子叶展开且真叶尚未长出时剪下子叶,在A1培养基中暗培养2d后进行侵染,侵染的子叶继续在A1培养基中培养2d,经A2培养基诱导得到愈伤组织和分化的芽,A3培养基分化培养得到小苗,最后由A4培养基生根培养得到生根小苗后移栽至中国农业科学院蔬菜花卉研究所北圃场玻璃温室。
1.6转基因番茄的PCR检测和种子荧光鉴定
提取转基因番茄植株的DNA,根据表达载体中TagRFP两侧序列设计引物O+R-7(F:GGTA-AGCGTG17ATCGTGGA:R:ATGAAGACGAGCCTCG-TAGC),PCR检测To代植株中是否插入TagRFP基因序列。将To代阳性转基因植株自交授粉收获的干燥种子,置于徕卡体视荧光显微镜(Leica MZ10F)下进行荧光成像和拍照。挑选呈现荧光和无荧光的种子分别催芽,真叶长出后提取DNA,进一步通过PCR验证番茄转基因后代中是否含有TagRFP基因。
2结果与分析
2.1番茄油体蛋白基因分析
在SGN数据库中检索到番茄中有9个油体蛋白编码基因,分别是Solyc029086490、Solyc039112440、Solyc039119820、Solyc069034040、Solyc069060840、Solyc069069260、Solyc089066040、Solyc089078160和Solyc129010920。在NCBI数据库中检索到6个水稻油体蛋白编码基因:Os0190643900、Os03949190、Os0490546500、Os0590576700、Os0690473800和Os0990324000。
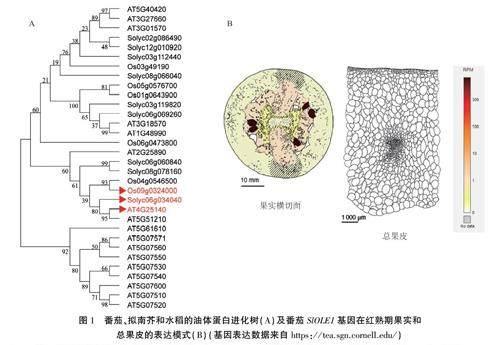
为了明确番茄、拟南芥和水稻油体蛋白之间的亲缘关系,对番茄9个、拟南芥17个和水稻6个的油体蛋白构建分子进化树(图1A)。在进化树中,番茄油体蛋白基因Solyc069034040与拟南芥中的AT4G25140(AtOLEI)和AT5G51210基因以及水稻的Os0990324000(OsoLE4)和Os0490546500(OsOLE3)基因亲缘关系最近。AT4G25140(AtOLEl)是拟南芥种子中最丰富的油体蛋白。利用自身启动子分别驱动AT4G25140(AtOLEI)、Os0990324000(OsOLE4)基因与GFP基因的融合蛋白基因表达,可使转基因拟南芥和水稻的种子呈现出绿色荧光。随后检索番茄基因表达数据库SGN-TEA(https://tea.sgn.cornell.edu/),发现Solyc039112440、Sotyc129010920和Solyc069034040基因在红熟期种子中的表达量居前三,RPM值分别为2114.94、1719.41和1185.15。由于Sotyc069034040在番茄红熟期果实的其他组织中几乎不表达(图1B),且其编码蛋白与拟南芥AT4G25140( AtOLEl)和水稻Os0990324000(OsOLE4)蛋白序列相似性最高,因此,推测Solyc069034040基因(以下简称SIOLEJ)启动子可以驱动融合蛋白在番茄种子中表达。
2.2种子红色荧光表达载体pSlOLEl-TagRFP构建
以番茄‘Rio Grande的DNA为模板,经引物Ole002扩增得到长度为4004 bp的SlOLE1基因全长片段(图2A),其中含启动子和终止子序列长度各约1.7 kb。利用引物ZP311扩增TagRFP编码区序列(715 bp,图2B)。PCR扩增片段分别连人Blunt Zero克隆载体,测序后获得序列正确的克隆载体Blunt-SIOLEl和Blunt-TagRFP。
将质粒Blunt-SIOLEl通过反向PCR线性化(引物Line005,表2),与质粒Blunt-TagRFP为模板的PCR产物的胶回收纯化产物(引物Insen005,表2)进行无缝克隆,成功在SlOLE1基因终止密码子前面插入TagRFP编码区序列,获得重组质粒Blunt-OR。
将pB1121双元载体的EcoR I和HindⅢ双酶切产物,与质粒Blunt-OR为模板的PCR产物的胶回收纯化产物无缝克隆(引物Insert010,表2)。无缝克隆产物转化大肠杆菌DH5a,利用引物ZP311进行菌液PCR鉴定,以Blunt-TagRFP质粒为阳性对照,有9份菌液扩增出条带,大小约为715 bp,其中菌液SIOLEl-TagRFP-14的测序结果完全正确,成功构建种子红色荧光表达载体pSlOLEl-TagRFP(图2C、图3)。
2.3SlOLEl-TagRFP转基因阳性番茄植株获得
将pSIOLEl-TagRFP质粒转入农杆菌GV3101,以番茄MM的子叶为外植体进行侵染,经组织培养共获得45个再生植株。以转基因植株的DNA为模板,使用引物O+R-7对TagRFP基因进行PCR检测。以pSIOLEl-TagRFP质粒为阳性对照,野生型MM为阴性对照,水为空白对照,结果(图4)显示,有8株To代番茄植株的扩增产物与野生型MM一致,为单一条带,表明没有插入TagRFP基因;其余37株均扩增出杂合条带,表明成功插入了TagRFP编码区序列,遗传转化效率达到82.22%。
2.4转基因番茄后代种子红色荧光观察及PCR鉴定
挑选9株生长健壮的SlOLEl-TagRFP阳性转基因番茄幼苗,移栽至玻璃温室,振荡自交授粉。将To代植株自交种子干燥后置于徕卡体视荧光显微镜下,以野生型MM的种子为阴性对照,分别在明场和荧光光源下检测TagRFP信号。结果(图5)显示,在明场下,野生型MM和转基因干燥种子颜色无明显差异:在荧光下,野生型种子均无红色荧光,转基因种子部分呈现明亮红色荧光。表明TagRFP可以作为一种标记筛选出转基因番茄后代中的红色荧光种子。
为了验证通过种子红色荧光检测番茄转基因后代的准确性,进一步播种转基因植株的红色荧光种子和无荧光种子各96粒,使用引物O+R-7进行PCR,鉴定后代植株是否存在TagRFP序列。结果表明,96株红色荧光种子萌发的幼苗扩增产物均为杂合条带,表明含有TagRFP编码区序列,而96株无荧光种子萌发的幼苗扩增产物均为单一条带,表明不含TagRFP编码区序列。荧光种子和无荧光种子各12粒萌发的幼苗PCR检测结果如图6所示,表明通过种子红色荧光鉴定番茄转基因后代的准确率达100%。
3讨论与结论
荧光蛋白有多种类型,其中,绿色荧光蛋白(GFP)、蓝色荧光蛋白(BFP)、青色荧光蛋白(CFP)和黄色荧光蛋白(YFP)等的发射波长局限在440-529nm,在细胞内成像时会激发某些内源物质产生荧光,对结果造成不同程度的干扰。本研究所使用的TagRFP的发射波长为584 nm,细胞内成像背景低,更容易检测。通过PCR分子标记检测发现,利用TagRFP作为筛选标记,区分转基因与非转基因种子的准确率可达100%。此外,使用荧光蛋白标记筛选转基因种子时,可利用荧光种子分选设备进一步提高鉴定效率。
种子特异性表达启动子驱动荧光蛋白基因,可用于转基因种子的可视化鉴定。在拟南芥中使用种子储藏蛋白基因napin启动子驱动荧光蛋白进行表达,可以在荧光显微镜下识别转基因种子。拟南芥和水稻油体蛋白基因AtOLEI、Os-OLE4与GFP的融合蛋白基因表达,可使转基因种子呈现绿色荧光,从而实现快速简便鉴定转基因后代。本研究发现SlOLE1(Solyc069034040)是拟南芥AtOLE1基因和水稻OsOLE4基因的同源基因,而且在番茄种子中特异、高水平表达。利用SlOLE1基因启动子驱动SlOLEl-TagRFP融合蛋白表达,成功实现了通过种子红色荧光快速简便鉴定转基因番茄后代的目的。由此推测,利用植物自身种子特异性启动子驱动油体蛋白与荧光蛋白融合基因的表达,可以应用于其他含油体蛋白种子植物的转基因后代鉴定。
种子荧光标记系统已经逐步应用于基础研究和育种领域。基因编辑通常借助稳定的遗传转化,需要对阳性转基因植株和不舍转基因成分的突变后代进行筛选,工作量大,费用高。玉米种子荧光报告系统辅助的CRISPR/Cas基因编辑技术通过胚中的红色荧光标记准确鉴别转基因或非转基因籽粒,显著提高了基因编辑工作效率。水稻智能不育育种技术将育性恢复基因、花粉失活基因和种子特异启动子驱动的红色荧光蛋白基因串联,一起导入到水稻雄性不育突变体中,自交可产生无荧光的雄性不育种子和有荧光的可育种子。玉米雄性不育制种新技术也可根据红色荧光实现不育系和保持系种子的分选。利用单倍体诱导系的单倍体育种技术可以加快纯系选育进程,提高育种效率。然而单倍体诱导效率一般不高,鉴别也较为困难。利用基因编辑技术创制拟南芥、玉米和马铃薯等植物的单倍体诱导系,结合种子荧光标记,提高了单倍体鉴别效率。Zhong等报道的番茄单倍体荧光鉴别技术体系中使用的是拟南芥FAST-Red标记。
本研究克隆了番茄自身的种子特异性表达基因StOLEJ,并构建了StOLEl-TagRFP嵌合基因。稳定遗传转化SIOLEI-TagRFP嵌合基因的番茄种子在激发光照射下发出明亮的红色荧光,准确率可达100%。该种子红色荧光标记筛选体系将有望应用于番茄基因编辑、智能雄性不育系创制和单倍体鉴别等领域,具有一定基础研究和育种应用价值。

