棉花CMS-D2和CMS-D8不育系线粒体全基因组比较分析
葛李爽 冯娟娟 张梦 郭立平 戚廷香 张学贤 李永旗 唐会妮 乔秀琴 邢朝柱 吴建勇


摘要:【目的】探究哈克尼西棉不育胞質(CMS-D2)和三裂棉不育胞质(CMS-D8)的不育系线粒体基因组之间的序列结构差异,为筛选鉴定不育相关基因奠定基础。【方法】根据D2A和D8A这2个不育系的线粒体基因组测序组装结果,使用Synteny and Rearrangement Identifier(SyRI)软件鉴定结构变异,用Plotsr可视化分析包含共线性及非共线性区域的重组变异位点。以D8A线粒体基因组注释结果为参考,用D2A线粒体基因组注释的开放阅读框(open reading frame, ORF)编码的氨基酸序列进行tblastn比对,筛选出D2A线粒体基因组中特有的ORF,并进行聚合酶链式反应(polymerase chain reaction, PCR)验证、相对表达量分析和生物信息学分析。【结果】D2A和D8A这2个不育系线粒体基因组之间存在2个部分重叠且相邻的倒易位区域。在D2A中发现17个特异的ORF,PCR验证了D2A中存在的6个特异ORF(orf114e、orf121b-1、orf121b-2、orf138b-2、orf186a-2和orf317a-2)。3~4 mm花蕾中orf121b-1和orf121b-2的相对表达量较高。orf114e、orf186a-2和orf317a-2具有典型的跨膜结构域和嵌合基因结构,符合不育基因的部分特征。【结论】D2A和D8A线粒体基因组间存在2个相邻的倒易位区域。D2A中存在6个特异的ORF,其中orf114e、orf186a-2和orf317a-2可能与棉花CMS-D2孢子体败育有关。
关键词:棉花;细胞质雄性不育;线粒体基因组;孢子体不育;配子体不育;开放阅读框
Comparative analysis of whole mitochondrial genomes between CMS-D2 and CMS-D8 sterile lines in cotton
Abstract: [Objective] This research aims to investigate the sequence structural differences between the mitochondrial genomes of Gossypium harknessii sterile cytoplasm (CMS-D2) and G. trilobum sterile cytoplasm (CMS-D8), which can establish a foundation for the screening and identification of sterility-related genes. [Methods] Based on the sequencing and assembled results of the mitochondrial genome of D2A and D8A, the Synteny and Rearrangement Identifier (SyRI) software was used to identify structural variations, and recombination variation sites containing collinear and non-collinear regions were visualized with Plotsr. The annotation results of the D8A mitochondrial genome were utilized as a reference, and the amino acid sequences encoded by the open reading frame (ORF) annotated in the D2A mitochondrial genome were used for tblastn comparison to screen out the unique ORF in the D2A mitochondrial genome. Subsequently, polymerase chain reaction (PCR) verification, relative expression level analysis, and bioinformatics analysis were performed. [Results] The mitochondrial genomes of the sterile lines D2A and D8A had two partially overlapped and adjacent inverted translocation regions. Seventeen specific ORF were found in D2A mitochondrial genome, and PCR verified the presence of 6 specific ORF (orf114e, orf121b-1, orf121b-2, orf138b-2, orf186a-2, and orf317a-2) in D2A. The relative expression levels of orf121b-1 and orf121b-2 were higher in 3-4 mm flower buds. orf114e, orf186a-2, and orf317a-2 have typical transmembrane domains and chimeric gene structures, which are consistent with some characteristics of sterility genes. [Conclusion] Two adjacent inverted translocation regions were identified between the mitochondrial genomes of D2A and D8A. There are six specific ORF in D2A, among which orf114e, orf186a-2, and orf317a-2 may be potentially related to sporophytic abortion of CMS-D2.
Keywords: cotton; cytoplasmic male sterility; mitochondrial genome; sporophyte sterility; gametophyte sterility; open reading frame
细胞质雄性不育(cytoplasmic male sterility, CMS)是指雌蕊正常发育,雄蕊丧失正常授粉功能的现象,不仅可以作为植物杂种优势利用的重要育种工具,也为研究细胞质-细胞核互作提供了重要材料[1]。目前,已发现至少150种植物中存在CMS现象[2]。利用CMS进行三系(不育系、保持系和恢复系)杂交育种具有操作简便、种子纯度高、成本较低等特点。因此三系法逐渐成为培育杂交种最便捷、有效的方法之一[3],已在水稻[4]、玉米[5]、甘蓝型油菜[6]等作物中广泛应用。
CMS是线粒体基因与核基因相互作用的结果[7]。线粒体是细胞内氧化代谢的主要场所,能够为细胞的生命活动提供能量,自身含有编码部分蛋白质的遗传物质。不同物种之间线粒体基因组(mitochondrial genome, mtDNA)大小具有较大差异,动物细胞中线粒体基因组一般仅为10~39 kb,而植物线粒体基因组可达100 kb~11 Mb。植物线粒体基因组含有大量的重复序列并具有整合其他基因组DNA序列的特性[8]。大量研究表明,CMS一般是由线粒体基因组结构异常引起,由于其自身基因组频繁发生重组与重排,易产生嵌合的开放阅读框(open reading frame, ORF),形成嵌合基因(新ORF),其编码产物能够通过不同形式影响线粒体的正常功能[9]。例如,在水稻CMS-BT中,嵌合基因orf79可以编码毒性蛋白,通过与线粒体内膜结合影响线粒体的功能,从而引起花粉败育[10]。在玉米CMS-C中,嵌合基因atp6c编码的蛋白通过与ATP6竞争作用,影响F1Fo-ATP合酶的组装,导致线粒体内膜质子过量积累,引发活性氧(reactive oxygen species, ROS)爆发、绒毡层过早程序性死亡,进而导致花粉败育[11]。
近年来,不育基因的挖掘一般是通过对线粒体基因组的序列进行比对,筛选出符合不育基因特征的ORF作为候选基因。大多数不育基因包含3个特征:(1)线粒体嵌合ORF;(2)至少具有1个跨膜结构域;(3)在不育系中特有或与其同核异质的保持系存在序列差异[12]。利用这种方法已经在许多作物中挖掘出一些CMS基因。例如,通过在D1-CMS型水稻中进行线粒体基因组比对,发现了不育基因orf182,实验验证发现orf182通过降低腺苷三磷酸(adenosine triphosphate, ATP)生成导致花粉败育[13]。在大豆中,发现了雄性不育候选基因orf178和orf261[14]。研究人员通过对棉花CMS-D2线粒体基因组进行组装,并与已公布的陆地棉(Gossypium hirsutum)CMS中保持系线粒体基因组比对分析[15],筛选出CMS-D2中的雄性不育候选基因orf610a,并对其功能进行了初步验证[16]。
花粉育性是由孢子体基因型或配子体基因型决定,由此CMS可分为孢子体不育和配子体不育共2种类型[17]。孢子体不育类型的育性受细胞核基因和细胞质基因控制,杂种1代可育花粉率为100%;配子体不育则是由配子体本身的基因型控制,杂种1代可育花粉率为50%[18]。哈克尼西棉(G. harknessii)胞质不育系CMS-D2属于孢子体不育,是目前棉花三系杂交种生产中的主要不育类型。三裂棉(G. trilobum)胞质不育系CMS-D8属于配子体不育,败育彻底且稳定遗传,CMS-D8三系材料是培育三系杂交种重要的种质资源。研究表明,许多导致植物CMS的线粒体基因的功能可以被1个或多个核基因编码的产物抑制或消除,这些核基因被称为恢复基因(Rf)[19]。CMS-D2与CMS-D8对应恢复系携带的恢复基因分别为Rf1[20]和Rf2[21],其中Rf1可以恢复CMS-D2和CMS-D8不育系的育性,Rf2只可以恢復CMS-D8不育系的育性。CMS-D8与CMS-D2这2种败育类型及对应的恢复基因的恢复功能的差异可能与线粒体基因组结构变异及特异的ORF有关。目前,关于植物孢子体不育和配子体不育不同败育类型的线粒体组学分析报道较少。本研究通过对D2A和D8A这2种不同棉种胞质不育系线粒体基因组进行比对分析,揭示了D2A和D8A线粒体基因组的结构差异,在线粒体基因组学层面比较分析了2种不同类型的不育系在进化过程中产生的差异特征。此外,通过对特异ORF的分析及验证,探究线粒体ORF和不同败育类型的潜在关系,为进一步筛选配子体败育和孢子体败育相关的线粒体ORF奠定基础。
1 材料与方法
1.1 植物材料
供试材料为2个不同类型的棉花细胞质雄性不育系,其中D2A为哈克尼西棉细胞质雄性不育系(CMS-D2),D8A为三裂棉细胞质雄性不育系(CMS-D8),详细的材料创制过程及来源参见本课题组前期的研究[22-24]。D2A和D8A材料均于2023年4月下旬种植于中国农业科学院棉花研究所东场试验基地(河南省安阳县),田间管理措施同当地普通棉田。
1.2 试验方法
1.2.1 提取总DNA。在苗期,取D2A和D8A的叶片,使用改良后的十六烷基三甲基溴化铵(hexadecyl trimethyl ammonium bromide, CTAB)法[25]提取总DNA,通过紫外分光光度计测量DNA浓度,并稀释至相同浓度。
1.2.2 总RNA的提取及反转录。在田间分别选取20株D2A和D8A,取其3~4 mm花蕾,在液氮中冷冻,于-80 ℃保存备用。取100 mg花蕾在液氮中迅速研磨成粉末,使用天根生化科技(北京)有限公司的RNAprep Pure Plant Plus Kit试剂盒进行总RNA提取。使用1.2%(质量分数)的琼脂糖凝胶电泳对提取的RNA进行纯度检测,同时使用NanoDrop 2000超微量紫外可见分光光度计测定RNA在不同波长下的吸光度,测定值应满足1.8≤OD260/OD280≤2.2、OD260/OD230≥1.8。检测后选取高纯度RNA使用PrimeScriptTM RT reagent Kit试剂盒(TaKaRa,北京)进行反转录合成cDNA,将cDNA稀释10倍后备用。
1.2.3 线粒体基因组测序和组装 。本课题组已经完成CMS-D2和CMS-D8不育系的线粒体基因组的测序、组装和基因注释,具体分析方法参照参考文献[16]。其中D2A线粒体基因组组装测序结果已经在美国国立生物技术信息中心(National Center for Biotechnology Information, NCBI)的序列读取档案(SRA数据库)中公开,登录号:SRR17406254[15]。D8A线粒体基因组的测序及组装结果暂未公开。
1.2.4 D2A和D8A线粒体基因组结构分析和验证。CMS现象的产生与线粒体基因组紧密相关。为探索D2A、D8A这2种不育胞质线粒体基因组的差异,本研究利用MUMmer软件(版本MUMmer/4.0.0beta2,https://github.com/mummer4/mummer/releases,nucmer全局比对参数: --max-match -c 100 -b 500 -l 50 refgenome qrygenome,delta-filter过滤参数:-m -i 90 -l 100)以D8A线粒体基因组作为参考,将D2A线粒体基因组进行比对,比对结果使用Synteny and Rearrange-ment Identifier(SyRI)软件(https://github.com/schneebergerlab/syri)鉴定结构变异,并用Plotsr软件(https://github.com/schneebergerlab/plotsr)进行可视化分析。线粒体基因组结构分析包含共线性及非共线性区域的重组变异位点,对经分析预测出的D2A的结构变异区域INV4和INV5,在D2A线粒体基因组上的2个边界位点上下游共400 bp区域(INV4-1、INV4-2、INV5-1、INV5-2)设计扩增引物(表1),以D2A、D8A的叶片DNA为模板进行聚合酶链式反应(polymerase chain reaction, PCR),用琼脂糖凝胶电泳方法进行验证。
1.2.5 D2A中特异ORF的预测、分析及验证。在D8A注释的ORF数据库中,利用BLAST软件(版本BLAST+/2.9.0)的tblastn(参数 -outfmt 7,其余参数均为默认值)对已经注释的D2A线粒体基因组的ORF编码的氨基酸序列进行比对分析。根据比对结果筛选D2A中特异的ORF。使用TMHMM Server 2.0软件(https://services.healthtech.dtu.dk/services/TMHMM-2.0/)对D2A中特异的ORF进行跨膜结构域预测,利用NCBI网站的BLAST在线分析差异的ORF是否为嵌合ORF,进一步分析D2A线粒体基因组中特异的ORF是否具有植物不育基因的特征。使用DNAMAN软件对D2A中特异的17个ORF全长序列设计引物(表2),并通过PCR扩增和琼脂糖凝胶电泳方法进行验证。
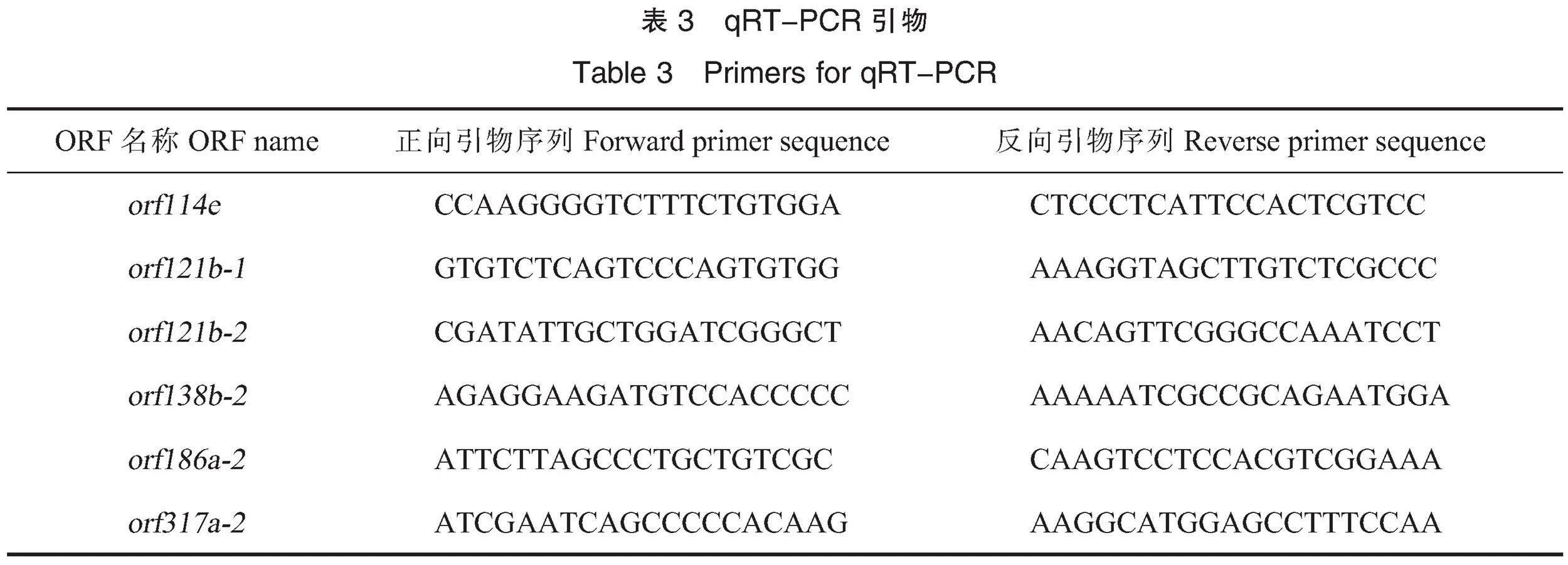
1.2.6 相对表达量分析。提取D2A 3~4 mm花蕾RNA,进行反转录合成cDNA,以此作为模版用于实时荧光定量聚合酶链式反应(quantitative real-time polymerase chain reaction, qRT-PCR)。RNA提取和反转录方法同1.2.2。在NCBI中设计特异引物(表3),以GhHIS3作为内参基因,使用TransStart○R Top Green qPCR SuperMix试剂盒(全式金,北京)进行qRT-PCR,反应体积为20 μL,扩增程序:94 ℃预变性30 s,94 ℃变性5 s,55 ℃退火15 s,72 ℃延伸10 s,40个循环。试验设计3次技术重复。所用仪器为Mastercycler ep realplex(Eppendorf,德国)。用 2-ΔΔCt法计算目的ORF的相对表达量。
2 结果与分析
2.1 D2A和D8A线粒体基因组重排
以D8A线粒体基因组作为参考,对D2A线粒体基因组进行共线性分析,结果表明D2A线粒体基因组在425 896~502 639 bp区间内存在2处倒易位区域,分别为INV4和INV5,INV4和INV5这2个区域相邻且部分重叠,重叠区长度为9 122 bp(图1A)。INV4和INV5在D2A中存在4个边界位点,分别在基因组425 896 bp、457 648 bp、466 769 bp和502 639 bp处。在4个边界位点上下游(INV4-1、INV4-2、INV5-1、INV5-
2,其中INV4-1、INV5-2在D2A和D8A共線性区域,INV4-2、INV5-1在非共线性区域)设计引物,INV4-1为D2A中425 896 bp处上下游400 bp区域,INV4-2为D2A中466 769 bp处上下游400 bp区域,INV5-1为D2A中457 648 bp处上下游400 bp区域,INV5-2为D2A中502 639 bp处上下游400 bp区域。在D2A和D8A的DNA中进行PCR扩增,琼脂糖凝胶电泳结果显示,在D2A和D8A中均扩增出INV4-1和INV5-2,在D2A中扩增出INV4-2和INV5-1,而D8A中未扩增出INV4-2和INV5-1(图1B)。这说明INV4-1、INV5-2在D2A和D8A中同时存在,而INV4-2、INV5-1仅在D2A中存在。结合以上分析结果,可以初步验证D2A和D8A线粒体基因组结构存在2个倒易位变异。
2.2 D2A和D8A线粒体基因组差异ORF分析
通过对D2A和D8A线粒体基因组组装、注释可知,D2A线粒体基因组大小为634 036 bp ,D8A线粒体基因组大小为634 407 bp,D2A和D8A的线粒体基因组分别注释到194个和154个ORF。D2A和D8A的线粒体基因组在大小上相差较小,但D2A比D8A多40个ORF。为探索孢子体不育系与配子体不育系的线粒体基因组之间存在的差异,以D8A线粒体基因组注释结果为参照,用注释后的D2A线粒体基因组ORF编码的氨基酸序列进行tblastn比对,分析D2A中特异的ORF。结果显示D2A中存在17个特异的ORF(编码的氨基酸数量相同但序列不同),分别为orf106b、orf109a、orf109b、orf111a、orf111c、orf114e、orf119b-1、orf119b-2、orf119c、orf121b-1、orf121b-2、orf138b-2、orf186a-2、orf208a、orf228a、orf270b和orf317a-2。研究表明,在已知基因附近的ORF可能与上游或者下游基因共同转录[19]。分析发现,在已知基因上游或者下游的ORF有4个(orf114e、orf186a-2、orf228a和orf317a-2),有可能与已知基因协同转录。由蛋白编码基因的同源序列组成的嵌合ORF有2个(orf208a和orf186a-2)。具有跨膜结构域的ORF有6个(orf114e、orf119b-1、orf119b-2、orf186a-2、orf208a和orf317a-2)(表4)。
利用NCBI數据库(NR库)和Swiss-Prot对测序结果进行注释,D2A线粒体基因组中5个特异的ORF即orf111a、orf114e、orf121b-1、orf121b-2和orf317a-2有功能注释信息。其中orf111a编码细胞色素b559亚基α,orf114e编码YMF16蛋白,orf121b-1、orf121b-2和orf317a-2均编码未知蛋白(表5)。1个完整的基因包括ORF序列以及非编码序列,ORF作为完整基因序列的一部分,可作为1个潜在蛋白编码基因的指示器,但是预测的ORF并不一定是编码蛋白,且部分ORF目前暂未注释到功能信息。
将D2A中特异的17个ORF在D2A和D8A的3~4 mm花蕾cDNA中进行PCR扩增验证,结果表明D2A中可扩增出6个ORF(orf114e、orf121b-1、orf121b-2、orf138b-2、orf186a-2和orf317a-2),其中orf114e、orf138b-2和orf317a-2的扩增条带较弱,可能与其表达量较低有关,而在D8A中未扩增出相应条带(图2)。4个ORF(orf121b-1、orf121b-2、orf138b-2和orf317a-2)位于D2A线粒体基因组倒易位区域。利用qRT-PCR分析这6个差异ORF在D2A 3~4 mm花蕾中的相对表达量,结果表明其中orf121b-1的表达量最高,orf121b-2的表达量次之,orf186a-2、orf317a-2和orf138b-2的表达量较低,orf114e的表达量最低(图3)。
经过对组装注释好的D2A线粒体基因组分析发现,orf114e是mttB基因序列的一部分,并且存在2个跨膜结构域,其中mttB编码膜转运蛋白(图4A、D)。orf186a-2位于atp8基因下游565 bp,与rps3基因形成嵌合基因,并且具有1个跨膜结构域,其中atp8编码ATP合成酶,rps3编码核糖体蛋白(图4B、D)。orf317a-2位于atp1基因(编码ATP合成酶)上游659 bp,存在3个跨膜结构域(图4C~D)。orf114e、orf186a-2和orf317a-2这3个ORF均具有CMS不育基因的部分特征。
3 讨论
植物线粒体基因组存在广泛的重复序列,易产生基因重组和重排现象[26]。Wang等[27]利用MUMmer对烟草的不育系和保持系的线粒体基因组进行了比对分析,发现了不育系和保持系线粒体基因组的高度同源区域,并分析了不育系线粒体基因组中存在的易位、倒位、倒易位变异现象。本研究使用MUMmer将D2A和D8A这2个不同胞质的线粒体基因组进行比对分析,发现D2A、D8A线粒体基因组在结构上存在差别,与前人研究结果相似,推测棉花D2A与D8A的不育类型不一致可能与倒易位变异有一定的关系。同时,本研究在2个线粒体基因组比对的基础上,通过PCR扩增边界序列进行初步验证,进一步证实D2A和D8A线粒体基因组之间存在2个具有重叠区且相邻的倒易位区域。
大量研究表明,CMS中不育系和保持系线粒体基因组结构的差异可能会导致CMS新基因(ORF)的产生。例如,在芹菜中,以W99A为参照进行基因组结构变异分析,发现W99A和W99B线粒体基因组之间存在23个易位区、5个倒位区和21个倒易位区。W99A的特异区域存在15个ORF,根据不育基因特征进行筛选,最终确定orf768a为芹菜W99A中雄性不育候选基因[28]。同样,在辣椒CMS-138A线粒体基因组的特异区域的35个ORF中,根据不育基因特征筛选出orf300a和orf314a这2个候选基因[29]。本研究以D8A线粒体基因组作为参考,对D2A线粒体基因组进行比对分析,在D2A中预测到17个特异的ORF。通过PCR扩增、琼脂糖凝胶电泳验证,结果表明D2A中存在6个特异的ORF,其中有4个ORF(orf121b-1、orf121b-2、orf138b-2和orf317a-
2)位于倒易位区域,可能为线粒体结构变异产生的新基因。以往研究表明CMS不育基因大部分为嵌合基因,与电子传递链相关基因协同转录,含有1个或多个跨膜结构域[30]。本研究经预测分析发现,D2A线粒体基因组中3个ORF即orf114e、orf186a-2和orf317a-2位于已知功能基因附近,可能与其上下游基因协同转录,并且3个ORF均存在跨膜结构域,这3个特异的ORF均符合不育基因的部分特征。本研究的结果丰富了CMS-D2不育基因的候选基因,但其在CMS产生过程中的功能及其作用机制仍需进一步验证。
4 结论
本研究利用课题组已有的D2A与D8A的线粒体基因组,通过MUMmer与SyRI对线粒体基因组序列进行分析,发现D2A和D8A存在2个具有重叠区域的相邻倒易位区域,分别为INV4和INV5。在D2A和D8A这2个材料中通过PCR扩增INV4、INV5边界序列初步证明D2A和D8A线粒体基因组在结构上存在差异。以D8A线粒体基因组为参考,比对分析发现D2A线粒体基因组中存在17个特异的ORF,并且PCR扩增出D2A中6个特异ORF,进一步分析发现其中4个ORF(orf121b-1、orf121b-2、orf138b-2和orf317a-2)位于INV4和INV5区域内,可能为线粒体结构变异产生的新基因。通过跨膜结构域与嵌合基因分析,D2A中特有的orf114e、orf186a-2和orf317a-2符合不育基因的部分特征,可能与CMS-D2孢子体不育有关。
参考文献:
[1] Chen L T, Liu Y G. Male sterility and fertility restoration in crops[J/OL]. Annual Review of Plant Biology, 2014, 65: 579-606[2024-01-30]. https://doi.org/10.1146/annurev-arplant-050213-040119.
[2] Xu F Y, Yang X D, Zhao N, et al. Exploiting sterility and fertility variation in cytoplasmic male sterile vegetable crops[J/OL]. Horticulture Research, 2022, 9: uhab039[2024-01-30]. https://doi.org/10.1093/hr/uhab039.
[3] 邢朝柱, 郭立平, 李威, 等. 棉花杂种优势利用研究十年成就和未来发展[J/OL]. 棉花学报, 2017, 29(S1): 28-36[2024-01-30]. https://doi.org/10.11963/1002-7807.xczxcz.20170825.
Xing Chaozhu, Guo Liping, Li Wei, et al. Ten-year achievements and future development of cotton heterosis utilization[J/OL]. Cotton Science, 2017, 29(S1): 28-36[2024-01-30]. https://doi.org/10.11963/1002-7807.xczxcz.20170825.
[4] 李宏, 周少川, 周德贵, 等. 以不育系为育种起点的水稻细胞质雄性不育系选育[J/OL]. 杂交水稻, 2013, 28(2): 7-10[2024-01-30]. https://doi.org/10.16267/j.cnki.1005-3956.2013.02.002.
Li Hong, Zhou Shaochuan, Zhou Degui, et al. Breeding of rice CMS lines started from utilization of CMS lines[J/OL]. Hybrid Rice, 2013, 28(2): 7-10[2024-01-30]. https://doi.org/10.16267/j.cnki.1005-3956.2013.02.002.
[5] 李晚忱, 榮廷昭, 雷本鸣, 等. 3个玉米细胞质雄性不育系的选育及分组鉴定[J]. 作物学报, 2001, 27(3): 308-312.
Li Wanchen, Rong Tingzhao, Lei Benming, et al. Breeding and classification of three cytoplasmic male sterile inbreds in maize[J]. Acta Agronomica Sinica, 2001, 27(3): 308-312.
[6] 陈大伦, 曾章丽. 甘蓝型油菜细胞质雄性不育系9171A的发现及育性特征[J/OL]. 种子, 2018, 37(11): 108-110[2024-01-30]. https://doi.org/10.16590/j.cnki.1001-4705.2018.11.108.
Chen Dalun, Zeng Zhangli. Discovery and fertility characteristics of cytoplasmic male sterile line 9171A in Brassica napus L.[J/OL]. Seed, 2018, 37(11): 108-110[2024-01-30]. https://doi.org/10.16590/j.cnki.1001-4705.2018.11.108.
[7] Zhao Z, Ding Z, Huang J J, et al. Copy number variation of the restorer Rf4 underlies human selection of three-line hybrid rice breeding[J/OL]. Nature Communications, 2023, 14(1): 7333[2024-01-30]. https://doi.org/10.1038/s41467-023-43009-4.
[8] 汪静, 荣廷昭, 潘光堂, 等. 植物线粒体遗传物质与细胞质雄性不育关系的研究进展[J]. 玉米科学, 2006(6): 78-82.
Wang Jing, Rong Tingzhao, Pan Guangtang, et al. Research progress of the relation between mitochondrial geneticmaterial and cytoplasmic male sterility in plants[J]. Journal of Maize Sciences, 2006(6): 78-82.
[9] 雷刚, 方荣, 周坤华, 等. 植物细胞质雄性不育与线粒体[J/OL]. 西北植物学报, 2020, 40(9): 1617-1626[2024-01-30]. https://doi.org/10.7606/j.issn.1000-4025.2020.09.1617.
Lei Gang, Fang Rong, Zhou Kunhua, et al. Cytoplasmic male sterility (CMS) and mitochondria in plants[J/OL]. Acta Botanica Boreali-Occidentalia Sinica, 2020, 40(9): 1617-1626[2024-01-30]. https://doi.org/10.7606/j.issn.1000-4025.2020.09.1617.
[10] Wang Z H, Zou Y J, Li X Y, et al. Cytoplasmic male sterility of rice with boro II cytoplasm is caused by a cytotoxic peptide and is restored by two related PPR motif genes via distinct modes of mRNA silencing[J/OL]. The Plant Cell, 2006, 18(3): 676-687[2024-01-30]. https://doi.org/10.1105/tpc.105.038240.
[11] Yang H L, Xue Y D, Li B, et al. The chimeric gene atp6c confers cytoplasmic male sterility in maize by impairing the assembly of the mitochondrial ATP synthase complex[J/OL]. Molecular Plant, 2022, 15(5): 872-886[2024-01-30]. https://doi.org/10.1016/j.molp.2022.03.002.
[12] Sang S F, Cheng H T, Hao M Y, et al. Mitochondrial localization of ORF346 causes pollen abortion in alloplasmic male sterility[J/OL]. The Crop Journal, 2021, 9(6): 1320-1329[2024-01-30]. https://doi.org/10.1016/j.cj.2021.01.008.
[13] Xie H W, Peng X J, Qian M J, et al. The chimeric mitochondrial gene orf182 causes non-pollen-type abortion in Dongxiang cytoplasmic male-sterile rice[J/OL]. The Plant Journal, 2018, 95(4): 715-726[2024-01-30]. https://doi.org/10.1111/tpj.13982.
[14] He T T, Ding X L, Zhang H, et al. Comparative analysis of mitochondrial genomes of soybean cytoplasmic male-sterile lines and their maintainer lines[J/OL]. Functional & Integrative Genomics, 2021, 21: 43-57[2024-01-30]. https://doi.org/10.1007/s10142-020-00760-x.
[15] Li S S, Liu G Z, Chen Z W, et al. Construction and initial analysis of five Fosmid libraries of mitochondrial genomes of cotton (Gossypium)[J/OL]. Chinese Science Bulletin, 2013, 58(36): 4608-4615[2024-01-30]. https://doi.org/10.1007/s11434-013-5962-4.
[16] Zhang Y J, Han Y, Zhang M, et al. The cotton mitochondrial chimeric gene orf610a causes male sterility by disturbing the dynamic balance of ATP synthesis and ROS burst[J/OL]. The Crop Journal, 2022, 10(6): 1683-1694[2024-01-30]. https://doi.org/10.1016/j.cj.2022.02.008.
[17] Li P B, Zhou H, Yang H Y, et al. Genome-wide association studies reveal the genetic basis of fertility restoration of CMS-WA and CMS-HL in xian/indica and aus accessions of rice (Oryza sativa L.) [J/OL]. Rice, 2020, 13: 11[2024-01-30]. https://doi.org/10.1186/s12284-020-0372-0.
[18] 周天理. 第二講孢子体不育和配子体不育[J]. 福建农业科技, 1986(5): 33-34.
Zhou Tianli. Sporophytic sterility and gametophytic sterility[J]. Fujian Agricultural Science and Technology, 1986(5): 33-34.
[19] Kim Y J, Zhang D B. Molecular control of male fertility for crop hybrid breeding[J/OL]. Trends in Plant Science, 2018, 23(1): 53-65[2024-01-30]. https://doi.org/10.1016/j.tplants.2017.10.001.
[20] Meyer V G. Male sterility from Gossypium harknessii[J/OL]. Journal of Heredity, 1975, 66(1): 23-27[2024-01-30]. https://doi.org/10.1093/oxfordjournals.jhered.a108566.
[21] Zhang J F, Stewart J M. CMS-D8 restoration in cotton is conditioned by one dominant gene[J/OL]. Crop Science, 2001, 41(2): 283-288[2024-01-30]. https://doi.org/10.2135/cropsci2001.412283x.
[22] Wu J Y, Gong Y C, Cui M H, et al. Molecular characterization of cytoplasmic male sterility conditioned by Gossypium harknessii cytoplasm (CMS-D2) in upland cotton[J/OL]. Euphytica, 2011, 181: 17-29[2024-01-30]. https://doi.org/10.1007/s10681-011-0357-6.
[23] Wu J Y, Zhang M, Zhang B B, et al. Genome-wide comparative transcriptome analysis of CMS-D2 and its maintainer and restorer lines in upland cotton[J/OL]. BMC Genomics, 2017, 18(1): 1-12[2024-01-30]. https://doi.org/10.1186/s12864-017-3841-0.
[24] Feng J J, Zhu H Y, Zhang M, et al. Development and utilization of an InDel marker linked to the fertility restorer genes of CMS-D8 and CMS-D2 in cotton[J/OL]. Molecular Biology Reports, 2020, 47: 1275-1282[2024-01-30]. https://doi.org/10.1007/s11033-019-05240-5.
[25] Zhang J F, Stewart J M. Economical and rapid method for extracting cotton genomic DNA[J/OL]. The Journal of Cotton Science, 2000, 4: 193-201[2024-01-30]. https://doi.org/10.3724/sp.j.1148.2009.00582.
[26] Fujii S, Bond C S, Small I D. Selection patterns on restorer-like genes reveal a conflict between nuclear and mitochondrial genomes throughout angiosperm evolution[J/OL]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(4): 1723-1728[2024-01-30]. https://doi.org/10.1073/pnas.1007667108.
[27] Wang R Y, Cai X H, Hu S N, et al. Comparative analysis of the mitochondrial genomes of Nicotiana tabacum: hints toward the key factors closely related to the cytoplasmic male sterility mechanism[J/OL]. Frontiers in Genetics, 2020, 11: 257[2024-01-30]. https://doi.org/10.3389/fgene.2020.00257.
[28] Cheng Q, Wang P, Li T T, et al. Complete mitochondrial genome sequence and identification of a candidate gene responsible for cytoplasmic male sterility in celery (Apium graveolens L.)[J/OL]. International Journal of Molecular Sciences, 2021, 22(16): 8584[2024-01-30]. https://doi.org/10.3390/ijms22168584.
[29] Wang P, Lu Q H, Ai Y X, et al. Candidate gene selection for cytoplasmic male sterility in pepper (Capsicum annuum L.) through whole mitochondrial genome sequencing[J/OL]. International Journal of Molecular Sciences, 2019, 20(3): 578[2024-01-30]. https://doi.org/10.3390/ijms20030578.
[30] Zhong X H, Chen D H, Cui J, et al. Comparative analysis of the complete mitochondrial genome sequences and anther development cytology between maintainer and Ogura-type cytoplasm male-sterile cabbage (B. oleracea Var. capitata) [J/OL]. BMC Genomics, 2021, 22(1): 1-15[2024-01-30]. https://doi.org/10.1186/s12864-021-07963-x.

