基于高通量基因表达数据库及生物信息学对坐骨神经痛差异表达基因的分析研究
曲艳平 贺文廷 王旭 董浩 范智东
基金项目:云南省科技厅科学研究基金项目(编号:2023Y0966)
作者简介:曲艳平,硕士研究生,医师,研究方向:慢性神经病理性疼痛的诊疗。
通信作者:范智东,大学本科,主任医师,研究方向:临床麻醉学。E-mail:fanzhidong68@163.com
【摘要】目的 基于高通量基因表达(GEO)数据库及生物信息学筛选坐骨神经痛相关差异表达基因(DEGs),分析其生物学功能,为临床治疗坐骨神经痛提供参考。方法 通过GEO数据库获取坐骨神经痛基因芯片(GSE124272)数据集,经仙桃学术软件异质性分析获取坐骨神经痛相关DEGs,对坐骨神经痛相关DEGs进行基因本体论(GO)、京都基因与基因组百科全书(KEGG)的通路富集分析。借助Cytoscape软件实施蛋白质互作网络(PPI)分析获取关键基因,经转录因子调控网络专用数据库(TRRUST)确定关键基因的主要调节转录因子及相应的调节关系。结果 415个坐骨神经痛相关DEGs经PPI分析共获取15个关键基因,经TRRUST确定共12个TF协同参与检查点激酶1(CHEK1)、含杆状病毒IAP重复序列蛋白5(BIRC5)、细胞周期蛋白B2(CCNB2)、胸苷酸合成酶(TYMS)、细胞周期蛋白B1(CCNB1)关键基因的调控。结论 CHEK1、BIRC5、CCNB2、TYMS、CCNB1可作为坐骨神经痛的新治疗靶点和预防标志物。
【关键词】高通量基因表达数据库;生物信息学;坐骨神经痛;差异表达基因
【中图分类号】R614 【文献标识码】A 【文章编号】2096-2665.2024.05.0041.03
DOI:10.3969/j.issn.2096-2665.2024.05.014
坐骨神经痛是慢性神经病理性疼痛中常见的周围神经痛,其主要表现为坐骨神经循环和分布区域的持续性或阵发性疼痛。有研究表明,45%的坐骨神痛病人症状会持续12个月,多发生于40~60岁人群[1]。该疾病不仅降低了患者晚年生活质量,也给社会带来严重的经济负担。坐骨神经痛的发病机制可能与坐骨神经根或其支配的感觉神经节受压迫与扭曲有关,也可能与局部炎性细胞因子的高表达有关。本研究基于高通量基因表达(gene expression omnibus,GEO)数据库及生物信息学,对8例坐骨神经痛患者和8例健康人的基因数据进行分析,旨在了解坐骨神经痛的发病机制,现报道如下。
1 资料与方法
1.1 数据的来源与获取 在GEO数据库点击“GEO Data Sets”,输入“SCIATICA”,根据研究样本及类别筛选坐骨神经痛基因芯片,即:坐骨神经痛基因芯片(GSE124272)数据集。选取GSE124272数据集为研究对象,其中包括
8例坐骨神经痛患者及8例健康人的基因数据。
1.2 筛选差异表达基因 运用仙桃学术软件对GSE124272数据集进行标准化预处理,获得16例研究对象的基因数据的表达矩阵,以|logFC|>1及矫正后P<0.05为阈值筛选坐骨神经痛相关差异表达基因(differential expressed genes, DEGs)。
1.3 富集分析 基因本体论(gene ontology,GO)数据库由基因本体联合建立,其从功能、生物学途径和细胞位置等方面提供基因的简单注释[2]。京都基因与基因組百科全书(kyoto encyclopedia of genes and genomes,KEGG)数据库主要用于存储不同物种的基因路径信息,分析DEGs所涉及信号通路[3]。运用仙桃学术软件对坐骨神经痛相关DEGs进行GO和KEGG通路富集分析,显著性基因富集临界值设定为P<0.05。
1.4 筛选关键基因 运用仙桃学术软件对坐骨神经痛DEGs进行蛋白质互作网络(PPI)分析,分析结果输入Cytoscape软件,运用CytoHubba插件中度数关联度算法获取节点最高的15个基因,选取较高连通度的基因为该疾病的关键基因。
1.5 转录因子(TF)的预测与验证转录因子调控网络专用数据库(transcriptional regulatory relationships unraveled by sentence-based text mining,TRRUST)是1个用于预测转录调控网络的数据库,目前包括人和小鼠2个物种[4]。通过TRRUST获取调控关键基因的TF,以及与关键基因之间的调控关系。
2 结果
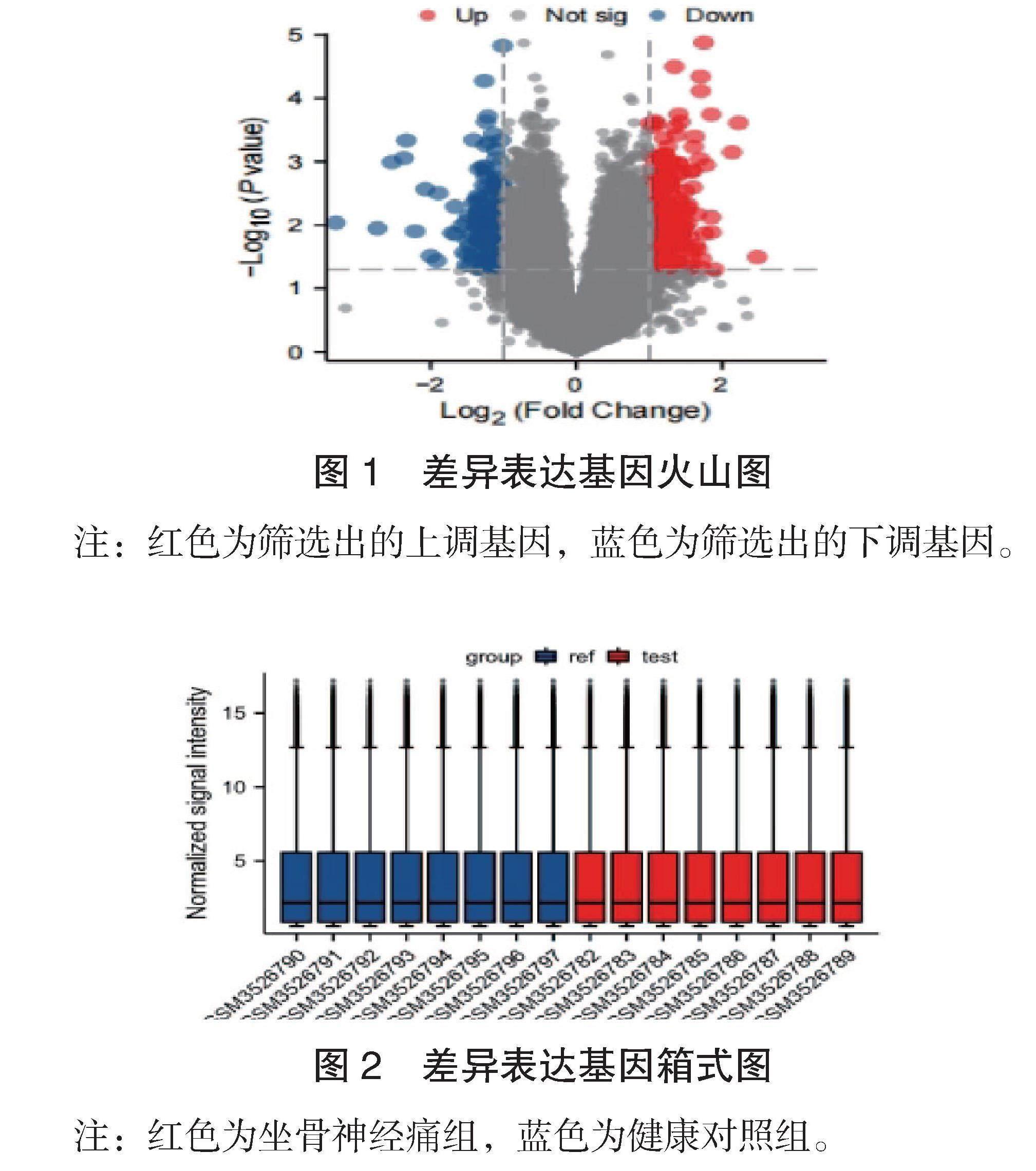
2.1 差异表达基因标准化预处理结果 GSE124272数据集标准化预处理结果显示:共415个坐骨神经痛相关DEGs,见图1。8个坐骨神经痛患者样本及8个健康人群的样本标准化预处理筛选结果以箱式图呈现,见图2。
图1 差异表达基因火山图
注:红色为筛选出的上调基因,蓝色为筛选出的下调基因。
图2 差异表达基因箱式图
注:红色为坐骨神经痛组,蓝色为健康对照组。
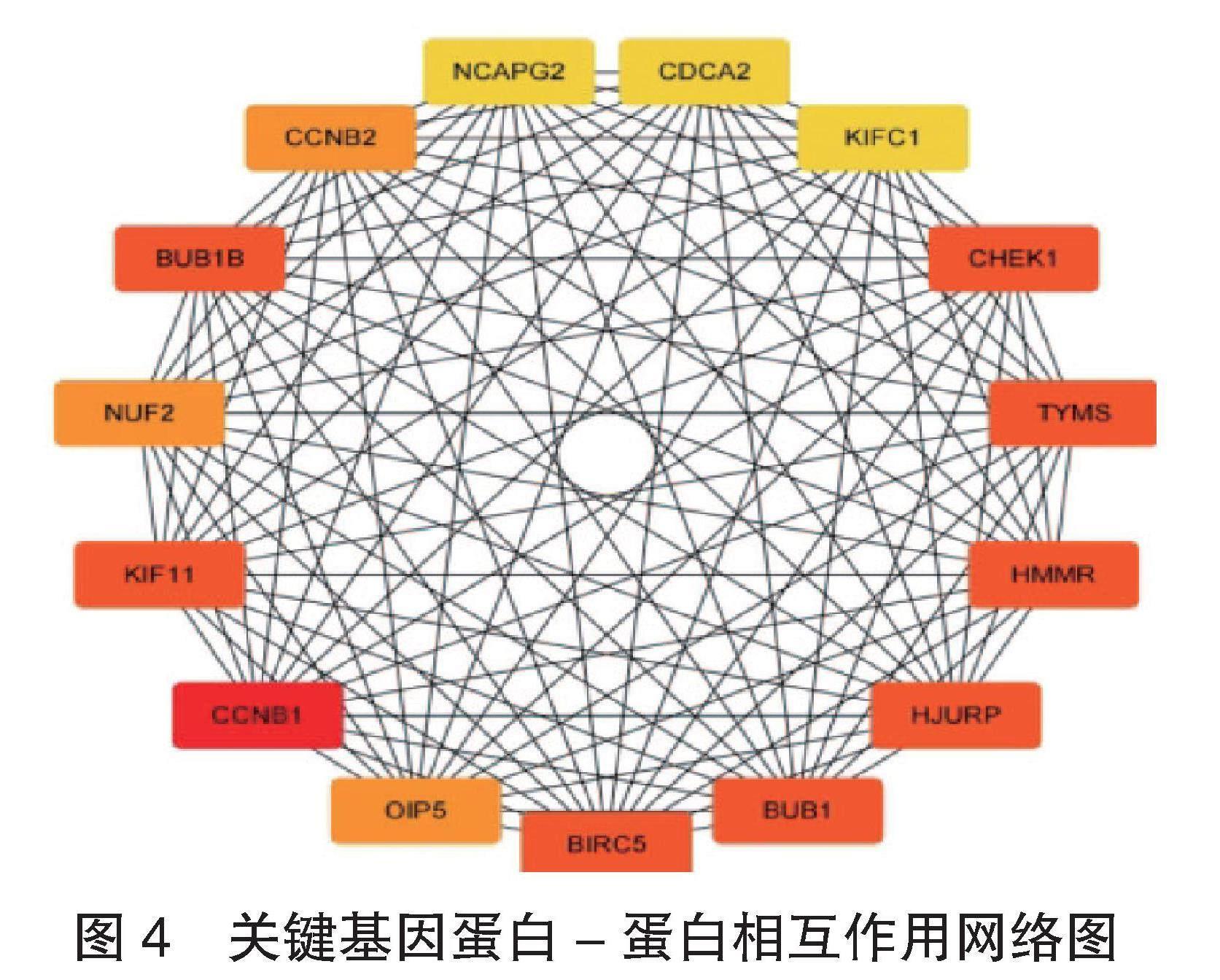
2.2 GO和KEGG通路富集分析结果 GO通路富集分析结果显示:坐骨神经痛相关DEGs主要富集于分泌颗粒管腔、特异性颗粒管腔、三级颗粒管腔、特异性颗粒管腔等部位,主要涉及有丝分裂核分裂的调节、有丝分裂核分裂的负调控、中性粒细胞介导的细胞毒性、嗜中性粒细胞介导的共生体细胞杀伤等过程。KEGG通路富集分析结果显示:坐骨神经痛相关DEGs主要与过氧化物酶体增殖物激活受体(PPAR)信号通路有关,PPAR信号通路主要调节基因的转录,见图3。
图3 GO和KEGG通路富集分析图
注:CC :细胞组分;BP:生物过程;KEGG:京都基因与基因组百科全书。
2.3 PPI分析结果 通过CytoHubba插件计算出15个节点,这15个节点称为关键基因,包含含杆状病毒IAP重复序列蛋白5(BIRC5)、检查点激酶1(CHEK1)、细胞周期蛋白B2(CCNB2)、胸苷酸合成酶(TYMS)、细胞周期蛋白B1(CCNB1)、Opa 相互作用蛋白 5(OIP5)、苯并咪唑出芽抑制解除同源物蛋白1(BUB1)、有丝分裂检查点丝氨酸/苏氨酸激酶 B(BUB1B)、细胞分裂周期相关 2(CDCA2)、驱动蛋白家族成员 C1(KIFC1)、Holliday交叉识别蛋白(HJURP)、驱动蛋白家族成员11(KIF11)、非 SMC Condensin II 复合物亚基 G2(NCAPG2)、透明质酸介导的运动受体(HMMR)及NDC80 着丝粒复合物的 NUF2 组分(NUF2),见图3。
图4 关键基因蛋白-蛋白相互作用网络图
2.4 关键基因的转录因子验证结果 基于TRRUST数据库发现12个调控关键基因的TF,分别为肿瘤蛋白P53(TP53)、上游转录因子1(USF1)、E1A结合蛋白P300(EP300)、Sp1转录因子(SP1)、E2F转录因子1(E2F1)、KLF 转录因子 4(KLF4)、核因子 Kappa B 亚基 1(NFKB1)、Y-Box 结合蛋白 1(YBX1)、RELA 原癌基因,NF-KB 亚基(RELA)、E2F转录因子4(E2F4)、MYC 原癌基因,BHLH 转录因子(MYC)、KLF 转录因子 5(KLF5),协同参与CHEK1、BIRC5、CCNB2、TYMS、CCNB1这5个关键基因的调控,见图5。
图5 转录因子调控网络图
3 讨论
近年来,人们对精准医疗的要求越来越高,许多新型的治疗手段被应用于临床诊疗中。目前,与疾病相关的基因研究成为研究热点,但多关于癌症方面,对于坐骨神经痛基因的研究较少。临床治疗坐骨神经痛主要为卧床休息、手术治疗、西药治疗。卧床休息能够缓解坐骨神经痛急性发作期因神经损伤所引起的疼痛及因机体活动所引起的神经牵拉。虽然长期卧床能够缓解一定程度的坐骨神经痛,但也会产生不良反应,如形成静脉血栓、肌肉萎缩、骨质疏松等;另外,长期卧床还会使患者心情烦躁,增加心理疾病发生的风险[5]。
研究显示,手术治疗与保守治疗相比,能够更快地缓解疼痛,但治疗1年和2年后疼痛缓解程度无明显差异[6]。西药治疗坐骨神经痛以镇痛与营养神经类药物进行对症治疗为主,研究表明目前临床治疗坐骨神经痛常用的非甾体类抗炎药并不能从根本上解决因坐骨神经受损引起的疼痛[7]。且在临床实践中,可观察到部分患者经过西药治疗后疼痛症状并未得到充分缓解[8]。因此,寻找新的坐骨神经痛治疗靶点和预防标志物对临床治疗具有重要意义。
本研究选取8个坐骨神经痛患者样本和8个健康人为研究对象,共筛选出了415个坐骨神经痛相关DEGs以及可能参与坐骨神经痛发生发展的15个关键基因。基于TRRUST数据库发现12个调控关键基因的TF协同参与CHEK1、BIRC5、CCNB2、TYMS、CCNB1这5个关键基因的调控。
CHEK1属于丝氨酸/苏氨酸蛋白激酶家族中的一员,在细胞DNA损伤时或未完全复制的DNA中表达,主要功能为参与调节细胞周期修复DNA的损伤[9]。有研究指出,CHEK1参与调节细胞周期及DNA的损伤修复[10]。同时,CHEK1的异常表达还可使细胞减数分裂进展缓慢[11]。高表达的BIRC5与趋化因子活性和免疫细胞趋化性相关[12]。TYMS是一种叶酸依赖性必需酶,可产生DNA合成和修復所需的tRNA[13]。细胞周期蛋白(Cyclin)家族成员是细胞周期进程的关键调节因子,CCNB1和CCNB2是Cyclin家族中非常重要的成员,在细胞周期的不同阶段发挥调节作用[14]。有丝分裂主要由CCNB1及其相关的催化活性伴侣所驱动[15]。以上5个关键基因在细胞的发育、组织分化等过程中发挥着重要的作用,因此,这5个关键基因有可能成为坐骨神经痛的新治疗靶点和预防标志物。
综上所述,坐骨神经痛发病与预后中发挥关键作用的基因为CHEK1、BIRC5、CCNB2、TYMS、CCNB1,可作为坐骨神经痛的新治疗靶点和预防标志物。
参考文献
JIANG C, LI Y,GUO M,et al. Acupotomy therapy for lumbar disc herniation: Protocol for a systematic review and meta-analysis[J].Medicine, 2018, 97(40): e12624.
GENE ONTOLOGY CONSORTIUM. The Gene Ontology resource: Enriching a GOld mine[J]. Nucleic Acids Res, 2021, 49(D1): D325-334.
KANEHISA M,FURUMICHI M, TANABE M, et al. KEGG: New perspectives on genomes,pathways, diseases and drugs[J]. Nucleic Acids Res, 2017, 45(D1): D353-D361.
SU W, ZHAO Y, WEI Y,et al. Exploring the pathogenesis of psoriasis complicated with atherosclerosis via microarray data analysis[J].Front Immunol, 2021, 12: 667690.
SHEN Y, ZHOU Q, QIU Z,et al. Acupotomy for calcaneodynia: A systematic review protocol[J]. Medicine, 2018, 97(14): e0143.
JACOBS W C, VAN TULDER M, ARTS M, et al. Surgery versus conservative management of sciatica due to a lumbar herniated disc: A systematic review [J]. Eur spine J, 2011, 20(4): 513-522.
RASMUSSEN-BARR E, HELD U, GROOTEN W J, et al. Non-steroidal anti-inflammatory drugs for sciatica [J]. Cochrane database Syst Rev, 2016, 10(10): CD012382.
老年慢性非癌痛藥物治疗中国专家共识[J]. 中国疼痛医学杂志, 2016, 22(5): 321-325.
TAN Z, CHEN M, WANG Y, et al. CHEK1: A hub gene related to poor prognosis for lung adenocarcinoma[J]. Biomark Med, 2022, 16(2):83-100.
TAN Z, CHEN M, WANG Y, et al. CHEK1: A hub gene related to poor prognosis for lung adenocarcinoma [J]. Biomark Med, 2022, 16(2): 83-100.
ABE H, ALAVATTAM K G,KATO Y,et al.CHEK1 coordinates DNA damage signaling and meiotic progression in the male germline of mice[J].Hum Mol Genet, 2018, 27(7): 1136-1149.
LIN T Y, CHAN H H, CHEN S H, et al.BIRC5/Survivin is a novel ATG12-ATG5 conjugate interactor and an autophagy-induced DNA damage suppressor in human cancer and mouse embryonic fibroblast cells[J]. Autophagy, 2020, 16(7): 1296-1313.
WILSON P M, DANENBERG P V, JOHNSTON P G, et al. Standing the test of time: Targeting thymidylate biosynthesis in cancer therapy[J]. Nat Rev Clin Oncol, 2014, 11(5): 282-298.
FANG L, LIU Q, CUI H,et al. Bioinformatics analysis highlight differentially expressed CCNB1 and PLK1 genes as potential anti-breast cancer drug targets and prognostic markers[J].Genes, 2022, 13(4): 654.
HAGAN I M, GRALLERT A. Spatial control of mitotic commitment in fission yeast[J].Biochem Soc Trans, 2013, 41(6): 1766-1771.

