人工智能在药物再利用治疗新型冠状病毒感染研究中的应用及问题分析
单士喆,文博,乔天慈,单光存
(1.中国中医科学院广安门医院,北京 100032;2.香港都会大学,香港 999077;3.上海中医药大学附属岳阳中西医结合医院,上海 200437;4.北京航空航天大学仪器科学与光电工程学院/北京大数据与精准医疗高精尖创新中心,北京 100191)
新型冠状病毒感染(Corona Virus Disease 2019,COVID-19)是由新型冠状病毒(severe acute respiratory syndrome coronavirus 2,SARS-CoV-2)引起的。SARS-CoV-2 是一种β 冠状病毒,传染性强,通过飞沫传播的能力及其在无生命物体表面的稳定性均很高,对全球健康造成威胁[1]。2023 年5 月5 日,世界卫生组织指出,COVID-19 不再构成国际关注的突发公共卫生事件,然而同时指出,这并不意味着COVID-19作为一种全球健康威胁已经结束,目前是一个既定和持续的卫生问题[2]。2020年以来,COVID-19 的出现和快速蔓延,对社会、经济和公众健康等构成了严重挑战。同时,在寻找有效的COVID-19 治疗药物的过程中,药物再利用引起广泛关注。药物再利用是发掘现有药物在原有适应证之外的新用途,比开发新药更快,成本更低,这些优势在COVID-19 全球大流行中尤其明显[3]。人工智能(artificial intelligence,AI)能够从庞大复杂的数据中挖掘出信息和规律,具有速度快和效能高的特点,成为药物再利用的有效工具,可加快研发速度,降低研发成本,在较短时间内开发更多的治疗策略和候选药物,展示出突出的应用潜力和发展前景。目前,AI 应用于药物再利用的方法分为基于网络的模型、基于结构的方法和机器/深度学习方法3 类,已在药物再利用治疗COVID-19 研究中取得一定进展。本文对该进展进行综述,以期为AI 技术进一步用于治疗COVID-19 药物研究提供参考。
1 Al在药物再利用治疗COVlD-19研究中的应用方法和原理
基于AI 的药物再利用策略已被证明是加速药物发现流程的一种高效途径。在寻求COVID-19治疗策略的过程中,已应用该方法开展了诸多研究。本文归纳了AI 在药物再利用治疗COVID-19 研究中应用的3类主要方法:基于网络的模型、基于结构的方法和机器/深度学习方法。如果研究中使用了混合方法,如同时使用了网络建模和机器学习的方法,则根据占主导地位的方法进行归类。例如,研究中网络建模的方法占主导地位,则该研究归为基于网络的模型。该3 类方法均依托于大型、高质量数据集,并依据各自方法特性而各有侧重的数据需求。三者在药物再利用计算过程中常用的数据集类型见图1。

图1 药物再利用治疗新型冠状病毒感染(COVlD-19)研究中3类人工智能(Al)驱动的方法——基于网络的模型、基于结构的方法和机器/深度学习方法常用的数据集(使用Figdraw绘制).SARS-CoV-2:新型冠状病毒;ACE2:血管紧张素转化酶2.
基于网络的模型因其整合多源数据的优势,在药物再利用研究中广泛应用,其原理是通过构建包含疾病、药物及蛋白质等生物医学实体间相互作用的复杂网络架构,涵盖药物-药物靶点相互作用网络、疾病相关基因网络和信号传导通路网络等层面[4]。在此框架下,通过解析网络中的节点连接关系和内在数据特征,实现对已批准药物针对特定疾病如COVID-19 潜在新用途的预测(图2)。该方法着重于挖掘潜在关联性,更多地关注药物作用在网络层次上的全局影响,可通过网络相似性分析、扩散算法和模块检测等方法寻找与已知疾病相关节点接近的药物靶点,从而捕获药物与疾病间的复杂相互作用,揭示药物在不同疾病之间的共同作用机制,为药物再利用研究提供新的研究线索。
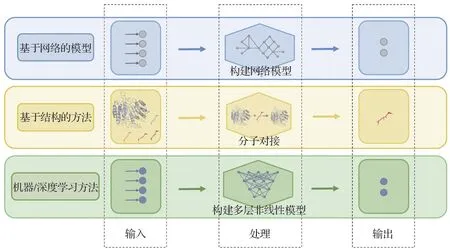
图2 基于网络的模型、基于结构的方法和机器/深度学习方法的核心原理.在基于网络的模型和机器/深度学习方法的原理图中,节点代表药物、疾病或其他相关生物医学实体,线段则表示这些实体之间关联性.基于网络的模型更多地关注药物作用在网络层次上的全局影响,基于结构的方法通过药物分子与靶标蛋白间的三维结构匹配以实现筛选,机器/深度学习方法侧重于对特征的多层分析.
基于结构的方法着重于分析药物化学结构与生物靶标的相互作用机制,其核心依赖于运用蛋白质和药物的三维结构信息进行计算模拟与虚拟筛选。该方法的原理建立在结构相似性原理基础上,即假定结构类似的药物可能具备相近的生物活性特征。通过构建和解析药物分子的立体构型模型,可预测其与目标蛋白质间的结合效能(图2)。此方法的核心策略在于通过比较药物分子间的结构相似性揭示其潜在的药效共性,从而发掘具有类似结构药物的再利用潜力[5]。
机器/深度学习方法的原理是立足于大规模数据集,通过深入挖掘疾病信息、药物属性数据、生物活性资料以及临床研究等大量数据,自动提取高阶抽象特征,实现对关键特征的有效提取和复杂模式的精准识别。以此为基础构建多层非线性模型,旨在揭示潜在的药物-疾病作用规律,并预测可能具有再利用价值的药物(图2)。该方法的核心优势在于运用大规模数据资源与复杂模型的强大学习能力,从多层次探寻关联性,从而发掘药物的再利用潜力,为药物在新疾病领域的应用提供前瞻性的预测依据[6]。
2 Al在药物再利用治疗COVlD-19研究中的优劣势分析
2.1 基于网络的模型
基于网络的模型适用于分析和挖掘药物-靶标之间的关联性和蛋白质相互作用。依托充足的实验数据库和临床试验成果,此类模型通过实施网络分析及算法预测手段,能够揭示潜在的药物与靶标的新型关联,并识别出新的药物靶点及共享相似作用机制的药物或药物组合,从而为药物再利用提供候选清单[7]。此外,该方法还可应用于探究与疾病相关的信号通路和基因调控网络,精准挖掘药物靶标及其代谢通路信息,进一步丰富药物再利用研究的理论依据。
基于网络的模型的优势在于可以从系统生物学的角度理解药物的作用机制,并且可能揭示出药物对整个生物网络的调控效果,从而找到新的治疗途径。然而,由于该模型依赖于相互作用关系而非具体的结构细节,故在预测准确性上存在固有局限性[8]。同时,其效能受到所用数据质量和现有知识库完备程度的显著制约,在应对如SARS-CoV-2 等新病毒或未充分了解的传染病场景时,预测效果尤为受限。此外,尽管该模型能挖掘出复杂关联模式,但这些关联并不直接等同于实际的生物化学相互作用,故所获结果需经过实验验证和深入研究加以确认。
2.2 基于结构的方法
基于结构的方法通过分析药物化学结构及其靶点间三维相互作用,能够在理论层面上高效鉴别出潜在候选药物。当目标蛋白的结构数据丰富且具有与已知药物相似结构的化合物库时,该策略尤显优势。此法适用于目标蛋白的三维结构已被充分解析且有关联药物结构数据库的情境,侧重于药分子构型及其交互特性,从而精确预测分子对靶点的结合能和亲和力,筛选可能具有效能的化合物[9]。此法凭借高通量计算模拟和结构导向筛选机制,在大规模药物筛选中体现出高效能与高精度特点,通过计算模拟基于蛋白-药物结构信息的筛选,可迅速缩小候选药物范围。借助详尽的蛋白质和药物结构信息,该方法能够深入挖掘化学空间中的多样性化合物,有助于新骨架药物的发掘以及创新治疗策略的设计[10]。然而,此方法的有效应用依赖于丰富而精确的靶标蛋白和药物结构信息,若针对的是结构未明确的新型病毒或蛋白,其效用可能受限。此外,考虑到基于结构的设计通常忽视生物体内环境的复杂性,导致其预测结果可能存在一定偏差。
2.3 机器/深度学习方法
机器/深度学习方法在处理大数据时展现出了显著优势,其通过整合多元类型的数据资源,能够从多维度训练模型以识别潜在的候选药物。该方法凭借高度的灵活性和自动化特性,擅长解析复杂、非线性的数据模式,从而有效地学习并揭示深藏其中的相关性和规律[11]。在药物再利用研究领域,此类方法通过对现有药物的结构特性和作用机制进行深入分析,能够快速筛选出具有潜在抗病毒活性的候选药物,并能用于预测特定药物的抗病毒效果、副作用及毒性等。尽管其具备强大的预测效能,能够挖掘庞大数据集中的隐性知识和特征,但机器/深度学习模型的预测性能高度依赖于高质量且充足的训练数据[12]。同时,这类方法的应用需要消耗大量的计算资源和时间来完成模型训练与优化过程。此外,模型复杂性与其解释性之间的平衡亦是关键挑战之一,虽然复杂的模型往往能在训练数据上表现出卓越性能,但可能牺牲了可解释性和可扩展性,使得评估模型的可靠性和泛化能力变得较为困难。
尽管该3类方法在某些方面存在交集且能相互补充,但它们在应用侧重点上仍有所区分。图3 比较了三者在不同应用场景下的性能优势及局限性。在实际研究或应用场景中,应依据具体任务需求和现有数据条件,选择合适的方法或将多种方法相结合,以优化药物再利用的研究策略。
3 Al在药物再利用治疗COVlD-19研究中的应用
3.1 基于网络的模型
基于网络的模型在药物再利用研究中占据重要地位,其能够整合多种医学实体间的相互作用关系,以发现潜在药物及药物组合。Zhou 等[13]提出了一种整合抗病毒药物再利用策略,使用网络方法量化了人类冠状病毒(human coronavirus,HCoV)与宿主之间的相互作用及人类蛋白质网络中药物靶点之间的相互作用。通过网络邻近性分析,筛选出16种潜在的抗HCoV 药物,并通过药物基因特征的富集分析进行验证。此外,该文还预测了潜在的药物组合。Sadegh 等[14]开发了一个用于SARSCoV-2 宿主相互作用组探索和药物靶点鉴定的在线交互平台,实现了基于网络的系统医学算法进行候选药物预测,识别出缓激肽B1 受体是SARSCoV-2 入侵宿主细胞的潜在作用目标,并指出其可由临床用于治疗COVID-19的血管紧张素转化酶抑制剂靶向,同时确定了6种靶向缓激肽B1受体的相关药物。Tomazou 等[15]设计了一种结合多组学数据的网络集成方法,以优先排序与COVID-19 高度相关的基因,据此重新评估并整理了候选药物清单。Choudhary 等[16]将基于网络的预测手段与倾向评分匹配的观察性研究相结合,提供了一个综合网络医学平台,用于预测COVID-19相关疾病表现,并推荐褪黑激素作为潜在预防和治疗COVID-19的药物选项。Alakwaa 等[17]开发了一种基于公共单细胞RNA 测序(scRNA-seq)数据和药物干扰数据库的“网络化细胞信号集成库”(LINCS),成功识别出数种有潜力治疗COVID-19 的候选药物,并特别推荐了4种优先考虑的药物。
3.2 基于结构的方法
基于结构的方法能够通过识别与大分子靶标紧密结合的小分子化合物,评估化合物与其生物配体的相互作用,从而发掘现有药物的新应用潜力[18]。Artese 等[19]运用基于结构的虚拟筛选技术成功识别了14 种针对SARS-CoV-2 的候选化合物,其中头孢洛赞(ceftolozane)和还原型辅酶Ⅰ(NADH)表现出优秀的多靶点属性,可能有助于减少耐药病毒株的产生。Feng等[20]介绍了一种新的计算机识别模式方法——分子复合物表征系统(MCCS),该方法通过考虑关键残基的作用提升了虚拟筛选的准确性,并推荐了若干抗病毒如人类免疫缺陷病毒和丙肝病毒的药物,以及对抗糖尿病的药物作为治疗COVID-19的潜在方案。Moovarkumudalvan等[21]运用高通量结构虚拟筛选方法对美国FDA 批准的LOPAC 药物库进行研究,发掘针对SARSCoV-2 细胞入侵机制的病毒进入抑制剂。Yadav等[22]利用分子对接和分子动力学模拟研究了2种已批准的胆汁盐——鹅去氧胆酸盐和熊去氧胆酸盐是否能结合SARS-CoV-2 包膜蛋白,进而探讨其用于COVID-19 治疗的可能性。Elfiky 等[23]则结合了序列分析、建模和对接手段,构建了COVID-19 RNA 依赖性RNA 聚合酶(RdRp)模型,并推荐索非布韦(sofosbuvir)、IDX-184、利巴韦林(ribavirin)和瑞德西韦(remidisvir)作为有潜力的治疗选项。以美国FDA 批准药物作为候选药物的研究为重点,表1汇总了应用基于结构的方法在药物再利用治疗COVID-19研究中获得的部分进展。
3.3 机器/深度学习方法
机器/深度学习作为连接特征与先验知识的有效工具,可以融合例如研究药物适应证、靶点和不良反应等大量数据,通过整合丰富的高质量数据以优化特定问题的识别与决策。Zeng 等[39]开发了1 种集成化网络深度学习方法——COV-KGE,该方法系统地结合了药物、疾病、蛋白质/基因、通路和表达数据间的复杂联系,并对SARS-CoV-2 感染人体细胞产生的转录组学和蛋白质组学数据进行深入分析,最终成功鉴别出41 种潜在治疗COVID-19 的候选药物。Giagulli 等[40]构建了一个基于随机森林的机器学习模型,筛选出了230 种可能有助于限制SARS-CoV-2 传播或降低COVID-19 死亡率的药物,其中皮质类固醇如布地奈德(budesonide)、地塞米松(dexamethasone)和倍他米松(betametha-sone)排名靠前。值得注意的是,雌二醇被确定为针对COVID-19的候选药物之一,而睾酮及其衍生物未被选中,这进一步支持了男性患者临床表现较差可能与睾酮水平有关的理论。Ke 等[41]提出了一种深度学习方法,用于快速高通量筛查具有抑制SARS-CoV-2 活性的化合物。他们建立猫冠状病毒体外复制细胞模型,采用AI鉴定的药物进行抗病毒活性验证,并将抗病毒活性结果反馈至AI系统进行再训练。经过多次循环学习预测,AI 系统最终确认了80种具有潜力的上市药物。Beck等[42]利用名为分子转换体-药物靶点相互作用(MT-DTI)的深度学习预测模型,筛选可能干扰SARS-CoV-2 病毒组分的抗病毒药物。Hsieh等[43]构建了一个全面的知识图谱,利用深度图神经嵌入技术连接病毒诱饵、宿主基因、通路、药物和表型之间的多重相互作用,并通过该方法鉴定了22 种候选药物。Han 等[44]则采用细胞图像特征和机器学习对药物作用机制进行建模,运用监督式学习算法——信息理论度量学习(ITML)以及聚类方法优化药物图像特征的度量,以发现具有相似作用机制的药物。例如,氯喹(chloroquine)通过改变细胞内环境、提高细胞内pH 值等方式抑制COVID-19 的内吞作用实现抗病毒效果,研究人员通过细胞图像确认克罗米芬(clomiphene)通过与氯喹类似的抑制内吞作用实现抗病毒效果,且已发现克罗米芬能够抑制埃博拉病毒侵入细胞,这进一步提示其可能具备针对COVID-19的潜在治疗潜力。
在药物再利用治疗COVID-19 的研究中,上述3 类方法已取得实质性应用成效,并展现出对其他疾病药物研发的潜在适用性。它们的核心原理在于运用大规模数据集成与学习算法预测化合物与疾病间的相互作用关系。当推广至不同疾病或药物时,应根据特定疾病特性、药物属性以及现有数据资源,针对性地调整并优化模型训练过程,确保模型在新疾病场景下的准确性和可靠性,以期获得更为精确的预测成果。
4 Al在药物再利用治疗COVlD-19研究中面临的问题及原因分析
AI在药物再利用治疗COVID-19研究中发挥着关键作用,然而迄今所取得的结果并未达到理想预期。尽管众多研究预测了药物潜在的有效性,但不同研究针对同一靶点的药物筛选结果往往不一致,实验验证层面的有效证据相对匮乏,且绝大多数候选药物尚未在临床环境中得到证实。尽管对药物再利用治疗COVID-19 抱有高度期待,但该领域仍面临严峻挑战。要全面理解AI 在药物再利用治疗COVID-19 过程中的应用瓶颈,需从药物发现和开发、非临床研究到临床试验3 个阶段进行深入分析和问题梳理。
4.1 药物发现和开发阶段
AI 模型的训练依赖大量数据,而数据的质量与数量直接影响模型在药物再利用中的准确性与有效性。目前,AI 在该领域的贡献似乎未达到预期水平,其原因可能在于纯数据驱动的策略存在局限性,尤其是在缺乏足够高质量数据的情况下,难以构建出性能优异、泛化能力强的模型。对于SARSCoV-2 而言,现有的认识及其与宿主间的相互作用尚处于初级阶段,可用的高质量数据与知识储备相对匮乏,这进一步凸显了基础研究的重要性,基础研究起到了“源头活水”的作用,在生物医学领域发展中具有不可替代的地位[45]。此外,基于机制推断的AI 结果并不能直接保证临床疗效。尽管AI 是一种强大的工具,但数据驱动的模型仅能提出潜在的候选药物供进一步研究,即使所提出的药物靶向病毒致病过程中的关键蛋白,其是否具有抗病毒活性仍需通过后续实验验证。例如,抑制某种辅助因子以防止病毒对宿主蛋白的操纵,也可能产生未预见的不良反应或前病毒效应。因此,对待AI所得的结果应持审慎态度,须在通过适当方法验证药物靶点后,由临床专家进行严谨审查,并按照规定的程序和临床试验进行评估。
4.2 非临床研究阶段
细胞及动物实验在模拟病毒于人体内感染的复杂宿主环境时存在固有限制。由于体外培养细胞与体内真实细胞在蛋白质表达、生物学特性上存在显著差异,这在评估药物渗透性、吸收性能和代谢途径时易引入偏差,尤其是对于涉及呼吸系统、消化系统以及免疫反应等多系统交互作用的生理过程无法进行全面再现。而动物模型,因其在解剖结构、生理功能和免疫系统上与人类的显著差异,难以准确反映病毒在人体内的复制、临床特征和病理反应。因此,可能出现AI预测药物有效性得到临床前实验验证支持,但当推进至临床试验阶段时,实际疗效却未达到预期的现象。
4.3 临床试验阶段
首先,候选药物在原适应证中的优化可能限制了其在其他方向的再利用潜力。候选药物可能已经针对初始适应证对特定靶点、剂量或组织分布进行了优化,这些优化的药物在应对COVID-19 时可能无法展现最佳的治疗效果。此外,AI 模型可能未充分考虑药物的吸收率和作用机制等可能存在的其他制约临床应用的关键特性。例如,洛哌丁胺(loperamide)作为潜在药物,已证实其能抑制中东呼吸综合征冠状病毒和SARS-CoV的体外复制[46]。然而,洛哌丁胺口服生物利用率低且不能穿透血脑屏障[47],这些因素在预测其对COVID-19 治疗效果时并未得到充分考虑。尽管体外研究显示出洛哌丁胺治疗COVID-19 的潜在价值,但其临床应用却面临挑战。同时,遗传异质性对临床治疗结果的影响亦不容忽视。SARS-CoV-2 感染表现出显著的个体间差异,从无症状到致命疾病不等,研究推测遗传背景可能是决定临床特征和药物反应的因素之一[48]。另一方面,COVID-19 是一种复杂的多系统疾病,其临床表现多样,从初期的呼吸系统疾病发展至涉及多个器官系统的复杂病理过程。Carlos等[49]将COVID-19 分为4 个临床分期:Ⅰ期病毒进入和复制(无症状)、Ⅱ期病毒传播(轻度或中度)、Ⅲ期多系统炎症(重度)及Ⅳ期内皮损伤、血栓和多器官功能障碍(危重),但各阶段之间的进展并非绝对线性,部分患者可直接发展为晚期。不同疾病阶段可能导致药物效应的变化,如RECOVERY 试验所示,地塞米松能够降低接受呼吸支持治疗的COVID-19 患者的28 d 死亡率,但对无需吸氧的患者并无明显益处[50],这提示药物使用的时机或许是导致AI 预测与临床效果之间存在差距的一个重要因素。
5 结语
尽管AI 应用于药物再利用治疗COVID-19 研究中面临多种挑战,但这些挑战同时也揭示了有待深入挖掘和完善的若干关键领域。具体而言,针对3 类主要的药物再利用方法,其在治疗COVID-19药物研究中的作用和局限性值得深入分析。基于网络的模型擅长整合多源数据,通过揭示疾病、药物及生物靶点间的复杂关系网络,发掘潜在的药物重定位线索。然而,此类方法的有效性受限于现有数据的质量和完整性,且对新型或未知病毒如SARS-CoV-2 的应用可能因缺乏足够的背景信息而受到制约。未来发展方向应注重优化数据集的构建与更新,并借助更高级别的网络算法提高预测准确度。基于结构的方法聚焦于药物分子与靶点蛋白之间的三维结构相互作用,以识别具有治疗潜力的化合物。尽管这类方法能够提供精确的药物筛选依据,但依赖于高质量的蛋白质三维结构数据,对于新出现的病原体如SARS-CoV-2,由于结构信息不足,可能导致预测结果的不确定性增大。为克服这一瓶颈,有必要加大对相关结构生物学研究的支持力度,并开发更为稳健和普适的结构预测工具。机器/深度学习技术凭借强大的数据处理能力和复杂模式识别优势,在药物发现与再利用中展现出巨大潜力。它们能有效利用大规模生物医学数据集进行训练,从而预测药物活性和潜在的新适应症。然而,当前AI 模型往往被视为“黑盒”,模型的可解释性、泛化能力及其预测结果的可靠性仍有待提升,这需要科研人员不断优化模型结构,引入更多维度的数据并建立严格的验证机制以确保计算结果的科学性和临床转化价值。
随着对SARS-CoV-2 生物学特性的深入理解,以及各类医学数据资源在质量、规模和开放获取方面的持续改进,AI 在挖掘新型候选药物方面的效能将进一步增强。坚定地推进AI技术的发展与优化,将有助于实现药物筛选和评估的更高精度,并有望在未来提供更多创新视角和解决方案,以应对各种复杂疾病的治疗需求。

