眼表外胚层与表面外胚层转录组差异分析
孙 露,张灿伟,宋玉雯,李建新,段 练,高 阳,谢月美,王露萍,党光福
•AIM:To identify transcriptional differences between the ocular surface ectoderm (OSE) and surface ectoderm (SE) using RNA-seq, and elucidate the OSE transcriptome landscape and the regulatory networks involved in its development.
•METHODS:OSE and SE cells were differentiated from human embryonic stem (hES) cells. Differentially expressed genes (DEGs) between OSE and SE were analyzed using RNA-seq. Based on the DEGs, we performed gene ontology (GO) analysis, Kyoto Encyclopedia of Genes and Genomes (KEGG) pathway enrichment analysis, and protein-protein interaction (PPI) network analysis. Transcription factors (TFs) and hub genes were screened. Subsequently, TF-gene and TF-miRNA regulatory networks were constructed using the NetworkAnalyst platform.
•RESULTS:A total of 4 182 DEGs were detected between OSE and SE cells, with 2 771 up-regulated and 1 411 down-regulated genes in OSE cells. GO-BP analysis revealed that up-regulated genes in OSE were enriched in the regulation of ion transmembrane transport, axon development, and modulation of chemical synaptic transmission. Down-regulated genes were primarily involved in nuclear division, chromosome segregation, and regulation of cell cycle phase transition. KEGG analysis indicated that up-regulated genes in OSE cells were enriched in signaling pathways such as cocaine addiction, axon guidance, and amphetamine addiction, while down-regulated genes were enriched in proteoglycans in cancer, ECM-receptor interaction, protein digestion and absorption, and cytokine-cytokine receptor interaction. Additionally, compared with SE, 204 TFs (including FOS, EGR1, POU5F1, SOX2, and PAX6) were up-regulated, and 80 TFs (including HAND2, HOXB6, HOXB5, HOXA5, and HOXB8) were down-regulated in OSE cells. Furthermore, we identified 6 up-regulated and 9 down-regulated hub genes in OSE cells, and constructed TF-gene and TF-miRNA regulatory networks based on these hub genes.
•CONCLUSIONS:The transcriptome characteristics of OSE and SE cells were elucidated through RNA-seq analysis. These findings may provide a novel insight for studies on the development and in vitro directed induction of OSE and corneal epithelial cells.
0 引言
眼表外胚层(ocular surface ectoderm,OSE)属于表面外胚层(surface ectoderm,SE),是眼表上皮(即角膜和结膜上皮)在胚胎时期的前体细胞[1],其对眼表正常发育及出生后正常视觉功能的维持至关重要。在胚胎发育过程中,前神经板发育而来的视网膜前体细胞向覆盖在其表面的表面外胚层延伸,然后该区域的表面外胚层增厚并产生假定的角结膜上皮[2]。因此,眼表外胚层在发育过程中受到表面外胚层以及来自视网膜前体细胞等神经外胚层的双重信号调控。目前表面外胚层细胞发育调控机制已较明确[3],分析眼表外胚层与表面外胚层细胞基因表达差异,有助于阐明眼表外胚层发育调控机制。基于此,本研究通过对人胚胎干细胞(human embryonic stem cells,hES)来源的眼表外胚层和表面外胚层进行转录组测序(RNA sequencing,RNA-seq)分析,筛选出眼表外胚层与表面外胚层的差异表达基因(differential expressed genes,DEGs)、转录因子(transcription factor, TF)及Hub基因,并进行基因本体(gene ontology, GO)分析、京都基因组百科全书(Kyoto Encyclopedia of Gene and Genomes, KEGG)分析及蛋白质-蛋白质相互作用(protein-protein interaction, PPI)分析,还针对Hub基因构建了TF-gene调控网络和TF-miRNA调控网络,在转录组水平揭示了二者表型及信号调控的差异,可为眼表外胚层转录组特征及发育调控机制研究提供指导。
1 材料和方法
1.1材料本研究中所用到的人胚胎干细胞均购自美国康宁公司,且相关实验工作均已获得山东第一医科大学第一附属医院医学伦理委员会批准,并按照相关规定进行。
1.2方法
1.2.1人胚胎干细胞培养及诱导分化人胚胎干细胞H1细胞株接种于1%浓度的matrigel包被的培养板,采用mTeSRTM 1或mTeSRTM plus培养基(StemCell Technologies)培养。每5-6 d按1∶10的比例传代一次。眼表外胚层细胞诱导参照以往文献报道的方法[4-5]。首先,将 hES 细胞接种于LN511包被的培养皿,mTeSRTM 1或mTeSRTM plus培养基培养4-7 d,然后将培养基改为分化培养基,成分见表1。培养3 wk后,在诱导的细胞中即可同时形成神经外胚层、眼表外胚层和其他表面外胚层细胞,在显微镜下手动刮除神经外胚层和其他表面外胚层后即可获取眼表外胚层细胞。表面外胚层细胞诱导:每平方厘米6.2×103个人胚胎干细胞接种于1%浓度的matrigel包被的培养板上。第2 d,将培养基改为含1 μmol/L RA(Sigma)和5 ng/mL重组人BMP-4(R&D Systems)的E6培养基培养,2 d换液1次,培养7 d。
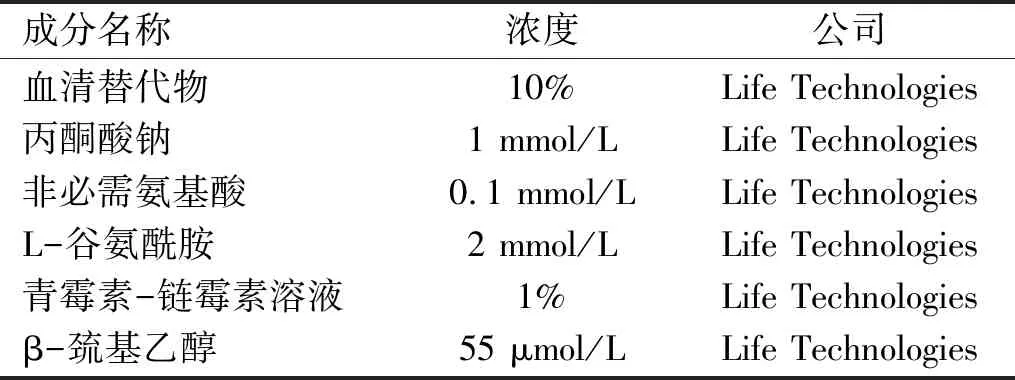
表1 细胞分化培养基成分表
1.2.2免疫荧光染色4%多聚甲醛固定细胞后,用PBS缓冲液冲洗。然后山羊血清孵育30 min,一抗在4 ℃孵育过夜。PBS缓冲液冲洗后加入二抗在37 ℃孵育1 h。PBS缓冲液冲洗后DAPI染色,PBS缓冲液冲洗后在荧光显微镜下采集细胞免疫荧光图像。
1.2.3定量实时逆转录酶PCR 使用RNeasy Mini Kit (QIAGEN)提取人胚胎干细胞及人胚胎干细胞诱导的眼表外胚层、表面外胚层细胞的总RNA。随后使用PrimeScript Real-Time Master Mix Kit (Takara)合成cDNA后进行定量实时逆转录酶PCR(qRT-PCR)。PCR程序:95 ℃变性3 min,然后进入热循环程序:95 ℃变性5 s和60 ℃退火和延伸30 s,重复40个循环。
1.2.4转录组测序及RNA-Seq数据分析提取的眼表外胚层和表面外胚层细胞的总RNA,检测质量合格后,使用 TruSeq Stranded mRNA Library Prep kit试剂盒构建文库。随后总RNA使用NextSeq 500(Illumina)进行转录组测序。差异基因及GO、KEGG分析:使用R4.1.2版进行基因表达分析,并应用DEseq2分析眼表外胚层、表面外胚层细胞中基因水平的差异表达,筛选出|log2(fold change)|>1和Padj<0.05的DEGs,使用R软件进行火山图谱绘制。随后,利用GO分析和KEGG分析对DEGs进行功能富集分析,以确定DEGs的主要生物学功能和相关的信号通路。转录因子分析:通过hTFtarget database 数据库(http://bioinfo.life.hust.edu.cn/hTFtarget)获取人类转录因子基因集,将差异基因与转录因子进行比较、筛选和校正。用R软件筛选|log2(fold change)|>1和Padj<0.05的转录因子,并绘制热图。
1.2.5PPI网络搭建及模块分析为了进一步探讨DEGs之间的相互作用,我们使用STRING数据库(http://string-db.org/)构建PPI网络,设置相关系数绝对值大于0.4[6-7]。使用Cytoscape 软件绘制 PPI 网络。另外,使用Cytoscape插件Cytoscape MCODE筛选关键功能基因模块。
1.2.6Hub基因的筛选应用Cytoscape软件中的cytoHubba 插件筛选Hub基因。然后,我们选择7种算法(MCC、MNC、Degree、EPC、Closeness、Radiality、Stress)的交集,最终得到Hub基因。最后,通过GeneMANIA10 (http://www.genemania.org/) 构建了Hub基因的共表达网络[6]。
1.2.7TF-gene调控网络和TF-miRNA调控网络的构建通过Networkanalyst数据库(https://www.networkanalyst.ca/)构建了TF-gene和TF-miRNA调控网络。随后通过Cytoscape软件可视化。
统计学分析:使用GraphPad Prism(GraphPad, San Diego, CA)软件进行统计分析。所有实验数据均以均数±标准差表示。采用单因素方差分析对各组间数据进行统计学检验,两组间比较采用独立样本t检验,以P<0.05为差异有统计学意义。
2 结果
2.1人胚胎干细胞诱导分化及相关标记物的鉴定人胚胎干细胞在分化培养基中被诱导,逐渐形成了3个同心细胞团(神经外胚层、眼表外胚层和其他表面外胚层细胞),见图1A,定向分化成表面外胚层细胞,见图1C。诱导的眼表外胚层细胞高表达PAX6、p63、K18、K8,而在神经外胚层细胞高表达PAX6,表面外胚层细胞高表达p63、K18、K8(图1B、D)。相对定量的qRT-PCR结果显示与免疫荧光染色的结果相一致(图1E)。选取一些眼表外胚层标记物,包括KRT8、KRT18、KRT19、PAX6、TP63、CDH1、TFAP2A、FOXC1,绘制成热图,见图1F。

图1 hES细胞诱导分化及特异性标记物鉴定图 A:hES细胞在角膜分化培养基分化后显微镜下照相; B:对分化的多胚层细胞团进行神经细胞标志物PAX6(红色)和角膜上皮发育相关标志物p63(绿色)、K18(红色)和 K8(绿色)的免疫荧光染色结果。细胞核蓝色;C:显微镜观察定向分化的SE细胞;D:对SE细胞进行神经细胞标志物PAX6(红色)和角膜上皮发育相关标志物p63(绿色)、K18(红色)和K8(绿色)的免疫荧光染色;E:以 hES 为对照组,OSE和SE细胞中TP63、PAX6、KRT8、KRT18基因的表达情况;F:常见眼表外胚层标记物基因热图。
2.2DEGs分析对眼表外胚层与表面外胚层之间的RNA-Seq数据进行了差异基因表达分析。相对于表面外胚层,眼表外胚层共有4 182个基因发生差异性表达,包含2 771个上调基因和1 411个下调基因(图2A),Padj值<0.05。按log2(fold change)值大小排序,前100位差异基因见图2B。

图2 RNA-Seq数据分析图 A:OSE与SE的 DEGs火山图。红色点代表上调,绿色点代表下调;B:OSE与SE的前100个差异基因热图;C:OSE与SE差异基因的GO功能富集的气泡图;D:OSE与SE差异基因的KEGG Pathway分析;E:OSE与SE差异基因的转录因子分析热图。
2.3GO和KEGG富集分析利用基因本体进行了GO分析。GO富集分析包括3个分支,即生物学过程 (biological process,BP)、细胞组分(cellular component,CC)和分子功能(molecular function,MF)。在GO-BP分析中,眼表外胚层上调的DEGs主要富集在离子跨膜转运的调节(regulation of ion transmembrane transport)、轴突发育(axon development)、调节化学突触传递(modulation of chemical synaptic transmission)等相关生物学过程,下调的DEGs主要富集在核分裂(nuclear division)、染色体分离(chromosome segregation)、细胞周期相变的调控(regulation of cell cycle phase transition)等生物学过程。在GO-CC分析中,眼表外胚层细胞上调的DEGs多定位在神经元细胞体(neuronal cell body)、含胶原蛋白的细胞外基质(collagen-containing extracellular matrix)、突触膜(synaptic membrane)等细胞结构中,下调DEGs主要定位在染色质区域(chromosomal region)、转轴(spindle)、浓缩染色质(condensed chromosome)等细胞结构中。在GO-MF分析中,眼表外胚层细胞上调的DEGs富集于信号受体调节活动(signaling receptor regulator activity)、信号受体激活剂活性(signaling receptor activator activity)、受体配体活性(receptor ligand activity)等分子功能,而下调的DEGs则更多地富集于微管蛋白结合(tubulin binding)、微管结合(microtubule binding)、细胞骨架运动活性(cytoskeletal motor activity)等分子功能(图2C)。在KEGG分析中,我们筛选了DEGs富集的20条通路(上调10条、下调10条),其中可卡因成瘾(cocaine addiction)、轴突导向(axon guidance)、苯丙胺成瘾(amphetamine addiction)、cAMP 信号(cAMP signaling pathway)等信号通路显著上调,癌症中的蛋白聚糖(proteoglycans in cancer)、ECM-受体相互作用(ECM-receptor interaction)、蛋白质消化和吸收(protein digestion and absorption)、细胞因子-细胞因子受体相互作用(cytokine-cytokine receptor interaction)等信号通路显著下调(图2D)。
2.4转录因子分析转录因子通过调控其他基因的表达在细胞表型决定及生物学功能中发挥关键调控作用。因此进一步分析了眼表外胚层和表面外胚层的差异转录因子,共筛选了284个转录因子。FOS、EGR1、POU5F1、SOX2以及PAX6等204核心转录因子在眼表外胚层中高表达,HAND2、HOXB6、HOXB5、HOXA5以及HOXB8等80个核心转录因子在表面外胚层中高表达。在眼表外胚层高表达转录因子中,FOS表达差异最大。图2E展示了眼表外胚层细胞相对于表面外胚层细胞表达上调及下调的前25位转录因子。
2.5PPI网络构建和模块分析将|log2(fold change)|>2的DEGs输入STRING数据库以获取蛋白互作信息,并进一步利用Cytoscape软件进行可视化分析,分别筛选出前6个Degree值最大的DEGs蛋白,见图3A。利用Ctoscape Mcode插件筛选DEGs相关功能模块基因(包含33个上调功能模块基因、13个下调功能模块基因),见图3B。发现上调功能模块基因在趋化作用(chemotaxis、taxis)、MAPK级联的正向调节(positive regulation of MAPK cascade)等生物过程中显著富集(图3C),富集于1型糖尿病(type I diabetes mellitus)、炎症性肠病(inflammatory bowel disease)等信号通路(图3D)。

图3 PPI分析及功能模块分析图 A:PPI网络图(差异表达基因之间的蛋白相互作用网络, 左为上调,右为下调); B:功能基因模块(左为上调,右为下调);C:上调功能模块基因GO-BP功能富集的气泡图(前十位);D:上调功能模块基因KEGG Pathway分析图(前五位)。
2.6Hub基因的选择与分析通过插件cytoHubba的7种算法,在眼表外胚层细胞上调和下调差异基因中分别计算出了排名前30的Hub基因(表2、3)。用venny进行交集后,我们发现了6个常见的上调Hub基因,包括FOS、IL1B、SOX2、IL6、JUN、MMP9,以及9个常见的下调Hub基因,包括COL1A1、POSTN、LUM、PDGFRA、VCAM1、IGF1、FGF10、ENG、BDNF。基于GeneMANIA数据库,我们分析了这些基因的共表达网络和相关功能,其中上调Hub基因物理相互作用为48.20%、共表达为26.40%、预测为23.35%、共定位为1.20%、途径为0.85%,下调Hub基因共表达为86.58%、共定位为9.19%、途径为2.31%、共享蛋白质结构域为1.92%,但未富集到已知的生物学功能(图4A)。

图4 Hub基因鉴定及TF-gene、TF-miRNA信号调控网络图 A:通过GeneMANIA分析Hub基因及其共表达基因图;B:TF-gene相互作用和TF-miRNA共同调控网络(左侧为上调基因,右侧为下调基因)。红色节点代表Hub基因,蓝色节点代表 TF; C:Venny图(OSE高表达TF与上调Hub基因互作TF取交集,筛选共表达TF);D:共表达TF热图;E:TF-miRNA 共同调控网络(左侧为上调,右侧为下调)。
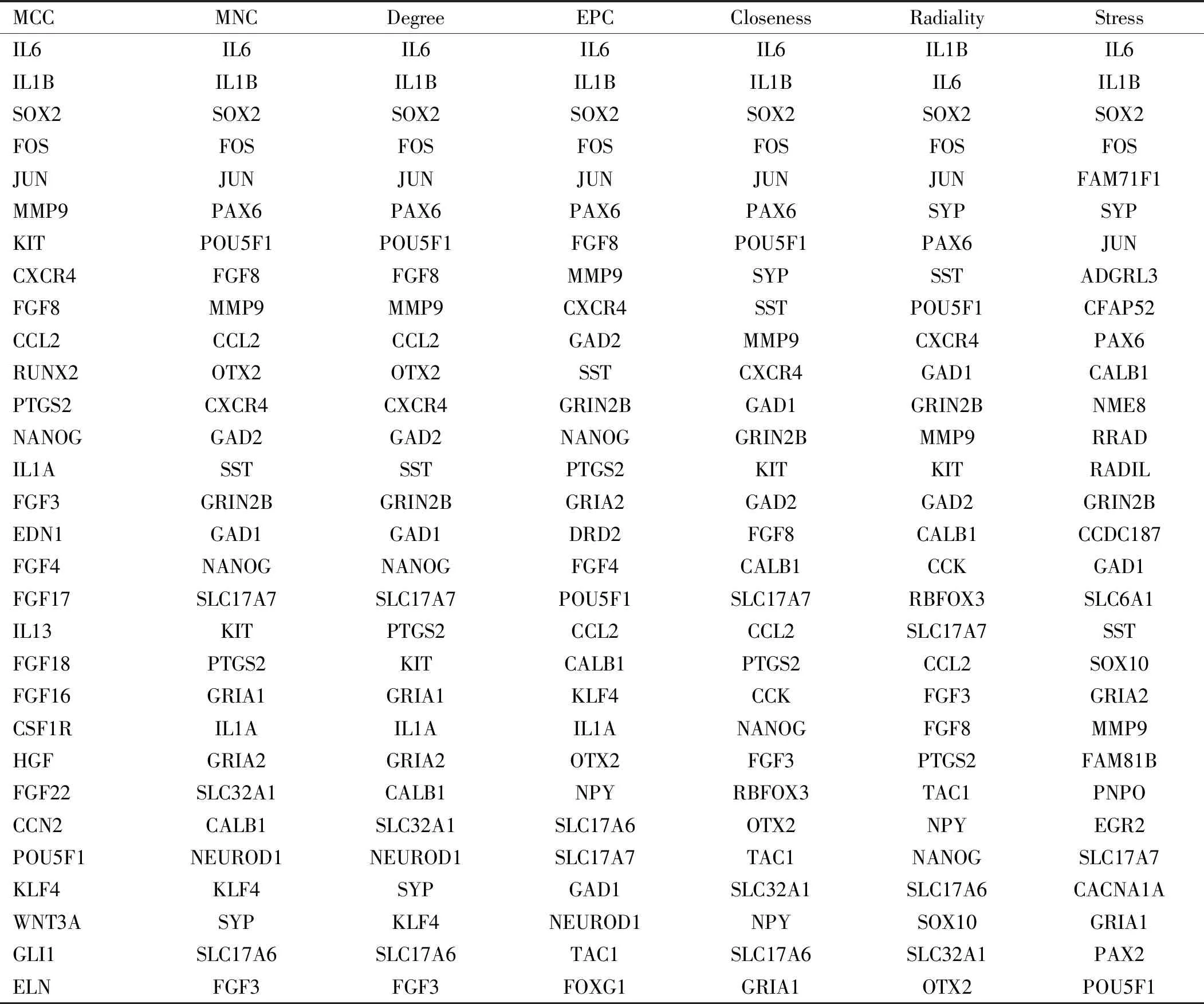
表2 cytoHubba 中上调DEGs前 30 个 Hub 基因排名

表3 cytoHubba 中下调DEGs前 30 个 Hub 基因排名
2.7TF-gene相互作用和TF-miRNA共同调控网络基于NetworkAnalyst数据库预测可与Hub基因相互作用的转录因子,设置最小网络数,然后使用Cytoscape对结果进行可视化(图4B)。上调网络包括150 个节点和 164个边, 其中受转录因子调节最多的基因是JUN,共有73个转录因子参与调控转录过程。随后,我们利用Venny将眼表外胚层高表达TF与上调Hub基因互作TF取交集,筛选出23个共表达TF(图4C),并制成热图见图4D。下调网络包括69个节点和72个边,其中受转录因子调节最多的基因是COL1A1,共有21个转录因子参与调控转录过程。此外,我们还使用NetworkAnalyst构建了TF-miRNA共调控网络,该网络预测了miRNA、TF和Hub基因之间的相互作用,设置最小网络数,最终筛选出由11个节点和24个边组成的上调网络图及由24个节点和40个边组成的下调网络图(图4E)。
3 讨论
眼表外胚层属于表面外胚层,其发育过程中受到表面外胚层发育调控信号和来自神经外胚层调控信号的双重调控。研究发现,眼表外胚层除了表达表面外胚层重要转录因子TP63外,还高表达神经外胚层核心转录因子PAX6[4],而角结膜上皮也与表面外胚层来源的皮肤表现出不同的表型[8]。近年来已有研究阐明了表面外胚层发育调控机制,而眼表外胚层和角结膜上皮发育调控机制仍不明确[3, 5]。
随着干细胞及眼发育领域研究的深入,人胚胎干细胞和诱导的多能干细胞(human induced pluripotent stem cells,hiPSCs)已被广泛应用于眼部器官发育的研究,其在眼部发育调控机制及遗传性眼病治疗方案的开发中发挥重要作用[9-11]。转录组测序是一种高通量测序技术,能够全面、快速地筛选差异表达基因,研究特定组织的基因表达情况,目前已成为疾病及胚胎发育机理研究不可或缺的技术手段。本研究通过对眼表外胚层和表面外胚层转录组数据进行分析,共筛选出4 182个DEGs, 包含2 771个上调基因和1 411个下调基因。从转录组水平阐明了眼表外胚层的转录组特征,明确了眼表外胚层细胞高表达的基因及信号通路。转录因子在决定细胞表型及命运中发挥关键用。既往研究发现神经细胞核心转录因子PAX6 通过促进角膜上皮标记物K3、K12等基因的表达决定角膜上皮细胞命运[12]。此外,FOS家族的成员(cFos、Fra-1、Fra-2及 FosB)与JUN家族(c-Jun、JunB和JunD)可以结合形成异二聚体AP1因子,参与调节角膜上皮细胞的分化及维持细胞稳态[13-14],在差异基因分析中,我们发现PAX6和FOS高表达于眼表外胚层细胞。据此,我们推测PAX6、FOS可能参与眼表外胚层发育调控,但其具体作用及机制仍需进一步研究。以往研究还发现神经细胞及干细胞标志物及重要转录因子SOX2在角膜上皮前体细胞中与p63共表达,参与角膜上皮细胞分化调控[15]。在本研究中,我们发现眼表外胚层细胞SOX2表达明显高于表面外胚层,这与以往研究结果相一致。另外,在眼表外胚层相对于表面外胚层高表达DEGs中,PRDM14表达量最大,以往研究发现PRDM14能够维持干细胞干性和多能性[16],但其在眼表外胚层中的作用尚未见报道,仍待进一步研究。
本研究还筛选了决定眼表外胚层和表面外胚层差异的Hub基因,共筛选出了6个上调的Hub基因,包括FOS、IL1B、SOX2、IL6、JUN、MMP9,9个下调Hub基因,包括COL1A1、POSTN、LUM、PDGFRA、VCAM1、IGF1、FGF10、ENG、BDNF。除FOS、JUN、SOX2外,上调 Hub基因IL1B、IL6、MMP9在外伤、炎症方面的研究较为深入,既往文献中提到它们参与角膜上皮损伤愈合过程,而其在角膜上皮发育中的作用尚未见报道[17-19]。此外,以往研究发现下调Hub基因多参与上皮细胞-间充质转化过程,在癌症转移和多种纤维化疾病中发挥了重要作用[20-21]。
GO分析结果显示,眼表外胚层细胞上调基因主要与神经发育、神经信号转导相关,下调的DEGs主要与细胞增殖过程相关。在KEGG分析中,我们发现眼表外胚层上调DEGs富集在轴突导向(axon guidance)、神经活性配体-受体相互作用(neuroactive ligand-receptor interaction)、cAMP信号(cAMP signaling pathway)通路等。有学者研究发现小鼠Gpr48基因缺失通过cAMP信号通路导致角膜上皮细胞增殖和分化不良[22],表明cAMP信号通路在角膜上皮发育中发挥调控作用。眼表外胚层下调差异基因主要富集在癌症中的蛋白聚糖(proteoglycans in cancer)、ECM-受体相互作用(ECM-receptor interaction)、蛋白质消化和吸收(protein digestion and absorption)等信号通路。既往研究表明,下调通路参与调节上皮细胞-间充质转化过程,与下调Hub基因功能基本相符,但它们在眼表外胚层发育中的调控机制尚未得到研究[21, 23]。
miRNA是基因表达的关键调节因子,具有通过抑制信使RNA(mRNA)翻译或促进mRNA降解的作用,在转录后水平调节基因表达。本研究中我们利用Hub基因构建了TF-miRNA共同调控网络,在上调网络中筛选出1个高相关miRNA,即has-miR-21。Kalaimani等[24]已在角膜缘干细胞中发现has-miR-21-5p高表达,该miRNA可能在角膜上皮细胞再生中发挥作用,但其在角膜上皮发育及再生中的具体通路尚未阐明。在下调网络中,我们筛选出4个高相关miRNA,即has-miR-586、has-miR-301、has-miR-181a、has-miR-30e。目前,下调网络中miRNA的研究多见于肿瘤细胞中,它们通过促进细胞增殖和转移来发挥致癌基因的作用,但在眼表外胚层及表面外胚层发育中的作用仍尚不清楚[25-27]。
综上所述,本研究通过转录组测序分析了人胚胎干细胞来源的眼表外胚层和表面外胚层转录组表达差异,筛选了眼表外胚层和表面外胚层差异基因富集的GO terms及信号通路,并筛选了决定这种表型差异的Hub基因以及相关的信号调控网络。这可为眼表外胚层及角结膜上皮发育调控机制研究及角膜上皮相关遗传性疾病的防治提供理论参考。我们将在以后的研究中进一步探索二者的差异基因及相关信号通路在眼表外胚层和表面外胚层发育调控中的作用,力争阐明眼表外胚层发育调控信号网络。

