酶介导的邻近细胞标记方法探究细胞间相互作用
沙昱彤, 李景超, 易 文
(浙江大学生命科学学院生物化学研究所肿瘤与干细胞生物学实验室, 杭州 310058)
自19世纪中期细胞学说被提出以来,细胞-细胞相互作用界面一直备受研究者们的关注。大量研究表明,可以通过细胞间的接触来介导参与包括免疫应答、上皮层形成、神经细胞信号传导和成肌细胞融合等多种生命活动[1]。长久以来,细胞-细胞相互作用(cell-cell interactions, CCIs)一直是多种疾病治疗的靶标。细胞间的直接相互作用在多细胞生物的生长发育中发挥至关重要的作用。这些相互作用可以通过细胞表面的蛋白质、聚糖、脂质等介导的质膜间的物理接触而发生。这些关键的相互作用驱动细胞信号的传递,对机体健康产生重要影响。在许多情况下,异常的细胞间相互作用是导致严重疾病和机体紊乱的主要原因[2]。
这些CCIs是复杂的,涉及许多不同的细胞表面和细胞内分子的参与。理清这些复杂的相互作用以及相关的信号通路,将会极大地提高对细胞接触介导的生物过程的认知,并能促进新的细胞疗法的开发[3]。因此,随着基础研究和转化医学的不断发展,如何准确识别细胞间相互作用并对其进行表征和定量,引起了各界的广泛关注。近年来,邻近标记是一种十分有前景的研究细胞间相互作用的方法。其中,酶介导的邻近细胞标记方法在检测和表征CCIs上具有重要的应用价值。该综述将标记过程中涉及有酶参与的方法定义为酶介导的邻近细胞标记方法,旨在归纳与总结近年来开发的酶介导的邻近细胞标记方法的原理、优缺点及现有应用。
1 邻近标记方法
近年来,研究CCIs的技术手段不断推陈出新,而邻近标记方法已经成为一种十分有前景的研究细胞间相互作用的化学生物学方法。这种方法不仅能够识别复杂系统中未知的细胞间相互作用,还能够发现与此类相互作用相关的特定分子[4-6]。
目前,主要有两种类型的标记策略。一种是依赖基因工程操作,在“诱饵”细胞表面表达外源酶,通过其与相邻细胞上的受体底物的直接结合,以便发生细胞间邻近标记(接触依赖性邻近标记方法)。另一种是使用酶或小分子催化剂(例如光催化剂),通过基因工程重组表达或借助化学(酶)方法将它们偶联在“诱饵”细胞表面,在适当的刺激或激活后,靶向递送感兴趣的标记分子,实现邻近标记(接触非依赖性邻近标记方法)。常用将含生物素的探针标记到邻近底物上,然后通过链霉亲和素与生物素的亲和力对被标记的底物进行富集,之后借助质谱、流式细胞术或测序等方法进行下游检测分析[3, 7-9]。
邻近标记方法在理解生物分子相互作用方面已经取得了重大进展,这种方法首先在蛋白质组学领域被引入,用于研究蛋白质-蛋白质、蛋白质-RNA和蛋白质-DNA相互作用[7, 9]。例如,Rhee等[10]使用APEX(ascorbate peroxidase)鉴定得到人类线粒体基质内的495种蛋白质,其中包括31种以前未与线粒体关联的蛋白质。Padrn等[11]开发的APEX-seq技术,其可以解析细胞内RNA的定位,并确定它们在关键RNA结合蛋白质附近的富集或缺失。Villaseor等[12]开发的ChromID,即使用生物素连接酶来识别与特定染色质标记相关的蛋白质。细胞间通讯通常由细胞表面蛋白质之间的相互作用来调控,尽管存在许多检测蛋白质-蛋白质相互作用(protein-protein interactions, PPIs)的方法,但是精确探测膜蛋白之间的相互作用仍然是一个难题[6]。
2 酶介导的邻近细胞标记方法
细胞间的相互作用是动态而复杂的,近年来已经出现了一些酶介导的邻近细胞标记的新兴方法来识别和检测这些相互作用。这种方法的一个明显优势是,由于酶和受体底物之间直接的物理接触或酶催化产生高反应性标记分子而实现小的标记半径范围,所以能够进一步特异性地精准定位研究目标细胞间的相互作用。下面本文对近年来开发的不同酶介导的邻近细胞标记方法进行概述(Table 1)。
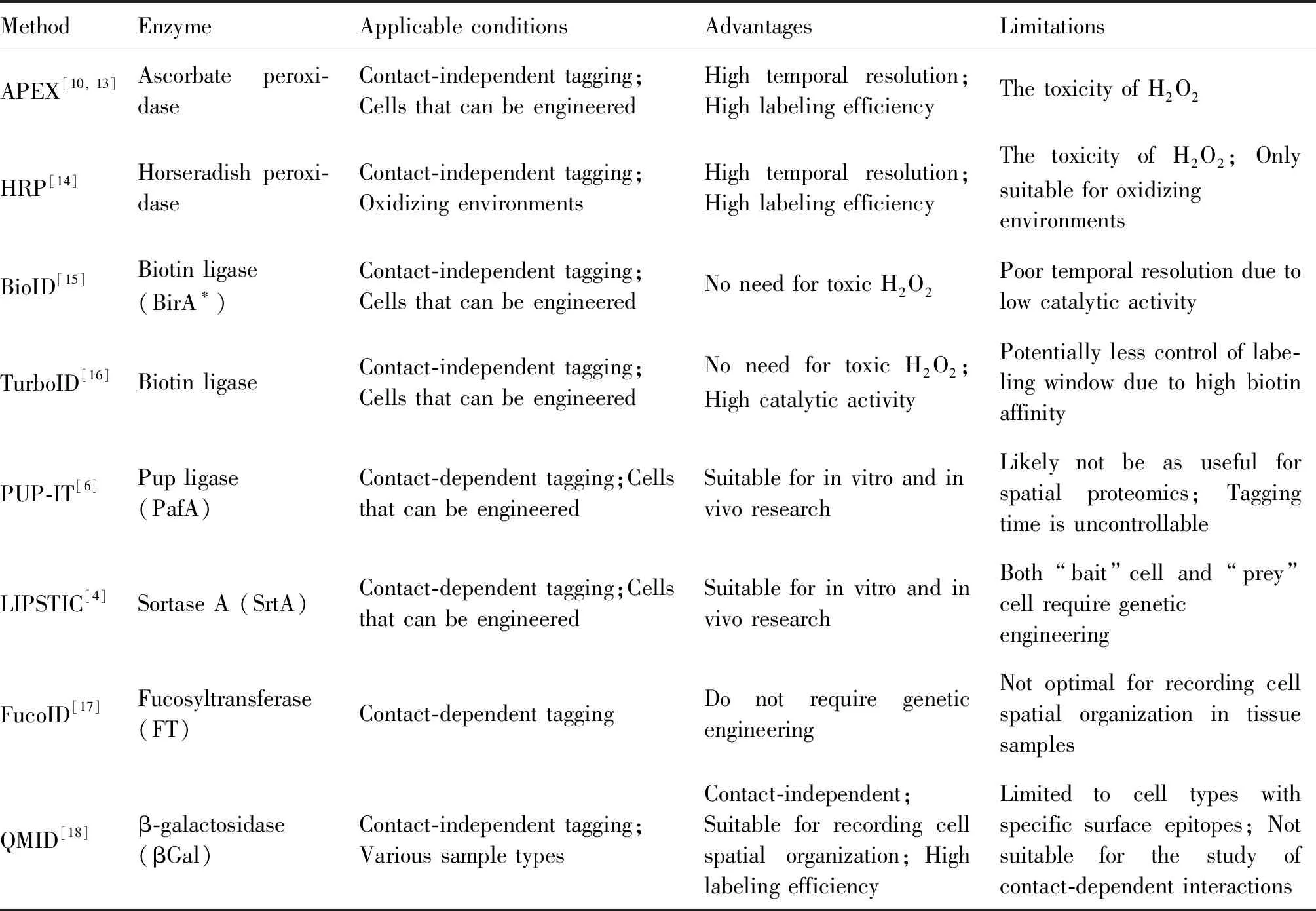
Table 1 Overview of enzyme-mediated proximity cell labeling methods
2.1 基于过氧化物酶的邻近细胞标记方法
一种源自植物的工程化抗坏血酸过氧化物酶(ascorbate peroxidase, APEX),现已成为进行邻近标记的重要工具。APEX在H2O2存在的情况下,能够将生物素酪胺转化为活性酪胺基自由基,然后攻击邻近细胞表面富含电子的氨基酸残基例如酪氨酸,并与其偶联,实现邻近细胞生物素化标记(Fig.1A)。活性酪胺基自由基的半衰期短(t1/2<1 ms)[10],标记半径小(<20 nm)[19]的特性,使得只有靠近APEX部分的蛋白质才会被标记。该反应能通过去除H2O2和添加淬灭缓冲液来停止,随后可以使用偶联有链霉亲和素的磁珠进行富集,并借助质谱法进行后续的检测鉴定[13, 20]。APEX还能被用于鉴定与特定RNA[21]或DNA[22]序列相互作用的蛋白质。
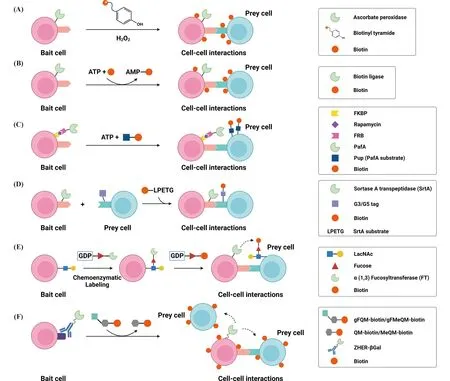
Fig.1 Schematic illustration of different enzyme-mediated proximity cell labeling methods (A) Schematic diagram of proximity cell labeling method based on ascorbate peroxidase (APEX): In the presence of H2O2, APEX can convert biotin-phenol to biotin-phenoxyl radicals, which biotinylate electron-rich amino acids within a radius of several nanometers; (B) Schematic diagram of the proximity cell labeling method based on biotin ligase (BioID): In the presence of ATP, BirA* catalyzes the conversion of biotin to reactive biotin-AMP, which reacts with neighboring nucleophilic lysine side chains; (C) Schematic diagram of pupylation-based interaction tagging (PUP-IT):By fusing the Pup ligase (PafA) to a “bait” protein, PafA catalyzes the phosphorylation of the C-terminal Glu on biotinylated Pup, in the presence of ATP, which then conjugates to the lysine residue on target proteins; (D) Schematic diagram of labeling immune partnerships by sortagging intercellular contacts (LIPSTIC): An engineered sortase A (SrtA) from Staphylococcus aureus was developed and fused to a cell surface ligand. After ligand-receptor interaction, SrtA can catalyze the transfer of biotin-containing sorting peptides (LPETG peptides) to an oligoglycine (G3/G5) acceptor peptide expressed on a complementary receptor of a neighboring cell; (E) Schematic diagram of interaction-dependent fucosyl-biotinylation (FucoID): Coupling fucosyltransferase (FT) with its natural substrate GDP-fucose, and self-installing FT to N-acetyllactosamine (LacNAc) on the surface of “bait” cells and then FT will catalyze the transfer of its substrate GDP-fucose-biotin to LacNAc glycans on the surface of the “prey” cells; (F) Schematic diagram of proximity cell labeling method based on β-galactosidase (QMID): A chimeric enzyme ZHER-βGal was constructed using the nanobody ZHER that recognizes HER2 and targeted to HER2-positive cells. βGal can convert gFQM-biotin or gFMeQM-biotin to QM-biotin and MeQM-biotin respectively, which then effectively react with nucleophiles, including amines and thiols on proteins of proximal cells. (Created with BioRender.com)
辣根过氧化物酶(horseradish peroxidase, HRP)的标记原理与APEX类似,但是它在哺乳动物胞质环境中表达时是无活性的,因为HRP的三维结构由4个二硫键和2个Ca2+结合位点来维持,该结构在还原条件下易被破坏[20, 23],这也限制了它用于研究细胞内的相互作用。但是HRP在氧化环境中具有活性,例如内质网腔或高尔基体腔和细胞外区域,因此,它已被用于活细胞表面的蛋白质组学研究[20]。而APEX相对于HRP的主要优势在于它缺乏二硫键和钙结合位点,因此,可以在细胞的还原胞质环境中表达而不会丧失活性[20]。
2.2 基于生物素连接酶的邻近细胞标记方法
基于APEX和HRP的邻近细胞标记方法虽然时间分辨率高,标记效率高,但是由于H2O2的毒性,在体内应用上受到了限制。因此,研究者开发了一种基于生物素连接酶的邻近细胞标记方法。
通过野生型生物素连接酶(biotin ligase, BirA)进行邻近标记的问题在于它对其底物具有严格的选择性[24],因此,已经开发出另一种被称为邻近标记生物素识别(proximity labeling biotin identification, BioID)的方法。该方法使用来自大肠杆菌的BirA突变体(BirA mutant R118G, BirA*)介导生物素与靶蛋白质的结合,从而实现对邻近蛋白质的生物素化标记,之后使用质谱分析进行鉴定[15, 24-28]。在ATP存在的情况下,BirA*催化生物素转化为反应活性生物素-AMP,它会与邻近细胞表面的赖氨酸残基侧链发生反应(Fig.1B)。虽然生物素-AMP的半衰期(t1/2≈1 min)比过氧化物酶产生的酪胺基自由基中间体长得多,但BioID不需要添加外源性的会造成细胞损伤的H2O2,可用于体内研究。
BioID的主要缺点是其缓慢的反应动力学,这使得此方法标记效果缓慢(18~24 h),时间分辨率差。为了解决这个问题,研究者们开发了一种类似的更快的系统,称为TurboID,其标记只需10 min[16]。TurboID比上述生物素连接酶相关邻近标记方法具有更高的反应活性,可以实现更高的时间分辨率和更广泛的体内应用[29]。这种生物素连接酶方法最近才被应用于研究细胞-细胞间相互作用,Shafraz等[30]使用与E-钙粘蛋白的细胞外结构域融合的TurboID来鉴定上皮细胞连接中的跨膜结合伙伴。
此外,还开发出了一些变体方法,例如split-BioID[31]、Contact-ID[32]和split-TurboID[29, 33],它们也已成为研究细胞间相互作用的有前景的方法。尽管APEX和BioID已被广泛用于许多生物系统的邻近标记,但目前主要用于分子层面的相互作用鉴定(即主要通过蛋白质组学),对于细胞层面的相互作用鉴定有待进一步研究。
2.3 基于Pup连接酶的邻近细胞标记方法
一种基于小型原核类泛素蛋白(prokaryotic ubiquitin-like protein, Pup)的相互作用标记方法(pupylation-based interaction tagging, PUP-IT)与BioID、HRP和APEX不同,它是通过将Pup连接酶(pup ligase, PafA)与“诱饵”蛋白质进行融合,适用于体内外研究。在细菌中,Pup蛋白C-末端的Gln首先脱氨基为Glu,在ATP存在的情况下,PafA催化Pup上C-末端Glu的磷酸化,然后将C-末端Glu与目标蛋白质上的赖氨酸残基侧链结合[6, 34-37]。已有报道,借助化学诱导二聚化的FK506结合蛋白(FK506 binding protein, FKBP)-FKBP12-雷帕霉素复合物结合结构域(FKBP12-rapamycin binding domain, FRB)系统,嵌合FRB的PafA可以被靶向附着在细胞表面的“诱饵”蛋白质上,然后对邻近相互作用细胞进行生物素化标记(Fig.1C),借此成功鉴定得到已知的和多种潜在的CD28相互作用蛋白[3, 6]。
因为赖氨酸残基在人类蛋白质中几乎是普遍存在的,因此,这种方法可以催化生物素化的Pup标记相互作用细胞的配体-受体对和其他邻近的蛋白质。PUP-IT方法标记的主要目标是细胞表面蛋白质的赖氨酸残基侧链,这与基于过氧化物酶催化的邻近标记方法中以酪氨酸残基为目标不同。一般来说,蛋白质中赖氨酸残基的含量高于酪氨酸残基[38]。因此,当可用酪氨酸残基的数量有限时,可能无法使用APEX和HRP来鉴定潜在的靶蛋白质。但是,PUP-IT的标记时间较难控制,可能不适用于空间蛋白质组学研究。
2.4 基于分选酶的邻近细胞标记方法
不同于使用高反应活性标记分子的方法,研究者也开发了很多依赖酶、供体底物和受体底物的邻近细胞标记方法。在这些方法中,酶和底物之间的距离通常在约10 nm以内,适用于直接接触的细胞-细胞相互作用研究。
在“通过依赖于细胞间接触的分选酶催化标记”(labeling immune partnerships by sortagging intercellular contacts, LIPSTIC)的方法中,研究开发了一种工程化改造的金黄色葡萄球菌的分选酶A(sortase A, SrtA),并将其通过基因工程技术融合到细胞表面配体上。在配体-受体相互作用后,SrtA可以催化含生物素的分选肽(LPETG肽)转移到邻近细胞表面表达N-末端甘氨酸(G3或G5)受体肽的互补受体上(Fig.1D)[4, 39]。这种使用小肽作为底物的方法易于捕获瞬时的配体-受体相互作用,并在体外和体内都有成功的应用。有研究报道,LIPSTIC可用于在体内外T细胞活化过程中检测树突状细胞和CD4+T细胞之间的相互作用。证明LIPSTIC是一种高效、特异性,可以用于体外和体内以酶促方式标记细胞间相互作用的方法[4],在免疫学领域有着巨大的应用前景。但是这种方法依赖基因工程技术改造细胞,操作复杂,并不是通用的方法。
由于野生型SrtA只能标记通过基因工程操作而具备在细胞表面表达N-末端甘氨酸受体肽的细胞,因此,一种类似的基于分选酶催化的方法被开发出来。该方法通过设计一种定向进化后的SrtA突变体(SrtA variant, mgSrtA)来催化标记相互作用细胞表面蛋白质上的N-末端单甘氨酸残基,从而在LIPSTIC的基础上进行了扩展。由于细胞表面蛋白质上暴露的N-末端单甘氨酸残基比三甘氨酸丰富得多,因此不需要预先处理“猎物”细胞,这提供了一种标记细胞间相互作用的通用方法[5]。
2.5 基于岩藻糖基转移酶的邻近细胞标记方法
尽管已经开发了很多基于酶介导的邻近标记方法,但大多都需要通过基因工程进行额外的细胞操作来引入标记酶,这将会阻碍这些方法在研究细胞间相互作用方面的广泛应用。因此,研究者们也对此进行了优化,开发出了一种依赖于相互作用的岩藻糖基生物素化标记方法(interaction-dependent fucosyl-biotinylation, FucoID),适用于研究直接接触的细胞间相互作用。该方法克服了需要通过遗传学操作的限制,它是通过使用无基因工程策略——化学酶方法,将来自于幽门螺旋杆菌的α(1,3)-岩藻糖基转移酶(fucosyltransferase, FT)与其天然底物GDP-岩藻糖[40, 41]进行偶联,然后通过自催化反应实现了FT在“诱饵”细胞表面N-乙酰-D-乳糖胺(N-acetyl-D-lactosamine, LacNAc)和/或α(2,3)-唾液酸LacNAc上的定位附着,该方法克服了需要通过遗传学操作的限制。然后FT会催化其底物GDP-岩藻糖-生物素转移到“猎物”细胞表面的LacNAc上,实现邻近细胞生物素化标记,以此来鉴定“诱饵”细胞和“猎物”细胞之间的相互作用(Fig.1E)。
LacNAc是一种常见的位于细胞表面的二糖,由于LacNAc和唾液酸化LacNAc在大多数细胞类型中均存在大量表达[17, 42],因此,该标记方法具有普遍适用性,可以实现在2个相互作用细胞间的高灵敏度标记。
已有研究使用抗原引发的偶联了FT的树突细胞,证明了FucoID可以从小鼠肿瘤模型中成功鉴定和分离出内源性肿瘤抗原特异性T细胞[17]。此外,通过这种方法分离得到的肿瘤特异性CD8+T细胞,显示出更高的肿瘤杀伤力及与T细胞活化相关的基因表达谱的增加,突出了这种方法具有从肿瘤微环境中直接获得肿瘤反应性T细胞用于下游治疗应用的潜力。但是,在从临床样本中无法分离富集得到所需的“诱饵”细胞时,这种方法无法探测特异性的CCIs。
近期另有研究表明,利用FT的定点化学偶联开发了位点特异性细胞-酶偶联体探针(site-specific cell-based fucosy-ltransferase conjugate, Cell-sFT)和抗体-酶偶联体探针(site-specific antibody-based fucosyltransferase conjugate, Ab-sFT),以使FucoID能够应用于更广泛的细胞环境[42]。研究发现,Cell-sFT不仅可以用于探测DC-T细胞相互作用,还可以应用于探测癌细胞-T细胞以及DC-B细胞相互作用。这些CCIs受嵌合抗原受体-抗原相互作用、程序性细胞死亡蛋白1(programmed cell death protein 1, PD-1)-程序性死亡配体1(programmed death-ligand 1, PD-L1)相互作用或未知的相互作用分子对控制。此外,使用新开发的Ab-sFT,以内源性癌细胞作为“诱饵”细胞,在人类患者临床样本中实现了FucoID。对FucoID鉴定出的未知CCIs系统中的“猎物”细胞,通过RNA-seq或流式细胞术分析进行表征,以剖析不同的分子特征,为研究复杂系统中更广泛的CCIs开辟了道路。
2.6 基于β-半乳糖苷酶的邻近细胞标记方法
为了拓展研究不需要直接接触的细胞间相互作用,近期开发了一种被称为亚甲基醌辅助细胞空间组织识别鉴定(quinone methide-assisted identification of cell spatial organization, QMID)的邻近细胞标记方法[18],该方法利用一类具有微米级标记半径的反应性亲电子物质——亚甲基醌(quinone methide, QM),它可以由β-半乳糖苷酶(β-galactosidase, βGal)[43-45]酶促脱笼反应原位生成。生成的QM在微米半径范围内扩散,并可以共价标记邻近细胞上的膜蛋白质。
利用识别人表皮生长因子受体2(human epidermal growth factor receptor 2, HER2)的纳米抗体ZHER,构建一种嵌合酶ZHER-βGal并靶向定位到HER2阳性细胞上,βGal能够将gFQM-生物素和gFMeQM-生物素转化为QM-生物素和MeQM-生物素,从而有效地标记“诱饵”细胞和邻近“猎物”细胞(Fig.1F)。此外,在共培养体系中,应用QMID对与肿瘤细胞邻近的巨噬细胞的基因表达情况分析发现,其多种基因的表达受到2种细胞的邻近性的显著调控。也能将QMID与scRNA-seq结合,系统地对小鼠脾中CD4+和CD8+T细胞的邻近细胞进行分析,结果显示,邻近CD8+T细胞的成熟的树突细胞中脂质分解代谢过程和WNT信号通路均上调[18]。QMID方法标记效率高、适用于各种类型样品,提供了一种分析各种生物系统中细胞空间组织的通用方法。
但是在QMID的方法中,由于βGal是通过抗体引入。因此,QMID 仅适用于表面具有特定表位的细胞类型。
3 问题与展望
综上,酶介导的邻近细胞标记方法借助基因工程操作在细胞表面上表达外源酶,或通过化学酶法在细胞表面上偶联酶后与其受体底物结合,或催化供体底物产生高反应活性标记分子,从而实现细胞间邻近标记。这些方法由于酶和底物之间直接的物理接触或酶催化产生高反应活性标记分子,因此,可以实现小的标记半径范围。但是,由于大多需要借助基因工程技术引入标记酶和/或受体底物,这可能会使得一些方法只适用于某些适合工程化改造的细胞系。因此,也出现了可以绕过此限制的新的标记方法,例如FucoID、mgSrtA。除此之外,还有克服依赖“诱饵”细胞和“猎物”细胞紧密接触的方法,例如QMID。
在产生高反应活性标记分子的邻近细胞标记方法中,例如像APEX,由于H2O2的毒性,其在体内的应用受到限制;HRP方法仅限于分泌途径和细胞外机制研究;而BioID标记效率低导致时间分辨率差;TurboID虽然催化活性高,但是它也由于生物素亲和力高,标记窗口的控制可能较少。而在依赖酶、供体底物和受体底物之间直接的物理接触的邻近细胞标记方法中,例如LIPSTIC、PUP-IT和FucoID的方法,由于其底物特异性,依赖基因工程技术改造细胞或依赖于相互作用细胞直接接触,只适用于接触依赖性的细胞间相互作用的研究。而在QMID的方法中,虽然研究不需要直接接触的细胞间相互作用,但是βGal是通过抗体引入的,以这种方式,QMID仅限于具有特定表面表位的细胞类型。
不同酶介导的邻近细胞标记方法都有各自的优缺点,因此,酶介导的邻近细胞标记方法仍需进一步改进,未来需要开发更为通用、简便、高效特异的方法,更加精准探测细胞-细胞间的相互作用。
众所周知,糖基化是最常见的蛋白质翻译后修饰之一,可发生在至少50%哺乳动物蛋白质上[46, 47]。糖基化产生了丰富、多样和高度调控的细胞聚糖,这些聚糖经常附着在蛋白质和脂质上。细胞表面的糖蛋白在细胞间相互作用中发挥着至关重要的作用,正如开发出的FucoID那样,对活细胞表面的糖蛋白进行特定修饰可用于探究细胞间相互作用,并有助于未来的疾病诊断和治疗[17, 42]。细胞表面糖蛋白的表达水平与肿瘤的发生、发展和转移密切相关,许多类型的糖蛋白被证实能作为诊治癌症的靶标。例如,唾液酸结合免疫球蛋白样凝集素家族(sialic acid binding Ig like lectin, Siglecs)可以通过识别肿瘤细胞表面的唾液酸(sialic acid, Sia)来介导细胞与细胞之间的相互作用,从而在免疫反应中发挥重要的调节作用[48, 49],未来有望开发一种利用Siglec和Sia之间特异性相互作用的邻近细胞标记方法。此外,研究者们旨在寻找一种可以快速、灵敏、准确检测肿瘤的新型生物标志物,试想未来可以着重探索更多与细胞表面特定糖蛋白识别的酶,拓展开发出新的酶介导的邻近细胞标记方法,靶向识别相互作用细胞,提高检测细胞间相互作用的时空分辨率,更为深入全面地理解癌症的发生发展机制,期望可以早发现、早诊断、早治疗癌症。
在免疫细胞的细胞间通讯中,B淋巴细胞、树突细胞(dendritic cells, DCs)和巨噬细胞都能摄取和加工处理抗原,并且可以将抗原信息暴露在细胞表面,以便呈递给其他免疫细胞,因此,这些细胞统称为抗原呈递细胞(antigen-presenting cells, APC)。T淋巴细胞是细胞免疫中的主要效应细胞,在免疫反应中产生细胞因子以介导炎症反应并调节其他类型的免疫细胞[50, 51]。免疫反应的激活依赖于抗原呈递细胞和T细胞之间的相互作用,对抗原特异性T细胞的检测和表征,对于理解免疫反应以及开发新的免疫疗法至关重要。酶介导的邻近细胞标记方法可以发挥其高灵敏度和特异性的优势来标记检测免疫细胞间的相互作用。甚至可以进一步将酶介导的邻近细胞标记方法应用于临床研究,高效鉴定特异性识别肿瘤特异性抗原(tumor specific antigen, TSA)的肿瘤浸润淋巴细胞,为开发新的免疫治疗策略提供重要的理论和实验基础。
尽管目前已经报道了很多研究CCIs的方法,但是探测细胞之间的相互作用仍然是一项艰巨的任务。由于突触形成的空间限制和所涉及的细胞类型较多,这对于在这种环境中特异性地识别细胞间相互作用带来了巨大的挑战。现今,研究者仍在不断地扩展开发新型的邻近标记方法,以进一步提高在探测细胞接触区域的时空分辨率。

