霜霉病菌胁迫下藜麦内参基因的筛选及其稳定性验证
解宇洁, 薛 婧, 姜晓东, 殷 辉, 赵晓军, 冯 铸, 李新凤
(1.山西农业大学植物保护学院,山西 太谷 030801;2.山西农业大学农学院,山西 太谷 030801; 3.山西省农业种子总站,山西 太原 030006)
藜麦(ChenopodiumquinoaWilld),属藜科藜属一年生草本植物,原产于南美洲安第斯高地,是一种全营养单体植物,因其特殊的营养价值及耐寒、耐旱、耐盐碱等特性,被越来越多的人青睐[1]。由PeronosporavariabilisGaüm引起的霜霉病是各藜麦产区常见的病害,严重影响藜麦的产量和品质,给藜麦产业造成了较大经济损失[2]。目前,对该病害的防治仍然以传统的化学药剂防治为主,既存在安全隐患,又提高了生产成本;抗病品种的合理利用是防治植物病害最经济、有效的措施[3]。了解藜麦抗霜霉病菌基因的表达模式和作用机制,进而选育抗病品种,对藜麦霜霉病的防治乃至藜麦产业的健康发展具有重要意义。
常见的基因表达量分析方法有Northern blots、实时荧光定量PCR(quantitative real-time PCR, qRT-PCR)、微阵列(microarray)法、基因表达连续分析(serial analysis of gene expression, SAGE)法、微球法等[4]。其中,qRT-PCR由于具有高重复性、高灵敏性和高特异性等特性,已被广泛用于微生物检测、单核苷酸多态性分析和基因表达研究等方面[5-6]。相对定量研究要求选择在不同试验条件下都能相对稳定表达的内参基因,对目标基因的表达水平进行校正,内参基因的稳定性对qRT-PCR结果的准确性具有决定性[7]。控制生物细胞基本结构和代谢功能的持家基因常被用作内参基因,目前用于植物研究的内参基因有编码肌动蛋白(actin, act)、α-微管蛋白(α-tubulin,α-tub)、18S核糖体RNA(18S ribosomal RNA, 18S rRNA)、翻译延伸因子(translation elongation factor, ef-1)、β-微管蛋白(β-tubulin,β-tub)和泛素连接酶(ubiquitin-ligase enzyme, ubc)等的基因[8-10]。但越来越多的研究发现,在不同组织、器官或试验条件下都能稳定表达的内参基因几乎不存在[11-12]。因此,为确保qRT-PCR结果的准确性,需要根据不同处理选择相应稳定表达的内参基因[13-14]。
基于qRT-PCR中基因表达的Ct值,一系列专门用于分析内参基因稳定性的程序被开发,目前应用最多的有geNorm[15]、NormFinder[16]、BestKeeper[17]和RefFinder[18]等软件。其中,geNorm和NormFinder软件用于计算各候选内参基因的表达稳定(M)值,M值越小,内参基因稳定性越强。geNorm程序以M=1.5为阈值,若M值大于1.5则不适合作为候选内参基因。同时,geNorm可以通过计算配对变异(V)值确定试验所需的最适内参基因数目,以提高结果可信度,该程序以0.15为阈值,当Vn/Vn+1小于0.15时,则最适内参基因数目为n个[19]。BestKeeper通过计算各候选内参基因Ct值之间的标准偏差(standard deviation, SD)判定候选内参基因的稳定性,SD值越小,基因表达越稳定,若SD>1,则认为该基因不稳定[19]。RefFinder将当前主流的计算程序(geNorm、NormFinder、BestKeeper和ΔCt法[20])进行整合,根据每个程序的排名,为单个基因赋予合适的权重,并计算其权重的几何平均值,以获得最终的总体排名[18]。这4款软件的工作原理各不相同,常组合使用以综合评估内参基因的稳定性[21]。
目前,国内外关于藜麦内参基因筛选的研究十分有限,并且主要集中在非生物胁迫下藜麦内参基因的筛选。例如:贾冰晨等[22]研究发现,藜麦在盐胁迫下的最佳内参基因为ACT-1和TUB-6;Fiallos-Jurado et al[23]研究发现,藜麦在茉莉酸甲酯胁迫下的最佳内参基因为MON1。但未见生物胁迫下藜麦内参基因的相关报道。本试验选取编码藜麦18S rRNA(CqRPS18)、ubc(CqUBC)、α-tub(CqTUA-1)、真核翻译起始因子(CqELF3)、act(CqACT1)、β-tub(CqTUB-6)、ef-1(CqEF-1a)、液泡融合蛋白(CqMON1)的8个基因[23]作为霜霉病菌胁迫下藜麦的候选内参基因,利用qRT-PCR技术及geNorm、NormFinder、BestKeeper和RefFinder软件分析和评价其在藜麦霜霉病菌胁迫下的表达稳定性,旨在为深入开展藜麦抗霜霉病菌相关基因表达的研究提供理论依据。
1 材料与方法
1.1 材料
供试藜麦材料74421和2403分别为感霜霉病菌和抗霜霉病菌品系。供试藜麦霜霉病菌为本实验室纯化、繁殖,保存于室内盆栽藜麦上,并结合形态学和分子生物学方法鉴定为P.variabilis的菌株。纯化、繁殖方法:2021年在山西省太谷区藜麦种植基地采集自然发病的典型藜麦霜霉病罹病叶片,用无菌水洗净叶片表面霉层,斜插于水琼脂平板上,待长出新霉层后,将其接种至苗龄为6周的盆栽藜麦(74421)上,表现出与田间相同的发病症状。
1.2 材料处理
将供试藜麦种子用35%(质量分数)甲霜灵可湿性粉剂按种子质量的0.2%~0.3%拌匀后,播于装有无菌营养土的花盆内,6周后接种霜霉病菌。自预先准备好的病株叶片上刮取霜霉病菌孢子囊,置于添加0.05%(体积分数)Tween-20的无菌水中,制成每毫升含1×106个孢子的悬浮液,在3 h内将其喷施到抗病和感病的藜麦材料上,用白色透明塑料袋覆盖,放入人工气候箱内,在23 ℃、相对湿度100%的条件下,先黑暗培养24 h,再改为12 h光照/12 h黑暗交替培养。分别于接种0、2、6、12、24、48、72 h后收集叶片,液氮速冻后用于RNA提取,每个处理设3株生物学重复。
1.3 藜麦叶片总RNA提取及cDNA合成
藜麦叶片总RNA提取参照TaKaRa MiniBEST Plant RNA Extraction Kit试剂盒(Code No.9769S)说明书,之后利用超微量蛋白核酸分析仪(BioDrop μlite+)测定RNA浓度及光密度(D)值,通过D260 nm/D230 nm值和D260 nm/D280 nm值评价RNA的质量。基因表达分析所用的cDNA(complementary DNA)模板均按照TaKaRa PrimeScriptTMRT Master Mix(Perfect Real Time)试剂盒(Code No.RR036A)说明书合成。
1.4 候选内参基因与目标基因的引物设计
参照逆境胁迫下常用的植物内参基因,并根据NCBI网站(https://www.ncbi.nlm.nih.gov/)发布的藜麦全基因组序列,选取CqRPS18、CqUBC、CqTUA-1、CqELF3、CqACT1、CqTUB-6、CqEF-1a、CqMON1共8个基因作为候选内参基因。除CqACT1、CqTUB-6和CqMON1基因序列利用已报道引物[22-23]外,其余基因序列的特异性引物均自行设计。先通过NCBI网站搜索藜麦全基因组序列,并下载藜麦各内参基因与目标基因相应的CDS序列,再在Primer-BLAST网站(https://www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi?LINK_LOC=BlastHome)在线设计特异性引物。参数设置如下:扩增长度80~200 bp,引物序列长度17~25 bp,上游引物与下游引物的解链温度(melting temperature, TM)相差5 ℃以内,限定物种为藜麦(C.quinoaWilld.taxid:63459),以保证引物的特异性。引物序列见表1,设计好的引物送至生工生物工程(上海)股份有限公司合成。
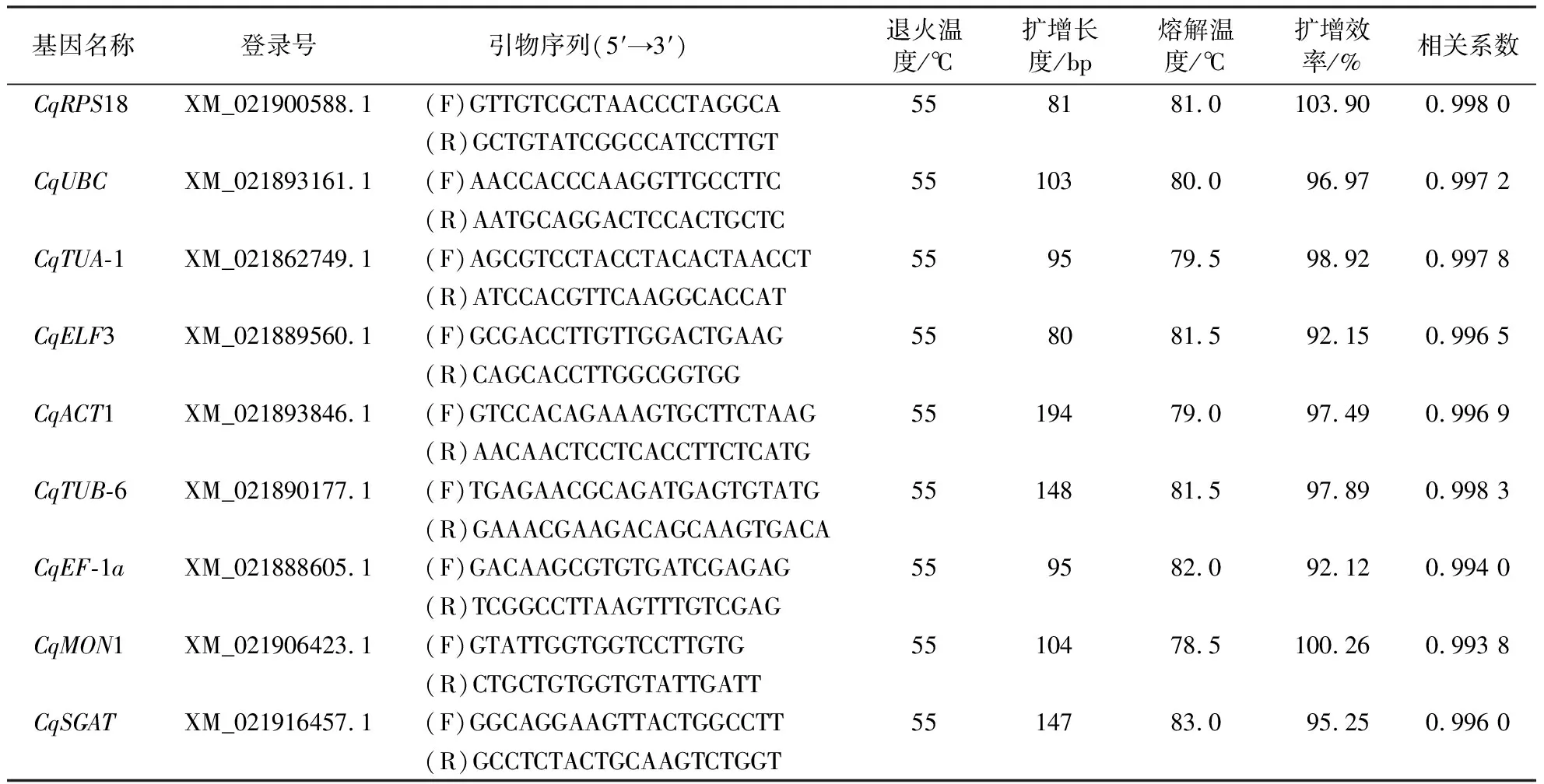
表1 候选内参基因和目标基因信息及其扩增效率和相关系数Table 1 Information, amplification efficiency and correlation coefficient of candidate reference genes and target gene
1.5 候选内参基因标准曲线的制备及引物扩增效率的测定
等量取42个样本的cDNA模板,混合后用ddH2O稀释6个浓度,每个浓度梯度稀释5倍,利用合成的引物于CFX96 qRT-PCR仪(Bio-Rad)中对相应内参基因与目标基因序列进行qRT-PCR扩增。根据所得Ct值绘制标准曲线,得到斜率K和线性相关系数r2,根据公式E=(5(-1/K)-1)×100%[24],计算扩增效率E。
采用TaKaRa TB Green®Premix ExTaqTMⅡ (Tli RNaseH Plus)荧光染料(Code No.RR820A)进行qRT-PCR反应定量分析。每个样品设置3个技术重复,以未加cDNA模板作为阴性对照。qRT-PCR反应体系总体积为20 μL:2×TB Green Premix ExTaqTMⅡ (Tli RNaseH Plus)10 μL、上游引物(10 μmol·L-1)0.8 μL、下游引物(10 μmol·L-1)0.8 μL、稀释后的cDNA模板2 μL、ddH2O 6.4 μL。反应程序:95 ℃,30 s;95 ℃,5 s,55 ℃,30 s,72 ℃,20 s,40个循环。反应结束后获得65~95 ℃熔解曲线。
1.6 候选内参基因引物特异性鉴定
分析候选内参基因的熔解曲线,并将qRT-PCR扩增产物经1.5%(质量分数)琼脂糖凝胶(100 V)电泳25 min,分析扩增长度、电泳条带数及是否有非特异性扩增。
1.7 候选内参基因稳定性分析
通过geNorm和NormFinder软件计算各候选内参基因的M值,geNorm以M=1.5为阈值,并计算Vn/Vn+1值,确定最佳候选内参基因数目,阈值为0.15[19];采用ΔCt法计算标准偏差数值(STDEV)[20];通过BestKeeper软件计算各候选内参基因Ct值之间的SD值,阈值为1[19];用RefFinder软件综合评估最适内参基因[18]。
1.8 候选内参基因稳定性验证
以CqSGAT为目标基因对藜麦霜霉病菌胁迫下8个内参基因的稳定性进行验证,采用2-ΔΔCt法[25]分析相对表达量数据,并利用Excel 2016软件作图。
2 结果与分析
2.1 藜麦叶片RNA的质量
提取的RNA样品的D260 nm/D230 nm值和D260 nm/D280 nm值均在2.0~2.1之间,说明提取的RNA纯度较高,符合后续试验的要求。
2.2 候选内参基因的扩增效率及扩增特异性
绘制各候选内参基因的标准曲线,计算引物扩增效率和相关系数,结果如表1所示。8个候选内参基因的相关系数均大于0.99,扩增效率均介于90%~110%之间,表明cDNA模板量与相应的Ct值具有良好的线性关系,符合qRT-PCR对引物扩增效率的要求。
各候选内参基因qRT-PCR扩增产物的琼脂糖凝胶电泳结果显示,扩增片段与目的片段大小吻合,无非特异性扩增及引物二聚体(图1)。进一步分析熔解曲线,发现8个候选内参基因均为单一熔解峰,各样品扩增曲线具有良好的重复性(图2)。以上结果表明,8个候选内参基因的引物特异性好,qRT-PCR反应专一性强,结果准确、可靠。

M.Marker;1.CqRPS18;2.CqUBC;3.CqTUA-1;4.CqELF3;5.CqACT1; 6.CqTUB-6;7.CqEF-1a;8.CqMON1。
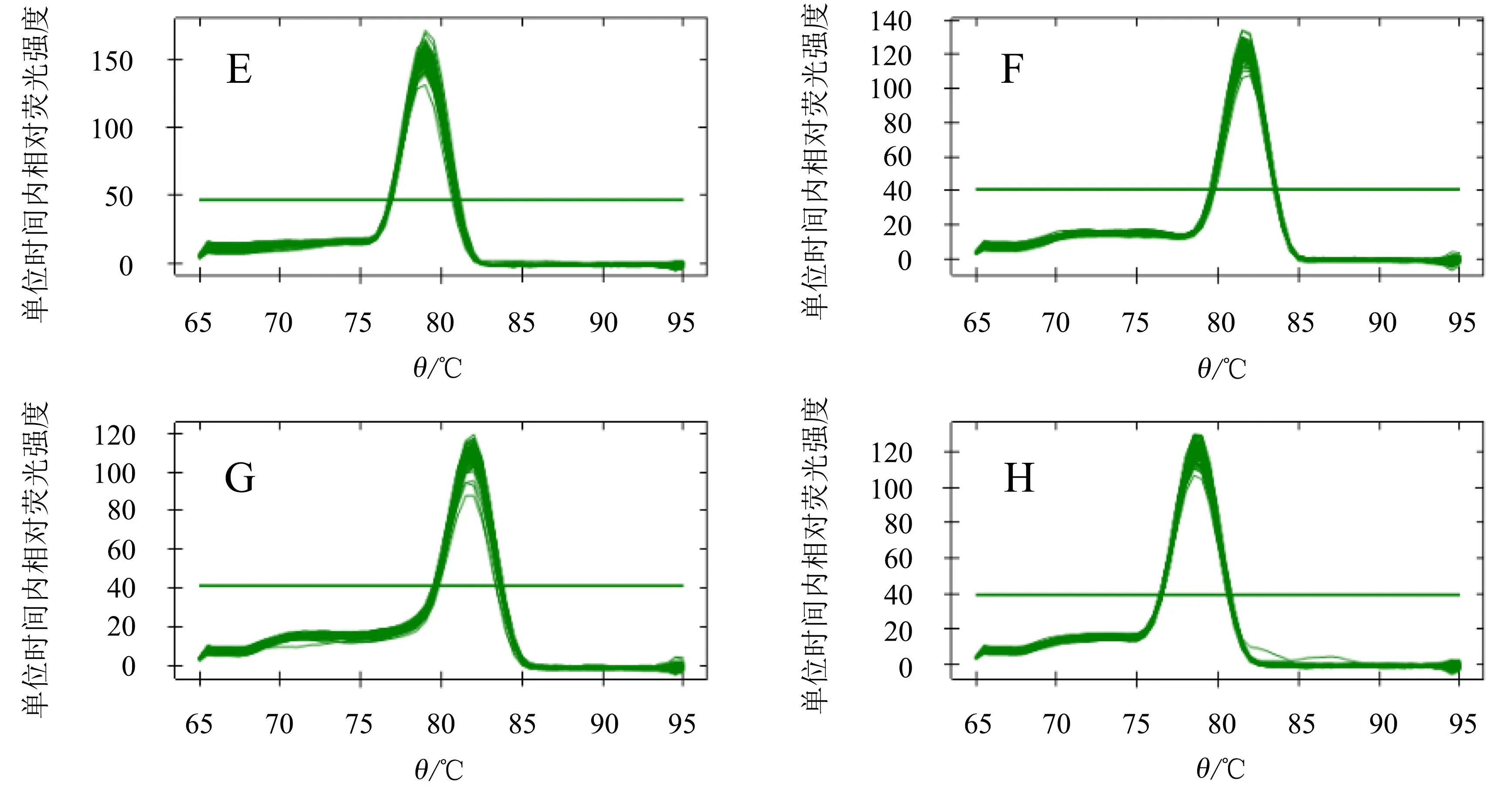
A.CqRPS18;B.CqUBC;C.CqTUA-1;D.CqELF3;E.CqACT1;F.CqTUB-6;G.CqEF-1a;H.CqMON1。
2.3 候选内参基因的表达丰度
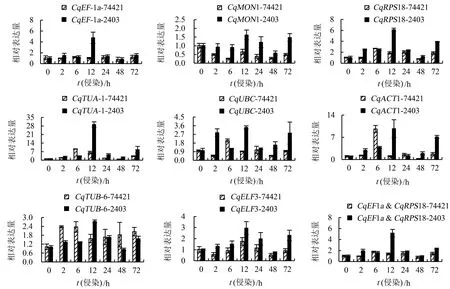
8个候选内参基因的Ct值介于19~31之间,其中,CqEF-1a基因的Ct值最小(20~24),CqELF3基因的Ct值最大(25~31),未加模板的对照组未检测到荧光信号(图3)。
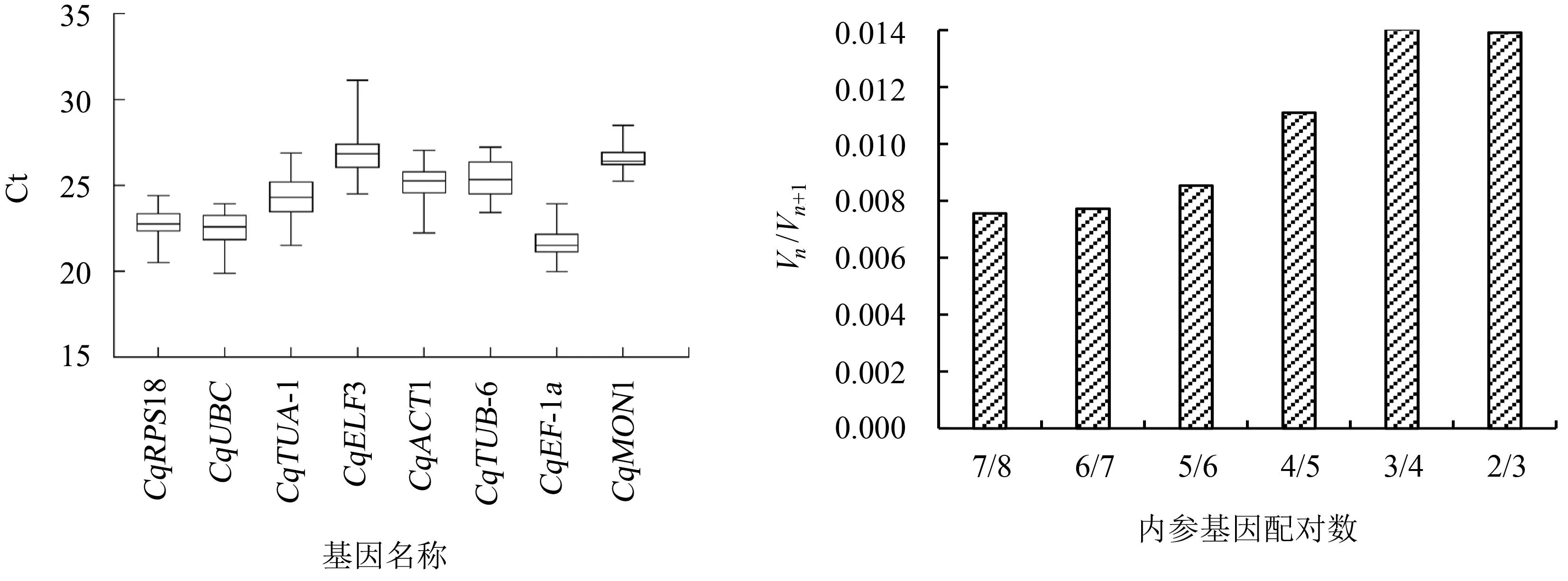
图3 候选内参基因的Ct值分布
2.4 候选内参基因的表达稳定性
geNorm软件分析结果如图4和表2所示。V2/V3=0.014<0.15(图4),表明本研究中最适候选内参基因数目为2个。8个候选内参基因的M值均小于1.5,说明所有基因表达都比较稳定。M值表现为CqEF-1a=CqMON1 表2 内参基因表达的稳定性Table 2 Expression stability of reference gene NormFinder软件分析结果(表2)表明,8个候选内参基因的M值表现为CqEF-1a BestKeeper软件分析结果(表2)表明,除CqELF3外的7个候选内参基因的SD值均小于1.00,说明这些基因的表达都比较稳定。SD值表现为CqMON1 ΔCt法得到的基因稳定性排名如表2所示,表现为CqEF-1a>CqRPS18>CqMON1>CqTUA-1>CqUBC>CqACT1>CqTUB-6>CqELF3,即最适合作为内参基因的是CqEF-1a和CqRPS18,该结果与NormFinder软件的评估结果一致。 RefFinder软件得到的稳定性综合排名如表2所示,表现为CqEF-1a>CqMON1>CqRPS18>CqTUA-1>CqUBC>CqACT1>CqTUB-6>CqELF3,即最适合作为内参基因的是CqEF-1a和CqMON1,该结果与geNorm软件的评估结果一致。综合4个软件和ΔCt法的分析结果可知,CqEF-1a、CqMON1和CqRPS18的稳定性好。 图5显示,以除CqUBC外的7个候选基因作为参照时,CqSGAT基因在藜麦抗病品系2403中的表达趋势基本一致:表达量均在霜霉病菌侵染后波动上升,12 h时表达量达到最高,后呈下降趋势,但在72 h时略有回升。此外,在不同内参基因处理下,CqSGAT的表达量存在较大差距。 74421为藜麦感病品种,2403为藜麦抗病品种。 分别以CqEF-1a或CqRPS18作为内参基因以及二者作为双内参基因时,CqSGAT基因在藜麦感病品种74421中的表达趋势一致,表达量均在霜霉病菌侵染后6 h内逐渐升高,在12 h时下降,24 h时再次升高,之后波动下降;而以其余6个候选基因作为内参基因时CqSGAT在藜麦感病品种中的表达趋势与上述情况有所不同。综上,选择CqEF-1a和CqRPS18组合作为藜麦在霜霉病菌胁迫下的内参基因。 本研究分析了CqRPS18、CqUBC、CqTUA-1、CqELF3、CqACT1、CqTUB-6、CqEF-1a、CqMON1共8个候选内参基因在藜麦霜霉病菌胁迫下的表达稳定性,并以CqSGAT基因作为目标基因,进一步验证了候选内参基因的表达稳定性,确定藜麦在霜霉病菌胁迫下稳定表达的最适内参基因为CqEF-1a和CqRPS18。 通过比较geNorm、NormFinder、BestKeeper软件和ΔCt方法的分析结果发现,geNorm、NormFinder软件和ΔCt法得到的内参基因的稳定性排序基本一致,而BestKeeper软件得到的稳定性排序与其他3种方法有明显差异。在盐胁迫下藜麦内参基因的筛选中也发现,BestKeeper软件的分析结果不同于其他算法[22]。此差异可能是由试验数据的误差和不同软件的统计学算法不同引起的。因此,使用目标基因验证候选内参基因的表达稳定性是十分必要的。 丝氨酸∶乙醛酸氨基转移酶(serine∶glyoxylate aminotransferase,SGAT)存在于植物、动物、微生物中,此酶参与高等植物光呼吸代谢过程,是过氧化物酶体中的标志酶,主要催化丝氨酸和乙醛酸之间的转氨基反应,生成羟基丙酮酸和甘氨酸[26]。该酶对植物的碳代谢和氮代谢循环都起着重要作用。Taler et al[27]从印度野生甜瓜的霜霉病抗性品种中克隆得到两个具有SGAT活性的甜瓜霜霉病抗性基因At1和At2,且过表达At1和At2基因的甜瓜对霜霉病的抗性增强。也有研究表明,SGAT的表达调控与植物生长发育、生物胁迫、非生物胁迫及抗衰老方面有密切关系[28]。因此,结合前期该基因在藜麦抗病和感病材料中的表达差异,选择其作为藜麦抗霜霉病的候选基因。 分别以不同候选基因作为参照,比较目标基因的表达量变化趋势,若候选基因稳定表达,则目标基因的表达量变化趋势一致,相反,则可能出现目标基因表达量变化趋势不一致的现象。本研究中,4种软件分析结果均认为CqEF-1a、CqMON1和CqRPS18基因表达稳定性好,但以CqMON1为内参基因时,CqSGAT基因在藜麦感病品种中的表达量变化趋势与CqEF-1a和CqRPS18作为内参基因时不一致。其原因可能是该基因的荧光阈值设置不合适,导致Ct值过高,进而使试验结果不能准确反映目标基因的实际表达趋势,故CqMON1基因是否适合作为藜麦在霜霉病菌胁迫下的内参基因,还有待调整荧光阈值后再进行验证。此外,本课题组前期研究认为藜麦抗霜霉病的性状可能是由多基因控制的数量性状[29],所以候选内参基因在霜霉病菌胁迫下的表达稳定性还有待使用其他抗病基因进行进一步验证。 贾冰晨等[22]研究发现,藜麦在盐胁迫下的最佳内参基因为ACT-1和TUB-6;Fiallos-Jurado et al[23]研究发现,藜麦在茉莉酸甲酯胁迫下的最佳内参基因为MON1。本研究结果表明,藜麦在霜霉病菌胁迫下的最适内参基因为CqEF-1a和CqRPS18。这说明同一内参基因在同一物种不同胁迫条件下表达的稳定性不尽相同,所以需要根据不同处理条件选择相对稳定表达的内参基因。此外,生物体中持家基因数目庞大,今后还有待进一步挖掘表达水平更稳定的内参基因。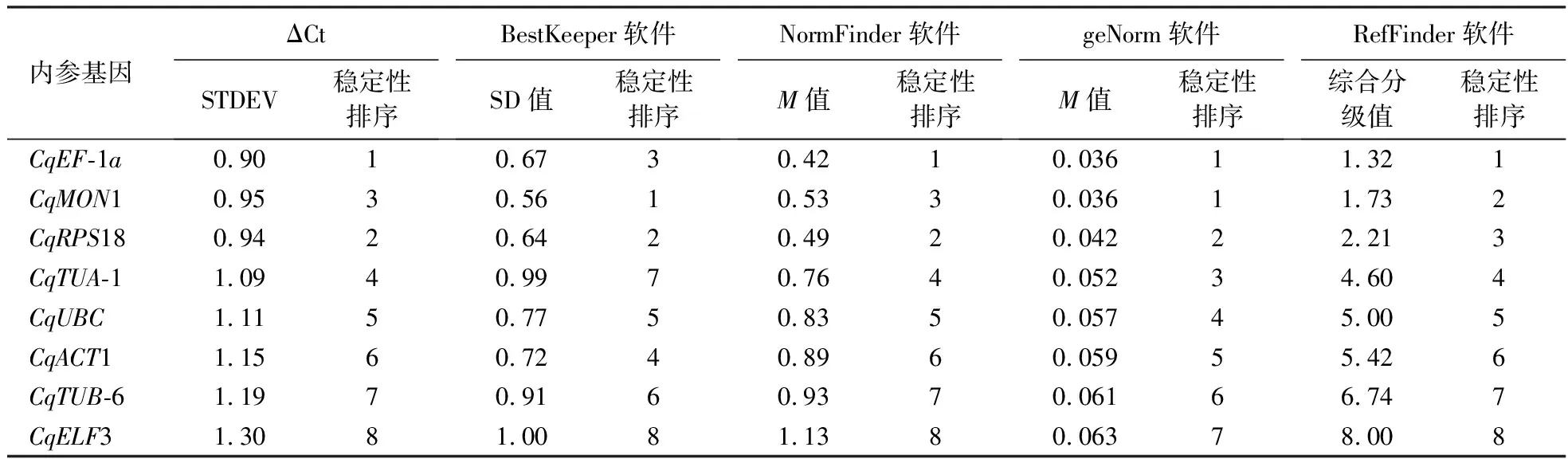

2.5 候选内参基因稳定性验证
3 讨论

