禾谷炭疽菌中剪接因子SR蛋白的生物信息学分析及基因克隆
张艺美,代亚锋,叶云英,陈 震,宫安东
(信阳师范大学 生命科学学院/河南省茶树生物学重点实验室, 河南 信阳 464000)
0 引言
玉米是我国三大主粮之一,其种植面积和总产量仅次于水稻和小麦,在保证我国粮食安全方面发挥着不可替代的作用。近年来,随着气候和农耕方式的变化,玉米炭疽病(Maize anthracnose)已成为威胁全球粮食安全的新兴病害[1]。玉米炭疽病是一种真菌性病害,在玉米种植区普遍发生,早期主要危害玉米叶片,出现梭形病斑,后期病斑融合致叶片枯死;发病严重时,危害茎秆,引起玉米茎基腐,造成玉米大量减产[2]。禾谷炭疽菌(Colletotrichumgraminicola)是引起玉米炭疽菌病的病原物,该病菌还能侵染小麦、高粱等禾本科作物引起炭疽病,给各国农业生产造成巨大的经济损失[3-4]。目前对玉米炭疽病的防治主要依赖苯并咪挫类化学药剂,而化学防治易带来病原抗药性、农药残留和环境污染等问题[5]。从长远角度考虑,深入解析禾谷炭疽菌致病相关因子,可以为开发安全有效的新型药剂提供特异性靶标,为作物抗病育种提供新理论依据和思路。
前体mRNA剪接是真核生物大多数基因表达的基础,并且可变剪接使单一基因产生多样蛋白,丰富了高等真核生物的蛋白多样性[6]。SR (serine and arginine-rich)蛋白对真核生物前体mRNA的组成型剪接及可变型剪接都起重要作用[7]。SR蛋白是一类具有高度保守结构域的剪接因子,N端含有一个或两个RNA识别结构域(RNA recognition motif, RRM),C端含有重复数量不同、排列可变的精氨酸/丝氨酸二肽结构域(Arginine/Serine rich domain)[8]。剪接复合体E的形成依赖SR蛋白和外显子加强子(ESE)的互作[9-10];复合体E转变为复合体A主要是通过RS结构域和磷酸二酯骨架的非特异性结合,进而促进U2 snRNP和分支点序列碱基的紧密结合[11]。SR蛋白在复合体A招募U4/U6-U5三联体形成“B-like”复合体以及复合体C的活性催化方面都有作用[12-13]。同时,SR蛋白还调节转录的延伸以及剪切和转录的共偶联活动[14-16]。除此之外,SR蛋白还参与mRNA的其他代谢活动,如mRNA的核输出,mRNA的衰减和翻译等[17]。
在真菌中,目前关于SR蛋白的研究相对较少。酿酒酵母的SR蛋白Npl3是减数分裂时期调控基因剪接所必需的,并且对于减数分裂细胞周期的正确执行是必不可少的[18]。FgSgh1和FgSrp1都能调控禾谷镰刀菌的生长、分生孢子的产生、植物侵染及mRNA的剪接[19-20]。在模式生物稻瘟菌中,MoSrp1的缺失导致病原菌生长速率减慢、产孢量下降、分生孢子畸形、致病力显著下降等表现及菌丝时期上千基因的可变剪接发生异常并进一步发现MoSrp1直接结合RNA的GUAG基序[21]。作为全球十大植物病原真菌,炭疽菌属寄主广泛,可侵染多种重要粮食作物和经济作物[22-23]。迄今为止,在炭疽属真菌中还未发现关于SR蛋白家族的生物信息学研究报道,严重制约SR蛋白的功能研究。
裂殖酵母(Schizosaccharomycespombe)作为真核模式生物,体内存在2个SR蛋白[24-25]。本研究基于裂殖酵母2个SR蛋白的氨基酸序列,利用禾谷炭疽菌的蛋白数据库通过Blastp比对分析及关键词搜索,获得与裂殖酵母SR蛋白同源的序列,并通过生物信息学分析明确保守结构域、理化性质、疏水性、二级和三级结构预测、亚细胞定位及核定位信号预测等特征,为进一步开展禾谷炭疽菌中SR蛋白的功能研究、同属炭疽菌但其基因组序列尚未公布的其他炭疽菌的研究,提供重要的理论指导。
1 材料与方法
1.1 材料
数据库信息:根据真菌模式生物裂殖酵母Schizosaccharomycespome数据库(http://fungi.ensembl.org/Schizosaccharomyces_pombe/Info/Index)中含有的2个经典SR蛋白氨基酸序列,利用禾谷炭疽菌Colletotrichumgraminicola蛋白质数据库(http://fungi.ensembl.org/Colletotrichum_graminicola/Info/Index)在线Blastp比对,参数选择为默认值,获得Colletotrichumgraminicola中含有的SR蛋白,明确此菌内编码SR蛋白的基因登录号和蛋白登录号信息。
真菌菌株:禾谷炭疽菌CgM2(Colletotrichumgraminicola)由信阳师范学院生命科学学院应用与环境微生物学实验室提供,存于-80 ℃冰箱。
PDA培养基:土豆200 g,葡萄糖20 g,琼脂15 g,UP水定容至1 L,用于禾谷炭疽菌的培养。
CTAB提取液:CTAB 8 g,0.5 mol/L EDTA (pH8.0) 16 mL,0.5 mol/L Tris-HCl (pH8.0) 80 mL, NaCl 32.72 g,UP水定容至400 mL。
引物设计与合成:参考https://fungi.ensembl.org/Colletotrichum_graminicola/Info/Index
数据库的序列信息,设计CgSRP1和CgSRP2基因序列的特异性引物。引物由安徽通用生物股份有限公司合成。
1.2 方法
1.2.1 保守结构域预测
利用SMART网站在线分析禾谷炭疽菌中SR蛋白所具有的保守结构域特征,同时,利用ClustalX对上述SR蛋白进行保守结构域分析。
1.2.2 系统进化树构建
为了分析CgSRP1和CgSRP2基因的系统进化,利用NCBI数据库进行蛋白序列比对,在果生炭疽菌(Colletotrichumfructicola)、粗糙脉孢菌(Neurosporacrassa)、球孢白僵菌(Beauveriabassiana)、禾谷镰刀菌(Fusariumgraminearum)、黑曲霉(Aspergillusniger)、构巢曲霉(Aspergillusnidulans)、粟酒裂殖酵母(Schizosaccharomycespombe)、禾柄锈菌(Pucciniagraminis)、玉米黑粉菌(Ustilagomaydis)等真菌蛋白数据库及小麦(Triticumaestivum)、水稻(Oryzasativa)、高粱(Sorghumbicolor)和玉米(Zeamays)等植物蛋白数据库中分别找到CgSrp1和CgSrp2的同源蛋白,利用MEGA7软件建立CgSrp1和CgSrp2系统进化树,多序列比对采用ClustalW,构建进化树采用Neighbor-Joining Tree。
1.2.3 蛋白质理化性质预测
利用蛋白质数据库(http://www.expasy.ch/tools/protparam.html)在线对禾谷炭疽菌中SR蛋白的分子质量、等电点及氨基酸组成等特征进行分析预测。
1.2.4 蛋白质疏水性预测
利用Protscale程序(http://web.expasy.org/protscale)对禾谷炭疽菌中SR蛋白在线分析。
1.2.5 蛋白质亚细胞定位分析
利用Cell-PLoc(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)对禾谷炭疽菌中的SR蛋白进行在线亚细胞定位预测;并利用核定位预测网站NLStradamus(http://www.moseslab.csb.utoronto.ca/NLStradamus/)对SR蛋白进行在线分析。
1.2.6 蛋白质二、三级结构预测
利用PSIPRED(http://bioinf.cs.ucl.ac.uk/psipred/)在线预测禾谷炭疽菌中SR蛋白的二级结构;利用Phyre2(http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)预测SR蛋白的三级结构,并用PDB(https://www.rcsb.org/#Category-visualize)对SR的三级结构的预测结果进行3D视图。
1.2.7 CgM2基因组DNA的提取
将存于-80 ℃冰箱的CgM2活化于PDA平板上,28 ℃静置培养4 d后,挑取气生菌丝到650 μL CTAB中,加入等体积的CI溶液(氯仿和异戊醇的体积比为24∶1)和钢珠1粒打磨菌丝。待菌丝充分打磨后,12 000 r/min离心7 min,吸取上清液到1.5 mL无菌离心管内,并加入50 μL的3 mol/L NaAc和500 μL的异丙醇,混匀后静置30 min,12 000 r/min离心10 min,弃上清,加入500 μL 75%的乙醇洗涤沉淀,离心弃上清,通风条件下吹干沉淀,加入50 μL无菌水将白色沉淀溶解,此即禾谷炭疽菌CgM2的基因组DNA,存于-20 ℃冰箱备用。
1.2.8CgSRP1和CgSRP2基因的克隆
用primer5软件设计克隆CgSRP1和CgSRP2基因的引物,其引物序列见表4。以1.2.7步骤中提取的CgM2基因组DNA为模板,以CgSRP1-F/R和CgSRP2-F/R为引物分别扩增CgSRP1和CgSRP2基因片段。
2 结果与分析
2.1 禾谷炭疽菌中含有2个SR蛋白
通过比对禾谷炭疽菌蛋白质数据库,发现该菌中存在2个候选的SR蛋白,其ID分别是GLRG_09205-1和GLRG_08760-1(表1)。根据S.pome中SR蛋白的命名规则分别将禾谷炭疽菌中GLRG_09205和GLRG_08760命名为CgSrp1和CgSrp2。经过SMART分析和与裂殖酵母SR蛋白序列比对发现,CgSrp1蛋白序列的N端含有一个保守的RNA结合结构域,C端含有丝氨酸/精氨酸二肽结构域(图1,表1);CgSrp2蛋白序列的N端含有2个保守的RNA结合结构域,C端含有2个精氨酸/丝氨酸区二肽结构域(图1,表1)。
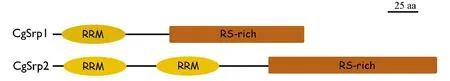
图1 禾谷炭疽菌中SR蛋白保守结构域分析

表1 禾谷炭疽菌中SR蛋白基本信息及保守结构域分布
2.2 2个SR蛋白系统进化树及差异序列分析
通过CgSrp1和CgSrp2的系统进化树发现,禾谷炭疽菌与同是炭疽菌属的果生炭疽菌亲缘关系最近,与担子菌门的玉米黑粉菌亲缘关系较远,与植物的亲缘关系最远(图2a和2b)。

注:a. Srp1的系统进化树;b. Srp2的系统进化树;c. Srp1的在真菌和植物之间的显著差异序列;d. Srp2的在真菌和植物之间的显著差异序列。
通过蛋白序列分析发现,SR蛋白在上述几个物种中较为保守,特别是RNA结合结构域高度保守,在不同中物种序列基本一致,这与SR蛋白能识别结合RNA,促进前体mRNA的剪接功能相符合。尽管SR蛋白在不同物种中其序列有较高的相似性,但在真菌和植物内其序列还存在一定的差异性。红色方框内分别显示Srp1和Srp2蛋白序列在真菌中是一样的,但与植物的序列存在较大差异或在植物中不存在类似的序列(图2c和2d)。
2.3 蛋白质理化性质分析
对Colletotrichumgraminicola中的2个SR蛋白的理化性质预测发现,因CgSrp1和CgSrp2所含氨基酸数目不同,它们的相对分子质量、分子式及原子数量均差别较大。二者相比,含氨基酸数量较多的CgSrp2相对分子质量、原子数量最大。CgSrp1和CgSrp2的理论等电点分别是9.32和9.38,均属于碱性氨基酸,而蛋白质内正负电荷氨基酸残基数量的多少影响等电点,当正电荷氨基酸残基多于负电荷氨基酸残基时,其等电点偏向于碱性(表2)。CgSrp1和CgSrp2的半衰期均为30 h,尽管两个SR蛋白的稳定系数相差较大,但均大于40,都属于不稳定蛋白;总平均亲水性均为负值(表2),表明CgSrp1和CgSrp2均属于亲水性蛋白。

表2 禾谷炭疽菌中SR蛋白的基本理化性质
2.4 蛋白质疏水性分析
利用Protscale程序分别对CgSrp1和CgSrp2蛋白进行疏水性分析,氨基酸标度选择默认的Hphob./Kyte&Doolittle,计算窗口数值设置为9,采用线性加权模型,输出结果显示CgSrp1中位于130位的精氨酸(R)亲水性最强,47位的亮氨酸(L)疏水性最强;CgSrp2中位于231位的丝氨(S)亲水性最强,169位的精氨酸(R)疏水性最强;CgSrp1、CgSrp2的亲水性氨基酸总和分别为-346.642、-415.807;疏水性氨基酸总和分别为8.276、12.866(图3)。CgSrp2蛋白的总平均亲水性是-1.735。尽管CgSrp1和CgSrp2在亲水性最强氨基酸、疏水性最强氨基酸、亲水性氨基酸残基数值总和、疏水性氨基酸残基数值总和等结果不相同,但它们均为亲水性蛋白。经计算,CgSrp1和CgSrp2蛋白的总平均亲水性分别是-1.735、-1.291,与利用protparam程序计算的结果-1.745、-1.316相近,说明此分析结果可信度较高。
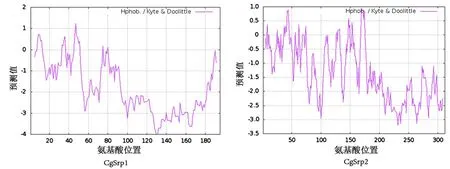
图3 禾谷炭疽菌SR蛋白疏水性情况
2.5 亚细胞定位及核定位信号分析
将SR蛋白的fasta格式的氨基酸序列提交到Cell-PLoc2.0的Euk-mPLoc2.0内,结果显示CgSrp1和CgSrp2均定位于细胞核。进一步利用核定位信号预测网站NLStradamus在线预测SR蛋白内的核定位信号,将Prediction Cutoff设置为0.9,CgSrp1蛋白序列的101—118 APRRSSPRRGRSPSPRRS是核定位信号。将Prediction Cutoff设置为0.5,CgSrp1蛋白序列的95—154 GRDRERAPRRSSPRRGRSPSPRRSTRDYSPKKDDRRDRDRDYDRSDRRDTRERSRSPDRR是核定位信号。将Prediction Cutoff 设置为0.5,CgSrp2蛋白序列的188—198 ARSRSPGGGRR是核定位信号,Prediction Cutoff设置为0.9时,没有核定位序列,说明CgSrp1具有更强的核定位信号,其蛋白定位很可能定位在细胞核。
2.6 SR蛋白的二级结构中无规则卷曲所占比例较高
利用PSIPRED在线预测SR蛋白的二级结构,发现CgSrp1蛋白有3个α螺旋、5个β折叠和一些无规则卷曲结构(图4a);CgSrp2蛋白有4个α螺旋、9个β折叠和一些无规则卷曲结构(图4b)。分析发现,在CgSrp1蛋白中,α螺旋和β折叠所占比例都是12.8%,无规则卷曲结构较多,占比为74.4%(图4c);α螺旋占CgSrp2的氨基酸比例是12.5%,β折叠所占比例是14.1%,无规则卷曲结构较多,所占比例为73.4%(图4c)。结果表明,虽然CgSrp1和CgSrp2的氨基酸长度不相同,但α螺旋、β折叠和无规则卷曲等二级结构所占比例相差不大。
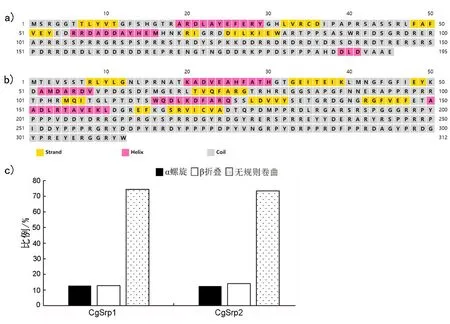
注:a. CgSrp1的二级结构情况;b. CgSrp2的二级结构情况;c. CgSrp1和CgSrp2所含的α螺旋、β折叠和无规则卷曲在各自二级结构中所占比例情况。
2.7 SR蛋白的三级结构
用Phyre2预测CgSrp1和CgSrp2的三级结构,图5分别是CgSrp1的第2—112位氨基酸的三级结构图,置信值是99.9%;和CgSrp2的第5—179位氨基酸三级结构图,置信值是100%,其余的氨基酸是无序区域,不能预测其三级结构。用PDB对CgSrp1和CgSrp2的三级结构进行3D view发现,CgSrp1蛋白有3个α螺旋、6个β折叠和一些无规则卷曲结构,CgSrp2蛋白有5个α螺旋、9个β折叠和一些无规则卷曲结构。CgSrp2氨基酸残基的第90、91位都是天冬酰胺,这二者之间的连接方式未能预测出来,因此,CgSrp2三级结构展示图是不连续的两个区域(图5)。
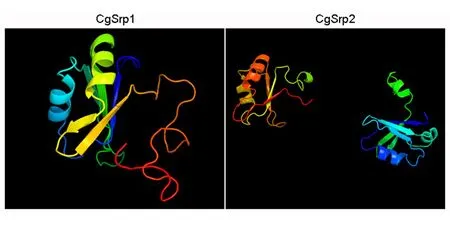
注:CgSrp1的三维坐标是:X:47.943, Y:40.851, Z:37.510;CgSrp2的三维坐标是:X:77.873, Y:41.636, Z:59.900;蛋白质N端到C端的顺序依彩虹颜色而定。
2.8 CgSRP1和CgSRP2的基因克隆
以CgM2为模板,分别以CgSRP1-F/R和CgSRP2-F/R为引物PCR扩增CgSRP1和CgSRP2的基因片段,此片段包括基因5’和3’端的转录非编码序列(表3)。琼脂糖凝聚电泳结果显示,PCR扩增出来的CgSRP1和CgSRP2基因片段大小与预期一致,如图6所示,经DNA测序验证扩增出来的基因片段序列与禾谷炭疽菌数据库中的碱基序列是一致的(https://fungi.ensembl.org/Colletotrichum_graminicola/Info/Index),说明禾谷炭疽菌基因组中存在CgSRP1和CgSRP2基因。

图6 CgSRP1和CgSRP2基因的扩增片段
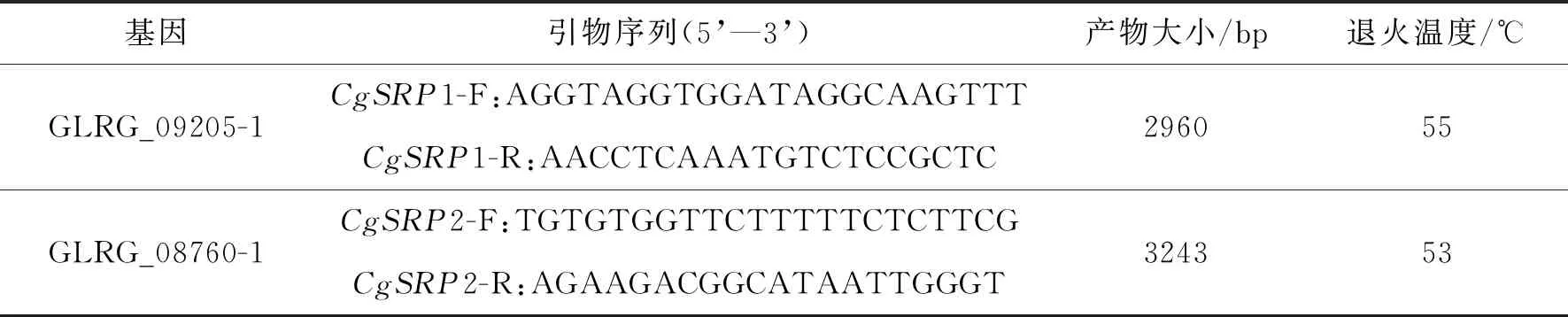
表3 引物信息
3 讨论
SR蛋白家族广泛存在于动植物和真菌中,是调控真核生物可变剪接的一类重要蛋白。研究表明,可变剪接与人类多种疾病的产生密切相关,而SR蛋白家族成员的表达量在不同肿瘤中都存在异常。目前已证实SRSF9是一个原癌基因,且在结肠癌、脑癌和肺癌等肿瘤组织中的表达量显著高于正常组织[26]。植物基因特别是与逆境相关的基因在干旱、高温和盐害等胁迫下更易发生可变剪接事件,说明可变剪接参与植物响应逆境胁迫过程。除了和植物响应逆境胁迫相关,SR蛋白还参与调控植物的生长发育。在裂殖酵母中,srp1基因的敲除突变体对低温更敏感,当过表达Srp1蛋白中的RNA结合结构域时,酵母生长停止[27];srp2是裂殖酵母的必需基因,同时突变Srp2蛋白中RS1和RS2导致酵母不能生长[20]。禾谷镰刀菌中,Fgsrp1突变体生长速率变慢、分生孢子产量严重下降、DON合成量降低以及致病力显著下降,RNA-seq分析FgSRP1的缺失影响其他基因的可变剪接[17]。不同于srp2是裂殖酵母的必需基因,Fgsrp2突变体生长速率稍微降低[28]。尽管SR蛋白家族成员的功能在人类和植物细胞中已有相当多的文献报道,但在植物丝状病原真菌中揭示SR蛋白作用机制的研究少有涉及,特别是关于炭疽属病原菌的SR蛋白功能研究未见报道。
中外学者对SR蛋白在动、植物和真菌中的作用已有揭示,结合丝状病原真菌禾谷镰刀菌中SR蛋白作用机制研究,有理由推测SR蛋白调控禾谷炭疽菌的生长发育及在玉米侵染过程中发挥重要作用。本研究利用生物信息学对Colletotrichumgraminicola中SR蛋白家族的系统进化、蛋白序列、理化性质、疏水性、亚细胞定位、二级及三级结构等方面进行分析。系统进化树分析发现SR蛋白在真菌和植物中都比较保守,特别是RNA识别结构域序列基本一致,说明SR蛋白作为前体mRNA的剪接因子在不同物种中发挥重要功能。尽管SR蛋白比较保守,但氨基酸序列比对结果显示,在真菌和植物内其序列还存在一定的差异性。红色方框内分别显示Srp1和Srp2蛋白序列在不同的真菌中是一样的,保守性很高,预测此区域序列可能调控真菌的生长发育。红色方框内的植物SR蛋白序列与真菌序列存在很大差异或植物中不存在类似的序列(图2C和2D),因此,此序列可作为真菌SR蛋白的潜在药物靶点,只抑制病原菌而不影响植物的生长发育,为新型药物的开发提供理论依据。
蛋白序列比对结果显示GLRG_09205(CgSrp1)和GLRG_08760(CgSrp2)的N端含有RNA结合结构域,C端包含RS双肽结构,符合典型的SR蛋白结构特征,因此,CgSrp1和CgSrp2都属于SR蛋白家族成员。疏水性分析发现CgSrp1和CgSrp2均是亲水性蛋白,说明CgSrp1和CgSrp2不含跨膜结构,不属于膜蛋白。亚细胞定位对研究蛋白功能非常重要,只有蛋白质定位正确,才可以执行正常的生物学功能,本研究发现CgSrp1和CgSrp2均定位于细胞核并含有核定位信号,这和SR蛋白作为剪接因子调控mRNA剪接功能相一致。蛋白质二级和三级结构分析发现,CgSrp1和CgSrp2都有α螺旋、β折叠和无规则卷曲。由于二级和三级结构预测软件不同,二级结构采用人工神经网络、遗传算法等技术构建的预测方法,三级结构是利用同源建模法对蛋白序列进行分析,所以,预测结果显示CgSrp1和CgSrp2含有α螺旋和β折叠的数量略有不同。通过上述分析对Colletotrichumgraminicola中SR蛋白的基本特征有了初步分了解,本研究同时给炭疽菌属中的其他病原菌SR蛋白生物信息学分析提供参考,但SR蛋白如何调控Colletotrichumgraminicola生长发育及具体的致病机制还需要深入解析探索。
4 结束语
禾谷炭疽菌中存在两个典型的SR蛋白CgSrp1和CgSrp2,分别含195和312个氨基酸,均为亲水性蛋白,均定位于细胞核,C端均有RS结构域,但CgSrp1的N端有一个RRM结构域,而CgSrp2的N端有两个RRM结构域。二者的二级和三级结构中α螺旋、β折叠和无规则卷曲的占比相似,且无规则卷曲具有较高的比例。本文通过构建SR蛋白的系统进化树并比对SR蛋白在真菌和植物中的序列差异性,挖掘真菌SR蛋白的特有序列。尽管如此,关于CgSRP1和CgSRP2基因调控禾谷炭疽菌生长发育及侵染致病的分子机制还不清楚,需要今后实验逐一解决。本研究成果将为进一步解析禾谷炭疽菌中SR蛋白的功能提供重要的理论依据;同时以本文中发现的真菌特有SR蛋白序列为药物靶标,为防治禾谷炭疽菌及其他炭疽菌属病原菌新型药剂的开发起到重要的理论指导作用。

