PEG 模拟干旱条件下黄瓜种子发芽前后转录组分析
谢洋 周国彦 苏航 邢雨蒙 闫立英
(河北科技师范学院园艺科技学院 河北省高校特色园艺植物生物育种应用技术研发中心,秦皇岛 066004)
黄瓜(Cucumis sativusL.)是世界性蔬菜作物,属于葫芦科黄瓜属一年生蔓生草本植物[1]。受全球气候变暖的影响,我国干旱、半干旱化的耕地面积持续增加,全国大部分地区干旱水平呈上升趋势[2]。黄瓜根系浅(吸水能力弱)、叶片大(蒸腾作用强),喜湿且不耐旱[3]。干旱胁迫对黄瓜的生长发育、产量和品质形成会产生诸多不良影响,如种子发育不良[4]、植株萎蔫[5]、开花结果异常[6],甚至死亡[7]等现象。因此,解析黄瓜干旱胁迫响应分子作用机制对于改良其品质和产量具有重要意义。
一定干旱条件下,植物体自身具有维持细胞水分平衡从而保持生存的策略。一方面,植物受到干旱胁迫后,体内产生活性氧,当活性氧超过植物体可处理能力后就会启用抗氧化防御系统,主要是超氧化物歧化酶(SOD)、过氧化物酶(POD)和过氧化氢酶(CAT)等抗氧化酶的调节作用,通过几种抗氧化酶的动态变化清除植物体内过量的活性氧,以维持植物体自身平衡[8];另一方面,干旱胁迫还会对植物产生直接的水分胁迫,而植物细胞会通过积累渗透调节物质如K+、Na+、Ca2+等无机离子,脯氨酸、甜菜碱、可溶性糖和多元醇等有机溶质及诱导产生的渗透调节蛋白等以降低细胞的渗透势和防止细胞脱水[9]。此外,ABA 途径是调节植物干旱响应并合理利用水分的重要策略,干旱胁迫促使植物器官中的ABA 产生和积累进而激活下游信号传导[10]。
植物对干旱胁迫的应答是一个涉及多基因、多信号途径和代谢过程的复杂反应机制[11]。转录组测序可以从mRNA 水平全面地研究植物基因功能及特定的生物学过程,为园艺植物重要性状基因挖掘和分子标记开发提供强大技术支持[12]。
本研究以抗旱型旱黄瓜‘KS30’与敏感型旱黄瓜‘KS30’为试材,分别通过种子萌发期的PEG 模拟干旱诱导进行发芽前期和发芽后期取样,构建8个cDNA 文库,利用转录组测序技术和生物信息学分析技术挖掘旱黄瓜抗旱相关基因,并基于PEG 模拟干旱诱导下抗旱型与敏感型黄瓜转录组数据开发全转录组水平的SNP 分子标记,进一步通过抗旱相关差异基因与SNP 变异位点信息,筛选旱黄瓜抗旱相关SNP 标记。
1 材料与方法
1.1 材料
本研究选用的抗旱型旱黄瓜‘KS30’(DT)和敏感型旱黄瓜‘KS30’(DS)为杂种一代品种。
1.2 方法
1.2.1 样品准备与转录组测序 将DT 和DS 种子分别进行去离子水(CK)和15% PEG6000 溶液(T)处理36 h 后进行第1 批次取样,样品命名为DT1‑T、DT1‑CK、DS1‑T、DS1‑CK;继续观察12 h 后进行第2 批次取样,命名为DT2‑T、DT2‑CK、DS2‑T、DS2‑CK。样品取样后立即存入超低温冰箱,备用。每个处理15 粒种子,3 次生物学重复。
根据总RNA 提取试剂盒(TIANGEN)操作说明,分别提取8 份样品总RNA。利用琼脂糖凝胶电泳分析RNA 降解程度和是否有污染,Nanodrop检测RNA 纯度(OD260/280)。总RNA 样品检测合格后,用磁珠寡核苷酸Oligo(dT)(Invitrogen)提取mRNA 并用裂解缓冲液将其片段化;以mRNA 为模板,用随机引物合成第一条cDNA 链,随后加入buffer、dNTPs 和DNA 聚合酶I 合成第二条cDNA。双链cDNA 纯化和末端修复以及3'末端单核苷酸A(腺嘌呤)添加;将此片段连接到测序接头上,通过PCR 扩增富集获得最终的cDNA 文库;最后,利用Illumina HiSeqTM2500 平台进行文库测序[13]。RNA提取、cDNA 文库构建与上机测序委托北京诺禾致源科技股份有限公司。
1.2.2 参考基因组比对与新基因预测 将测序获得的原始数据raw reads 经过滤、测序错误率检查、GC含量分布检查,获得后续分析使用的clean reads。使用HISAT2 软件将clean reads 与参考基因组进行快速精确的比对,获取reads 在参考基因组上的定位信息[14]。
1.2.3 定量分析与差异基因鉴定 根据基因比对在参考基因组上的位置信息,采用subread 软件中的featureCounts 工具统计每个基因从起始到终止范围内覆盖的readscount。采用FPKM(expected number of fragments per kilobase of transcript sequence per millions base pairs sequenced)方法进行基因表达定量[15]。基因表达定量完成后,对其表达数据进行统计学分析,获得假设检验概率(P‑value)和多重假设检验校正FDR 值(Padj),筛选样本在不同状态下表达水平显著差异的基因。利用edgeR 软件,筛选标准设定为|log2(FoldChange)|≥2 且Padj ≤0.05[16]。
1.2.4 差异基因韦恩图与富集分析 利用在线网站(https://bioinfogp.cnb.csic.es/tools/venny/index.html)制作不同比较组合间的韦恩图,展示比较组合共有或独有的差异基因。采用clusterProfile 软件分别对差异基因集进行GO(gene ontology)功能富集和KEGG(Kyoto encyclopedia of genes and genomes)通路富集分析,均以Padj 小于0.05 作为显著性富集的阈值,并分别选取最显著的20 个通路绘制散点图进行结果的展示[17-18]。
1.2.5 SNP 变异位点检测与分析 利用PEG 模拟干旱诱导下抗旱型与敏感型黄瓜转录组数据开发全转录组水平的SNP 分子标记。使用GATK 进行变异位点的检测,进而根据SnpEff 注释信息对每个变异位点进行变异位点功能统计(同义突变、错义突变、无义突变)、变异位点区域统计(EXON、INTRON、INTERGENIC)以及变异位点影响统计(HIGH、MODERATE、LOW、MODIFIER)等方面进行分析和绘图[19]。联合转录组挖掘到的重要候选基因和SNP 位点检测信息,筛选抗旱相关SNP 分子标记。
1.2.6 荧光定量PCR 分析 选取转录组中差异倍数大、表达丰度高且含有SNP 变异位点的基因进行荧光定量PCR(RT‑qPCR)分析。采用Beacon Designer 7.0 软件设计RT‑qPCR 引物,内参基因序列信息参考前人研究报道[20],均由中美泰和生物技术(北京)有限公司合成。总RNA 提取(型号DP432)、cDNA反转录(型号KR116)与RT‑qPCR 加样与扩增程序(型号FP313)均参考天根生化科技(北京)有限公司试剂盒说明书,每个基因3 次重复。利用美国伯乐bio‑rad CFX connect 实时荧光定量PCR 仪进行扩增,基因相对表达量计算采用2-∆∆CT方法,导出数据经Excel 和DPS 软件作图与差异显著性分析。
2 结果
2.1 PEG模拟干旱诱导下黄瓜种子萌发特征分析
从图1 可知,PEG 模拟干旱诱导下,敏感型(DS)旱黄瓜种子萌发力较对照变差,其芽长和芽长+根长分别下降90.90%和42.29%;而抗旱型(DT)旱黄瓜较对照其种子萌发力增强,其芽长和芽长+根长分别增加93.33%和24.50%(图1)。结果表明,基因型的差异导致抗旱型(DT)和敏感型(DS)旱黄瓜种子响应干旱的机制不同,且抗旱型(DT)更能适应干旱胁迫环境。
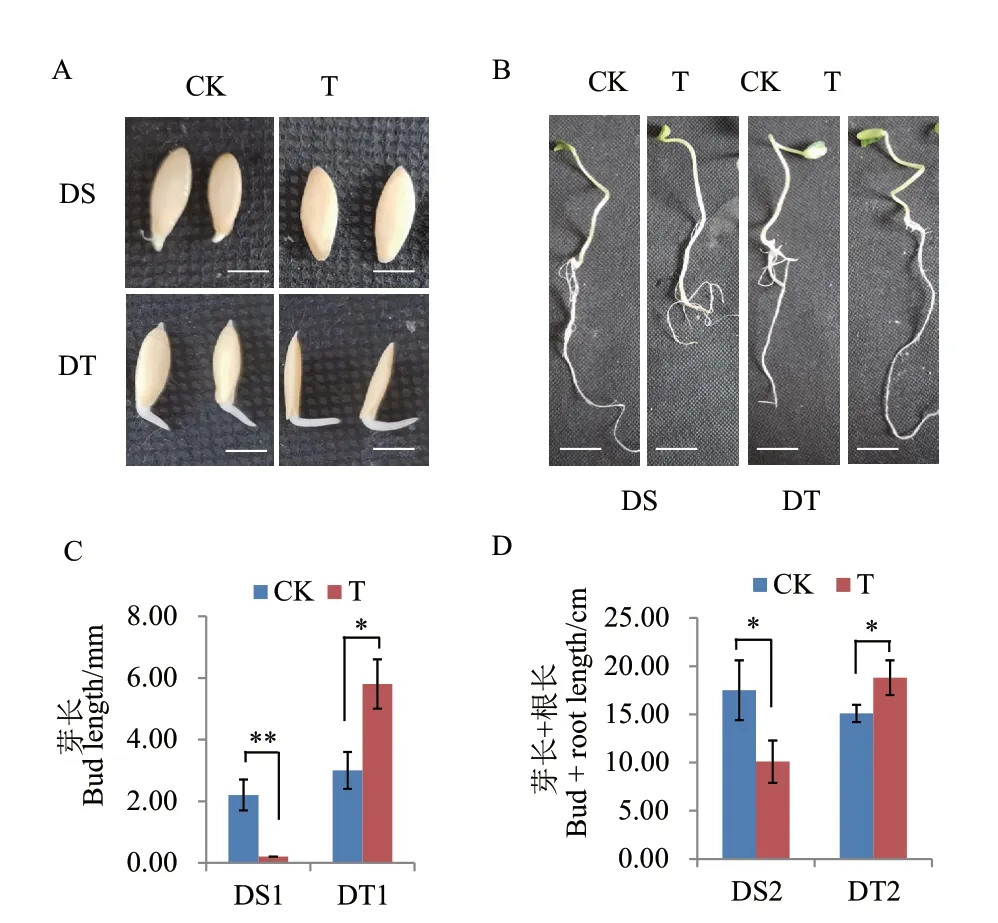
图1 PEG 模拟干旱诱导下旱黄瓜种子萌发特征Fig. 1 Seed germination characteristics of dry cucumber under PEG drought simulation
2.2 测序数据质量分析
从8 个样本文库DT1_CK、DT1_T、DS1_CK、DS1_T、DT2_CK、DT2_T、DS2_CK、DS2_T 中分别获 得43.83、39.02、40.69、42.27、40.79、43.82、41.55 和43.06 M 的clead reads, 与raw reads 比值介于97.94%-99.18%,且其Q20 值分别为97.20%、97.58%、97.10%、97.30%、97.13%、97.23%、97.34%和97.22%,说明测序reads 成功比对到基因组的比例非常高,能够获得高比例的reads 在参考基因组上的定位信息(表1)。
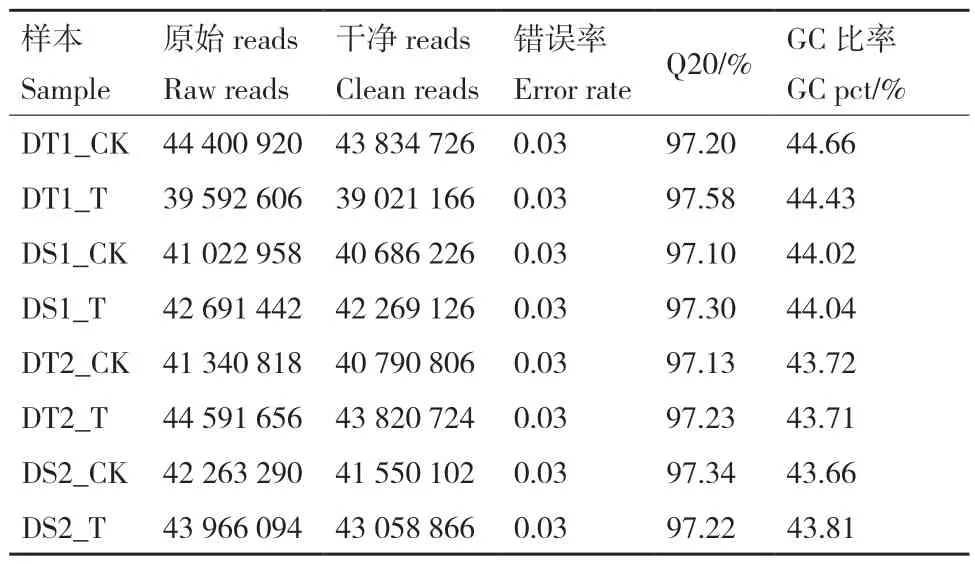
表1 样本测序数据质量汇总Table 1 Quality summary of samples sequencing data
2.3 差异表达基因鉴定
从4 组比较组差异基因数量可看出,抗旱型材料发芽前期PEG 模拟干旱诱导较对照(DT1_TvsDT1_CK)鉴定到727 个上调和345 个下调差异基因,敏感型材料发芽前期PEG 模拟干旱诱导较对照(DS1_TvsDS1_CK)鉴定到1 226 个上调和111个下调差异基因;抗旱型材料发芽后期PEG 模拟干旱诱导较对照(DT2_TvsDT2_CK)鉴定到333 个上调和480 个下调差异基因,敏感型材料发芽后期PEG 模拟干旱诱导较对照(DS2_TvsDS2_CK)鉴定到600 个上调和470 个下调差异基因。同一时期,抗旱型较敏感型的差异基因数量偏少,并且在上调基因数量上尤为明显。如发芽前期抗旱型诱导与对照相比,上调基因727 个,下调基因345 个;而敏感型上调基因1 226 个,是抗旱型1.69 倍。同样,发芽后期敏感型上调基因是抗旱型的1.80 倍(图2)。结果说明,敏感型旱黄瓜对PEG 模拟干旱诱导响应表现更为剧烈。

图2 比较组合差异基因数目统计柱状图Fig. 2 Statistical bar chart for comparison of the number of combinatorial differential genes
2.4 差异基因韦恩图分析
发芽前期抗旱型和敏感型2 个材料,PEG 模拟干旱诱导较处理组共有差异基因数量为89 个,分别有212 和1 202 个差异基因特异于抗旱型和敏感型比较组中。在发芽后期抗旱型和敏感型2 个材料,PEG 模拟干旱诱导较处理组共有差异基因数量为132 个,分别有681 和938 个差异基因特异于抗旱型和敏感型比较组中(图3)。从差异基因数量来看,发芽前期响应PEG 模拟干旱诱导的差异基因在抗旱型和敏感型旱黄瓜中差异数目较悬殊,可作为后续抗旱相关基因挖掘的重要数据基础。
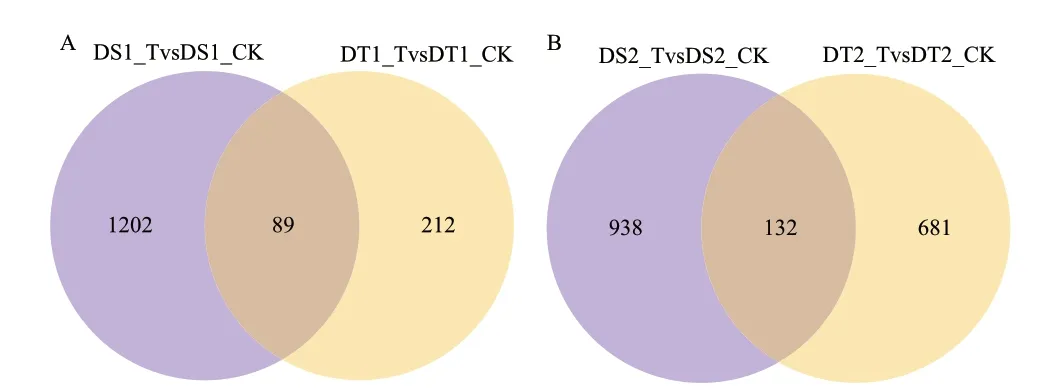
图3 比较组合差异基因韦恩图Fig. 3 Venn diagram of comparing the combined differential genes
2.5 差异基因富集分析
发芽前期抗旱型材料,PEG 模拟干旱诱导较处理组差异基因主要显著富集的GO 条目有氧化还原过程(GO:0055114)、细胞壁(GO:0005618)、氧化还原酶活性(GO:0016491)、阳离子结合(GO:0005506)等;发芽期敏感型材料,PEG 模拟干旱诱导较处理组差异基因主要显著富集的GO 条目有氧化还原过程(GO:0055114)、响应刺激(GO:0050896)、膜(GO:0016020)、氧化还原酶活性(GO:0016491)、转运活性(GO:0005215)、DNA 结合转录因子活性(GO:0003700)等(图4)。
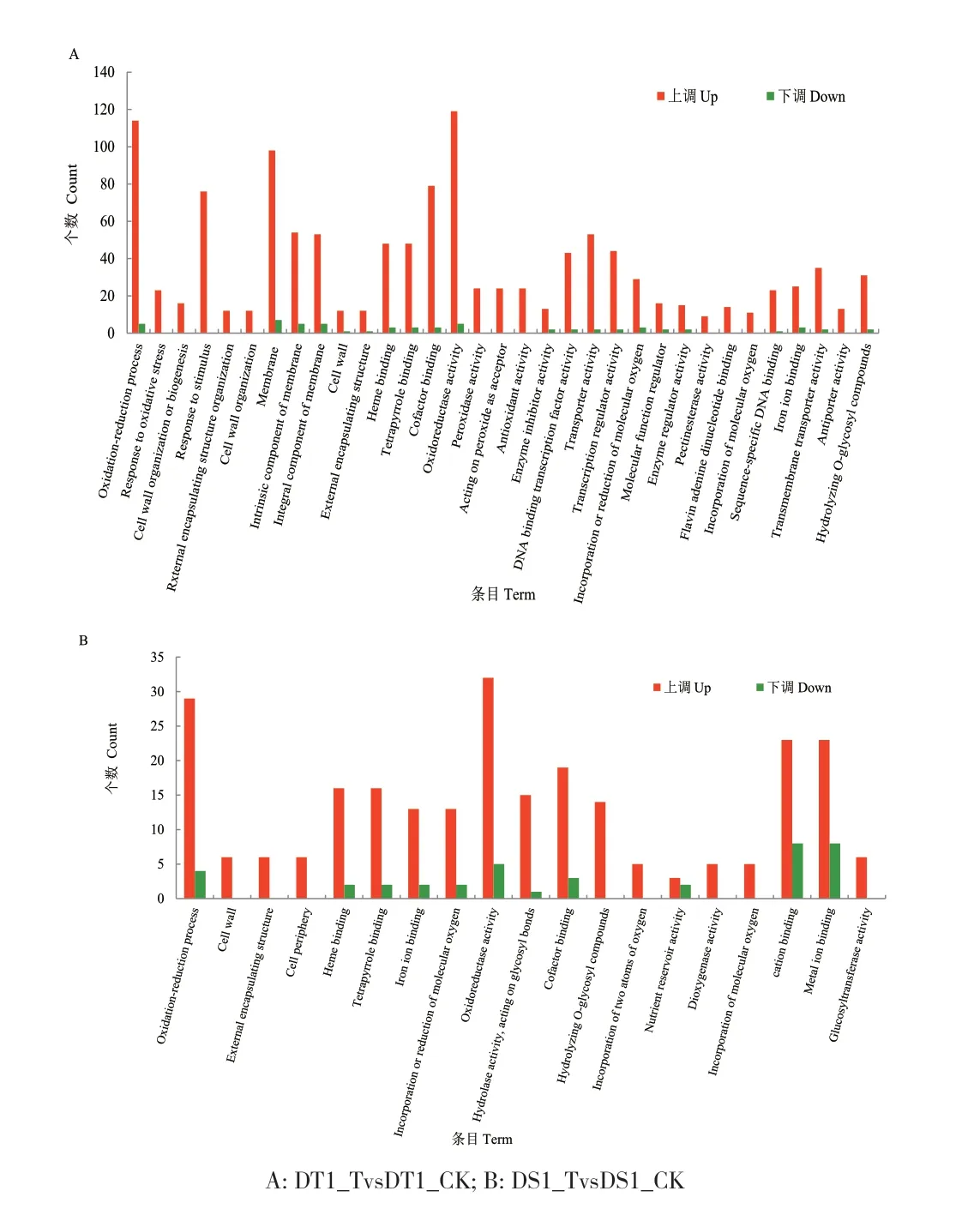
图4 GO 富集分析柱状图Fig. 4 Bar diagram of GO enrichment analysis
发芽前期抗旱型材料,PEG 模拟干旱诱导较处理组差异基因主要显著富集的KEGG 通路有苯丙素的生物合成(csv00940)、戊糖、葡萄糖醛酸转换(csv00040)和亚油酸代谢(csv00591)(图5);发芽期敏感型材料,PEG 模拟干旱诱导较处理组差异基因主要显著富集的KEGG 通路有苯丙素的生物合成(csv00940)、植物激素信号转导(csv04075)、戊糖、葡萄糖醛酸转换(csv00040)、α-亚油酸代谢(csv00592)、玉米素生物合成(csv00908)和亚油酸代谢(csv00591)等(图5)。
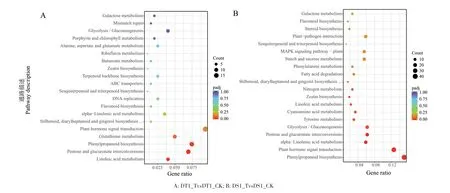
图5 KEGG 富集分析散点图Fig. 5 Point diagram of KEGG enrichment analysis
将发芽前期的抗旱型与敏感型2 个材料,PEG模拟干旱诱导与对照的差异表达基因进行关联分析,筛选到亚油酸代谢通路中3 个,分别是CsaV3_4G 023820、CsaV3_2G006420 和CsaV3_4G023810;戊糖、葡萄糖醛酸转换(csv00040)通路中4 个,分别 是CsaV3_2G025090、CsaV3_3G011280、CsaV3_3G028700 和CsaV3_2G014800;苯丙素的生物合成(csv00940)通路中6 个,分别是CsaV3_1G042480、CsaV3_6G006890、CsaV3_3G030410、CsaV3_2G03 5150、CsaV3_2G009070 和CsaV3_6G039690;以及植物激素信号转导(csv04075)通路中7 个,分别是CsaV3_2G013210、CsaV3_5G023170、CsaV3_6G 007970、CsaV3_7G027610、CsaV3_1G030250、Csa V3_2G004130 和CsaV3_3G005590(表2)。

表2 KEGG 显著富集的差异基因分析Table 2 Differential gene analysis of KEGG enrichment
2.6 基于转录组SNP分子标记开发与筛选
8 个转录组文库DT1_CK、DT1_T、DS1_CK、DS1_T、DT2_CK、DT2_T、DS2_CK、DS2_T 分别检测到外显子区域的变异数量为5 018、5 101、7 340、7 972、5 252、5 819、8 471 和8 228 个;检测到同义突变、错义突变、无义突变3 种突变类型,其中,无义突变数量分别为25、26、29、26、24、27、29和26 个,错义突变数量分别为2 114、2 166、2 898、3 173、2 132、2 375、3 315 和3 211 个,同义突变数量分别为2 905、2 936、4 441、4 810、3 118、3 443、5 175 和5 033 个;变异位点影响高的数量分别为41、43、49、48、40、47、49 和44 个(图6)。
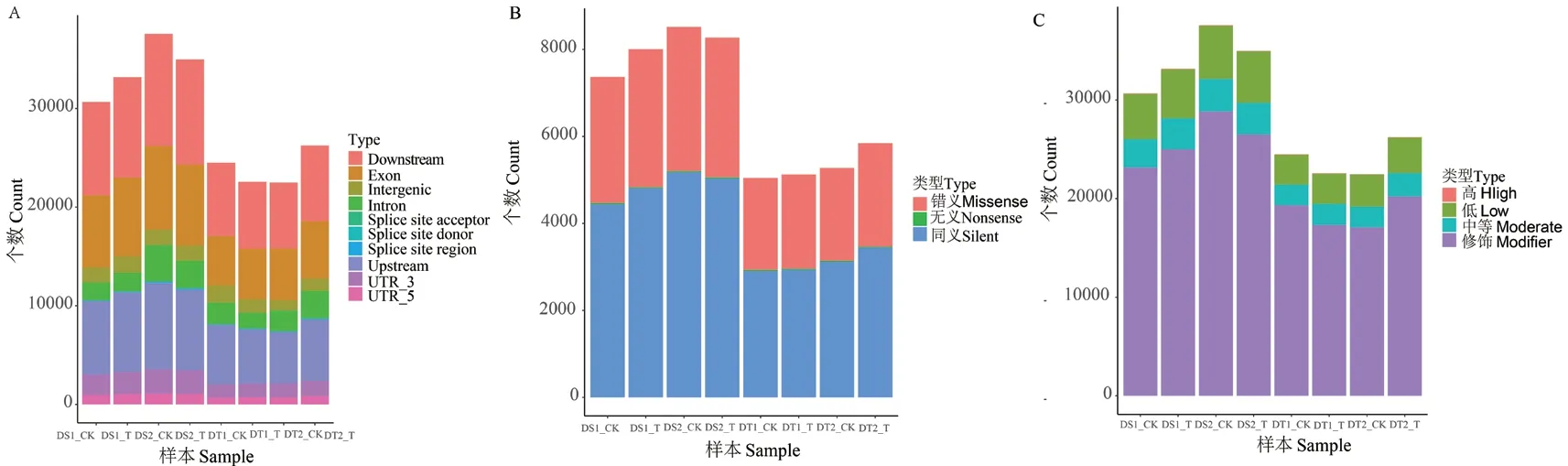
图6 SNP 变异位点统计分析Fig. 6 Statistical analysis of SNP variation sites
4 个 文 库DS1_T、DS1_CK、DT1_T、DT1_CK 检测到含SNP 变异位点基因数目分别为6 934、6 427、4 962 和5 141 个。将基于转录组中KEGG 显著富集通路上的差异基因与SNP 变异位点数据进行整合分析,筛选在抗旱关键候选基因中含SNP 变异位点的分子标记。结果(表3)显示,亚油酸代谢通路中CsaV3_4G023820、CsaV3_2G006420,以及戊糖、葡萄糖醛酸转换通路中CsaV3_2G025090 均检测到SNP 变异位点,说明该基因和SNP 分子标记可能为旱黄瓜抗旱关键基因。
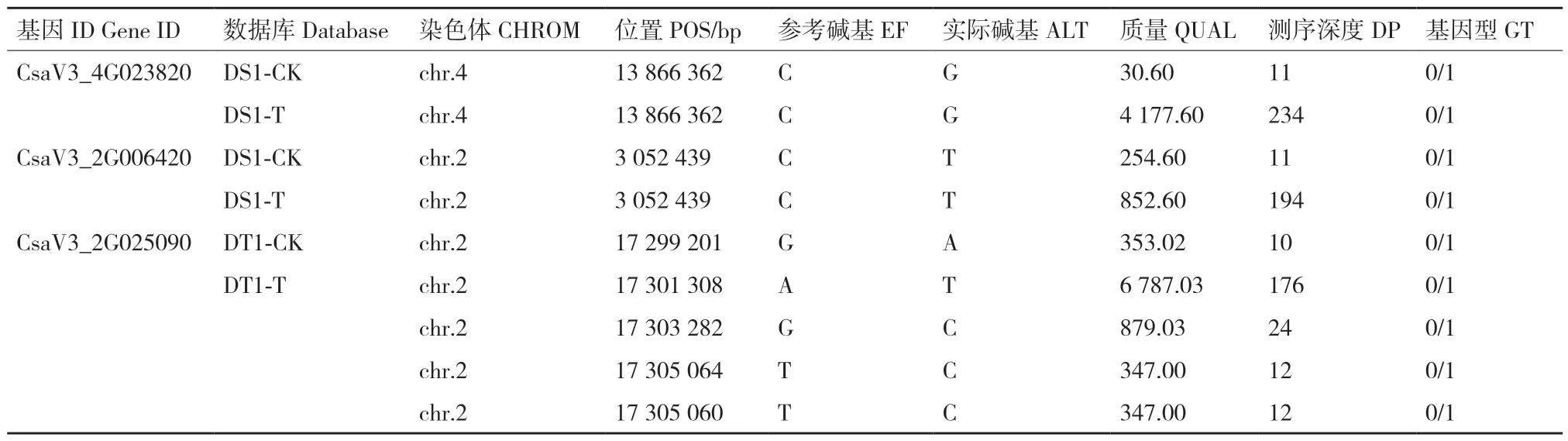
表3 SNP 变异位点与差异基因整合分析Table 3 Integration analysis of SNP variation sites and differential genes
2.7 RT‑qPCR分析
通过SNP 变异位点与差异基因整合分析筛选到CsaV3_4G023820、CsaV3_2G006420 和CsaV3_2G02 5090 共计3 个候选基因。利用Beacon Designer 7.0软件设计出RT‑qPCR 引物(表4)。RT‑qPCR 分析发现,发芽前期CsaV3_4G023820、CsaV3_2G006420在抗旱型材料PEG 模拟干旱诱导较对照上调表达,而敏感型材料PEG 模拟干旱诱导较对照下调表达;CsaV3_2G025090 在敏感型材料PEG 模拟干旱诱导较对照上调表达,而在PEG 模拟干旱诱导下抗旱型材料中未检测到相应表达量(图7)。结果说明,抗旱型材料和敏感型材料干旱响应基因表达模式不同,并且CsaV3_4G023820 和CsaV3_2G006420 可作为响应干旱的候选基因。

表4 SNP 变异位点与差异基因整合分析Table 4 Integration analysis of SNP variation sites and differential genes
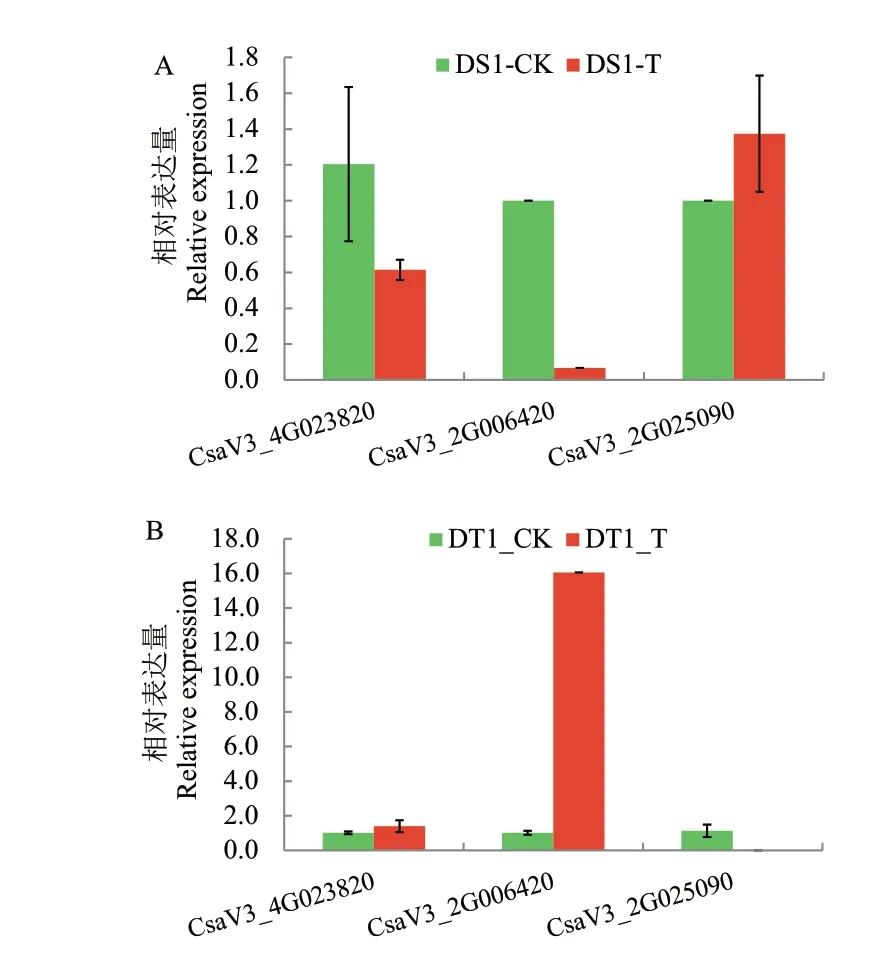
图7 候选基因RT-qPCR 分析Fig. 7 RT-qPCR analysis of candidate genes
3 讨论
高通量测序技术为园艺植物重要性状基因挖掘和分子标记开发提供了强大技术支持。近十年,基因组、转录组、蛋白组、代谢组等组学技术迅猛发展,大量相关研究和报道应运而生。如何从海量的组学数据中,整合或筛选有效的生物基因信息,将成为高效且低成本挖掘重要性状基因、开发分子标记和解析植物复杂生物学机制的一种有效手段。
转录组测序可以从mRNA 水平全面地研究植物基因功能及特定的生物学过程,利用该技术能研究植物在干旱下所有基因的表达水平情况,有助于揭示植物对干旱的应答机理[21]。张蕾等[22]以甘蓝型油菜‘湘油15’为试材,利用转录组分析干旱胁迫下差异基因和关键代谢通路。张婷茹等[23]利用转录组测序技术分析了角果碱蓬干旱胁迫下差异表达基因,鉴定到426 个上调表达基因和294 个下调表达基因。卢坤等[24]利用转录组测序技术鉴定到甘蓝型油菜叶片干旱胁迫响应相关基因,从转录水平解析了油菜适应干旱环境的分子机制。王玉斌等[25]以高粱干旱敏感材料和耐旱材料为试材,利用转录组技术鉴定到耐旱材料4 032 个差异基因,敏感材料8 928 个差异基因,并且推测苯丙素生物合成是高粱耐旱性的主要KEGG 通路。本研究也采用以上转录组测序技术方法,以抗旱型和敏感型旱黄瓜为试材,鉴定到PEG 模拟干旱诱导下抗旱型旱黄瓜中1 072 个差异表达基因,敏感型旱黄瓜中1 337 个差异表达基因,并挖掘到亚油酸代谢(csv00591)、戊糖、葡萄糖醛酸转换(csv00040)、苯丙素的生物合成(csv00940)和植物激素信号转导(csv04075)等4 个KEGG 显著富集通路。其中苯丙素的生物合成(csv00940)通路是在很多物种中广泛被鉴定到为干旱响应通路,这与高粱研究结果相一致[25]。
基于转录组数据开发旱黄瓜SNP 分子标记,可为旱黄瓜建立一种高效和低成本的分子标记开发方法。本研究以8 个转录组数据库为基础开发SNP 分子标记,结合4 个KEGG 富集通路中20 个关键候选基因定位信息,筛选出9 个旱黄瓜抗旱性状相关SNP 分子标记。值得说明的是,亚油酸代谢通路中CsaV3_4G023820、CsaV3_2G006420 在抗旱型和敏感型两类材料中,检测到变异位点信息完全一致,均为“C”突变成“G”和“C”突变成“T”,而CsaV3_2G025090 检测到变异位点信息在抗旱型和敏感型两类材料中不完全一致,进一步暗示着亚油酸代谢通路及CsaV3_4G023820、CsaV3_2G006420 在旱黄瓜抗旱性方面起重要调控作用,为旱黄瓜分子设计育种提供理论支持。
4 结论
本研究利用转录组测序鉴定到富集于亚油酸代谢(csv00591)通路、戊糖、葡萄糖醛酸转换(csv00040)通路、苯丙素的生物合成(csv00940)通路以及植物激素信号转导(csv04075)通路上20 个旱黄瓜抗旱相关基因。开发了全转录组水平的SNP 分子标记,筛选出抗旱基因CsaV3_4G023820、CsaV3_2G006420及其2 个相关SNP 分子标记。