远海梭子蟹TTK 基因测序与分析*
齐乐凤,苗贵东,2,钱庆忠,李 浩
(1.黔西南州生物遗传资源挖掘与分子育种重点实验室,贵州 兴义 562400;2.兴义民族师范学院生物与化学学院,贵州 兴义 562400)
远海梭子蟹(Portunus pelagicus) 俗称蓝花蟹,属于梭子蟹属的甲壳类动物。出肉率可观,肉质鲜甜,可供出口外汇,具有极高的发展前景[1],是我国重要的经济蟹类之一[2]。随着科技的发展,我国对远海梭子蟹的研究也迎来黄金发展时期,在远海梭子蟹的人工育苗[3-4]、生长发育[5]、生活习性[6]、繁殖[7]、生物学探究[8]、贮藏条件[9]、遗传物质核酸等都有基础的报道[10]。研究远海梭子蟹的生长发育、生物习性以及生物调控分子机制,不仅从生物学角度,而且从遗传机制角度,解释其发育规律,从而为远海梭子蟹资源的可持续开发以及利用,提供重要的遗传学基础。
苏氨酸酪氨酸激酶(Threonine and Tyrosine Kinase,TTK) 是一种双特异性激酶,与细胞增殖有关,在细胞的有丝分裂过程中对染色体的排列组合具有促进作用[11]。其基因转录产物是重要的转录因子,对生物体的发育、分化、性行为等有着调控作用[12],当分裂出错时,TTK 就起着纠正的功能,直到细胞正常分裂结束[13]。当前TTK 基因的研究报道丰富,但却并无远海梭子蟹TTK 基因的研究报道,因此开展远海梭子蟹TTK 基因基础研究具有较高的价值。
1 远海梭子蟹TTK 基因测序与分析的实验材料与方法
实验所用远海梭子蟹个体采自广西北海海域,按照蟹类样品取样规范,经处理后保存备用。在实验室远海梭子蟹cDNA 文库中找到TTK 基因的基因全长序列,利用软件Premier 5.0 设计上游、下游引物,通过PCR 反应进行验证口基因序列,从而进行分析。
2 远海梭子蟹TTK 基因测序与分析的实验结果
2.1 远海梭子蟹DNA 的提取及测序验证
使用天根海洋动物组织基因组DNA 提取试剂盒提取的远海梭子蟹基因组DNA 用于后续实验。由于远海梭子蟹TTK 基因序列长度为3260 bp,因此将其序列分为多个部分进行双向测序。对筛选得到的基因组DNA 通过引物进行PCR 扩增,琼脂糖凝胶电泳后经凝胶成像系统得到PCR 产物的凝胶电泳结果,见图1。将目标产物送生工生物工程(上海) 股份有限公司进行测序。测序结果返还后进行序列组装与拼接,与实验室已有TTK 基因序列进行比对,测序比对结果见图2。由图2 可知,比对一致性为99.45%,表明三代测序结果质量可靠,可用于后续基因结构与功能预测分析。
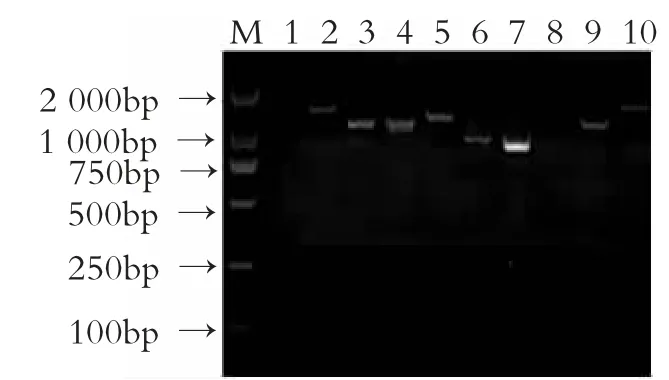
图1 PCR 产物的凝胶电泳结果

图2 测序比对结果
2.2 生物信息学分析
2.2.1 序列比对
将拼接序列上传至NCBI Blast 进行比对,确定结果的可信度并使用NCBI ORF finder 确定TTK 基因的开放阅读框,结果表明TTK 基因的开放阅读框长度为1752 bp,起始密码子ATG,终止密码子TGA,共编码了583 个氨基酸。运用DNAMAN 软件对CDS 序列进行碱基组成分析,分析显示TTK基因的CDS 序列中碱基A(26.0%)、T(18.3%)、G(27.6%)、C(28.2%),其中GC 含量超过50%,序列结构稳定性高。
2.2.2 氨基酸序列相似性比较及进化树
利用MegAlign 工具对TTK 基因进行氨基酸序相似性分析,远海梭子蟹蛋白质序列同源比对统计结果见图3。分析得出三疣梭子蟹、斑节对虾、南美白对虾的氨基酸序列相似性都超过90%,相似性高;四爪螯蟹、桑蚕、日本对虾、中国对虾、克氏原螯虾的氨基酸序列相似性较低,在80%以下。运用MEGA11 软件作出远海梭子蟹TTK 基因系统进化树,见图4。分析得出远海梭子蟹与三疣梭子蟹亲缘关系最近,与中华绒螯蟹、角藻亲缘关系较近,与中国对虾、日本对虾、斑节对虾、四爪螯蟹、南美白对虾、克氏原螯虾亲缘关系较远,符合传统进化规律。

图3 远海梭子蟹蛋白质序列同源比对统计结果
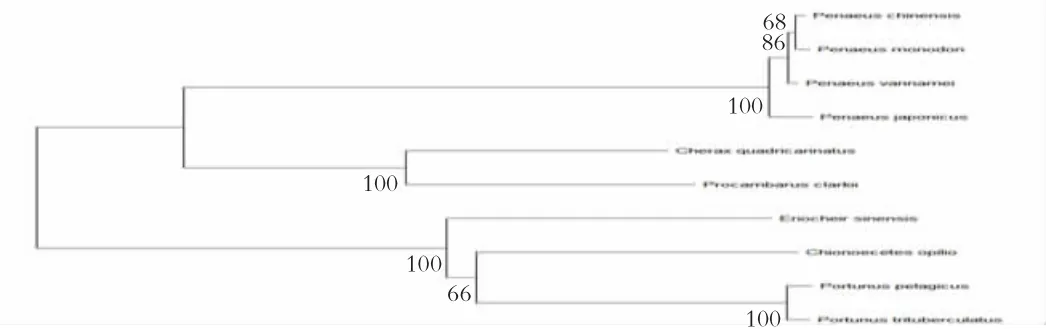
图4 远海梭子蟹TTK 基因系统进化树
2.2.3 TTK 基因蛋白表达预测分析
通过ExPASy-ProtParam 在线工具分析可知,TTK 序列带负电荷的氨基酸数(Asp+Glu) 85;带正电荷的氨基酸数(Arg+Lys) 61;氨基酸组成中丝氨酸占比最多,为10.1%;色氨酸占比最少,仅有0.5%。
运用TMHMM2.0 对TTK 蛋白质进行跨膜区预测,见图5,结果显示该蛋白质不存在跨膜区。运用SignaIP6.0 在线软件对TTK 蛋白质进行信号肽预测,见图6,结果显示TTK 蛋白质不存在信号肽。
图5 TTK 蛋白质跨膜区预测
图6 TTK 蛋白质信号肽预测
通过NetPhos3.1 在线软件预测远海梭子蟹TTK蛋白质发现,预测结果显示该蛋白质的磷酸化位点有64 个,其中丝氨酸磷酸化位点47 个,苏氨酸磷酸化位点17 个,见图7。

图7 TTK 蛋白质磷酸化位点预测
使用在线工具SOPMA,对TTK 蛋白质进行二级结构预测,见图8。结果表明α-螺旋、延伸链、β-折叠、无规则卷曲占比分别为30.87%、7.38%、6.00%、55.75%。使用SWISS-MODEL 在线工具进行三级结构的建模分析得出TTK 蛋白质的GMQE值为0.10,QMEAN 值为-3.17。TTK 蛋白三级结构预测显示该蛋白的空间结构主要由α-螺旋和无规则卷曲组成,见图9。
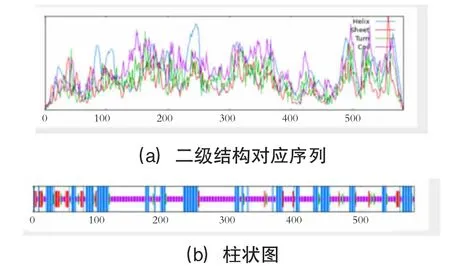
图8 TTK 蛋白质二级结构预测结果
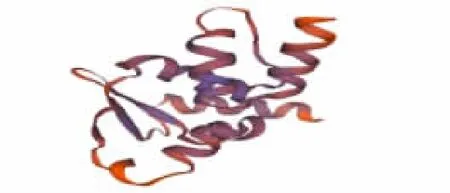
图9 TTK 蛋白质三结构预测
3 讨论与结论
本实验对远海梭子蟹中的TTK 基因进行基础研究。得到TTK 基因CDS 序列为1752 bp,CDS 同源性分析得出与三疣梭子蟹的同源性最高;进化树分析得到远海梭子蟹与三疣梭子蟹的同源性最高,与中国对虾、日本对虾、斑节对虾、四爪螯蟹、南美白对虾、克氏原螯虾亲缘关系较远,符合传统进化规律;序列编码氨基酸583 个,其中负电荷的氨基酸数(Asp+Glu) 85,带正电荷的氨基酸数(Arg+Lys) 61,氨基酸组成中丝氨酸占比最多,为10.1%,色氨酸占比最少,仅有0.5%;TTK 蛋白质不存在跨膜区和信号肽;序列的理论等电点5.19,氨基酸平均亲水性-0.808,是亲水蛋白质;序列中磷酸化位点64 个,其中丝氨酸磷酸化位点47 个,苏氨酸磷酸化位点17 个;蛋白质二级结构中α-螺旋、延伸链、β-折叠、无规则卷曲占比分别为30.87%、7.38%、6.00%、55.75%。本实验通过高通量测序对远海梭子蟹TTK 基因序列进行验证和基础分析,对其蛋白质的结构和功能进行了初步的验证和分析,为进一步研究该基因的功能奠定了基础并提供了一定参考,对甲壳类生物TTK 基因的后续研究具有较高意义。

