花鲈半乳糖凝集素基因家族的鉴定及在不同环境因子胁迫下表达响应❋
郑 圆, 温海深, 李吉方, 方 秀, 王灵钰 ,张 冲, 陶泽鑫, 张永航, 李 昀❋❋
(1. 海水养殖教育部重点实验室(中国海洋大学), 山东 青岛 266003; 2. 福建省花鲈育种重点实验室, 福建 福鼎 355200)
凝集素(Lectin)是一类具有广泛生物学功能的碳水化合物结合蛋白,在病毒、细菌和动植物中均广泛分布,其介导的蛋白质-碳水化合物间的相互作用在先天免疫如病原体识别中扮演着重要角色[1-3]。半乳糖凝集素(Galectins)是凝集素超家族中的一个较为保守的β-半乳糖苷结合蛋白家族,其特点为具有一个保守、可特异性识别β-半乳糖苷的碳水化合物识别结构域(Carbohydrate-recognition domain,CRD),但缺少信号肽,是一类非经典的分泌型蛋白质[4]。根据CRD 的结构和数量特征,人的Galectins可划分为三种类型:原型(Prototype),仅含一个CRD,多以单体或同型二聚体的形式存在,包括Galectin-1(LGALS1)、Galectin-2(LGALS2)、Galectin-5(LGALS5)、Galectin-7(LGALS7)、Galectin-10(LGALS10)、Galectin-11(LGALS11)、Galectin-13(LGALS13)、Galectin-14(LGALS14)、Galectin-15(LGALS15)、Galectin-16(LGALS16)、Galectin-17(LGALS17)以及Galectin-related protein(GRP)[5-7];串联重复型(Tandem repeat-type),为单体凝集素,通过短链多肽连接两个CRD,包括Galectin-4(LGALS4)、Galectin-6(LGALS6)、Galectin-8(LGALS8)、Galectin-9(LGALS9)和Galectin-12(LGALS12);嵌合型(Chimeric type),同为单体凝集素,在N-末端具有富含脯氨酸和甘氨酸的结构域,在C-末端含有CRD,其连接到一个非凝集素氨基末端区域,可形成五聚体结构,仅包括Galectin-3(LGALS3)[8]。
诸多研究表明Galectins可参与多种生物学过程,如癌细胞的转化、迁移、侵袭与转移[9],炎症和免疫反应[10],以及个体发育等[11]。在哺乳动物中,Galectins广泛分布于免疫器官和免疫细胞中,参与免疫反应和肿瘤发生[12]。近年来,越来越来多的研究表明水生生物中Galectins可作为抗菌和非自我识别分子在先天性免疫中发挥着至关重要的作用[13]。例如,在斑马鱼(Daniorerio)中,当传染性造血器官坏死病病毒(Infectious hematopoietic necrosis virus,IHNV)感染时,可在细胞外观察到Galectins以碳水化合物结合的方式直接与上皮细胞表面的IHNV的糖基化包膜和多糖相互作用,从而显著降低病毒的粘附[14]。当尼罗罗非鱼(Oreochromisniloticus)感染无乳链球菌(Streptococcusagalactiae)5 d后,LGALS8在脾脏中的表达显著上调,表明其参与了抗细菌感染的免疫反应[15]。条石鲷(Oplegnathusfasciatus)的重组蛋白Gal2具有血凝性,对乳糖和半乳糖表现出亲和力,可凝集和结合潜在致病菌和纤毛虫,增强免疫能力[16]。
此外,Galectins在响应环境胁迫中发挥着重要作用,被认为是细胞应激反应的生物标志物[17]。例如,在体外和体内试验中,证明了小鼠LGALS3在缺氧和营养匮乏的微环境应激中可通过上调表达来提高胶质瘤细胞的存活能力[18]。在氧化胁迫(30% H2O2,37 ℃培养16 h)下,管圆线虫(Angiostrongyluscantonensis)的LGALS10的表达水平显著增加,且体外原核表达的管圆线虫GAL10蛋白可在小鼠小胶质细胞系中激活其小鼠小胶质细胞来响应氧化胁迫[19]。通过RNA干扰技术来沉默中华蜜蜂(Apisceranacerana)LGALS1的表达,发现在氧化应激下相关抗氧化基因出现显著差异表达,表明LGALS1参与了氧化应激响应,能够提高抗氧化损伤的抵抗力[20]。鱼类作为生活在水环境中的低等脊椎动物,易遭受多种类型的环境因子胁迫,养殖过程中常见的有温度、盐碱度、溶解氧等[21]。胁迫会打破个体与环境间的平衡状态,导致鱼体生理机能紊乱,免疫防御能力下降,严重时可导致死亡[22]。因此,研究Galectins对于提高机体免疫能力和解析环境胁迫应答机制具有重要意义。自从在电鳗(Electrophoruselectricus)的发电器官中发现了第一个硬骨鱼的半乳糖凝集素-电凝集素(Electrolectin)以来[23],Galectins在许多鱼类中被鉴定出来。到目前为止,分别在大菱鲆(Scophthalmusmaximus)、斑点叉尾鮰(IctalurusPunctatus)和红鳍东方鲀(Takifugurubripes)中完成了对Galectin基因家族的鉴定[24-26],然而这些研究多集中于该家族在免疫应答功能方面的研究,对其在响应环境因子胁迫中的功能机制尚未见报道。
花鲈(Lateolabraxmaculatus)又称中国花鲈,俗称海鲈、寨花、七星鲈等,隶属于鲈形目(Perciformes)鮨科(Serranidae)花鲈属(Lateolabrax)。其广泛分布于中国、越南与韩国沿海地区,具有营养价值高、口感好的特点,是中国养殖产量高的海产经济鱼类之一[27-28]。随着近年来养殖规模的不断扩大,花鲈养殖被愈来愈多的环境因素所制约,严重影响了其养殖产量和经济效益。本研究对花鲈Galectin基因家族进行了全基因组水平的系统鉴定,并开展了拷贝数、系统发育、共线性及基因结构特征等分析。此外,利用转录组数据绘制了花鲈Galectin基因家族的组织表达图谱,并分析了其在低氧、盐度、碱度及高温胁迫下的基因表达特征。本研究结果为阐明在多种环境因子胁迫下花鲈Galectins的功能机制提供理论基础,并为花鲈抗逆性状的良种选育工作的开展提供了候选分子。
1 材料与方法
1.1 花鲈Galectin基因的全基因组鉴定和序列分析
从NCBI(http: //www.ncbi.nlm.nih.gov/)数据库中检索下载人(Homosapiens)、斑马鱼和尼罗罗非鱼Galectin家族全部成员的氨基酸序列,通过TBtools软件的TBLASTN(1×10-5)方法对花鲈全基因组数据库(PRJNA408177)和转录组数据库(SRR4409341和SRR4409397)进行比对,初步获得花鲈候选Galectin基因的核苷酸序列。通过ORF Finder(https: //www.ncbi.nlm.nih.gov/orffinder/)工具对花鲈候选Galectin的氨基酸序列进行预测,在NCBI非冗余蛋白数据库(NR)中通过BLASTP工具对获得的预测序列进行确认。此外,利用ExPASy Prot-Param(https: //web.expasy.org/protparam/)工具预测Galectin蛋白的分子量(Molecular Weight, MW)和理论等电点(isoelectric point, pI),通过DTU Health Tech(https: //www.healthtech.dtu.dk/english)中的NetPhos-3.1工具对Galectin蛋白的磷酸化位点进行预测。
1.2 花鲈Galectin基因系统发育和同源性分析
为构建系统发育树,在NCBI数据库中检索下载人、小鼠(Musmusculus)、鸡(Gallusgallus)、牛(Bostaurus)、斑马鱼、尼罗罗非鱼、大黄鱼(Larimichthyscrocea)、大西洋鳕鱼(Gadusmorhua)、红鳍东方鲀和斑点叉尾鮰的Galectin氨基酸序列。通过MEGA11.0软件(https: //www.megasoftware.net/)中ClustalW算法(默认参数)进行多重序列比对,运用Neighbor-Joining(NJ)法和Jones-Taylor-Thornton(JTT)模型构建系统发育树,每个分支上的bootstrap值重复1 000次[29]。利用在线网站ITOL(http: //itol.embl.de/)对发育树进行可视化。
通过共线性分析来进一步确定基因的同源性。使用多重共线性扫描工具包(MCScanX)的默认参数[30]分析花鲈与斑马鱼、尼罗罗非鱼物种间的共线性关系,利用Circos[31]对花鲈基因组内的共线性基因进行可视化,并将Galectin基因定位到花鲈染色体上。斑马鱼(GRCz11)和尼罗罗非鱼(UMD_NMBU)参考基因组数据从NCBI数据库中下载,物种间的共线性分析通过Multiple Synteny Plot工具进行(https: //github.com/CJ-Chen/TBtools)。
1.3 花鲈Galectin基因的基因结构与保守基序分析
通过NCBI的CDD数据库(https: //www.ncbi.nlm.nih.gov/cdd/)预测花鲈Galectin蛋白的保守结构域。利用在线网站MEME(https: //meme-suite.org/meme/tools/meme)对花鲈Galectin蛋白的保守基序(Motif)进行分析。将上述所获结果通过TBtools软件进行可视化[32]。
1.4 花鲈Galectin基因的组织表达谱分析
为探究花鲈Galectin基因在不同组织中的表达模式,从NCBI中下载1龄花鲈7个组织的RNA-seq数据集(肝-SRR7528886、鳃-SRR7528883、脾-SRR752888、胃-SRR7528884、脑-SRR7528887、精巢-SRR7528885、卵巢-SRR2937376)。下载原始测序数据(Raw data)后,通过FastQC v0.11.9软件进行质检(http://www.bioinformatics.babraham.ac.uk/projects/fastqc/),利用trimmomatic v0.39对原始数据进行过滤,去除接头和低质量序列,获得高质量clean reads[33]。使用Hisat2 v2.2.1的默认参数[34]将clean reads比对到花鲈参考基因组(PRJNA407434)中,利用samtools 1.6进行排序[35],通过StringTie v2.1.7进行基因表达量标准化,结果以FPKM(Fragments per kilobase of transcript per million fragments mapped)的形式表示[36]。取log2(FPKM+1),通过TBtools软件绘制Galectin基因的表达量热图。
1.5 环境因子胁迫试验
为探究花鲈Galectin基因在不同环境因子胁迫下的表达特征,对已有的4个环境胁迫试验:低氧、盐度、碱度及高温胁迫处理后测得的转录组数据库进行基因表达模式分析。胁迫试验流程如下文所示。
1.5.1 低氧胁迫试验 试验前将60尾花鲈(体质量为(178.25±18.56) g,体长为(48.76±4.26) cm)进行2周暂养(溶解氧为(6.89±0.25) mg/L)。试验开始前30 min向3个400 L平行圆桶连续快速注入氮气降低溶氧量,当溶氧量下降并稳定在预实验所获临界值(1.10±0.14) mg/L(溶氧仪YSI DO200)时,将花鲈迅速转移至3个圆桶内(每桶随机均分20尾)进行低氧胁迫试验。试验过程中,通过气石输入空气,使花鲈呼吸消耗的氧气与通入的空气维持平衡,保持溶解氧相对稳定。于胁迫前(0 h)及胁迫后3、6和12 h共4个时间点各随机选取9尾花鲈采集鳃组织样品(每个平行圆桶随机采集3尾)。
1.5.2 盐度胁迫试验 试验前将108尾花鲈(体质量为(127.35±15.31) g)随机均分于9个120 L平行方桶进行一周盐度驯化(盐度为30)。暂养结束后将其中3个方桶盐度于12 h内逐渐调整至0(设为淡水FW组),3个方桶盐度调至12(设为半咸水BW组),剩余3个方桶盐度保持30(设为海水SW组)。试验周期为30 d,期间其余试验条件保持不变,每日喂食一次。养殖结束后每组各取18尾鱼的鳃组织样品(每个平行方桶各随机采集6尾)。
1.5.3 碱度胁迫试验 试验前将36尾1龄花鲈(体质量为(140.32±2.56) g)进行2周暂养,然后立即将其转移到3个400 L装有碱度水体的平行圆桶(碳酸盐浓度为(18.0±0.2) mmol/L,pH=9.0±0.2)进行急性碱度胁迫试验。碱度水体由NaHCO3(12.8 mmol/L)和Na2CO3(2.6 mmol/L)配制而成,在试验前对水体进行24 h曝气处理。于胁迫前(0 h)及胁迫后12、24和72 h共4个时间点各采集9尾鱼的鳃组织样品(每个平行圆桶各随机采集3尾)。
1.5.4 高温胁迫试验 试验前将60尾花鲈(体质量为(38.96±2.01) g,体长为(13.33±0.24) cm)随机均分于3个120 L平行方桶中进行2周暂养。试验开始前3个平行方桶水温为25 ℃,随后以1 ℃/h升温速率升至目标温度32 ℃并保持至试验结束。分别于0 h(升温前)、10 h (热应激后3 h)、13 h (热应激后6 h)、19 h(热应激后12 h)以及31 h(热应激后24 h)共5个时间点各采集9尾鱼的肝组织样品(每个方桶内各随机采集3尾)。
1.6 花鲈Galectin基因在环境因子胁迫下的表达模式分析
采用TRIZOL法(Invitrogen,美国)对以上样品总RNA进行提取,并用NanoDrop 2000核酸定量仪(Thermo Scientific,美国)和琼脂糖凝胶电泳对总RNA进行质量检测。质检合格后,低氧胁迫试验中将来自同一圆桶的3 尾鱼的 RNA 样本进行等质量混合,共构建12个测序文库(4个采样时间点×3个重复),利用Illumina HiSeq 4000测序平台对各文库进行测序(PRJNA408177);盐度胁迫试验中将来自同一方桶的3 尾鱼的 RNA 样本进行等质量混合,共构建9个测序文库(3个试验组×3个重复),利用Illumina NovaseqTM6000测序平台对各文库进行测序(PRJNA515986);碱度胁迫试验中将来自同一圆桶的3 尾鱼的 RNA 样本进行等质量混合,共构建12个测序文库(4个采样时间点×3个重复),利用Illumina HiSeq X Ten 测序平台对各文库进行测序(PRJNA611641);高温胁迫试验中将来自同一方桶的3尾鱼的RNA样本进行等质量混合,共构建15个测序文库(5个采样时间点×3个重复),利用Illumina NovaseqTM6000测序平台对各文库进行测序(未发表数据)。
下载原始测序数据后,参考1.4中RNA-seq数据分析流程,得到花鲈特定组织在4种不同环境因子胁迫下Galectin基因的FPKM值。其中在clean reads比对到花鲈参考基因组后,使用featurecount v2.0.1统计基因reads数[37],随后将原始计数数据(Raw counts data)导入DESeq2 v1.30.1(http: //www.bioconductor.org/packages/release/bioc/html/DESeq2.html)进行差异表达统计分析,以P<0.05表示差异显著。取log2(FPKM+1),通过TBtools软件展示Galectin基因的聚类表达热图。
2 结果与分析
2.1 花鲈Galectin基因鉴定及序列分析
本研究在花鲈基因组中共鉴定出14个Galectin基因,根据现有的分类方式进一步分为3类(见表1):(1)原型,包括LGALS1、LGALS2a、LGALS2b、LGALS17、GRPa、GRPb和GRPc;(2)串联重复型,包括LGALS4、LGALS8a、LGALS8b和LGALS9;(3)嵌合型,包括LGALS3a、LGALS3ba和LGALS3bb。各Galectin基因序列的ORF长度范围为396~1221 bp,编码蛋白质的氨基酸数目为131~406,预测分子量为15.90~44.86 kDa,等电点为4.69~9.69,磷酸化位点个数为8~41。以上基因信息及Genbank序列号在表1中列出。

表1 花鲈Galectin基因家族成员的序列特征Table1 Summary ofsequence characteristics of Galectin gene family members in spotted sea bass
2.2 花鲈Galectin基因系统发育分析
为进一步验证花鲈Galectin基因注释的准确性,选取人、鼠、鸡、牛、斑马鱼、尼罗罗非鱼、大黄鱼、大西洋鳕鱼、红旗东方鲀、斑点叉尾鮰以及花鲈Galectin氨基酸序列构建系统发育树。如图1所示,根据系统发育关系,Galectin基因可聚为8个分支,分别为LGALS1、LGALS2、LGALS3、LGALS4、LGALS8、LGALS9、LGALS17和GRP。其中硬骨鱼同种Galectin基因聚为一枝,后与其他高等脊椎动物的相应基因共聚为一枝。花鲈Galectin基因与在分类地位上亲缘关系较近的硬骨鱼聚为一枝,聚类效果良好(见图1)。

(分支节点上的数字表示bootstrapping值,红色圆圈表示花鲈Galectin基因,不同颜色圆环表示Galectin基因不同的亚家族。Numbers on node denote the bootstrapping values, red circles denote Galectin genes of spotted sea bass, and different colored rings denote different subfamilies of Galectin genes.)图1 Galectin基因系统发育树Fig.1 Phylogenetic tree of Galectin genes
2.3 花鲈Galectin基因拷贝数分析
对Galectin基因在人、小鼠、鸡、牛、斑马鱼、尼罗罗非鱼、大黄鱼、大西洋鳕鱼、红鳍东方鲀、斑点叉尾鮰和花鲈共11种脊椎动物中的拷贝数进行了比较(见表2)。结果显示,不同物种的Galectin基因数量从8到16不等。具体而言,人和斑马鱼有16个Galectin基因,为检测物种中数量最多,鸡的数量最少,只有8个。LGALS7和LGALS12为哺乳动物所特有。在硬骨鱼中,均含有2个LGALS8基因和3个GRP基因,而高等脊椎动物中均各只含一个。此外,LGALS2、LGALS3在硬骨鱼的基因组也普遍存在多拷贝现象,而高等脊椎动物仅含有一个基因拷贝。值得一提的是,尼罗罗非鱼的Galectin基因数量与本研究中花鲈鉴定得到的相同,这也印证了两个物种在分类地位上的相近。
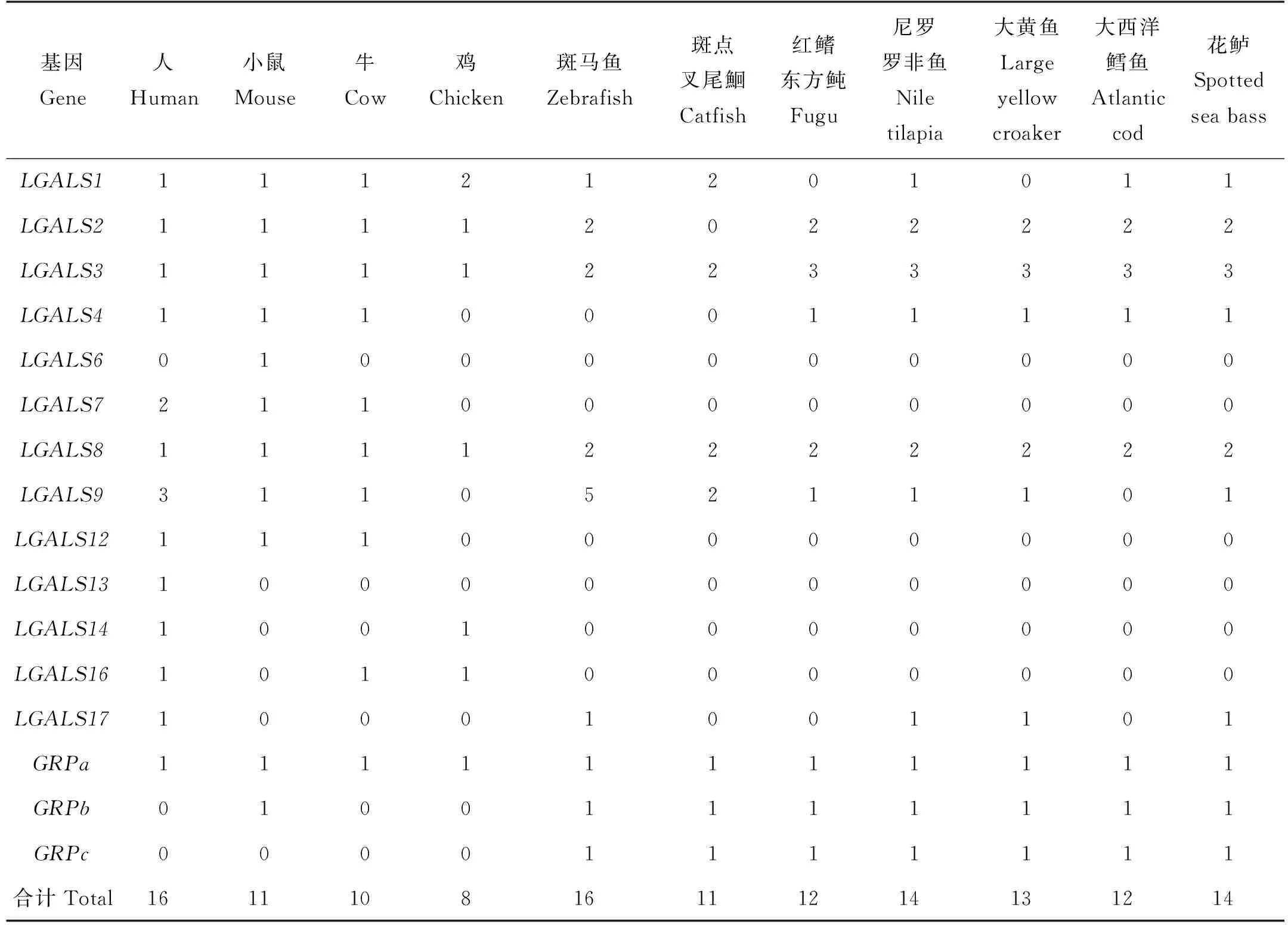
表2 几种代表性脊椎动物中Galectin基因的拷贝数Table 2 Copy numbers of Galectin genes among several representative vertebrate species
2.4 花鲈Galectin基因染色体分布及共线性分析
通过Circos对花鲈Galectin基因在染色体上的分布进行展示。如图2所示,花鲈的Galectin基因分别分布于chr1、chr3、chr10、chr11、chr17、chr22、chr24和未能挂载在染色体的scaffold_158上。其中,chr22上含有3个Galectin基因:LGALS3ba、LGALS8b和GRPb,是所有染色体中最多的。chr1、chr3、chr10和chr17均包含2个Galectin基因,chr11、chr24和scaffold_158仅含1个Galectin基因。
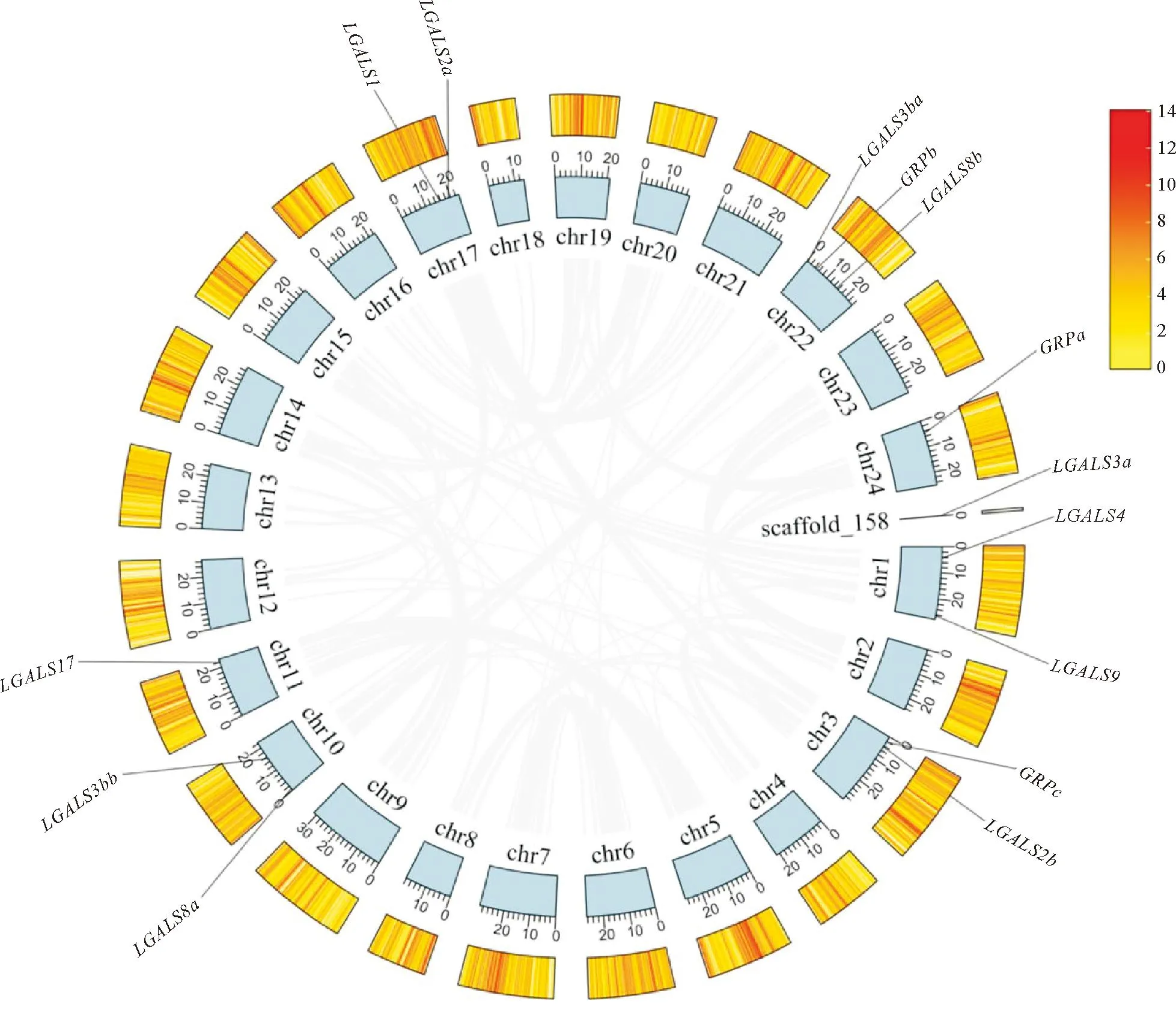
(灰线表示花鲈基因组的共线性区块,蓝色方块表示花鲈染色体,外环表示基因密度。图示的标尺中“0~14”表示基因组中基因数量与基因组长度的比值。 The gray line denotes the genome collinearity block of spotted sea bass, the blue square denotes the chromosome of spotted sea bass, and the outer ring denotes the gene density. The ratio of gene number to genome length is presented by “0~14” of scale legend.)图2 花鲈Galectin基因的染色体分布和共线性关系Fig.2 Chromosomal distribution and homologous relationships of Galectin genes in spotted sea bass
如图3所示,在尼罗罗非鱼与花鲈的共线性分析中,共得到22个尼罗罗非鱼和花鲈的Galectin基因有直系同源关系的共线性区块,分别位于尼罗罗非鱼的chr4、chr6、chr13、chr14、chr15和chr19以及花鲈的chr1、chr3、chr10、chr11、chr17和chr22染色体上。而在斑马鱼与花鲈的共线性分析中,共得到11个斑马鱼和花鲈的Galectin基因有直系同源关系的共线性区块,分别位于斑马鱼的chr1、chr3、chr6、chr13、chr15和chr17以及花鲈的chr1、chr3和chr22染色体上。
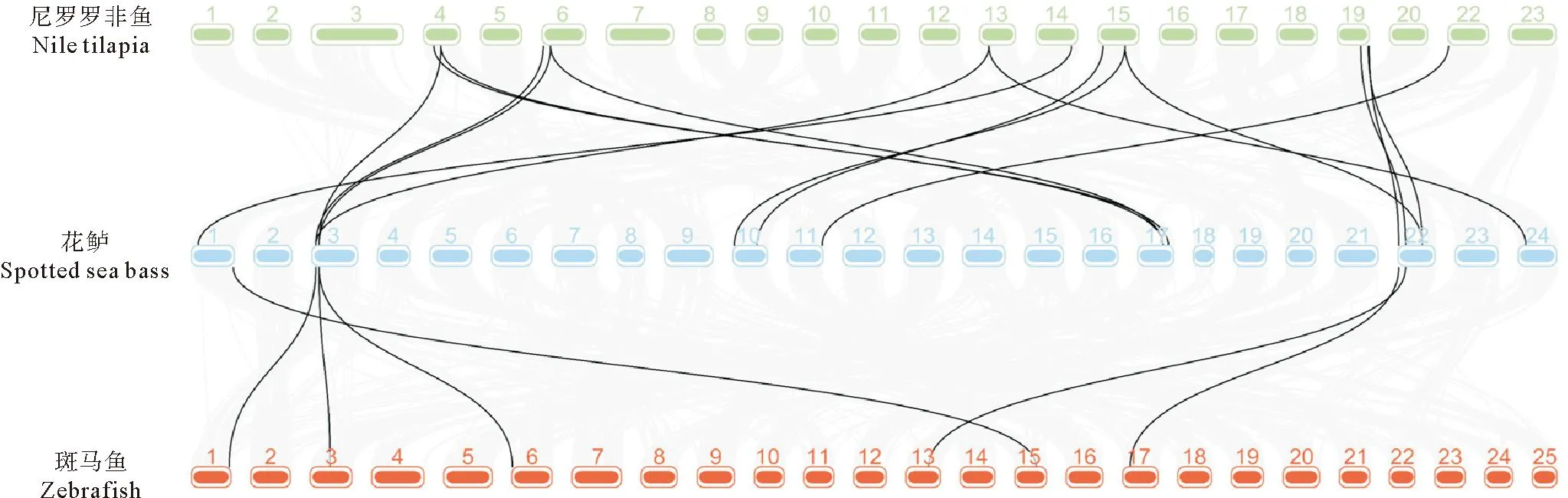
(灰线表示物种间的共线性区块,黑线表示花鲈Galectin基因的共线性区块,不同颜色方块表示不同物种的染色体。The gray lines denote the collinearity block between species, the black lines denote the collinearity block of spotted sea bass Galectin genes, and different colored ring squares denote the chromosomes of different species.)图3 尼罗罗非鱼、斑马鱼和花鲈Galectin基因的共线性分析Fig.3 Synteny analysis of Galectin genes among Nile tilapia, zebrafish and spotted sea bass
2.5 花鲈Galectin基因结构分析
图4对花鲈Galectin基因的预测保守结构域(Domain)和保守基序(Motif)进行可视化。利用CDD数据库预测得到6种类型的保守结构域,包括GLECT、Gal-bind_lectin、PRK07764超家族、Ig超家族、IG_like和Cytadhesin_P30超家族结构域。其中GLECT结构域在LGALS1、LGALS2b和LGALS9中预测获得;LGALS2a、LGALS3a、LGALS3ba、LGALS3bb、LGALS4、LGALS8a、LGALS8b、LGALS9、GRPa、GRPb和GRPc含有Gal-bind_lectin结构域;仅LGALS17预测出Ig超家族和IG_like结构域;PRK07764超家族和Cytadhesin_P30超家族结构域分别在LGALS3bb和LGALS9中预测获得。通过MEME数据库预测共获得7个Motif,其中LGALS1、LGALS2a和LGALS2b均含有Motif 2、3、4、5;LGALS3a、LGALS3ba、LGALS3bb、GRPa、GRPb和GRPc均含有Motif1、2、3、4;而LGALS4、LGALS8a、LGALS8b和LGALS9都含有这7个Motif(见图4)。

图4 花鲈Galecitn基因结构可视化分析Fig.4 Visualization analysis of Galectin genes structure of spotted sea bass
2.6 花鲈Galectin基因组织表达分析
本研究对花鲈(1龄)7个成体组织(肝、鳃、脾、胃、脑、精巢和卵巢)中Galectin基因的表达量进行了聚类分析(见图5)。如图5所示,LGALS8a、LGALS9和LGALS17在7个组织中广泛表达,且在鳃、脾、胃、肝脏和精巢中均呈现高表达,且LGALS9和LGALS17在脾中表达最高,鳃中次之。除此之外,LGLS3ba在鳃中表达量最高,胃和脾中次之,LGALS4仅在胃中高表达,GRPa仅在脾和脑中表达较高,而其余Galectin基因在上述7个组织中表达量较低。
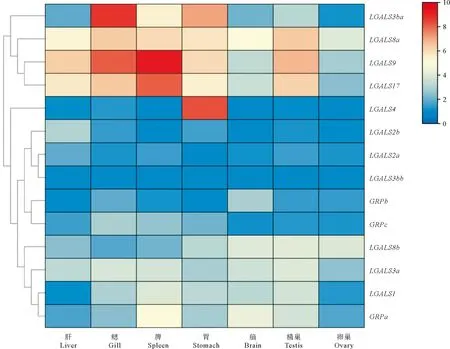
(图示的标尺中“0~10”表示log2(FPKM+1)值。The value of log2(FPKM+1) is presented by “0~10” of scale legend.)图5 花鲈Galectin基因的组织表达模式Fig.5 Expression patterns of Galectin genes in different tissues of spotted sea bass
2.7 花鲈Galectin基因在多种环境因子胁迫下的表达分析
为研究花鲈Galectin基因在多种环境胁迫下的表达模式,对多个胁迫试验的特定组织的转录组数据进行分析,结果表明,在低氧胁迫下的花鲈鳃组织中LGALS3a、LGALS17、GRPb和GRPc显著差异表达。
其中,LGALS17的表达量呈短暂降低的趋势,且在3 h时显著降低(为0 h的62%),并在12 h恢复到了0 h的水平;LGALS3a、GRPb和GRPc的表达量呈短暂升高的趋势,LGALS3a在6 h时表达量显著升高(为0 h的1.11倍),并在12 h恢复到了0 h的水平;GRPb在12 h时表达量显著升高(为0 h的1.71倍);GRPc在3和6 h时表达量显著升高(分别为0 h的1.34和1.50倍)。而LGALS1、LGALS2a、LGALS2b、LGALS3bb、LGALS4、LGALS8b和GRPa在整个过程中一直保持较低的表达水平 (FPKM值在各时间点均小于10且无显著差异);LGALS3ba、LGALS8a和LGALS9虽然表达水平较高(FPKM值在各时间点均大于70),但在各时间点间不存在显著差异(见图6A)。
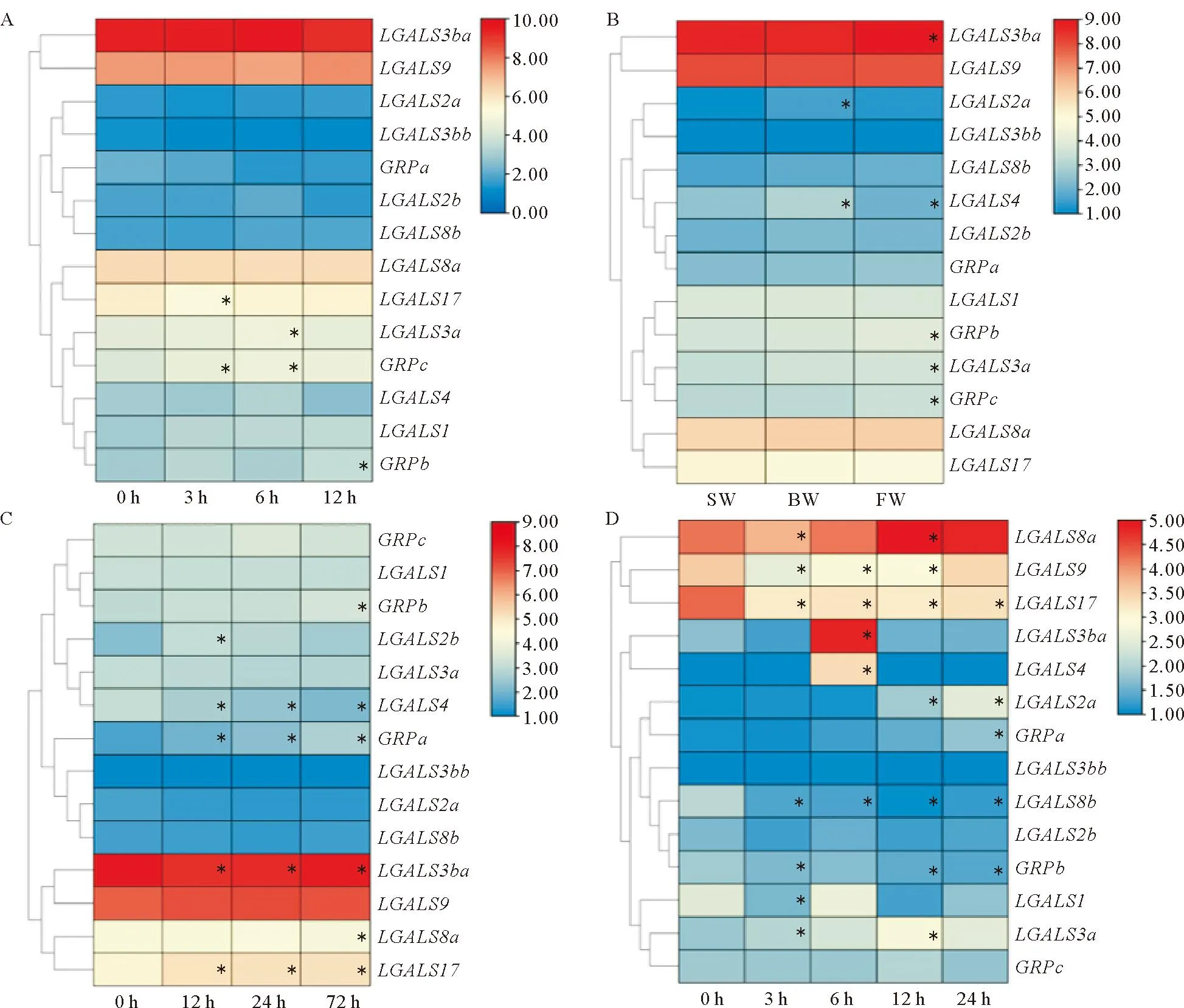
(A:低氧胁迫下鳃中的表达模式;B:不同盐度胁迫下鳃中的表达模式;C:碱度胁迫下鳃中的表达模式;D:高温胁迫下肝中的表达模式。图示的标尺表示log2(FPKM+1)值; 表示显著差异(P<0.05)。A: Expression patterns of gill under hypoxia stress; B: Expression patterns of gill under different salinity stresses; C: Expression patterns of gill under alkalinity stress; D: Expression patterns of liver under heat stress. The value of log2(FPKM+1) is presented by the scale legend; denotes the significant differences (P<0.05).)图6 花鲈Galectin基因在多种环境胁迫下的表达模式Fig.6 Expression patterns of Galectin genes in spotted sea bass under various environmental stresses
在不同盐度环境下的花鲈鳃组织中,相较于海水组(SW),半咸水组(BW)中的LGALS4表达量显著升高(为SW组的1.61倍),淡水组(FW)的LGALS4表达量显著降低(为SW组的60%),而LGALS3a、LGALS3ba、GRPb和GPRc的表达量显著升高(分别为SW组的1.22、1.17、1.31和1.35倍)。LGALS2a和LGALS3bb几乎检测不到表达(FPKM<1),而LGALS1、LGALS2b、LGALS8a、LGALS8b、LGALS9、LGALS17和GRPa表达水平在各盐度组并未出现明显差异(见图6B)。
在碱度胁迫下的花鲈鳃组织中,LGALS17和GRPa的表达量在整个胁迫过程中显著升高,并在72 h仍处于持续升高的状态。其中GRPa的上调程度最显著,72 h表达量为0 h的4.42倍。LGALS2b在12 h时的表达量呈短暂升高的趋势(为0 h的2.47倍),并在24 h恢复到初始水平。LGALS3ba和LGALS4的表达量则在整个胁迫过程中均处于显著降低的状态。其中LGALS4的降低程度最显著,72 h表达量为0 h的34%。除此之外,GRPb和LGALS8a在72 h时才表现出表达量的显著改变(分别升高为0 h的1.37倍和降低为0 h的88%,见图6C)。
在高温胁迫下的花鲈肝脏组织中,LGALS8b、LGALS17以及GRPb的表达量在整个胁迫过程中显著降低,且在24 h时仍处于显著降低的状态。而LGALS8b降低程度最显著,在12 h时降低为0 h的5%。LGALS2a、LGALS3a、LGALS3ba、LGALS4和GRPa的表达量在胁迫过程中呈短暂升高的趋势。其中,LGALS3a分别在3和12 h发生显著升高(分别为0 h的1.38和3.31倍);LGALS3ba和LGALS4在6 h时达到最高点(分别为0 h的20.5和8.5倍),且在24 h恢复到了0 h的水平;LGALS2a分别在12和24 h发生显著升高(分别为0 h的1.65和3.71倍);GRPa则在24 h发生显著升高(为0 h的1.41倍)。LGALS1和LGALS9则恰好相反,在胁迫过程中出现了短暂的降低趋势,均在3 h达到最低点(分别为0 h的29%和39%),并在24 h恢复到0 h的水平。此外,LGALS8a的表达量呈先降低后升高趋势,其中3 h的表达量显著降低(为0 h的69%),而12 h的表达量显著升高(为0 h的1.69倍)。LGALS2b,LGALS3bb和GRPc在整个高温胁迫过程中几乎检测不到表达(FPKM<1)(见图6D)。
3 讨论
Galectins是一个较为保守的β-半乳糖苷结合蛋白的凝集素家族,从真菌到脊椎动物中均有发现[38]。然而,大多数的基因都是通过数据库比对分析来进行注释的,这使得许多基因的命名变得混乱,尤其是在Galectin家族的鉴定中[24]。例如,在斑马鱼中三个LGALS3基因都带有类似Galectin-3-like的注释,这使得进一步的基因功能分析变得十分困难[13]。本研究在花鲈中对Galectin基因家族进行完整的系统鉴定,为阐明Galectins在响应环境胁迫过程中的功能机制提供理论基础。
本研究共鉴定了14个Galectin基因,系统发育分析结果验证了基因注释的准确性(见图1)。此外,拷贝数分析结果表明,花鲈Galectin基因数量与分类地位较近的尼罗罗非鱼相同,且与其他硬骨鱼类相近(见表2)。同时在硬骨鱼中,LGALS2、LGALS8和GRP数量一致。除此之外,Galectin的多拷贝基因并不是串联重复排列,而是分布在不同的染色体上(见图2),这表明花鲈中Galectin基因的多拷贝现象可能是由于硬骨鱼中额外的全基因组复制(Whole genome duplications, WGD)事件产生的[39]。共线性分析中,花鲈Galectin基因与尼罗罗非鱼、斑马鱼具有良好的直系同源关系(见图4):22个尼罗罗非鱼和花鲈Galectin基因共线性区块、11个斑马鱼和花鲈Galectin基因共线性区块,这进一步说明了其基因注释的正确和结构上的保守。
Galectins广泛存在于大多数生物体中[40],能识别病毒、细菌和原生动物寄生虫表面的碳水化合物[41-42]。本研究中,组织表达模式分析显示14个Galectin基因在花鲈7个组织(肝、鳃、脾、胃、脑、精巢和卵巢)中广泛表达。Galectin基因这种广泛的组织表达分布在其他鱼类中也有发现。比如,大菱鲆中13个Galectin基因在脑、皮肤、肠、鳃、肝、脾、血以及头肾中均有表达[24]。在斑点叉尾鮰中,12个Galectin基因广泛表达于脑、体肾、肌肉、肠、头肾、脾、肝、鳃和皮肤中[25]。红鳍东方鲀中12个Galectin基因在脑、肾、心脏、皮肤、鳃、肌肉、脾、肠和肝中均有表达[26]。同时,LGALS3ba、LGALS8a、LGALS9和LGALS17在花鲈脾脏中高表达,而脾脏为鱼类的主要免疫器官,表明这些基因可能在花鲈免疫应答时发挥着重要作用。而LGALS3、LGALS8和LGALS9的免疫功能在草鱼(Ctenopharyngodonidella)、尼罗罗非鱼和鳜(Sinipercachuatsi)等硬骨鱼中也得到了验证[15,43-44]。
鱼类对环境胁迫的响应大致分为三个阶段[45-46]:初级阶段(如神经内分泌反应和皮质类固醇-儿茶酚胺释放),次级阶段(如代谢、细胞、血液和免疫反应)和三级阶段(整个生物体的生理和行为应激反应)。Galectin家族是在先天免疫系统中发挥重要作用的一个家族,本文针对其在花鲈响应环境因子胁迫的表达情况进行了分析。LGALS17的表达水平在短期胁迫(高温、低氧和碱度)中均发生显著变化,且均出现在胁迫12 h之内甚至3 h以前,而长期盐度试验中未发现其表达量变化。在基因结构分析中,对LGALS17预测得到Ig superfamily和IG-like保守结构域。IG作为具有抗体活性的免疫球蛋白,在免疫系统中对防御致病菌和病毒具有着重要作用[47],推测LGALS17是在短期胁迫中进行快速的免疫应答来完成胁迫响应。LGALS4的表达模式在短期胁迫(高温和碱度)和长期盐度胁迫中存在显著差异。LGALS4作为串联重复型凝集素,其两个结构域(CRD)可同时与不同的配体结合,在许多生物学过程中成为关键的交联剂和调节因子,具有增强脂筏稳定性、参与糖蛋白转运、促进肠道创伤愈合和促进神经元轴突发育及髓鞘形成等生理功能,对维持生理平衡起着重要作用[48]。推测LGALS4在短期和长期胁迫中能通过维持生理平衡来进行胁迫响应。有研究表明LGALS3和GRPb在细菌感染过程中可作为细胞表面的附着点或促进粘附的交联因子[49-52]。在尼罗罗非鱼中,LGALS3和GRPb可通过调节单核巨噬细胞的活性来完成免疫应答[52-53]。本研究中LGALS3a、LGALS3ba和GRPb在短期和长期胁迫中均差异表达,其可能通过免疫应答来实现胁迫响应。综上分析发现,LGALS3a、LGALS3ba、LGALS4、LGALS17和GRPb可能在进行环境因子胁迫的应答时发挥着潜在作用。在关于Galectin基因的研究中,也发现其在响应胁迫中扮演着重要角色。比如,小鼠LGALS3可通过协调内质网和线粒体来保证线粒体网络形态的完整性,以启动细胞应激反应来提高上皮细胞对环境变化的适应与防御能力[54]。在鹿角杯形珊瑚(Pocilloporadamicornis)中,LGALS1通过调节病原体与共生藻的识别来响应高温胁迫下的珊瑚白化[55]。虽然Galectin基因在环境因子胁迫下的表达调控机制尚未完全阐明,但其可能以不同的调控模式在抗逆胁迫中发挥着潜在作用。
4 结语
本研究在花鲈中共鉴定出14个Galectin基因,通过系统发育、拷贝数、共线性和基因结构分析验证了基因注释的准确性和结构上的保守性。Galectin基因在花鲈多个组织中广泛表达,且有6个基因(LGALS3ba、LGALS4、LGALS8a、LGALS9、LGALS17和GRPa)表现出了组织特异性。在不同环境因子胁迫中,花鲈Galectin基因在不同组织中的表达量发生变化,这表明其参与了胁迫响应并发挥了潜在作用。其中,LGALS3a、LGALS3ba、LGALS4、LGALS17和GRPb变化更显著、更迅速,其可能在响应环境胁迫中起着更重要的作用。本研究提供了花鲈Galectin基因的结构与表达信息,为进一步研究硬骨鱼中Galectin基因的功能提供了重要的基础数据。
——以多重共线性内容为例

