黄颡鱼硒蛋白基因的克隆与分析





摘要 为探讨黄颡鱼硒蛋白selenow2a、selenop2 和selenot2 基因之间的关系,采用3′/5′ RACE PCR 克隆得到3 个基因的cDNA 全长,分别为891、1 998 和1 432 bp,其中ORF 长度分别为288、828 和600 bp,编码95、275 和219 个氨基酸。在线工具SECISerach3 对3 个基因的cDNA 序列分析结果显示,它们都含有可以编码硒代半胱氨酸的终止密码子,以及在3′非编码区存在SECIS 元件。通过氨基酸序列比对和系统发育树分析,发现sele⁃now2a、selenop2 和selenot2 基因预测得到的氨基酸序列与斑马鱼(Danio rerio)氨基酸相似性分别为82.24%、66.19% 和79.45%,而与斑点叉尾鮰(Ictalurus punetaus)的氨基酸相似性分别为94.74%、68.50% 和90.95%,在发育树上则显示为树杈相接近。采用实时荧光定量PCR 检测3 个硒蛋白基因的mRNA 在黄颡鱼心脏、肝脏、肌肉、脑、肠、脾脏、精巢和卵巢组织中的表达,结果显示其mRNA 表达水平呈现组织特异性。表明3 个基因拥有硒蛋白家族的特征,但在组织表达上具有特异性。
关键词 硒蛋白; 基因克隆; 分子特征; 黄颡鱼; 组织表达
中图分类号 Q959.4 文献标识码 A 文章编号 1000-2421(2024)01-0185-09
硒(selenium)是脊椎动物维持细胞功能的正常行使并参与多种代谢活动必需的微量元素之一[1-2]。体内过高或过低的硒水平会引起水产动物机体免疫力[3]、生长性能[4]的降低以及硒毒性[5]等负面影响。因此,在水产饲料中添加适量的硒用于保持鱼体健康成长是必需的[6]。
硒代半胱氨酸(selenocysteine,Sec) 是细胞中硒的主要形式,于 20 世纪 70 年末被发现且被归于第21 种氨基酸,由终止密码子UGA 编码,并通过特殊的共翻译机制嵌入到多肽链中。硒蛋白是含有Sec的蛋白,也是硒参与体内生理调控的关键生物分子,其特征是至少含有1 个Sec,Sec 的存在使得硒蛋白能够参与氧化还原反应。至今,在小鼠和人类中分别发现了24 种和25 种硒蛋白[7]。通常情况下,UGA密码子在翻译中执行终止信号。然而,当硒半胱氨酸插入序列(selenocysteine insertion sequence,SECIS)出现在3′非翻译区(3′ untranslated region,3′UTR)时,UGA 会被翻译成Sec[8]。
在斑马鱼中已经鉴定18 个含Sec 的硒蛋白基因[9],此外,部分硒蛋白基因也在其他鱼类中被发现。例如,在蓝鳍金枪鱼(Thunnus maccoyii)和光唇鱼(Acrossocheilus fasciatus)发现硒蛋白gpx1 基因[10-11];在鲤(Cyprinus carpio)和蓝鳍金枪鱼得到了gpx4 基因的信息[10,12]。硒蛋白基因selenou、selenow和selenop 的信息也分别在河豚(Tetraodontidae)、鲫(Carassius auratus)和罗非鱼(Oreochromis niloticus)中获得[13-15]。最近,笔者所在实验室已经在黄颡鱼中克隆获得了7 种硒蛋白(gpx1、gpx3、gpx4、sele⁃now、selenop、t xnrd2 和txnrd3)基因的信息,并描述了这些硒蛋白基因的分子特征[16]。在此基础上,本研究克隆获得黄颡鱼3 个硒蛋白基因(selenow2a、selenop2 和selenot2)的全长cDNA 序列,使用生物信息学等方法分析其氨基酸序列同源性、结构域和进化关系,并探讨了它们的组织表达分布,以期为深入了解硒蛋白基因之间的结构和功能的异同及它们在生物进化上的联系提供基础资料。
1 材料与方法
1.1 试验鱼及试剂
试验用鱼购自武汉市当地农贸市场。在不投喂处理24 h 后,选取健康、体形一致的体质量为(24.2±2.4) g 的黄颡鱼分2 组进行采样分析:第1 组黄颡鱼取肝脏和卵巢组织用于基因cDNA 克隆。第2 组黄颡鱼样品用于测试心脏、肝脏、脑、脾脏、肌肉、精巢、卵巢和肠组织中黄颡鱼基因的表达水平。取样方法参照Zhao 等[17]方法,所有样品的获取均在冰上进行,样品总RNA 的分离和提取在取样后迅速进行,于-80 ℃保存样品和RNA 提取物。
凝胶回收试剂盒(Omega)、TRIzol 试剂、逆转录试剂盒、3′/5′RACE PCR 试剂盒、RT-PCR 试剂盒,均购自TaKaRa 公司;RNA 提取实验中用到的异丙醇、乙醇等化学试剂均为分析纯(上海国药集团化学试剂有限公司)。
1.2 硒蛋白基因cDNA序列的克隆
序列克隆参照文献[17]的研究方案进行。总RNA 的提取按照TRIzol 说明书进行。使用琼脂糖凝胶电泳和Nanodrop ND 2000 分光光度计(ThermoFisher Scientific,USA)检测总RNA 的质量和浓度,以提取的总RNA 为模板,按照逆转录试剂盒(Prime‑ScriptTM II 1st Strand cDNA Synthesis Kit)说明书进行,合成第一链cDNA。
根据GenBank 数据库及转录组测序数据,设计扩增黄颡鱼硒蛋白基因序列的引物(表1)。包括目的cDNA 核心片段及3′和5′末端,采用巢式PCR 反应扩增目的片段,其中Outer-PCR 反应参数为:95 ℃预变性5 min;然后95 ℃ 30 s,55 ℃ 30 s,72 ℃ l min,共25 个循环;最后72 ℃延伸5 min。Inner-PCR 反应参数为:95 ℃预变性5 min;然后95 ℃ 30 s,57 ℃30 s ,72 ℃ 1 min ,共30 个循环;最后72 ℃ 延伸5 min[18]。扩增所得目的片段,采用Omega 公司胶回收试剂盒进行回收提纯,然后将目的片段克隆到pMD™19-T( TaKaRa)载体,转化到DH5α感受态细胞中,后续将转化的菌液均匀涂在含氨苄的LB 平板上,随机挑选菌落进行PCR 检测,将带有目的条带的菌液送至武汉擎科生物有限公司测序。
1.3 序列分析
使用DNAStar 软件拼接序列片段,获得cDNA全长。拼接好的cDNA 序列通过NCBI 进行Blast 分析,以确定该序列命名是否正确。使用DNAStar 软件,找出目的基因的开放阅读框(open reading frame,ORF),将其翻译成氨基酸序列,并使用Clustal-W 软件进行氨基酸多序列比对和氨基酸相似性分析。在GUIDANCE 网站 (http://guidance.tau.ac.il/)上对氨基酸多序列进行比对、剪切操作[19],并采用MEGA 6.0 软件的邻接法(NJ)构建构建进化树,每个节点的可信值设置为1 000 次重复计算。使用SECISearch3(http://gladyshevlab. org/selenoproteinpredictionserver)预测得到硒蛋白的SECIS 元件,使用SECISln (http://genome. crg. es/software/SECISaln/)获得SECIS 元件的标准模型[18]。
1.4 硒蛋白基因的组织表达分析
采用实时荧光定量PCR(qPCR)检测黄颡鱼硒蛋白基因的组织表达情况[18]。各组织样品的总RNA 提取方法同本文“1.2”,按照逆转录试剂盒(Ta‑KaRa)说明书进行操作。荧光定量PCR 引物如表2所示。本试验用GeNorm 进行标准化选出在黄颡鱼不同组织中稳定表达的hprt 和b2m 作为内参基因,以2−ΔΔCt法计算硒蛋白基因的组织表达水平。
1.5 数据处理
试验数据用SPSS 26.0 软件(SPSS,MichiganAvenue,Chicago,IL,USA)进行处理,结果以“平均值±标准误”( mean±SEM,n=3) 表示,使用单因素方差分析(one-way ANOVA)和Duncan’s 多重比较检验进行不同组间指标差异性的统计检验。统计分析之前用Kolmogorov-Smirnov 检验所有数据的正态分布性,以Levene 检验分析方差齐性,显著性水平为α= 0.05[17-18]。
2 结果与分析
2.1 selenow2a、selenop2 和selenot2 基因的分子特征和进化关系
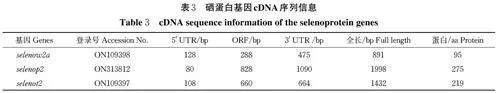
本研究使用RT-PCR 和RACE 的方法获得了selenow2a、selenop2 和selenot2 的cDNA 全长序列,长度分别为891、1 998 和1 432 bp,其中ORF 长度分别是288、828 和600 bp,能够编码的氨基酸个数分别为95、275 和219 个(表3)。将黄颡鱼selenow2a、sele⁃nop2 和selenot2 基因预测得到的氨基酸序列与斑马鱼(Danio rerio)进行比较,发现氨基酸相似性分别为82.24%、66.19% 和79.45%,而与斑点叉尾鮰(Ictal⁃urus punetaus)的氨基酸相似性分别为94.74%、68.50% 和90.95%。
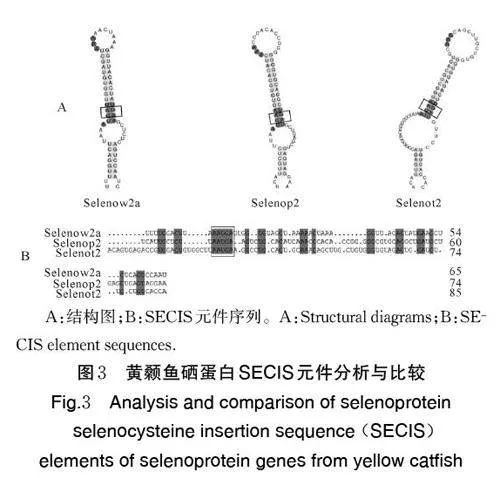
cDNA 序列中存在编码Sec 的终止密码子,在3′非编码区有SECIS 元件序列(图1)。黄颡鱼sele⁃now2a、selenop2 和selenot2 基因预测的氨基酸结构域中存在保守的氧化还原序列CXXU(U 为Sec,X 为任意氨基酸)。其中Selenow2a 结构域中还包括有硒蛋白Rdx 结构域和硫氧还蛋白样超家族,Selenop2 结构域包括信号肽区域和富组氨酸区域,Selenot2 结构域包括硒蛋白Rdx 结构域(图2)。AAUGA 保守序列存在于黄颡鱼Selenow2a、Selenop2 和Selenot2 的SECIS 元件上(图3)。
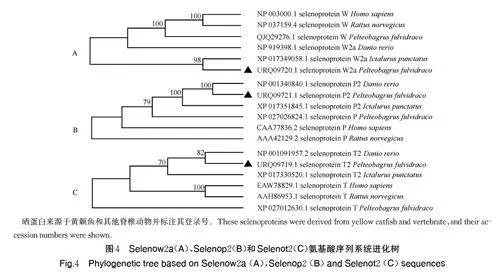
在进化关系上,黄颡鱼Selenow2a 与斑点叉尾鮰的Selenow2a 进化关系更加密切,而与斑马鱼Selenow2a、黄颡鱼同源蛋白Selenow、人类Selenow 和大鼠Selenow 关系较远(图4A)。Selenop2 则与斑马鱼Selenop 的同源蛋白Selenopb(Selenop2)在进化上更为亲近,而与其他物种关系较为疏远(图4B),黄颡鱼Selenot2 的进化关系与Selenop2 较为类似(图4C)。
2.2 selenow2a、selenop2 和selenot2 基因的组织表达分析
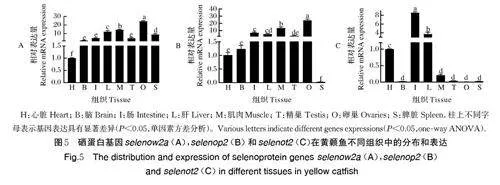
通过实时荧光定量PCR 检测克隆的黄颡鱼sele⁃now2a、selenop2 和selenot2 基因在8 种组织中的mRNA 表达水平,结果如图5 所示。黄颡鱼sele⁃now2a mRNA 在各个组织中广泛表达,表达水平最高的是卵巢,而心脏中mRNA 表达相对较低(图5A)。selenop2 在各个组织中广泛分布,其mRNA 表达最高和最低分别是卵巢和脾脏( 图5B)。selenot2的心脏、肠、肝脏中mRNA 表达水平较高,最高的是肠,而脑、精巢、卵巢、脾脏中很低(图5C)。总的来说,表达水平具有组织特异性。
3 讨论
本研究克隆并获得3 个黄颡鱼硒蛋白基因sele⁃now2a、selenop2 和selenot2,并在克隆得到的3 个硒蛋白基因的cDNA 序列中发现编码Sec 的终止密码子和在3′非编码区发现SECIS 元件,确定了克隆得到的基因属于硒蛋白家族[8]。比较和分析预测得到的氨基酸序列和结构域,发现3 个克隆的基因与斑马鱼、斑点叉尾鮰都有比较高的氨基酸相似性和相似结构域,暗示它们在进化过程中是保守的[9-10,20-21]。通过比对不同物种之间硒蛋白基因以及亚型基因之间的进化关系,进一步说明了这种保守的现象。最后,检测了3 个硒蛋白基因在黄颡鱼8 种测试组织中的表达分布,结果表明3 个硒蛋白基因具有组织特异性。
克隆得到的selenow2a 是黄颡鱼selenow 的亚型基因。已有的研究报道了selenow 在黄颡鱼多个组织中表达分布,并且发现在黄颡鱼的鳃中表达量最高[16]。但是本研究结果显示selenow2a 在卵巢中表达水平最高。有研究人员发现雌性塞内加尔鳎(Solea senegalensis)的卵母细胞生长中会增加sele⁃now2a 的表达,这是一种抗氧化保护措施[22],因此,我们推测selenow2a 基因在黄颡鱼卵巢中的高mRNA 表达可能和雌性黄颡鱼后续的卵巢发育有关。本研究在预测的结构域中发现了与氧化还原调节相关的CXXU 序列和硫氧还蛋白样折叠结构,暗示Selenow2a 能够参与机体的抗氧化反应[9],这与之前报道的Selenow 类似[16],这可能与2 个基因具有相近的ORF 长度和氨基酸数目有关。氨基酸同源性比较和系统发育树分析的结果表明黄颡鱼Selenow2a和斑点叉尾鮰Selenow2a 在进化关系中最接近。
Selenop 是唯一已知的含有多个Sec 的硒蛋白,主要将硒从肝脏传递到其他器官[23],在人类中有10个,而在斑马鱼中发现了17 个[9],之前的研究报道了在黄颡鱼的selenop 基因上发现了13 个Sec[16]。然而,在本研究中克隆得到selenop2 的cDNA 序列中仅发现了1 个Sec,以及3'非编码区也仅含有1 个SECIS元件,这和斑马鱼中的基因selenop2 是类似的结果[9],这可能表明Selenop2 不参与机体中硒的传递。
本研究克隆的selenop2 的cDNA 序列相较于数据库中selenop2 基因多出了SECIS 元件,证明了克隆得到的selenop2 属于硒蛋白家族。结构域分析发现了保守区域UXXC 氧化还原序列,这种保守序列暗示Selenop2 能够参与黄颡鱼的氧化还原稳态调节。此外,氨基酸同源性和系统发育树的分析结果显示黄颡鱼Selenop2 和斑马鱼Selenopb(Selenop2)在进化上最近。有趣的是,黄颡鱼的selenop 基因和selenop2基因在不同的组织表达水平是不同的[16],表现出基因表达的特异性。Selenop2 和Selenop 生物组织定位、生化特异性和进化关系之间的差异可能有助于理解硒蛋白P 的独特。
Selenot2 和黄颡鱼Selenot 具有同源关系,与Selenot 具有类似的硒蛋白Rdx 结构域和保守的CXXU 氧化还原序列,暗示其具有氧化还原功能。氨基酸同源性和系统发育树的分析结果表明黄颡鱼Selenot2 和斑马鱼Selenot2 在进化中关系最近。黄颡鱼selenot2 基因主要在黄颡鱼的心脏、肠、肝脏和肌肉中表达。相关研究表明,Selenot 在保护金鱼(Carassius auratus)的心脏免受缺血再灌注(Ischemia-Reperfusion,IR)损伤时发挥了重要作用[24],因此,我们猜测selenot2 基因在黄颡鱼心脏中的高mRNA 水平可能也有这种作用。
本研究通过RACE 克隆获得3 个黄颡鱼硒蛋白基因(selenow2a、selenot2 和selenop2)并使用生物信息学技术推测出它们的氨基酸序列。通过与斑马鱼、斑点叉尾鮰对比分析,发现3 个硒蛋白基因的核苷酸序列和氨基酸序列在鱼类中存在保守的序列,并且可以在人类和大鼠中找到,表明这些保守序列可能在不同物种的硒蛋白功能的稳定发挥中扮演重要角色。实时荧光定量PCR 结果显示,3 个硒蛋白基因在黄颡鱼体内均有表达,但不同的组织中表达水平差异显著,这可能与硒蛋白功能有关,还需要进一步深入的研究。
参考文献References
[1] LU J,HOLMGREN A.Selenoproteins[J].Journal of biological
chemistry,2009,284(2):723-727.
[2] LABUNSKYY V M,HATFIELD D L,GLADYSHEV V N.
Selenoproteins:molecular pathways and physiological roles
[J].Physiological reviews,2014,94(3):739-777.
[3] GAO X J,TANG B,LIANG H H,et al.Selenium deficiency
induced an inflammatory response by the HSP60 - TLR2-
MAPKs signalling pathway in the liver of carp[J]. Fish amp;
shellfish immunology,2019,87:688-694.
[4] LIN Y H,SHIAU S Y.Dietary selenium requirements of juvenile
grouper,Epinephelus malabaricus[J].Aquaculture,2005,
250(1/2):356-363.
[5] ZHANG B L,DUAN G Q,FANG Y Y,et al.Selenium(Ⅳ)
alleviates chromium(Ⅵ)-induced toxicity in the green alga
Chlamydomonas reinhardtii[J/OL].Environmental pollution,
2021,272:116407[2022-11-02]. https://doi. org/10.1016/j.
envpol.2020.116407.
[6] 郭正富,李军,杨小琴. 不同硒水平对鲈鱼生长性能及抗氧化
能力的影响[J]. 中国饲料,2018(6):88-92.GUO Z F,LI J,
YANG X Q.Effects of dietary selenium content on growth performance
and antioxidant capacity of juvenile Japanese seabass
(Lateolabrax japonicus)[J]. China feed,2018(6):88-92 (in
Chinese with English abstract).
[7] KRYUKOV G V,CASTELLANO S,NOVOSELOV S V,
et al.Characterization of mammalian selenoproteomes[J].Science,
2003,300(5624):1439-1443.
[8] SQUIRES J E,BERRY M J.Eukaryotic selenoprotein synthesis:
mechanistic insight incorporating new factors and new functions
for old factors[J].IUBMB life,2008,60(4):232-235.
[9] KRYUKOV G V,GLADYSHEV V N.Selenium metabolism
in zebrafish:multiplicity of selenoprotein genes and expression
of a protein containing 17 selenocysteine residues[J].Genes to
cells,2000,5(12):1049-1060.
[10] THOMPSON J L,SEE V H L,THOMAS P M,et al.Cloning
and characterization of two glutathione peroxidase cDNAs
from southern bluefin tuna (Thunnus maccoyii)[J].Comparative
biochemistry and physiology part B:biochemistry and molecular
biology,2010,156(4):287-297.
[11] LIU G D,SHENG Z,WANG Y F,et al.Glutathione peroxidase
1 expression,malondialdehyde levels and histological alterations
in the liver of Acrossocheilus fasciatus exposed to cadmium
chloride[J].Gene,2016,578(2):210-218.
[12] HERMESZ E,FERENCZ Á.Identification of two phospholipid
hydroperoxide glutathione peroxidase (gpx4) genes in common
carp[J]. Comparative biochemistry and physiology part
C:toxicology amp; pharmacology,2009,150(1):101-106.
[13] CASTELLANO S,NOVOSELOV S V,KRYUKOV G V,
et al.Reconsidering the evolution of eukaryotic selenoproteins:
a novel nonmammalian family with scattered phylogenetic distribution
[J].EMBO reports,2004,5(1):71-77.
[14] CHEN W B,ZHANG Z,DONG H Y,et al.Molecular cloning
and sequence analysis of selenoprotein W gene and its mRNA
expression patterns in response to metabolic status and cadmium
exposure in goldfish,Carassius auratus[J]. Comparative
biochemistry and physiology part B:biochemistry and molecular
biology,2015,184:1-9.
[15] THUMMABANCHA K,ONPARN N,SRISAPOOME P.
Molecular characterization and expression analyses of cDNAs
encoding the thioredoxin-interacting protein and selenoprotein
P genes and histological changes in Nile tilapia (Oreochromis
niloticus) in response to silver nanoparticle exposure[J].Gene,
2016,577(2):161-173.
[16] XU X J,ZHANG D G,ZHAO T,et al.Characterization and
expression analysis of seven selenoprotein genes in yellow catfish
Pelteobagrus fulvidraco to dietary selenium levels[J/OL]. Journal
of trace elements in medicine and biology,2020,62:126600
[2022-11-02].https://doi.org/10.1016/j.jtemb.2020.126600 .
[17] ZHAO T,WU K,HOGSTRAND C,et al.Lipophagy mediated
carbohydrate-induced changes of lipid metabolism via oxidative
stress,endoplasmic reticulum (ER) stress and ChREBP/
PPARγ pathways[J]. Cellular and molecular life sciences,
2020,77(10):1987-2003.
[18] 杨水波. 糖诱导SUMO 化修饰对黄颡鱼脂质代谢调控机理
的研究[D]. 武汉:华中农业大学,2021. YANG S B. Sumoylation
and its regulatory mechanism in carbohydrate influencing
lipid metabolism of yellow catfish Pelteobagrus fulvi⁃
draco[D]. Wuhan:Huazhong Agricultural University,2021
(in Chinese with English abstract).
[19] PENN O,PRIVMAN E,LANDAN G,et al. An alignment
confidence score capturing robustness to guide tree uncertainty
[J].Molecular biology and evolution,2010,27(8):1759-1767.
[20] SCHWEIZER U,STEEGBORN C. New insights into the
structure and mechanism of iodothyronine deiodinases[J].Journal
of molecular endocrinology,2015,55(3):R37-R52.
[21] XIE X Z,CHEN M N,ZHU A Y.Identification and characterization
of two selenium-dependent glutathione peroxidase 1
isoforms from Larimichthys crocea[J].Fish amp; shellfish immunology,
2017,71:411-422.
[22] TINGAUD-SEQUEIRA A,CHAUVIGNÉ F,LOZANO J,
et al. New insights into molecular pathways associated with
flatfish ovarian development and atresia revealed by transcriptional
analysis[J/OL]. BMC genomics,2009,10:434[2022-
11-02].https://doi.org/10.1186/1471-2164-10-434.
[23] SCHOMBURG L,SCHWEIZER U,HOLTMANN B,et al.
Gene disruption discloses role of selenoprotein P in selenium
delivery to target tissues[J]. The biochemical journal,2003,
370(2):397-402.
[24] MAZZA R,GATTUSO A,IMBROGNO S,et al.Selenoprotein
T as a new positive inotrope in the goldfish,Carassius au⁃
ratus[J/OL]. The journal of experimental biology,2019,222
(11):jeb201202[2022-11-02]. https://doi. org/10.1242/
jeb.201202.
(责任编辑:边书京)

