基于内质网应激相关基因构建胰腺癌预后预测模型
吕胜男, 彭新宇, 张 健, 刘 欢, 魏 锋
吉林大学第一医院普通外科中心肝胆胰腺外科, 长春 130021
胰腺癌是消化系统恶性程度最高的肿瘤之一,5年生存率不到9%,目前的治疗手段对患者生存期的改善仍十分有限[1],大多数患者死于肿瘤复发或转移,因此寻找新的标志物对于评价胰腺癌的预后具有重要的临床意义。
内质网应激是细胞常见的一种适应性反应,当细胞受到内外不良环境的影响时,内质网应激被激活,通过激活未折叠蛋白质反应和回收错误折叠的蛋白质分子,使细胞恢复稳态,维持细胞存活,而当细胞稳态无法被恢复时,内质网应激也会诱导细胞凋亡。内质网应激在多种肿瘤中具有重要功能[2],其既可以调控肿瘤细胞本身的生存状态,影响肿瘤细胞的信号通路改变及侵袭、迁移表型的变化,也可以调控肿瘤微环境内巨噬细胞[3]、自然杀伤细胞(NK 细胞)等免疫细胞的功能状态[4],进一步影响肿瘤进展。内质网应激在胰腺癌的发生发展中具有重要的调控作用[5],但其与胰腺癌预后的关系尚未明确。
因此,本研究利用了生物信息学方法,分析内质网应激基因在胰腺癌组织和正常胰腺组织中的表达特征,并据此划分预后不同的胰腺癌亚群,进一步筛选亚群间的差异表达基因作为预后标志分子,构建了胰腺癌的预后预测模型,最后通过生物学实验验证预后标志分子在胰腺癌组织内的表达情况。该研究证实了内质网应激基因影响胰腺癌预后,并筛选出最可能作为预后标志物和治疗靶点的基因,为此后基于内质网应激的胰腺癌治疗的相关研究提供了重要思路。
1 材料与方法
1.1 材料与试剂
1.1.1 组织样本 利用生物信息学分析方法,对人类癌症基因组图谱(TCGA)及基因型-组织表达(GTEx)数据库收录的177 例胰腺癌样本和170 例正常胰腺组织样本的转录组数据中的260 个内质网应激基因的表达特征进行分析,并依据差异表达且影响预后的内质网应激基因划分胰腺癌亚群。实验所用的40 对胰腺癌组织和癌旁正常胰腺组织标本来源于吉林大学第一医院普通外科中心肝胆胰外科,经术后病理确诊为胰腺导管腺癌。
1.1.2 主要试剂和仪器 实时荧光定量PCR 仪购自美国Roche 公司;研究所用的RNA 采用总RNA 提取试剂盒(Invitrogen,美国)提取,利用PrimeScript RT reagent Kit (Takara)及通用引物对RNA 进行反转录,应用SYBR Primix Ex Taq 酶(Takara)进行实时荧光定量,引物序列在表1中列出。

表1 引物序列Table 1 Primer sequences
1.2 实验方法
1.2.1 研究数据获取及处理 通过基因组数据共享数据门户(https://portal. gdc. cancer. gov/)平台下载了TCGA和GTEx数据库收录的胰腺癌和正常胰腺组织样本的转录组测序数据。利用R 分析软件(R-4.2.1)的limma(3.52.4)包进行归一化处理,应用Sva(3.46.0)包进行去批次效应。
1.2.2 内质网应激基因的获取 通过MsigDB 网站(https://www. gsea-msigdb. org/gsea/msigdb)获取到与内质网应激相关的260个基因。
1.2.3 差异表达分析 通过R 分析软件DESeq2(1.36.0)包进行差异表达分析,并计算表达量倍数(fold change,FC),筛选条件为:|log2FC|≥1 且校正后的P值(Padj)<0.05。
1.2.4 共识聚类分析 利用R 分析软件的ConsensusClusterPlus(1.60.0)包对TCGA 胰腺癌队列的177例样本进行共识聚类分析,迭代次数为50次。
A:如果这位老师扮演的角色就只是“管工”,那么,孩子即使很努力,也无法“用心学习”的。如果加上家长只会等着“收割”,那就是不断制造压力,让孩子失去学习兴趣。一年级的孩子需要半年的“磨合期”,对学校的制度、对老师的教学、对时间的管理,都需要重新适应,老师和家长的帮助必不可少。
1.2.5 总RNA 提取及反转录 按照Invitrogen 总RNA 提取试剂盒的说明书提取细胞总RNA 并测定RNA 浓度,按照逆转录试剂盒的操作步骤将RNA 反转录为cDNA。
1.2.6 实时荧光定量PCR 以反转录获得的cDNA为模板进行实时荧光定量PCR,以GAPDH 作为内参基因,采用Folds=2-ΔΔCT公式计算实验组与对照组的相对倍数关系。每个实验重复3次,取平均值。
1.3 统计学方法 采用SPSS 20.0 统计软件以及GraphPad Prism10进行数据分析。计量资料以±s表示,两组间比较采用成组t检验。计数资料两组间比较采用χ2检验。通过R 分析软件的survival(3.5-0)包进行单因素Cox 回归分析,判断单个变量与患者预后的关系,对有统计学意义的变量应用R 软件glmnet(4.1-6)包进行Lasso回归分析筛选,以得到系数不为0的特征变量。基于R分析软件的survival 和 surviminer包进行生存分析,并绘制Kaplan-Meier生存曲线,利用Log-rank 检验进行生存期的比较。应用R 软件caret(6.0-93)包进行预测模型的建立,并利用ROCR(1.0-11)包绘制受试者工作特征(ROC)曲线,通过评估ROC曲线下的面积以评价模型的预测价值。P<0.05 为差异有统计学意义。
2 结果
2.1 与胰腺癌预后相关的内质网应激基因 对在TCGA和GTEx队列中胰腺癌肿瘤组(n=177)和正常组(n=170)之间差异表达的内质网应激基因进行单因素Cox回归分析,结果显示,MARCKS为胰腺癌预后的保护因素,而CEBPB、PMAIP1、UBXN10 是胰腺癌预后的独立危险因素(图1)。

图1 胰腺癌预后相关内质网应激基因森林图Figure 1 Forest map of ER stress genes associated with prognosis in pancreatic cancer
2.2 两种预后不同的胰腺癌亚群 基于CEBPB、MARCKS、PMAIP1、UBXN10 的表达量对TCGA 胰腺癌队列中177 个肿瘤样本进行了共识聚类分析(图2a),确定聚类变量K=2 为最佳分簇数并最终获得两个胰腺癌亚群:簇A(n=106)和簇B(n=71),簇B 患者的总体生存期要明显长于簇A(χ2=12.232,P=0.01)(图2b)。
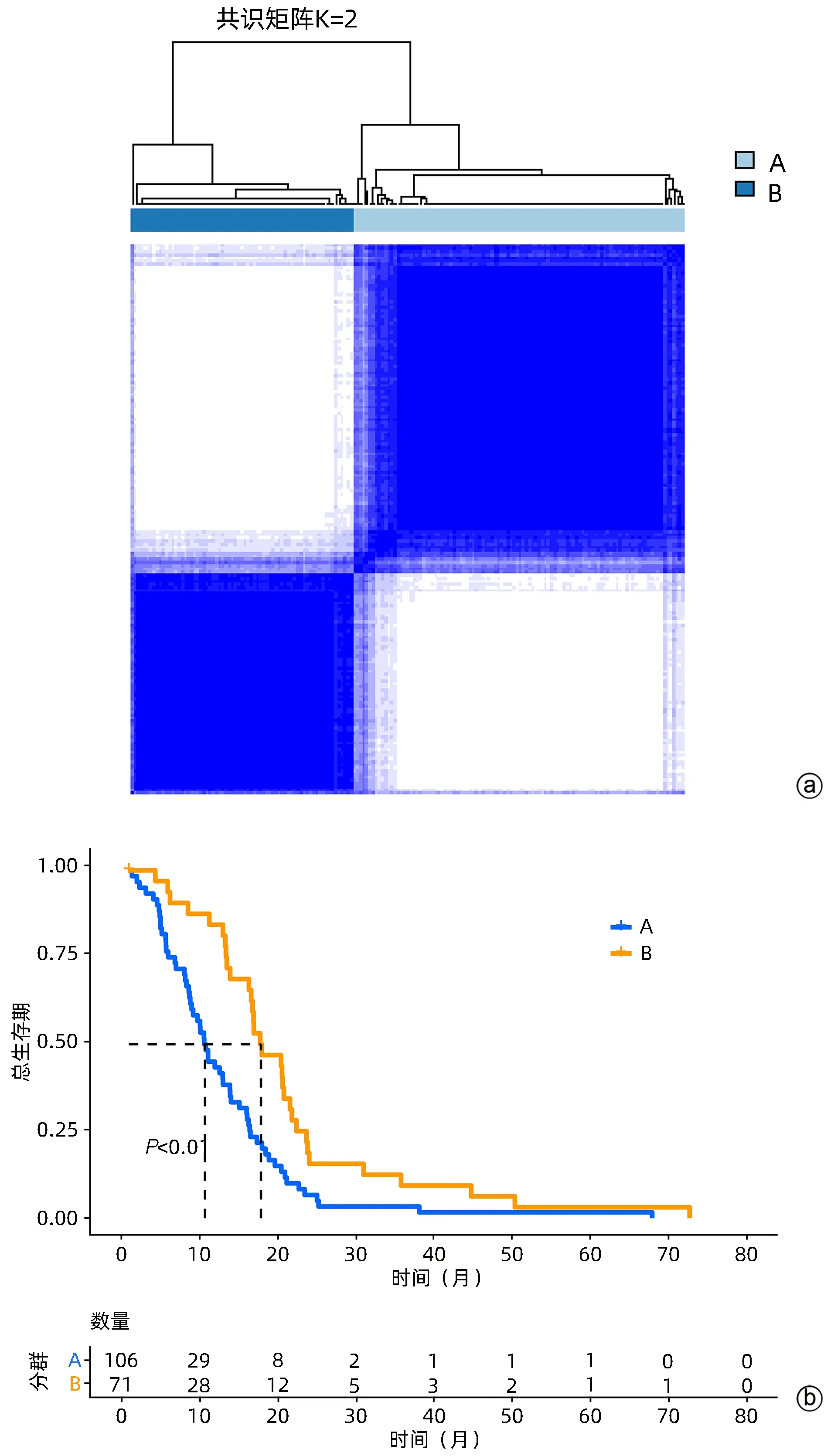
图2 依据胰腺癌内质网应激基因划分的两个亚群的聚类热图及生存曲线Figure 2 Cluster heat map and survival curve of two clusters of pancreatic cancer based on ER stress genes
2.3 胰腺癌亚群预后的独立危险因素 分析了簇A和簇B 的基因差异表达情况,依据|log2
FC|≥1 且Padj<0.05的筛选条件共获得了51个差异基因(附录A),通过单因素Cox 回归分析,鉴定出14个与总生存期相关的基因,且均为胰腺癌预后的危险因素(附录A)。
2.4 成功构建胰腺癌预后预测模型
2.4.1 构建胰腺癌预后预测模型 对14 个预后相关的差异基因进行分析,应用Lasso 回归分析方法,通过交叉验证实验得出log(λ)最小值为5(注:λ 表示惩罚系数)(图3a),确定构建模型的5 个最佳基因并计算了回归系数(图3b),风险评分方程为:风险评分=0.156×CDA+0.135×AHNAK2+0.020×RHOV+0.095×LY6D+0.054×SPRR1B。将TCGA胰腺癌队列的177个肿瘤患者根据中位风险值分为高风险或低风险,采用留出法进行训练集和测试集的划分,训练集∶测试集=7∶3,通过随机抽样方法将177 个样本划分为训练集(n=125)和测试集(n=52)以训练并测试模型,结果显示,该模型总体预测性能较好,预测患者1、3、5 年生存期的准确率分别为0.731、0.712、0.686(图3c),且该模型所预测的高风险患者的总体生存期与低风险组相比明显缩短(χ2=11.733,P=0.001)(图3d)。
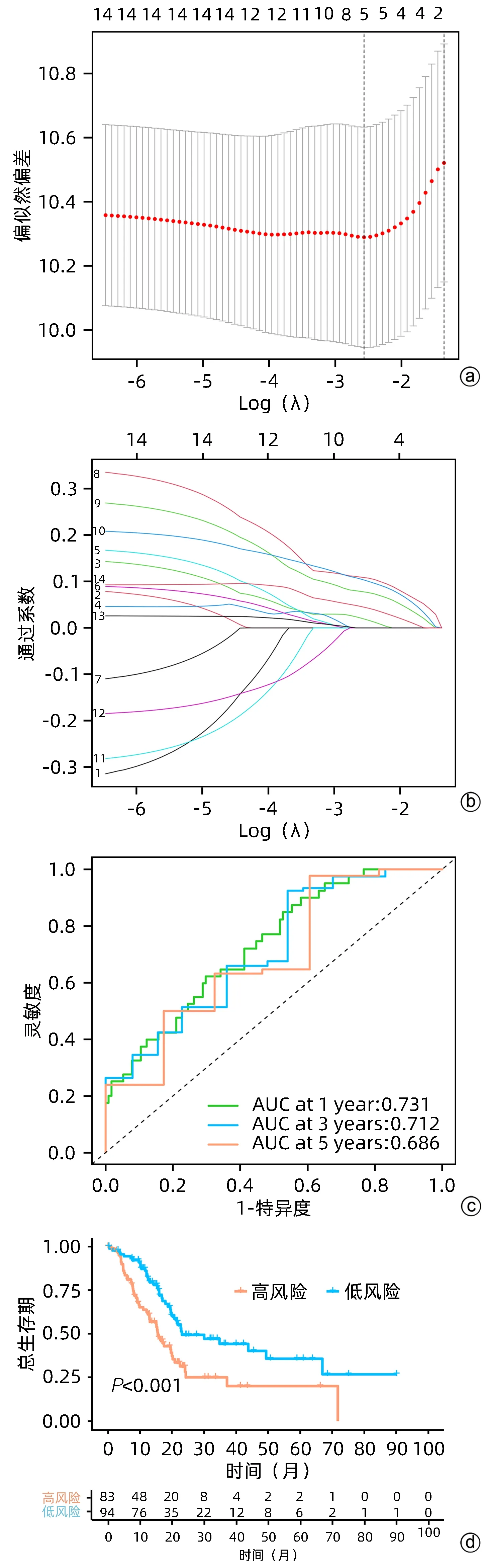
图3 Lasso回归结果及模型预测效果Figure 3 The results of Lasso regression and the effectiveness of this prediction model
采用χ2检验比较TCGA 胰腺癌队列高低风险组之间的临床特征(包括年龄、性别、肿瘤分期、T、N、M分期),结果显示两组之间的年龄、性别、肿瘤分期、T分期、N 分期均无显著差异,而M 分期存在差异(χ2=8.730,P=0.007),但可能是大量样本的远处转移情况无法评估导致的(附录B)。
2.4.2 外部数据集验证反映模型的预测效果良好验证了该模型对来自GEO 数据库的外部数据集GSE57495 的预测效果,结果显示,该模型表现出了良好的预测性能,预测患者1、3、5 年生存期的准确率分别为0.674、0.680、0.608(图4a),且该模型所预测的高风险患者的总体生存期与低风险组相比明显缩短(χ2=5.303,P<0.05)(图4b)。

图4 模型对外部数据集生存期的预测结果Figure 4 Survival prediction of external datasets using this model
2.5 CDA、AHNAK2、RHOV、LY6D、SPRR1B 在胰腺癌组织中的表达情况 检测了在40 对胰腺癌组织和癌旁正常组织中的基因表达量,结果显示,与癌旁正常组织相比,CDA(t=2.529,P=0.013 4)、AHNAK2(t=2.458,P=0.016 2)、RHOV(t=3.314,P=0.001 4)、LY6D(t=3.583,P=0.000 6)、SPRR1B(t=5.082,P<0.000 1)在胰腺癌组织中显著上调(图5)。
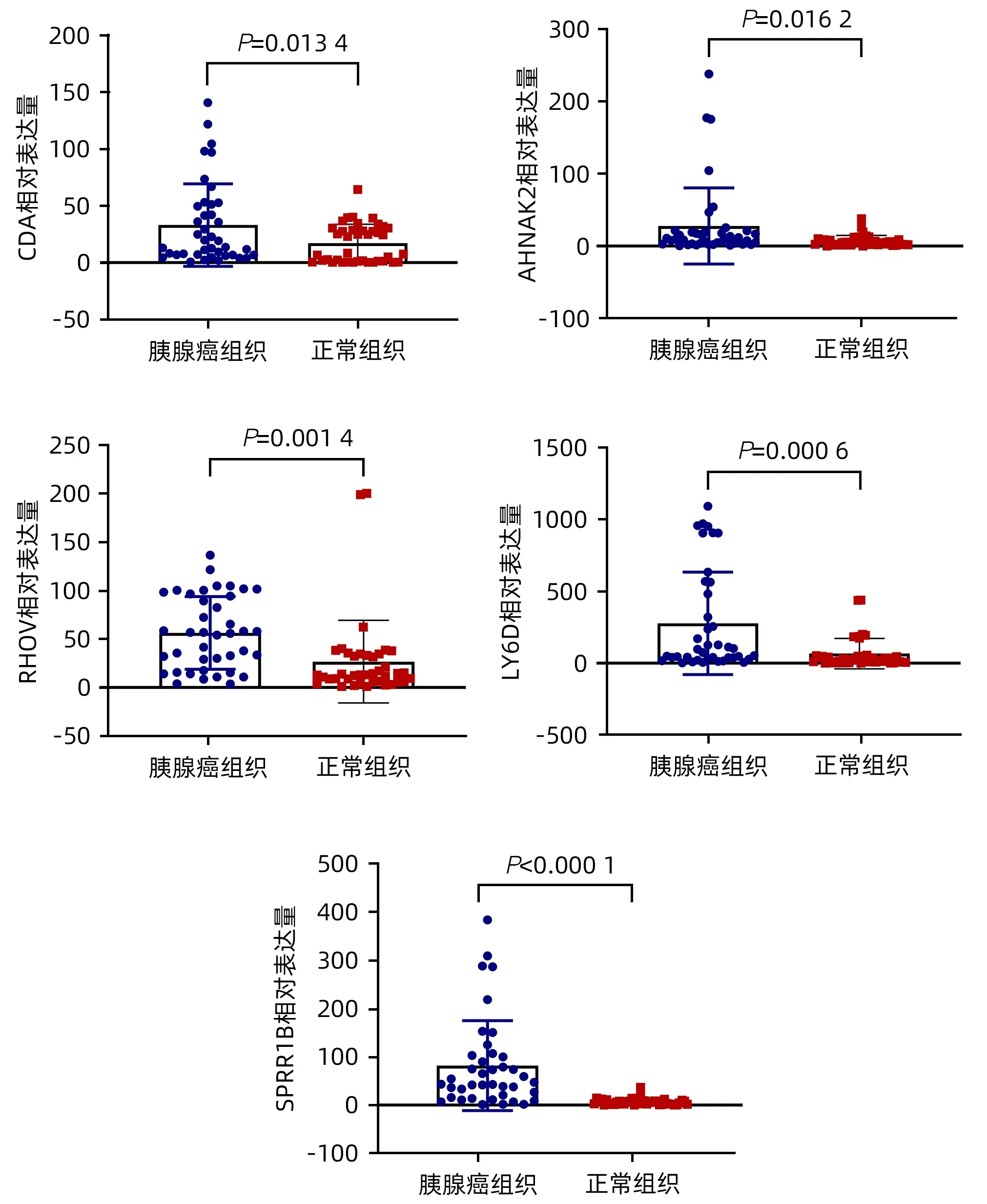
图5 CDA、AHNAK2、RHOV、LY6D、SPRR1B在肿瘤组织和正常组织的表达量Figure 5 The expression levels of CDA, AHNAK2,RHOV, LY6D, and SPRR1B in the tumor and normal tissues
3 讨论
胰腺癌是消化系统常见的恶性肿瘤,是全球所有癌症中第二大致死性病因,目前胰腺癌的5 年生存率约为9%[6]。近几年来胰腺癌的发病率仍呈逐年上升趋势,但病死率并没有因技术手段发展出现明显改善,仍和发病率基本持平[7-8]。其根本原因在于胰腺癌的早期诊断困难,大部分患者起病隐匿,且临床症状不典型,导致许多患者错过了最佳的治疗时机,且胰腺癌恶性程度高,容易发生神经、血管浸润及转移。目前针对胰腺癌患者的治疗策略仍以手术治疗为主[9],辅助吉西他滨等化疗药物治疗,由奥沙利铂、伊立替康、氟尿嘧啶、亚叶酸钙组成的Forfirinox 联合化疗方案也取得了良好的效果[10]。但尚未研制出有效的分子靶向药物,常规的免疫治疗方法在胰腺癌中也已达到理想的疗效[11]。如何找到有效的治疗药物,延长胰腺癌患者生存期,仍是医学界亟待攻克的难关。
内质网应激在胰腺癌的发生发展中具有重要作用。内质网应激基因BZW1 能够以PERK 通路依赖的形式促进胰腺癌细胞的Warburg效应进而促进动物模型体内的肿瘤生长[12],内质网氧化还原酶1 α在内质网应激条件下上调,并能够促进胰腺癌细胞的增殖[13]。此外,有研究[14]表明内质网应激可能与胰腺癌的不良预后相关,葡萄糖调节蛋白78(glucose-regulated protein 78,GRP78)是一种重要的内质网伴侣蛋白,它被视为内质网应激的标志物,来自180 例胰腺癌患者的基于组织微阵列的肿瘤和正常组织的免疫组化显示,与非肿瘤组织相比,癌细胞中的GRP78 表达显著增加(P<0.05),GRP78 的高表达与更高的T 分期和较差的总生存期有关(P<0.05)[15]。多变量分析显示,内质网跨膜蛋白ATF6α 高表达和磷酸化P38 低表达的组合与胰腺癌患者的不良结局(HR=2.705,P=0.023)以及肿瘤分级(HR=2.886,P=0.029)相关[16]。
但目前尚无研究阐述内质网应激基因能否作为胰腺癌的预后分子标志物,本研究适时地填补了这一领域的空白。笔者首次揭示了内质网应激基因作为胰腺癌预后的独立相关因素的重要价值,并成功依据它们的表达特征实现了胰腺癌亚群的划分。对两个亚群间的基因表达特征的进一步探究发现了对胰腺癌预后有重要影响的14 个差异基因,并从中筛选出最重要的5 个基因:CDA、AHNAK2、RHOV、LY6D、SPRR1B,并且依据该组核心基因成功构建了胰腺癌的预后预测模型,而这一组基因在胰腺癌中的重要作用此前鲜有报道。有研究表明CDA编码胞苷脱氨酶,在胰腺癌组织中高表达,能够灭活吉西他滨,降低其对肿瘤细胞的毒性[17],高表达CDA的巨噬细胞也与胰腺癌的吉西他滨耐药相关[18]。而AHNAK2 目前在胰腺癌中的研究较少,仅有一项荟萃分析[19]提示AHNAK2可以作为胰腺癌的诊断分子。既往有其他生物信息学分析提出LY6D与胰腺癌的不良预后相关[20]。仅有一项研究[21]表明SPRR1B是胰腺癌预后的危险因素。而RHOV在胰腺癌中的作用尚未被提及。
本研究的局限性在于,只能依据生物信息学分析推测变量与结局之间的相关性,并结合生物学背景进行一定的推测,但无法明确地阐明因果关系,这也是生物信息学的局限性所在。如需探究某变量变化与肿瘤发生发展的先后顺序,揭示其内在作用机制,仍需要在体内和体外实验中通过分子生物学实验具体研究。此外,本研究只利用了TCGA 数据库的胰腺癌队列数据进行模型训练,如果能纳入多中心、多样本数据,将有希望提升模型预测的准确性。
总之,本研究在构建胰腺癌内质网应激基因分型的基础上,依据亚群间基因的表达差异筛选出5 个核心基因用以构建胰腺癌的预后预测模型,最终在40对胰腺癌组织中验证了它们的高水平表达。本研究揭示了内质网应激在胰腺癌发生发展中的重要作用,为寻找胰腺癌治疗靶点、预测胰腺癌患者预后提供了重要的参考价值。
伦理学声明:本研究方案于2022年2月28日经由吉林大学第一医院伦理委员会审批,批号:2022-017。
利益冲突声明:本文不存在任何利益冲突。
作者贡献声明:吕胜男负责课题设计,数据收集与分析,实验设计及操作,撰写论文;彭新宇负责数据分析及撰写代码;张健负责修改文章;刘欢负责组织样本收集及处理;魏锋负责拟定研究思路,指导文章撰写并最后定稿。

